扩展功能
文章信息
- 梁莹, 海荣, 蔡虹
- LIANG Ying, HAI Rong, CAI Hong
- 鼠疫菌3种常见质粒的结构特征分析
- Structural features of three common plasmids in Yersinia pestis strains
- 中国媒介生物学及控制杂志, 2015, 26(1): 33-36,41
- Chin J Vector Biol & Control, 2015, 26(1): 33-36,41
- 10.11853/j.issn.1003.4692.2015.01.008
-
文章历史
- 收稿日期:2014-08-29
- 网络出版日期:2014-12-17
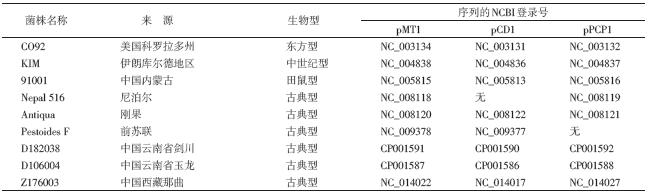
鼠疫耶尔森菌(Yersinia pestis,鼠疫菌)在自然界中以啮齿动物为主要宿主,通过媒介跳蚤的叮咬引起动物间鼠疫的传播和流行[1],人类若接触了染疫动物及其制品或被染疫蚤叮咬,也可能感染鼠疫菌,如果治疗不及时还会进展成能够通过气溶胶在人与人之间传播、严重威胁生命的肺鼠疫,历史上曾先后发生过3次世界范围的鼠疫大流行,夺去上亿人的生命,给人类社会带来了深重灾难[2]。毒力完整的鼠疫菌基因组中含有3种质粒:pMT1、pCD1和pPCP1,根据质粒上主要编码基因的名称和功能分别被命名为鼠毒素质粒、低钙反应质粒和鼠疫菌素质粒,3种质粒最早是由Ferber和Brubaker[3]于1981年发现。其中,pCD1质粒是3种致病性耶尔森菌所共有的(假结核耶尔森菌中称为pIB质粒,小肠结肠炎耶尔森菌中称为pYV质粒),主要编码Ⅲ型分泌系统。pMT1和 pPCP1质粒是鼠疫菌独有的2个质粒,在进化过程中通过基因水平转移而获得,主要编码F1抗原、鼠毒素、纤溶酶原激活蛋白和鼠疫菌素等一系列重要毒力决定因子,在鼠疫菌的致病性、抵抗力和传播过程中发挥了关键作用[4]。研究表明,由于插入序列(IS)介导的同源重组,导致大的DNA片段之间经常发生移位、逆转、逆转性移位等基因重排事件,从而使不同来源鼠疫菌株间的染色体结构差异较大[5]。在鼠疫菌的pMT1、pCD1和pPCP1三种常见质粒核酸序列中亦有一定数量的IS分布,本研究根据鼠疫菌质粒的序列信息,对9株已完成全基因组测序鼠疫菌株的质粒结构特征进行对比分析。 1 材料与方法 1.1 数据来源
4株鼠疫菌中国分离株(91001、D182038、D106004、Z176003)和5株国外鼠疫菌株(CO92、KIM、Nepal 516、Antiqua、Pestoides F)的3种常见质粒序列均下载自美国国立生物技术信息中心网站(http://www.ncbi.nlm.nih.gov/genome),其中,Pestoides F菌株缺失了pPCP1质粒[6],Nepal 516菌株的pCD1质粒序列未测定成功[7](表 1)。
CO92菌株作为参考株,其他8株作为对比株。将CO92菌株pMT1质粒上的所有编码基因(CDS)按照基因编号由小及大的顺序,依次排列在Excel表格的同一列内。然后应用Blast软件将每个CDS序列与其余8株菌的pMT1质粒序列分别进行比对,从中找到与其相似性最高的基因片段。将序列一致性最高的CDSs在各个菌株中的起止核苷酸位置记录在Excel表格的同一行内。pCD1和pPCP1质粒按照同样的方法进行基因比对和数据整理。 1.2.2 基因模块划分
根据CDSs相似性比较结果,将3个以上连续排列的CDSs划分为鼠疫菌质粒的一个基因模块。 1.3 软件自动分析质粒结构
应用Mauve软件包[8]分别对来自9株鼠疫菌的9条pMT1质粒序列、8条pCD1质粒序列和8条pPCP1质粒序列进行多重比对,采用软件默认的参数设置。 2 结 果 2.1 鼠疫菌pMT1质粒结构 2.1.1 基因模块划分结果
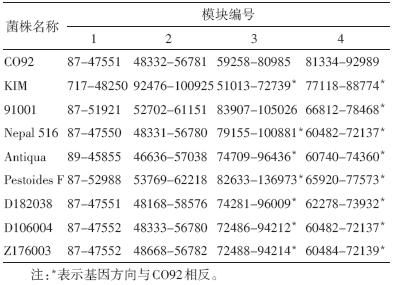
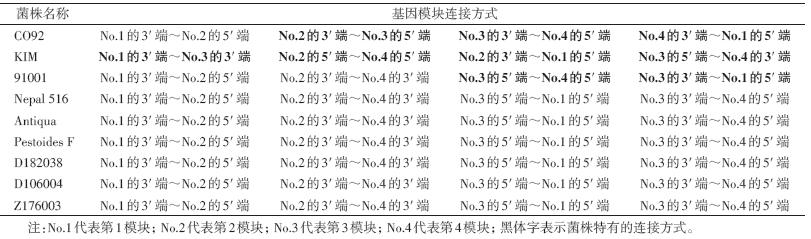
pMT1质粒被人为划分成4个基因模块,每个模块的起止核苷酸位置见表 2。模块内部的基因组成和排列顺序高度保守和稳定,而模块之间可以发生移位和逆转等基因重排事件。与参考株CO92的pMT1质粒相比,KIM菌株中的第3模块和第4模块分别发生了逆转性移位,移动到第1和第2模块之间;91001菌株的第4模块发生了逆转性移位,移动到第2和第3模块之间;Nepal 516、Antiqua、Pestoides F、D182038、D106004和Z176003六株菌的pMT1质粒结构基本相同,均是第3模块和第4模块作为一个整体发生逆转,但没有移位。基因模块在所有菌株中的连接情况见表 3,KIM菌株的4处模块连接方式均与其他菌株不同,CO92中有3处连接方式是该菌株独有,91001中有2处连接方式是其特有的;在Nepal 516等其余6株菌中,4处模块连接情况完全相同。
 |
 |
由图 1A可见,以CO92菌株pMT1质粒序列作为参照,所有菌株均包含4个主要的基因模块,它们在9株菌中的相对位置和方向与2.1.1中的人工划分结果完全一致。存在于CO92、KIM、91001、Nepal 516、Pestoides F菌株中的蓝色小条块,以及存在于KIM、91001、Nepal 516、Pestoides F菌株中的紫色小条块内的基因片段主要为插入序列,在人工划分时被分至基因模块之间的断裂区内。 2.2 鼠疫菌pCD1质粒结构 2.2.1 基因模块划分结果
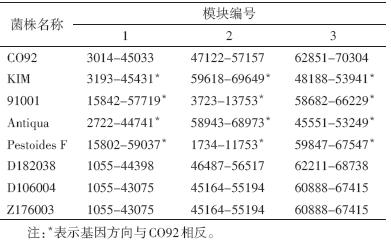
pCD1质粒被大体分成3个基因模块,分离自中国的3株古典生物型菌株(D182038、D106004、Z176003)与参考株CO92的pCD1质粒结构完全相同,而KIM、91001、Antiqua、Pestoides F与CO92相比,3个模块的基因方向均与CO92的相反(表 4)。由于自然状态下的质粒呈环状结构,因此这3个基因模块的连接顺序实际上在8株鼠疫菌中完全一致(缺少Nepal 516的pCD1质粒序列),连接方式均为:模块1的3′端与模块2的5′端相连,模块2的3′端与模块3的5′端相连,模块3的3′端与模块1的5′端相连。
 |
由图 1B可见,同样以CO92菌株pCD1质粒序列作为参照,所有菌株均包含3个主要的基因模块。除此以外,图 1B中还显示了另外3个只包含插入序列的基因片段(分别用红色、绿色和蓝色小条块表示)在每株菌中的位置。绿色和蓝色小条块比较稳定,与主要基因模块的相对位置未发生变化;而红色条块的插入位置在CO92、KIM、91001、Antiqua和Pestoides F 5株菌中均不相同,在D182038、D106004和Z176003菌株中则未插入。

|
| 图 1 鼠疫菌3种常见质粒序列的多重比对结果(Mauve软件自动生成) Figure 1 Multiple sequence alignment of three common plasmids in Y. pestis strains (generated automatically by Mauve software) |
除Pestoides F菌株缺失pPCP1质粒外,其余8株鼠疫菌的pPCP1质粒结构完全相同,即该质粒上所有编码基因的排列顺序和方向在8株菌中完全一致。 2.3.2 Mauve 软件分析结果
与人工划分结果相同,8条pPCP1质粒全部由一个完整的红色条块表示,其间未发现基因重排现象(图 1C)。 3 讨 论
通过对不同鼠疫菌株来源的9条pMT1质粒、8条pCD1和pPCP1质粒进行序列比对分析后发现,整体结构最为稳定的是pPCP1质粒,其次是pCD1质粒,变异程度最大的是pMT1质粒。而这3种常见质粒中,编码基因数目按由少到多的顺序依次是:pPCP1、pCD1和pMT1。因此,小而紧实的基因结构在遗传中更易保持稳定。pCD1质粒虽然被分成3个基因模块,但实际上并未发生基因大片段的重新排列,8条质粒序列间最大的不同在于插入序列IS100的插入位置和插入方向:除分离自中国的3株古典型鼠疫菌pCD1质粒上无IS100插入外,其余5株菌的pCD1质粒均含有1个拷贝的IS100,且在质粒中的位置或者方向各不相同。IS100是鼠疫菌基因组中最为活跃的可移动元件[9],2个方向相反的IS100很容易导致其间的基因片段发生逆转。菌株Pestoides F、D106004、Z176003的pMT1质粒上拥有1个拷贝的IS100;菌株CO92、KIM、Nepal 516的pMT1质粒上包含2个拷贝的IS100,且方向相反;菌株D182038的pMT1质粒上同样包含2个拷贝的 IS100,但方向相同;菌株91001和Antiqua的pMT1质粒上都包含3个拷贝的IS100,且方向不完全相同。结果显示,pMT1质粒具有4种组成结构,分别对应于4种生物型的鼠疫菌株。而导致pMT1质粒上的基因模块发生重排的主要原因是由于插入序列IS100介导的同源重组。
3种常见质粒结构特征的分析结果为研究鼠疫菌质粒的遗传多态性和微进化提供了可靠的数据支持。根据已知的pMT1质粒基因模块连接方式,在每个基因模块边界处设计特异性引物,通过普通PCR技术可以检测更多未测序鼠疫菌株的pMT1质粒结构,进而判断其与鼠疫菌生物型的关联性。此外,还可以对pCD1质粒上IS100的分布特征进行深入研究,也可以监测pPCP1质粒上IS100的分布情况,及时了解鼠疫菌的遗传变异情况。
| [1] | 卫生部卫生应急办公室,中国疾病预防控制中心. 鼠疫防控应 急手册[M]. 北京:北京大学医学出版社,2009:1-5. |
| [2] | Perry RD,Fetherston JD. Yersinia pestis-etiologic agent of plague[J]. Clin Microbiol Rev,1997,10(1):35-66. |
| [3] | Ferber DM,Brubaker RR. Plasmids in Yersinia pestis[J]. Infect Immun,1981,31(2):839-841. |
| [4] | 何电,董兴齐. 鼠疫耶尔森氏菌质粒的结构和功能研究进展[J]. 地方病通报,2008,23(3):64-65,68. |
| [5] | Liang Y,Hou XX,Wang YH,et al. Genome Rearrangements of completely sequenced strains of Yersinia pestis [J]. J Clin Microbiol,2010,48(5):1619-1623. |
| [6] | Garcia E, Chain P, Worsham P, et al. Pestoides F,an atypical Yersinia pestis strain from the former Soviet Union[J]. Adv Exp Med Biol,2007,603:17-22. |
| [7] | Chain PSG,Hu P,Malfatti SA,et al. Complete genome sequence of Yersinia pestis strains antiqua and Nepal 516:evidence of gene reduction in an emerging pathogen[J]. J Bacteriol,2006,188 (12):4453-4463. |
| [8] | Darling ACE, Mau B,Blattner FR,et al. Mauve:multiple alignment of conserved genomic sequence with rearrangements[J]. Genome Res,2004,14(7):1394-1403. |
| [9] | Song YJ,Tong ZZ,Wang J,et al. Complete genome sequence of Yersinia pestis strain 91001,an isolate avirulent to humans[J]. DNA Res,2004,11(3):179-197. |
 2015, Vol. 26
2015, Vol. 26