扩展功能
文章信息
- 吴荣泉, 张建庆, 方义亮, 肖武, 房长天, 刘宝英
- WU Rong-quan, ZHANG Jian-qing, FANG Yi-liang, XIAO Wu, FANG Chang-tian, LIU Bao-ying
- DNA条形码技术在福建省蝇类鉴定中的应用
- Application of DNA barcoding in identification of flies in Fujian province, China
- 中国媒介生物学及控制杂志, 2014, 25(6): 514-517
- Chin J Vector Biol & Control, 2014, 25(6): 514-517
- 10.11853/j.issn.1003.4692.2014.06.007
-
文章历史
- 收稿日期:2014-04-26
2 福建医科大学公共卫生学院, 福州 350108;
3 福建国际旅行卫生保健中心
2 Public Health School of Fujian Medical University, Fuzhou 350108, Fujian Province, China;
3 Fujian International Travel Healthcare Center
蝇类属双翅目昆虫,住区蝇类可机械性传播伤寒、痢疾等多种消化道疾病。蝇种类繁多,仅福建省就已发现405种[1],而且捕获数量和种类均居各类医学媒介生物之首;加之其自身形态特征复杂,形态相似种广泛存在,传统形态鉴定困难,多数雌性麻蝇无稳定的种间形态差异,迫切需要一种更为客观的鉴定方法。DNA条形码技术(DNA barcoding)是利用目的DNA片段序列区分生物物种的一种全新方法,该技术结合多种目的DNA片段,可对生物进行属种级别甚至是近缘种的鉴别,不受生物的生长状态以及形态完整性的影响,越来越多的研究表明DNA条形码技术可以广泛应用于生物的分类与鉴定,是一种简便、高效、准确的物种鉴定技术[2, 3]。细胞色素C氧化酶亚基Ⅰ(COⅠ)基因被证实能对多种动物物种进行高效区分,因而被广泛地应用于物种的分类鉴定中[4]。
目前,蝇类COⅠ基因分类研究的报道主要集中在对经济有影响的实蝇科种群[5, 6]和具有法医学意义的嗜尸性蝇类[7, 8],但与人类接触密切、能够传播部分传染病的住区蝇类研究相对较少。而且分子分类系统并未建立,差异达到多少才是种间差别、属间差别或科间差别仍是一个问题,而蝇类地理株之间差异的存在也给分子鉴定体系的建立增加了难度。因此,本文通过对福建省常见蝇类的COⅠ基因进行研究,初步建立福建省蝇类基因库并探讨该基因在福建省常见蝇类鉴定的可能,为后续的DNA条形码鉴定提供参考依据。 1 材料与方法 1.1 材料
所有标本均于2012-2013年在福建省部分地区采集,共计4科13属19种64只(表 1)。
三羟甲基氨基甲烷盐酸、乙二胺四乙酸铁、十二烷基磺酸钠,购于国药集团化学试剂有限公司;2×Taq PCR Master Mix (主要成分:0.1 U Taq 聚合酶/μl,500 μmol/L dNPT,20 mmol/L Tris-HCl pH 8.3,100 mmol/L KCl,3 mmol/L MgCl2等),购于天根生化科技(北京)有限公司。 1.3 方法 1.3.1 基因组DNA提取
按Collins等[9]方法,提取蝇类DNA。 1.3.2 PCR扩增
(1)引物:采用多细胞无脊椎动物COⅠ通用引物[10]。上游引物LCO1490:5′-GGT CAA CAA ATC ATA AAG ATA TTG G-3′,下游引物HCO2198: 5′-TAA ACT TCA GGG TGA CCA AAA AAT CA-3′。引物由生工生物工程(上海)股份有限公司合成。
(2)PCR 50 μl反应体系:上游引物2.5 μl,下游引物2.5 μl,2×Taq PCR Master Mix 25 μl,DNA模板2 μl,灭菌双蒸水18 μl,共计 50 μl。
(3)PCR反应条件: 94 ℃ 预变性 3 min,30次PCR循环(94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min),72 ℃延伸5 min,最后PCR产物保存在4 ℃条件下。 1.3.3 PCR扩增产物的电泳检测
称取2 g琼脂糖,溶解于100 ml 1×TAE缓冲液,在微波炉上加热成溶胶,并加入5 μl溴化乙锭(0.5 mg/ml),配置成2%的琼脂糖凝胶。取5 μl PCR扩增产物,130 V、30 min电泳,于凝胶成像系统检测观察扩增的条带。 1.3.4 PCR产物测序
采用双向测序,由生工生物工程(上海)股份有限公司完成。 1.3.5 结果分析
利用DNAStar软件的SeqMan程序对每一个体正反向测序的结果拼接,然后使用MegAlign程序进行同源性分析。使用Mega 4.0软件,采用目前应用广泛适合核酸序列经Gamma校正的Kimura 2-parameter模型[11],计算进化分歧,同时构建序列间的邻接系统进化树[12],并采用Bootstrap检验(1000次),计算系统发生树每一分支的支持值。 2 结 果 2.1 PCR 扩增蝇类线粒体COⅠ基因
利用所提取的蝇类DNA为模板扩增COⅠ基因,均获取大约700 bp的扩增片段,与预期结果相符。扩增的COⅠ基因除去引物后为658 bp,在所选的64条序列中,保守位点429个,变异位点231个,总突变数297个,单一突变位点37个,简约位点194个。平均碱基含量:T为38.4%,C为15.7%,A为30.3%,G为15.6%;序列中富含A/T,高达近70%。 2.2 同源性及进化分歧分析
对蝇类相应COⅠ核苷酸系列进行多序列比较分析显示,蝇类的种内同源性为96.4%~100%,其中花蝇科3种蝇类种内同源性为99.4%~100%,蝇科3种蝇类为99.8%~100%,丽蝇科7种蝇类为99.2%~100%,麻蝇科6种蝇类为96.4%~100%。而种间同源性除了丝光绿蝇与铜绿蝇之间高达99.1%~99.4%外,其余种间的同源性均<95.3%。进化分歧分析显示,各蝇种种内进化分歧均数在0~3.32%之间,最高的为紫绿蝇(3.32%),其次为黄须亚麻蝇(1.92%),其余均在1.00%以内。种间进化分歧均数除丝光绿蝇与铜绿蝇之间为0.69%外,其余种间进行分歧均数在5.26%~18.30%之间。 2.3 系统发育树的建立
采用 Kimura 2-parameter+ gamma模型对所选的序列进行分析后,用邻接法建立系统进化树,该树反映了蝇类的系统进化及分类阶元的关系(图 1),从科水平上看,并未像形态学一样形成4个大的独立分支,而是聚集成两大簇,各科间存在同源交叉。从属水平上看,麻蝇科的2属未形成独立的2个分支,短角亚麻蝇形成独立的单系,其他亚麻蝇属内种类与棕尾别麻蝇聚为一簇;丽蝇科中,丽蝇亚科的各属聚成大簇;金蝇亚科也聚成一簇,可信度为91%;各属均形成各自的单系,与形态学结果一致。从种的水平上看,除丝光绿蝇与铜绿蝇外,同种之间呈明显的聚集,且可信度高,均达96%以上。丝光绿蝇与铜绿蝇聚成一簇,达到100%的支持,2种存在同源性交叉。
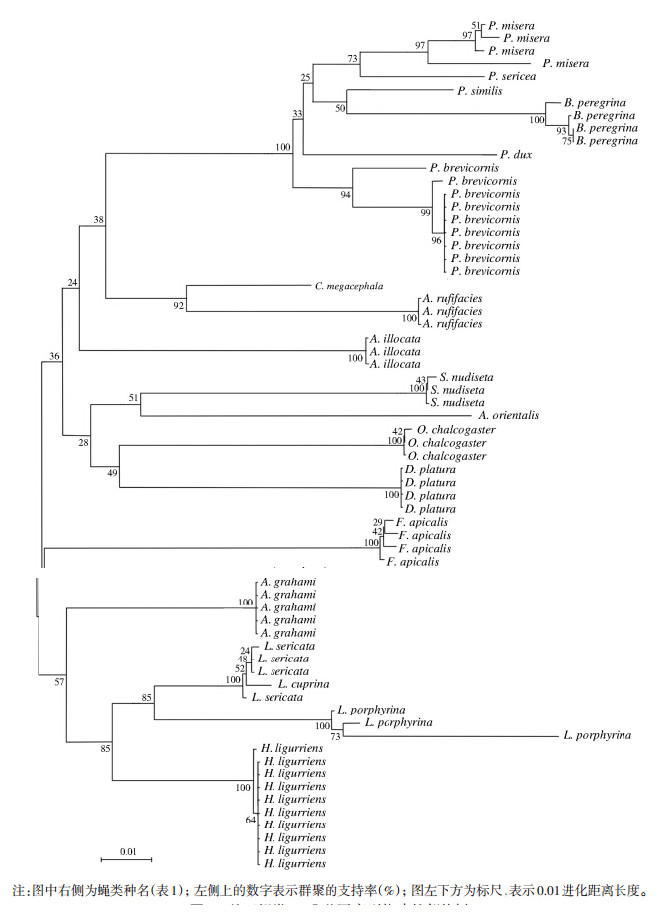
|
| 图 1 基于蝇类COⅠ基因序列构建的邻接树 Figure 1 Neighbor-joining tree based on flies’ COⅠ gene sequences |
有关研究显示,COⅠ适合用在亲缘关系比较近的昆虫分子系统研究,特别是较低阶元(如科内),但对于较高级阶元(如科与科之间)尚不明确[13]。本次结果显示,在科水平上,系统进化树与形态学不一致,各科未独立的完全聚集在一起,而是存在着一定的同源交叉,说明利用COⅠ基因片段尚无法完全将各种蝇类定科。蝇类鉴定困难主要体现在种及种下水平的鉴别,而科与科之间的鉴别特征稳定且明显,容易区分,本研究重点探讨种内水平的鉴别。丽蝇科中,各种种内同源性在99.2%~100%之间,种间同源性除丝光绿蝇与铜绿蝇外,其余均在94.2%以下;进化分歧结果,除紫绿蝇外,其余种内进化分歧均数在1.00%以内,而种间除丝光绿蝇与铜绿蝇外,其余均在5.00%以上。根据Wells和Sperling[14]报道,在丽蝇科中种内进化分歧均数<1.00%,种间分歧均数>3.00%,即可确信在其种内序列差异和进化分歧均数百分比范围内无重复,另外进化树结果显示丽蝇科内不同属分别聚类,属内各种除铜绿蝇与丝光绿蝇聚为一个单系外,其余各自聚成为一个单系,可信度高,说明依据COⅠ基因的658 bp片段可以将丽蝇科中的各种鉴别出来。紫绿蝇虽然其进化分歧均数为3.32%,但其种内同源性为99.2%~100%,且进化树中也完全成为一个单系,所以并不妨碍它的鉴别。铜绿蝇与丝光绿蝇同源性高(99.1%~99.4%),进化分歧小(0.69%),且进化树上不能完全分开,与国内外研究相似[15, 16],说明利用COⅠ基因的658 bp片段对丽蝇科中一些近缘种的鉴别存在缺陷。
麻蝇科,其形态极为相似,通常依据雄性尾器做出鉴别,但雌性麻蝇目前尚无统一的分类标准[17]。本研究显示了各蝇种亲缘关系与形态学分类较为一致,亚麻蝇属内各种同源性高,与棕尾别麻蝇同源性相对较低,进化树显示酱亚麻蝇与短角亚麻蝇、黄须亚麻蝇与褐须亚麻蝇亲缘程度最高,此与Song等[17]的研究结果相似。Kurt等[18]指出通常麻蝇科种内差异<1.50%,而种间一般>2.00%~2.50%,而国外的一些研究也证实麻蝇科种内差异和种间差异比较大,可以用COⅠ基因进行鉴定分类。本研究发现麻蝇科中种内同源性为96.4%~100%,而种间同源性<95.3%,没有重叠;进化分歧均数种内<1.92%,种间>5.00%;系统进化树显示,不同种都各自成为单系,可以区分开来。
肖金花等[19]认为利用COⅠ基因序列来区分物种的关键在于难以提供一个统一的区分标准。本研究也显示了有些种间和种内差异存在重叠,而且系统进化分析时有些种类不能形成强支持的分支,说明COⅠ基因序列用于种内阶元下的分类也存在一定的限制。在基因水平上讨论系统进化,需要大量的生物学信息,单基因难免存在缺陷,扩大基因选取的范围,尝试多个分子标记的结合,可能可以弥补这些缺陷。目前,许多研究证实不同种类间COⅠ基因序列存在足够的差异,但是由于模式标本不能提取DNA并制作成分子标签,因此尚无法做出统一的判断标准。而形态学上,分类学家已经给相当多的物种命名并确定其分类地位,因此将COⅠ基因序列的差异性与形态学相结合,可以达到对蝇类进行准确鉴别的目的。
| [1] | 徐宝海. 福建省蝇类新记录[J]. 中国人兽共患病学报,2009,25(1):73-77. |
| [2] | Hebert PD, Cywinska A, Ball SI, et al. Biological identifications through DNA barcodes[J]. Proc Biol Sci,2003,270(1512):313-321. |
| [3] | Hebert PD, Ratnasingham S, dewaard JR. Barcoding animal life:Cytochrome coxidase subunit 1 divergences among closely related species[J]. Proc Biol Sci,2003,270 Suppl 1:S96-99. |
| [4] | 窦向梅,肖晖,黄大卫. DNA分类概述[J]. 生物学通报,2008,43(6):23-26. |
| [5] | Han HY,Ro KE. Molecular phylogeny of the superfamily Tephritoidea (Insecta:Diptera):new evidence from the mitochondrial 12S,16S,and COⅠ genes[J]. Mol Phylogenet Evol,2005,34(2):416-430. |
| [6] | Harvey ML,Mansell MW,Villet MH,et al. Molecular identification of some forensically important blowflies of southern Africa and Australia[J]. Med Vet Entomol,2003,17(4):363-369. |
| [7] | 王新杰,王学海,刁立江,等. 潍坊地区6种常见嗜尸性蝇类mtDNA COⅠ区序列研究[J]. 法医学杂志,2006,22(2):93-94. |
| [8] | 蔡继峰,廖志钢. 嗜尸性蝇类线粒体DNA分子标记检测的研究进展[J]. 法医学杂志,2005,21(1):68-72. |
| [9] | Collins FH, Mendez MA, Rasmussen MO, et al. A ribosomal RNA gene probe differentiates member species of the Anopheles gambiae complex[J]. Am J Trop Med Hyg,1987,37(1):37- 41. |
| [10] | 岳巧云,邱德义,黄文艺,等. 应用DNA条形码技术鉴定未知双翅目蛹[J]. 中国国境卫生检疫杂志,2011,34(5):343-347. |
| [11] | Kimura M. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences[J]. J Mol Evol,1980,16(2):111-120. |
| [12] | Saitou N, Nei M. The neighbor-joining method:a new method for reconstructing phylogenetic trees[J]. Mol Biol Evol,1987,4(4):406-425. |
| [13] | Bernasconi MV, Pawlowski J, Valsangiacomo C, et al. Phylogeny of the Scathophagidae (Diptera: Calyptratae) based on mitochondrial DNA sequences[J]. Mol Phylogenet Evol,2000,16(2):308-315. |
| [14] | Wells JD, Sperling FAH. Molecular phylogeny of Chrysomya albiceps and C. rufifacies (Diptera:Calliphoridae)[J]. J Med Entomol,1999,36(3):222-226. |
| [15] | Wallman JF, Docnnellan SC. The utility of mitochondrial DNA sequences for the identification of forensically important Blowflies (Diptera:Calliphoridae) in southeastern Australia[J]. Forensic Sei Int,2001,120(1/2):60-67. |
| [16] | Jeffrey DW, Wall R, Stevens JM. Phylogenetic analysis of forensically important Lucilia flies based on Cytochrome oxidase I sequence: a cautionary tale for forensic species determination[J]. Int J Legal Med,2007,121(3):229-233. |
| [17] | Song ZK, Wang XZ, Liang GQ. Phylogenetic relationships among 15 sarcophagid fly species (Diptera:Sarcophagidae) based on partial sequences of Mitochondrial cytochrome b and Cytochrome oxidase subunit I genes[J]. Acts gntomologica Sinica,2008,51(3):298-306. |
| [18] | Kurt J, Gontran S, René R, et al. Identification of forensically important Sarcophaga species (Diptera: Sarcophagidae) using the mitochondrial COⅠ gene[J]. Int J Legal Med,2008,127(2):491-504. |
| [19] | 肖金花,肖晖,黄大卫. 生物分类学的新动向:DNA条形编码[J].动物学报,2004,50(5):852-855. |
 2014, Vol. 25
2014, Vol. 25



