扩展功能
文章信息
- 廖俊蕾, 岳巧云, 邱德义, 魏晓雅, 刘德星, 贾凤龙
- LIAO Jun-lei, YUE Qiao-yun, QIU De-yi, WEI Xiao-ya, LIU De-xing, JIA Feng-long
- 新截获中国未见分布种杜比亚丽蝇形态和DNA条形码鉴定
- Morphology and DNA barcoding of a newly intercepted fly species exotic in China, Calliphora dubia (Macquart, 1855)
- 中国媒介生物学及控制杂志, 2014, 25(6): 509-513
- Chin J Vector Biol & Control, 2014, 25(6): 509-513
- 10.11853/j.issn.1003.4692.2014.06.006
-
文章历史
- 收稿日期:2014-08-04
2 中山出入境检验检疫局, 广东 中山 528403
2 Zhongshan Entry-Exit Inspection and Quarantine Bureau, Zhongshan 528403, Guangdong Province, China
目前往来我国口岸的船只、集装箱很多,货物中难免会夹杂着诸如医学媒介生物等有害生物,尤其是一些在国内没有分布的外来物种,会对经济、生态以及人类健康造成威胁[1]。目前鉴定昆虫的方法主要是依据成虫的外部形态,并辅之以比较解剖学特征、地理分布等信息,依赖经验丰富的分类学专家;但在日常的口岸检疫工作中,面对的往往是形态不完整的虫体,甚至是幼虫、卵、蛹等非成虫虫态,即便是经验丰富的昆虫分类专家也很难进行准确的种类鉴定[2]。以往非成虫虫态的个体需要培养,耗时长,不适合检验检疫部门快速检疫通关的要求[3, 4]。
2003年Hebert等[5]首次倡导使用以细胞色素C氧化酶亚基Ⅰ(COⅠ)基因片段作为主要的DNA条形码基因,并推广其作为一种鉴定物种尤其是动物物种的标准片段,主要通过对标准的目标基因DNA序列进行分析来达到物种鉴定的目的,成功弥补了传统形态分类的不足。COⅠ基因保守度适中,能够保证足够变异的同时又很容易被通用引物扩增,且其DNA序列本身很少存在插入和缺失,适合用来进行物种的鉴定[6],并且能很好地解决不仅成虫还包括幼虫等非成虫阶段的物种鉴定问题[7]。生物条形码技术在全球生物保护计划iBol(the international barcode of life project)的推动下,在全球范围内被广泛应用[8],迄今为止,COⅠ基因在昆虫分类鉴定中被证明是行之有效的,在很多生物门类中都得到成功的应用[9]。在我国的检验检疫工作中,已经有很多应用DNA条形码进行截获媒介生物鉴定的成功案例,如岳巧云等[10]应用该技术鉴定出从英国进口废纸中截获的双翅目蛹的种类;王颖等[11]应用该技术成功鉴定从进口原木中截获的蚊种;贺骥等[12]将该技术应用于口岸吸血蠓类的分子鉴定,提高鉴定效率和准确率等。
本研究对2013年9月中山出入境检验检疫局神湾口岸从澳大利亚进口的水果中截获的丽蝇科雌性成虫进行形态与DNA条形码的鉴定,并结合与澳大利亚University of Wollongong的James Wallman教授赠予标本的形态及DNA条形码比对结果,确定其为杜比亚丽蝇(Calliphora dubia),经科技查新确定为中国未见分布的蝇种。再将其与形态极其相似的螳丽蝇(Calliphora augur)在形态和分子上进行比较,通过二者的相互验证,再次证实DNA条形码在快速准确鉴定外来媒介生物的实用性。 1 材料与方法 1.1 材料
样本为2013年9月中山出入境检验检疫局从澳大利亚进口水果中截获的2只丽蝇科雌性成虫,将其制作成针插标本。为确保结果的正确性,特向澳大利亚University of Wollongong的James Wallman教授索要杜比亚丽蝇和螳丽蝇各2只,作为研究的比对参照标本。研究样本和比对参照标本均保存于中山出入境检验检疫局检验检疫技术中心标本室。 1.2 形态鉴定
使用体视显微镜(型号:ZEISS Discovery. V8)对标本进行检视。依据James Wallman的检索表[13],并与参照标本进行比对。将雄性成虫放入回软器回软24 h后,解剖取出杜比亚丽蝇和螳丽蝇的尾器,用Adobe Illustrator CS6软件对二者的雄性外生殖器和尾叶进行描绘,并用Adobe Photoshop CS6软件进行后期处理。 1.3 DNA条形码鉴定 1.3.1 DNA提取、PCR扩增及产物测序
分别从样本和参照标本上各取1条后足,采用TIANGEN公司生产的Universal Genomic DNA Extraction试剂盒(编号:DP302-02),按照试剂盒的使用手册进行基因组DNA提取。
PCR扩增所用引物为多细胞无脊椎动物COⅠ基因的通用引物:LOC1490 5′-GGT CAA CAA ATC ATA AAG ATA TTG G-3′,HCO2198 5′-TAA ACT TCA GGG TGA CCA AAA AAT CA-3′[14],由宝生物工程(大连)有限公司合成。Ex?Taq DNA聚合酶、dNTP等PCR试剂均从TIANGEN公司购置。
PCR反应体系(50 μl):10×PCR buffer 5 μl;正向引物(20 μmol/L)2.5 μl;dNTP(10 mmol/L)1.5 μl;Ex?Taq DNA聚合酶 1 μl;模板DNA 2 μl;无菌水定容到50 μl。扩增条件:94 ℃变性5 min;94 ℃ 40 s,54 ℃ 40 s,72 ℃ 1 min,反应40个循环;72 ℃延伸10 min。
将经纯化的PCR产物用TIANGEN公司生产的pGM?T克隆试剂盒(编号:VT202-02)进行克隆转化,操作方法按照试剂盒的说明书进行。再随机挑选3个阳性转化子送上海立菲科技有限公司测序。 1.3.2 序列分析与比对
测序结果经校对无误后用于后续分析。将所获得的同一个体的3条序列进行一一比对,因获得的3个克隆序列完全一致,因此仅保留1条序列用于分析。所得序列上传至GenBank,将序列在GenBank和BOLD System v3中进行比对。在GenBank中选取亲缘关系较近的丽蝇属、金蝇属和绿蝇属的序列共6条,另选家蝇(Musca domestica)作为外群。用Mega 6.06软件进行分析[15],以Kimura 2-parameter模式构建邻接法系统发育树。NCBI和BOLD中共有杜比亚丽蝇的COⅠ序列17条,螳丽蝇序列9条,其中与本实验所测序列具有重叠区域的杜比亚丽蝇序列6条,螳丽蝇序列3条,除螳丽蝇的1条序列变化太大未被采用,其他序列均采用与截获样本以及参照样本的序列进行校准,重叠区域仅397 bp,对该397 bp片段以Kimura 2-parameter模式构建邻接法系统发育树。 2 结 果 2.1 形态特征
由于杜比亚丽蝇的形态鉴定缺乏相关的中文资料,为方便检疫或其他人员在实际工作中较容易鉴别杜比亚丽蝇,现将其外部形态描述如下。
体长 8.0~10.0 mm
雄性
头部:复眼裸,间额、侧额、侧颜及颊橙黄色,间额的最小宽度比前单眼横径的2倍还宽,颊的上半部毛黑色,下半部毛黄色,后头的毛亦为黄色;触角第2节及第3节基部橙黄色,第3节灰黑色,上有淡淡的白色粉被;触角芒长羽状,仅端部1/4近裸,褐色;下颚须橙黄色,棒状,端部略粗,前颏黑色。
胸部:背板蓝黑色,具白色粉被,肩胛黄白色,中鬃2+3,背中鬃3+3,肩后鬃3,翅内鬃1+2,沟前翅内鬃不发达;腹侧片鬃2∶1,前、后气门大,淡黄色。胸部侧片几乎全为蓝黑色,除中侧片着生黑色的毛外,其余均着生橙黄色长毛。翅透明,翅肩鳞及前缘基鳞黄色;m1+2脉端段近直角,2R5室开放;下腋瓣几乎全覆盖黄色毛。各足除跗节黑褐色外,全为黄色,具黑色鬃毛。
腹部:背板中央具1条甚宽有金属光泽的紫蓝色纵条,两侧及腹板黄色;第4、5背板具梯形的紫蓝色斑,且整个第5背板均具白粉被。雄性外生殖器:端阳体基部的膜状结构朝内张开呈钝角(侧面观);肛尾叶与侧尾叶末端尖细。
雌性
额宽率为0.35;腹部第4背板的色斑向后缘延伸比雄性明显,第5背板梯形斑两侧白粉被亦较雄性明显,其余与雄性相同。
观察标本,并与从澳大利亚得到的参照标本进行比对,符合上述所描述的形态学特征,初步判定该样本为杜比亚丽蝇。 2.2 序列结果
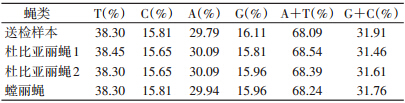
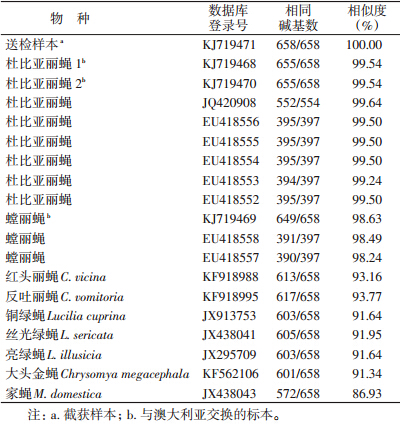
截获样本和参照样本的COⅠ片段均为658 bp,没有缺失和插入,可编码219个氨基酸。对所得COⅠ序列的碱基进行分析,其组成如表 1所示,可见A+T的比例均高于G+C的比例,这与其他关于昆虫COⅠ碱基构成的研究结果一致[16, 17, 18]。截获的样本COⅠ序列与NCBI中及BOLD System v3的杜比亚丽蝇COⅠ序列一致性高达99.64%(表 2)。
 |
 |
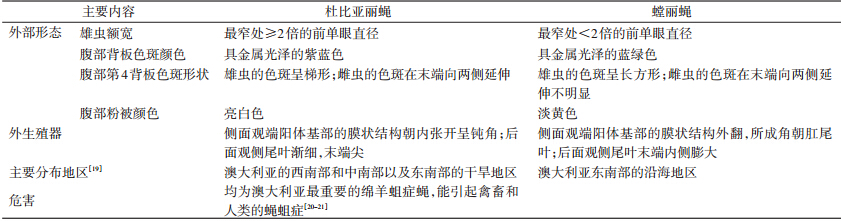
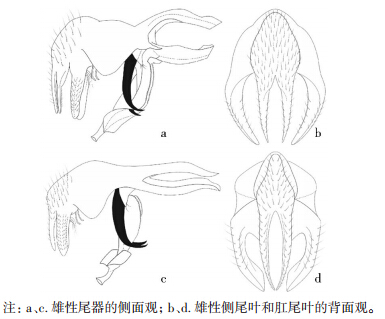
对杜比亚丽蝇和螳丽蝇的成虫在外部形态与地理分布等信息进行比较,其结果见表 3、图 1。
 |
|
| 图 1 杜比亚丽蝇(a、b)与螳丽蝇(c、d)的雄性尾器 Figure 1 Comparison in male terminalia between C. dubia (a,b) and C. augur (c,d) |
截获样本COⅠ序列与参照标本中的杜比亚丽蝇1及杜比亚丽蝇2序列相似度均为99.54%;而与参照标本中螳丽蝇的序列相似度为98.63%(表 2)。截获样本与参照标本的COⅠ序列碱基位点的差异见表 4,截获样本的COⅠ序列与参照标本中2条杜比亚丽蝇的序列有3个碱基不同;而与参照标本螳丽蝇的序列有7个碱基不同。用Mega 6.06软件以Kimura 2-parameter模式计算两两距离,截获样本与参照标本中杜比亚丽蝇的距离为0.46%和0.61%,而与参照标本中螳丽蝇的距离为1.39%。由此可见,样本COⅠ序列与杜比亚丽蝇序列更为相近。
 |
用Mega 6.06软件对所测的4条序列及所引入的7条其他蝇种的序列,选用Kimura 2-parameter模式以邻接法构建系统发育树(NJ树)。由图 2可见,本研究所测的蝇类样本与参照标本杜比亚丽蝇最先聚在一起,然后再与参照标本的螳丽蝇聚在一起;丽蝇属的蝇类均聚在一起,而绿蝇属的3个不同种亦聚成一支,家蝇明显单独为另一支。据此判断样本蝇类为杜比亚丽蝇,与螳丽蝇为亲缘关系很近。
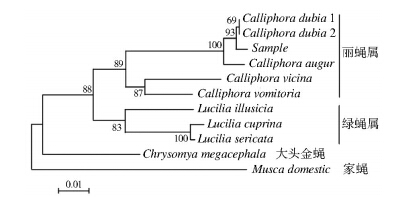
|
| 图 2 基于DNA条形码序列构建NJ系统发育树 (Kimura 2-parameter模式) Figure 2 Neighbor?joining tree based on 658 bp DNA barcodes (Kimura 2-parameter model) |
以GenBank中的杜比亚丽蝇及螳丽蝇序列相对应的长度为397 bp的序列片段及其他参照种类的序列为基础,同样用Mega 6.06软件以Kimura 2-parameter模式构建NJ系统发育树,由图 3可见,所得样本序列及参照标本中杜比亚丽蝇序列与GenBank中的杜比亚丽蝇的序列聚在同一个分支,所测参照标本中螳丽蝇序列与GenBank中螳丽蝇的序列聚在同一个分支,结果与用658 bp的分析结果一致。
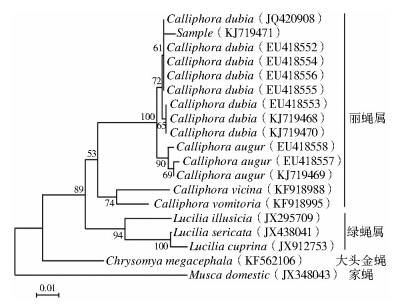
|
| 图 3 基于线粒体COⅠ序列397 bp重叠片段构建的NJ系统 发育树(Kimura 2-parameter模式) Figure 3 Neighbor?joining tree based on 397 bp overlapped COⅠ gene of mitochondria (Kimura 2-parameter model) |
对于外来物种,常常因缺乏相关的形态学资料而无法进行准确鉴定。而分子生物学技术,特别是DNA条形码技术在分类学中的应用,成为传统分类学方法的有力补充。将DNA条形码数据与传统形态特征相结合的“综合分类法(integrated taxonomy)”[22, 23]或更确切地说“条形码与形态分类法(barcodes and morphology taxonomy,也叫B & M分类法)”,被证明是很有成效的[24],而本研究再次验证其实用性与准确性。本研究在对所截获的蝇类样本进行形态学鉴定后,再运用DNA条形码技术进行分子复核鉴定,通过形态特征与分子证据相结合的方法确保鉴定结果的准确性,这一结果也表明DNA条形码可以作为物种鉴定的一种快速、高效、准确的方法,可以作为一种信息和资源缺乏的应对措施,来识别外来生物物种,以弥补传统分类学不足。
杜比亚丽蝇和螳丽蝇在形态上极为相近,在过去曾一度认为杜比亚丽蝇是螳丽蝇的一个亚种[25]。直到1932年Hardy[26]才根据二者腹部背板上的色斑及粉被的不同,以及雄性成虫额宽的不同将杜比亚丽蝇从螳丽蝇中区分出来。在本研究中再次对2个物种的外部形态及外生殖器进行比较,尽管外形上差异细微,但外生殖器上的差异还是较为明显。由于这2个种的亲缘关系很近,曾有关于二者杂交的报道,但所产生的后代不具有生育能力[27],这是二者不是同一个物种,而是近缘种最强有力的证据。
杜比亚丽蝇和螳丽蝇的COⅠ序列在658 bp的长度中仅有7个固定的核苷酸不同,但都未引起编码氨基酸的变化,相似度为98.78%。可见在这658 bp的COⅠ序列中2个物种差异很小,说明2种丽蝇的遗传关系很近,种间差异很小,在系统发育树上同属一单系的2个分支,与Wallman等[28, 29]、Harvey等[30]的研究结果相一致。对此,Wallman等[29]认为这种细微的差异可能是由于澳大利亚的气候在上新世和更新世发生巨大变化后所造成的物种分化。
传统分类学研究十分依赖形态特征完整的标本和分类学家的知识与经验,只有经过长期的专业训练才能培养出精通于某些特定生物门类的分类学专家。而在检疫工作中应用DNA条形码技术,则能够让即便只具备有限分类学知识的国境口岸卫生检疫机关工作人员也能对物种进行科学准确的鉴定,满足目前出入境检验检疫工作中提出的“检得出、检得快、检得准”的要求[31]。当面对中国没有记录的外来入侵物种时,可以通过查阅相关外文文献,与物种原产国的专家学者交流,并通过借阅已确定的标本来比对,从而保证鉴定的准确性。
尽管目前DNA条形码技术在生物分类鉴定上仍存在一定的局限性,并存在一定的争议[32, 33, 34],但与传统的形态分类相结合,可以提高物种鉴定的效率和准确性。相信随着DNA条形码技术及数据库的日趋完善,该技术在我国检验检疫领域的应用前景将更加广阔。
志谢 澳大利亚University of Wollongong的James Wallman教授和CSIRO?Ecosystem Sciences的Adam Slipiński教授在杜比亚丽蝇与螳丽蝇鉴定中给予帮助,特此志谢| [1] | 施宗伟,姚文国. 从口岸截获疫情浅析外来昆虫入侵特点和防范对策[J]. 昆虫知识,2004,41(4):371-374. |
| [2] | Wallman JF. Third-instar larvae of common carrion-breeding blowflies of the genus Calliphora in South Australia[J]. Invertebr Taxion,2001,15(1):37-51. |
| [3] | 谭玲,何建伟,王志杰,等. DNA条形码在国境卫生检疫中的应用[J]. 中国国境卫生检疫杂志,2009,32(5):425-431. |
| [4] | 岳巧云,邱德义,胡佳,等. DNA条形码:医学媒介生物快速准确鉴定的利器[J]. 检验检疫学刊,2013,23(5):49,60-63. |
| [5] | Hebert PDN, Cywinska A, Ball SL, et al. Biological identifications through DNA barcodes[J]. Philos Trans R Soc Lond B Biol Sci, 2003,270:313-321. |
| [6] | Schindel DE, Miller SE. DNA Barcoding a useful tool for taxonomists[J]. Nature,2005,435:17. |
| [7] | Gossner MM, Hausmann A. DNA barcoding enables the identification of caterpillars feeding on native and alien oak (Lepidoptera: Geometridae)[J]. Mitt Münch Ent Ges,2009,99:135-140. |
| [8] | Vernooy R, Haribabu E, Muller MR, et al. Barcoding life to conserve biological diversity: beyond the taxonomic imperative[J]. PLoS Biol,2010,8(7):e1000417. doi:10.1371/journal.pbio.1000417. |
| [9] | Taylor HR, Harris WE. An emergent science on the brink of irrelevance: a review of the past 8 years of DNA barcoding[J]. Mol Ecol Resour,2012,12(3):377-388. |
| [10] | 岳巧云,邱德义,黄艺文,等. 应用DNA条形码技术鉴定未知双翅目蛹[J]. 中国国境卫生检疫杂志,2011,34(5):343-347. |
| [11] | 王颖,王勇,高涛,等. DNA条形码技术用于原木携带蚊种的鉴定[J]. 中国国境卫生检疫杂志,2013,36(4):249-251. |
| [12] | 贺骥,朱福春,苏永宽,等. DNA条形码技术应用于口岸吸血蠓类快速分子鉴定的研究[J]. 中华卫生杀虫药械,2014,20(2):131-135. |
| [13] | Wallman JF. A key to the adults of species of blowflies in southern Australia known or suspected to breed in carrion[J]. Med Vet Entomol,2001,15(4):433-437. |
| [14] | Folmer O, Black M, Hoeh W, et al. DNA primers for amplification of mitochondrial cytochrome c oxidase subunitⅠ from diverse metazoan invertebrates[J]. Mol Mar Biol Biotechnol,1994,3(5):294-299. |
| [15] | Kumar S, Nei M, Dudley J, et al. MEGA: a biologist-centric software for evolutionary analysis of DNA and protein sequences[J]. Brief Bioinform,2008,9(4):299-306. |
| [16] | Renaud AK, Savage J, Adamowicz SJ. DNA barcoding of Northern Nearctic Muscidae (Diptera) reveals high correspondence between morphological and molecular species limits[J]. BMC Ecol,2012, 12:24. doi:10.1186/1472-6785-12-24. |
| [17] | Rivera J, Currie D. Identification of Nearctic black flies using DNA barcodes (Diptera:Simullidae)[J]. Mol Ecol Resour,2009,9 Suppl 1:S224-236. |
| [18] | Schuehli GS, Carvalho CJB, Wiegmann BM. Molecular phylogenetics of the Muscidae (Diptera: Calyptratae): new ideas in a congruence context[J]. Invert Syst,2007,21:263-278. |
| [19] | Wallman JF, Adams M. Molecular systematics of Australian carrion-breeding blowflies of the genus Calliphora (Diptera: Calliphoridae)[J]. Aust J Zool,1997,45:337-356. |
| [20] | Mackerras IM, Fuller ME. A survey of the Australian sheep blowflies[J]. J Csir,1937,10:261-270. |
| [21] | Lee DJ. Human myiasis in Australia[J]. Med J Aust,1968,1(5):170-173. |
| [22] | Dayrat B. Towards integrative taxonomy[J]. Biol J Linn Soc Lond, 2005,85(3):407-415. |
| [23] | Padial JM, Miralles A, De la Riva I, et al. The integrative future of taxonomy[J]. Front Zoo,2010,7:16. |
| [24] | Bergsten J, Brilmyer G, Crampton-Platt A, et al. Sympatry and colour variation disguised well-differentiated sister species: Suphrodytes revised with integrative taxonomy including 5 kbp of housekeeping genes (Coleoptera: Dytiscidae)[J]. DNA Barcodes, 2012,1:1-18. |
| [25] | Kurahashi H. The tribe Calliphorini from Australian and Oriental regions.Ⅱ. Calliphora-group (Diptera: Calliphoridae)[J]. Pacific Insects,1971,13:141-204. |
| [26] | Hardy GH. Some Australian species of Calliphora (subgenera Neopollenia and Proekon)[J]. Bull Entomol Res,1932,23(4):549-558. |
| [27] | Monzu N. Coexistence of Carrion-breeding Calliphoridae (Diptera) in Western Australia[D]. Perth:University of Western Australia,1977. |
| [28] | Wallman JF, Donnellan SC. The utility of mitochondrial DNA sequences for the identification of forensically important blowflies (Diptera: Calliphoridae) in southeastern Australia[J]. Forensic Sci Int,2001,120(1/2):60-67. |
| [29] | Wallman JF, Leys R, Hogendoorn K. Molecular systematics of Australian carrion-breeding blowflies (Diptera:Calliphoridae) based on mitochondrial DNA[J]. Invert Syst,2005,19(1):1-15. |
| [30] | Harvey ML, Gaudieri S, Villet MH, et al. A global study of forensically significant calliphorids: implications for identification[J]. Forensic Sci Int,2008,177(1):66-76. |
| [31] | 岳巧云,石先来. DNA条形码技术“查入侵”又快又准[N]. 中国国门时报,2010-12-09(3). |
| [32] | Dasmahapatra KK, Elias M, Hill RI, et al. Mitochondrial DNA barcoding detects some species that are real, and some that are not[J]. Mol Ecol Resour,2010,10(2):264-273. |
| [33] | DeSalle R, Egan MG, Siddall M. The unholy trinity: taxonomy, species delimitation and DNA barcoding[J]. Philos Trans R Soc Lond B Biol Sci,2005,360:1905-1916. |
| [34] | Dupuis JR, Roe AD, Sperling FAH. Multi-locus species delimitation in closely related animals and fungi: one marker is not enough[J]. Mol Ecol,2012,21(18):4422-4436. |
 2014, Vol. 25
2014, Vol. 25


