服务
文章信息
- 王颖,鲁闽,李金庆,刘明杰,岳巧云
- WANG Ying, LU Min, LI Jin-qing, LIU Ming-jie, YUE Qiao-yun
- 基于COⅠ基因的媒介蝇类快速分子鉴定研究
- Study on rapid molecular identification of vector flies based on COⅠ gene
- 中国媒介生物学及控制杂志, 2014, 25(5): 427-431
- Chines Journal of Vector Biology and Control, 2014, 25(5): 427-431
- 10.11853/j.issn.1003.4692.2014.05.011
-
文章历史
- 收稿日期:2014-05-15
2 中山出入境检验检疫局
2 Zhongshan Exit-Entry Inspection and Quarantine Bureau
医学媒介生物的鉴定和识别是监测与控制疾病发 生的基础,是检疫、疾病预防与控制领域的核心步骤, 经典的鉴定方法为形态学鉴定法。而国境口岸的媒介 生物随交通工具和货物散播的过程中可导致形态缺 失,形态学鉴定法难以正确识别种类。为解决上述难 题,加快口岸通关速度,以蝇类单后足代表微量组织为 研究对象,拟建立基于线粒体细胞色素C氧化酶亚基Ⅰ (mitochondrial cytochrome c oxidase subunitⅠ,COⅠ)基 因的蝇类快速分子鉴定方法。
1 材料与方法 1.1 材料 1.1.1 试剂DNA 抽提试剂盒(DNeasy® Blood & Tissue Kit,QIAGEN),双翅目国际通用引物〔LCO1490: 5′-GGT CAA CAA ATC ATA AAG ATA TTG G-3′,HCO2198:5′-TAA ACT TCA GGG TGA CCA AAA AAT CA-3′,生工生物工程(上海)股份有限公司合 成〕,分子生物学试剂购置于TaKaRa公司。
1.1.2 仪器自动电泳仪(AE8130,ATTO)、凝胶成像 系统(Molecular Imager®,BIO-RAD)、生物安全柜 (SG-603,BAKER COMPANY)、冷冻高速离心机 (CT15RE,HITACHI)。
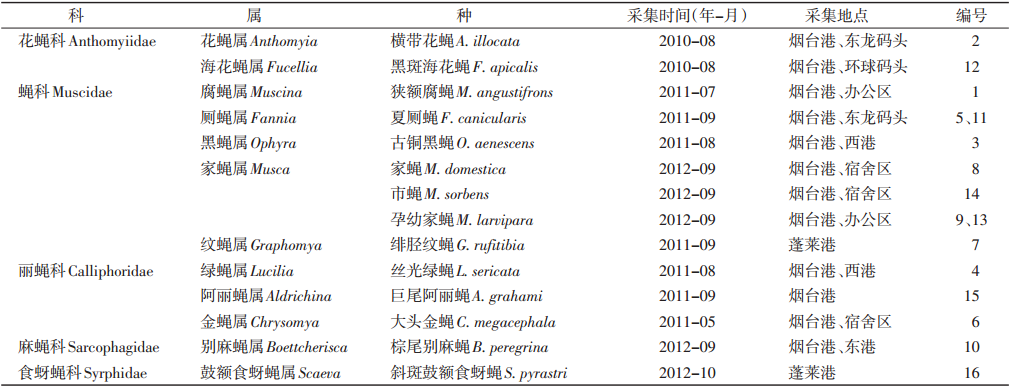
1.1.3 实验对象待测蝇类于烟台口岸使用诱蝇笼诱 捕法捕获,依据《中国常见蝇类检索表》[1]、《中国重要 医学昆虫分类与鉴别》[2],对捕获蝇类进行形态学鉴 定,将完成鉴定的标本装入冻存管内,做好标记,放 入-70 ℃冰箱内保存待用。待测蝇类详见表 1。
取蝇的单后足用DNeasy® Blood & Tissue Kit试剂盒进行基因组DNA抽提;建立总体积为25.0 μl 的反应体系:10×PCR buffer 2.5 μl,25 mmol/L MgCl2 0.5 μl;10 mmol/L each dNTP Mix 0.25 μl,10 μmol/L LCO1490 0.5 μl,10 μmol/L HCO2198 0.5 μl,5 U/ml Taq DNA 聚合酶0.5 μl,DNA模板2.0 μl;反应程序设 定为94 ℃ 3 min;94 ℃ 30 s,45 ℃ 30 s,72 ℃ 1 min,共 5个循环;94 ℃ 30 s,51 ℃ 1 min,72 ℃ 1 min,共35个 循环;72 ℃ 10 min,进行COⅠ基因扩增。
PCR产物经琼脂糖凝胶电泳及纯化回收后,送生 工生物工程(上海)股份有限公司进行序列测定,序列 结果于NCBI、BOLD等数据库进行比对,报告蝇种。并 应用Mega 5.05软件对蝇类COⅠ序列进行系统发育 分析。
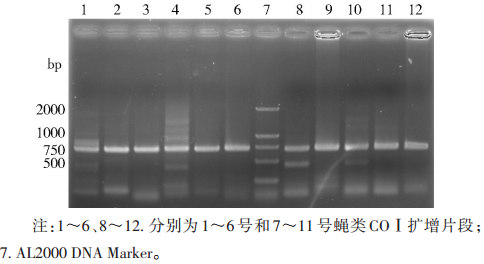
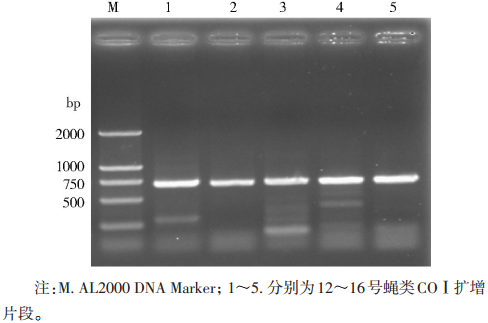
2 结果 2.1 COⅠ基因的PCR扩增和序列分析实验提取蝇 类基因组DNA,针对COⅠ序列进行有效扩增。由 PCR扩增结果可见(图 1、2),待检蝇种在略小于750 bp 下有特异性扩增条带,大小与文献中报道的COⅠ序列 相似[3]。
|
| 图 1 1~11号蝇类样品COⅠ片段PCR扩增结果 Figure 1 PCR amplification of COⅠ fragments from flies labeled 1-11 |
|
| 图 2 12~16号蝇类样品COⅠ片段PCR扩增结果 Figure 2 PCR amplification of COⅠ fragments from flies labeled 12-16 |
与脊椎动物mtDNA相比,昆虫mtDNA富含更多 的A+T,C+G含量偏低[4]。16只蝇类扩增的部分COⅠ 片段长度为690 bp,A、T、C、G碱基平均含量见表 2。COⅠ序列A∶T∶C∶G为29.30∶39.23∶15.45∶16.02,其中 A+T含量高达68.53%,与各文献报道的含量高度相似。
将16只蝇类的COⅠ序列在NCBI上进行比对,其 结果与形态学鉴定结果相同,应用COⅠ序列对蝇类进 行分类鉴定可行。
采用k2p+gamma 模型对16条COⅠ核苷酸序列 进行距离计算,矩阵结果见表 3。
 |
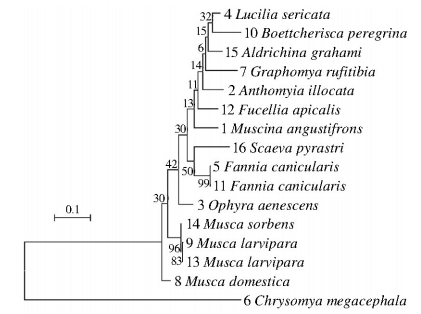
由图 3可见,同为 夏厕蝇的5、11号样品COⅠ基因同源性高达99%;9、 13号孕幼家蝇同源性为96%;孕幼家蝇与市蝇的基因 同源性为83%;但其他蝇种之间的COⅠ同源性均≤ 50%。
|
| 图 3 应用COⅠ数据通过ML法构建的蝇类严格一致树 Figure 3 The strict consensus phylogenetic tree of COⅠ gene in flies constructed with ML method |
与ML系统发育 树类似,NJ法构建的蝇类严格一致树,2只夏厕蝇(5、 11号)的同源性为97%,孕幼家蝇(9、13号)的同源性 为88%,其他蝇种间的同源性<56%(图 4)。

|
| 图 4 应用COⅠ数据通过NJ法构建的蝇类严格一致树 Figure 4 The strict consensus phylogenetic tree of COⅠ gene in flies constructed with NJ method |
MP法构建的系统 发育树与NJ树高度一致(图 4、5)。
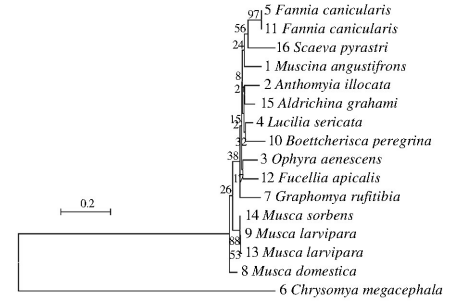
|
| 图 5 应用COⅠ数据通过MP法构建的蝇类严格一致树 Figure 5 The strict consensus phylogenetic tree of COⅠ gene in flies constructed with MP method |
综上所述,在应用COⅠ序列对蝇种进行分类鉴定 时,COⅠ序列种内差异多在5%以内,而种间差异多在 15%以外,种内差异远小于种间差异[5]。显然,COⅠ序 列可理想地将同种蝇类划分在一起。
3 讨论形态学鉴定是最为直观的分类方法,它借助分类 工作者的专业知识和记载形态学特征的论著直接得到 结论。但目前对蝇类进行形态学分类存在局限,如:蝇 类个体差异;蝇类存在隐存分类单元[6, 7],形态学鉴定 方法受蝇类发育阶段的限制[8],分类工作者经验是否 丰富,蝇类形态是否完整等。
线粒体基因的变异速度大约是核基因变异速度的 10倍。相比核基因,线粒体基因更合适于较低阶元的 系统发育关系的建立,每个物种都有唯一的COⅠ核苷 酸序列,种内的遗传差异很小,而种间差异又足够大用 以区分物种[8, 9]。COⅠ基因长度为648 bp(5′末端开始 的58~705位)的片段区域被认为是DNA条形码编码 理想区域。每种生物都具有独一无二的DNA条形码 区域,该区域的碱基排序有4648种可能。因此对4648的 识别完全可以用于对目前已知的1000万种生物的鉴 定[3]。并且,COⅠ基因本身存在很少的插入和缺失, 同时拥有蛋白质编码基因的共同特征——密码子第3 位碱基可以自由变异[2],足以包括所有的物种,而且种 间差异远超过种内差异[10]。目前,COⅠ基因已用于多 种动物及植物的分类研究,动物分类研究较植物更为 成功,可成功对鱼类、鸟类、昆虫等进行分类[11, 12, 13]。研 究显示,COⅠ对研究亲缘关系不是很远的昆虫系统发 育是十分适合的。
入境交通工具、货物中携带的媒介生物在运输途 中不易保持形态的完整性。如出现解剖结构的缺失, 将对形态学鉴定结果产生巨大影响,不同专家鉴定结果之间有可能出现差异。本研究结果显示,以单只蝇 的微量组织作为实验材料可有效扩增COⅠ基因,是解 剖结构缺失情况下进行分类鉴定的理想工具,准确性 高[12]。传统的形态学鉴定对媒介生物形态的完整性要 求高,非成虫期的高度相似性和有些特征在不同的发 育时期不够稳定更增加了形态学鉴定的难度,不能保 证其鉴定的准确性,有学者成功地将DNA 条形码技术 应用到媒介生物的非成虫期物种鉴定上[14, 15],如卵、幼 虫、成虫的样本,不再需要长达数周的培养,再对成虫 进行鉴定,大大缩短了检测周期。
本研究应用DNA条形码技术,建立了COⅠ序列 扩增程序,序列比对结果显示形态学鉴定结果与COⅠ 序列比对结果一致。而且,COⅠ序列种内差异多在 5%以内,而种间差异多在15%以外,种内差异明显小 于种间差异。
应用DNA条形码技术进行分类鉴定的关键环节 之一,即要对扩增到的序列提交到数据库进行比对。 NCBI网站(http://www.ncbi.nlm.nih.gov/)GenBank数据 库囊括的序列种类广泛,将需要比对的序列输入后能 提供较多的类似序列。但也由于内容的广泛,导致某 一领域的不专业性,如COⅠ序列高度类似的情况下, 检索得到的蝇种却不一致。数据库中信息的正确有效 性直接影响到比对结果是否准确。生命的DNA条形 码数据库(BOLD,http://barcodinglife.org/)是权威的条 形码数据库之一,是由加拿大Guelph大学构架和维护 的,供研究人员下载、管理和分析DNA条形码数据,数 据库不仅包括序列信息,还有完整的物种描述、标本图 片、地理分布信息等[16, 17]。基于BOLD以及相应的检 测方法和技术,生命条形码联盟(CBOL,the Consortium for the Barcode of Life)成立,截止到2012年 8月,BOLD共收录179万余条严格通过审核的序列信 息,囊括了12余万物种,为全球物种鉴定工作提供了 有力的数据支持。但相对于自然界庞大的物种群来 说,由于数据库成立时间较短,加之从事条形码工作的 科技工作者比较局限,数据库的内容还没有足够丰富 以进行比对,数据库收录的物种COⅠ序列仍相对较 少。例如,BOLD数据库中关于家蝇科黑蝇属的条形 码信息仅有5条(斑蹠黑蝇1条,厚环黑蝇3条,Ophyra albuquerquei 1条),仅记载黑蝇属3个蝇种,不足以用 于比对工作,建立一个数据可靠,内容丰富的数据库是 条形码工作的一个关键环节。因此,条形码数据库需 要更多、更为严谨的条形码数据为分类鉴定学者提供 比对依据。这就对分子鉴定工作者提出新的要求, 即积极提供实验室科学、精准的实验结果,丰富数据 库数据。
| [1] | 范滋德. 中国常见蝇类检索表[M]. 2 版. 北京:科学出版社, 1992:30-825. |
| [2] | 陆宝麟,吴厚永. 中国重要医学昆虫分类与鉴别[M]. 2版. 郑州: 河南科学技术出版社,2003:375-442. |
| [3] | Frezal L,Leblois R. Four years of DNA barcoding:current advances and prospects[J]. Infect Genet Evol,2008,8(5): 727-736. |
| [4] | Navajas M,Fournier D,Lagnel J,et al. Mitochondrial COⅠ sequences in mites:evidence for variations in base composition[J]. Insect Mol Biol,1996,5(4):281-285. |
| [5] | 肖金花,肖辉,黄大卫. 生物分类学的新动向:DNA条形编码[J]. 动物学报,2004,50(5):852-855. |
| [6] | Hebert PD,Cywinska A,Ball SL,et al. Biological identifications through DNA barcodes[J]. Proc Biol Sci,2003,270(1512): 313-321. |
| [7] | Smith MA,Wood DM,Janzen DH,et al. DNA barcodes affirm that 16 species of apparently generalist tropical parasitoid flies (Diptera:Tachinidae)are not all generalists[J]. Proc Natl Acad Sci USA,2007,104(12):4967-4972. |
| [8] | Ball SL,Hebert PDN. Biological identification of mayflies(Ephem-eroptera)using DNA barcodes[J]. J N Am Benthol Soc,2005,24 (3):508-524. |
| [9] | Herbert PD,Ratnasingham S,de Warrd JR. Barcoding animal life: cytochrome c oxidase subunitⅠ divergences among closely related species[J]. Proc Biol Sci,2003,270 Suppl 1:S96-99. |
| [10] | Tautz D,Arctander P,Minelli A,et al. A plea for DNA taxonomy[J]. Trends Ecol Evol,2003,18(2):70-74. |
| [11] | Smith PJ, McVeagh SM, Steinke D. DNA barcoding for the identification of somoked fish products[J]. J Fish Biol,2008,72 (2):464-471. |
| [12] | Hebert PD,Stoeckle MY,Zemlak TS,et al. Identification of birds through DNA Barcodes[J]. PLoS Biol,2004,2(10):e312. |
| [13] | Lunt DH,Zhang DX,Szymura JM,et al. The insect cytochrome oxidase Ⅰ gene:evolutionary patterns and conserved primers for phylogenetic studies[J]. Insect Mol Biol,1996,5(3):153-165. |
| [14] | 师永霞,相大鹏,李祖海,等. 广东国境口岸不同蚊种COⅠ序列分析和分子鉴定方法[J]. 中国国境卫生检疫杂志,2008,31(2): 103-107. |
| [15] | 岳巧云,邱德义,黄艺文,等. 应用DNA条形码技术鉴定未知双翅目蛹[J]. 中国国境卫生检疫杂志,2011,34(5):343-347. |
| [16] | 莫帮辉,屈莉,韩松,等. DNA条形码识别Ⅰ. DNA条形码研究进展及应用前景[J]. 四川动物,2008,27(2):303-306. |
| [17] | Ivanova NV, Zemlak TS, Hanner RH,et al. Universal primer cocktails for fish DNA barcoding[J]. Mol Ecol Notes,2007,7(4): 544-548. |
 2014, Vol. 25
2014, Vol. 25