扩展功能
文章信息
- 刘智通, 冯浩, 吴家红, 孙毅
- LIU Zhi-tong, FENG Hao, WU Jia-hong, SUN Yi
- 长须血蜱嗅觉相关蛋白的分子特征研究
- Molecular characteristics of olfaction-related proteins in Haemaphysalis aponommoides
- 中国媒介生物学及控制杂志, 2024, 35(3): 274-280
- Chin J Vector Biol & Control, 2024, 35(3): 274-280
- 10.11853/j.issn.1003.8280.2024.03.003
-
文章历史
- 收稿日期: 2024-01-29
2 贵州医科大学基础医学院寄生虫教研室, 贵州 贵阳 561113
2 Department of Parasitology, School of Basic Medicine, Guizhou Medical University, Guiyang, Guizhou 561113, China
蜱属于节肢动物门蛛形纲蜱目(Ixodida),是脊椎动物的专性非永久性外寄生虫,可传播多种病原体,对公共卫生和畜牧业发展产生影响。长须血蜱(Haemaphysalis aponommoides)隶属于硬蜱科血蜱属,多寄生于牦牛和黑熊,广泛分布于尼泊尔、印度以及我国西藏、青海、贵州、甘肃、湖南等省(自治区)。该蜱形态特殊,是进化中较为原始的类型,同时也是人类和动物疾病的潜在传播媒介[1]。目前,对于蜱类的防治以化学防治为主,而化学杀虫剂的长期使用容易造成环境污染、蜱类抗药性和化学药物残留等问题[2-3],因此,急需寻找一种新的经济有效、绿色可持续的防治方法来代替化学杀虫剂防治策略。
蜱类可利用其第1对足跗节上哈氏器(Haller’s organ)的化学感应功能实现对宿主的感知和定位[4-5],这种化学感知功能在蜱类躲避猎物、获取食物、交配和产卵场所选择等方面发挥重要作用[6]。了解蜱类的嗅觉感应分子基础和反应机制,可为新的驱蜱剂或引诱剂的开发提供新的思路。C型尼曼-匹克蛋白2(Niemann-Pick C2 protein,NPC2)是一种小的可溶性携脂蛋白,主要参与脊椎动物的脂质代谢,NPC2蛋白具有和气味结合样蛋白(odorant binding protein like,OBPL)、化学感受蛋白(chemosensory protein,CSP)类似结构及功能特征,表明它们可能在蜱、螨等节肢动物的化学感受中发挥气味结合蛋白作用[5, 7-10]。近年来,关于嗅觉相关蛋白的研究受到广泛关注,主要集中在蜘蛛、蚂蚁、蜜蜂、蚊虫等类群[7, 11-13],而对于蜱类嗅觉相关蛋白的研究鲜有报道。本研究旨在通过对长须血蜱转录组数据库的嗅觉相关基因进行筛选、同源建模和分子对接,初步了解其对潜在气味刺激的感应机制和嗅觉感应分子基础,为探索有效的蜱类控制技术,进而为促进蜱和蜱媒病的防治提供技术支撑[5]。
1 材料与方法 1.1 研究对象长须血蜱采自甘肃省天祝藏族自治县野外植物上,带回后经形态学鉴定,饲养于室温为(25±1) ℃、相对湿度为(70±10)%、自然光周期的实验室环境。成蜱饲血采用新西兰兔(普通级),幼蜱和若蜱采用昆明小鼠(清洁级)。
1.2 嗅觉基因的鉴定用不含核糖核酸酶(RNase)的水将未吸血的雌性成蜱清洗3次,加入液氮并研磨样品。使用RNA提取试剂盒(QIAGEN,货号:74136)从3组未饲血的雌蜱中提取总RNA(每组5只)。使用Nanodrop 1000分光光度计(Thermo Fisher Scientifc)测量核酸浓度。然后建立cDNA文库,并在Illumina HiSeq平台上进行转录组测序和组装(Novogene Technology,中国北京)。测序数据提交美国国立生物技术信息中心(NCBI)数据库,收录于蜱转录组项目(项目号:PRJNA1059273)SRR27389208,基因登录号:PP375530.1~PP375533.1。测序返回的数据进行整理分析,首先对原始测序读长(reads)进行去除接头序列和低质量读长后得到高质量读长,然后使用MEGAHIT将高质量读长进行从头(de novo)组装得到连续序列(contigs)。用基于局部比对算法的搜索工具(basic local alignment search tool,BLAST)的BLASTx将组装好的连续序列与NCBI非冗余蛋白质数据库(Nr)进行比较,为保持高灵敏度和低假阳性率,将期望值(E value)的阈值设为1×10-5,获得全部序列的分类信息,根据分类信息提取相关序列,使用Lasergene 17.3软件包中的SeqMan程序对获得的序列进行合并和去重,从而获得目标片段的完整核苷酸序列。为避免组装拼接错误,将读长映射到相应的参考序列上,同时利用Geneious Prime 2023软件检查映射结果,库中每百万读长(read per million mapped reads,RPM)来估计每个靶标基因的丰度。BLAST同源比对和基因注释使用7个大型数据库[Nr、核酸序列数据库(Nt)、蛋白质家族数据库(Pfam)、真核生物蛋白质直系同源簇(KOG)、Swiss Prot、京都基因与基因组百科全书(KEGG)和基因本体论(GO)][14]。使用开放阅读框(open reading frame,ORF)预测单基因的氨基酸序列。使用针对Pfam的基于隐马尔可夫模型的序列比对工具(hidden Markov model for molecular evolution,HMMER)搜索NPC2同源序列蛋白质数据库,并通过预测跨膜区编码进行鉴定。
1.3 系统发育树构建为了确定目标序列与其他节肢动物类NPC2序列之间的进化关系,利用BLASTp将目标蛋白序列与NCBI数据库比较,筛选来自不同节肢动物的NPC2同源序列,下载fasta格式序列。将所有氨基酸序列输入MEGA 11.0,进行序列对齐,按最大似然法构建进化树。建树参数:建树检验方法为自助法(bootstrap method)、bootstrap值调整为1 000、其他参数设置为默认。
1.4 分子建模与分子对接针对蛋白质数据库的NPC2、microplusin样蛋白(microplusin-like protein,ML)和OBPL序列,在PDB数据库(http://www.rcsb.org)搜索预先构建的位置特定评分矩阵(position-specific scoring matrix,PSSM)中蛋白质序列,以获得同源蛋白质模型。BLAST返回的配置文件使用Clustal W 2.0软件进行分析以获得多重序列比对和系统图。然后利用Swiss-Model软件进行同源建模,先根据序列相似性重新排列同源蛋白pro文件和二级结构,再通过同源性、进化、序列相似性、半胱氨酸(Cys)的数量系统图和结构参数选择最优模型。建模参数设置:“最大主链模型数量”为50、“侧链样品在300K”为5,“中间体”和“最终模型”设置为“优”、AMBER99参数设置为“原子力场”、其他参数设置为默认值。建模后,模型的立体结构优化和自由能最小化分别通过广义波恩-体积整合技术(generalized Born/volume integral methodology)和溶剂化自由能计算(electrostatic solvation energy)进行优化。
以优化后的蛋白三级结构同氨水、吲哚等挥发性气味分子进行分子对接。对接采用Sybyl-X 2.0中的Docking Suite标准模式进行。对接多通道曲面参数设置为原型分子摩尔生成,膨胀系数设置为2 Å,单分子额外起始构象增加为10,“搜索密度”设置为6。为提高对接精度,还考虑了环的灵活性,将最终构象之间的最小均方根偏差(root mean square deviation,RMSD)设置为0.5 Å,其他参数设置为默认值。
2 结果 2.1 嗅觉相关蛋白的序列分析与二级结构预测从长须血蜱中共获得嗅觉相关转录子6个,其中,3个转录子的全长ORF核苷酸长度分别为501、480和444 bp,编码氨基酸长度分别为166(NPC2-1)、159(NPC2-2)和147 aa(NPC2-3),经在线BLASTp比对,与长角血蜱(Ha. longicornis)NPC2序列高度同源;1个转录子全长ORF核苷酸长度为541 bp,编码氨基酸长度为180 aa,与长角血蜱OBPL序列高度同源;剩余2个转录子的全长ORF核苷酸长度分别为453和330 bp,编码氨基酸长度分别为150(ML-1)和110 aa(ML-2),与长角血蜱嗅觉相关的ML蛋白高度同源。与目标蛋白的序列相比,这些嗅觉相关蛋白NPC2(相对分子质量为17 742,等电点为9.03)、OBPL(相对分子质量为20 206,等电点为6.32)和ML(相对分子质量为16 185,等电点为7.32),在100%覆盖度下,氨基酸相似率均 > 90%。基于这些序列利用最大似然法构建的系统发育树结果显示,长须血蜱的上述嗅觉相关蛋白,与相近的长角血蜱、褐黄血蜱(Ha. flava)、青海血蜱(Ha. qinghaiensis)等血蜱属种类的相应蛋白序列聚为一支。见图 1。
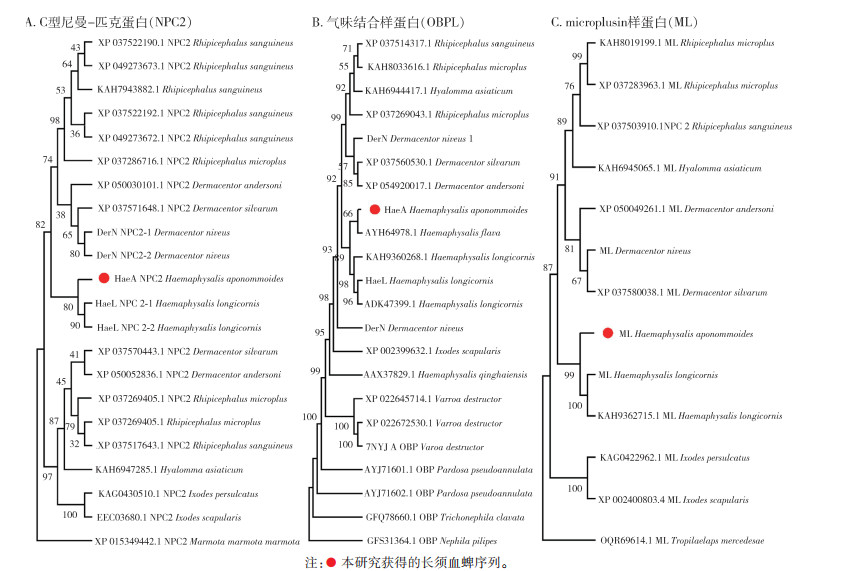
|
| 图 1 长须血蜱3个候选嗅觉相关蛋白与其他节肢动物候选蛋白的系统发育关系 Figure 1 Phylogenetic relationship between three candidate olfaction-related proteins of Haemaphysalis aponommoides and those of other arthropods |
| |
对上述蛋白的二级结构分析结果显示,经SignalP 5.0预测,NPC2的信号肽长度不等,为20~43 aa,结构域编码长度130 aa;含有6个Cys残基,9个β-折叠,5个转角,结构特征为X49-C1-X14-C2-X4-C3-X45-C4-X6-C5-X41-C6-X9(X代表任何氨基酸)。OBPL蛋白的信号肽长度为17 aa,结构域编码长度为148 aa。OBPL具有典型的6个Cys残基,具有2个β-折叠、5个α-螺旋,2个转角,属于X38-C1-X11-C2-X27-C3-X37-C4-X21-C5-X9-C6-X11(X代表任何氨基酸)OBP亚群。仅1条ML蛋白序列(ML-1)有信号肽,长度为20 aa,结构域编码130 aa;含有6个Cys残基,9个β-折叠,5个转角,结构特征为X26-C1-X14-C2-X5-C3-X45-C4-X5-C5-X40-C6-X9(X代表任何氨基酸)。见图 2。
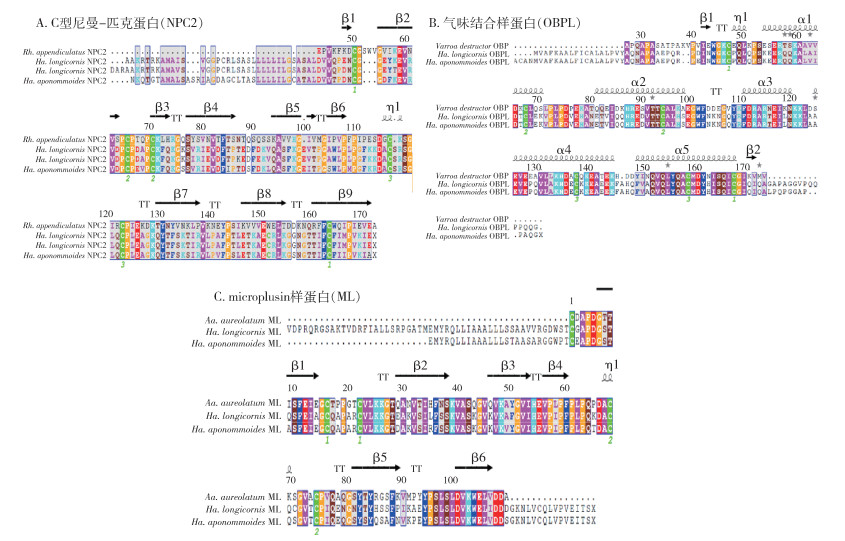
|
| 注:Rh. Rhipicephalus;Ha. Haemaphysalis;Aa. Amblyomma。 图 2 长须血蜱3个候选嗅觉相关蛋白的二级结构示意图 Figure 2 Schematic representation of secondary structure of three candidate olfaction-related proteins of Haemaphysalis aponommoides |
| |
使用Swiss-Model(https://swissmodel.expasy.org/)对所获得序列进行同源蛋白搜索,以相似度 > 50%为阈值获得了长须血蜱NPC2的同源结构蛋白12个,包括附节扇头蜱(Rhipicephalus appendiculatus)NPC2(A0A131YR94)、牛NPC2(2 hka,1nep)、人NPC1-NPC2(5kwy,6w5v)等,选择A0A131YR94作为同源建模的模型,长须血蜱NPC2蛋白模型较为理想预测[全局模型质量估计(global model quality estimation,GMQE)值为0.73],平均精度局部距离差测试(predicted local distance difference test,pLDDT)值为86.56%。通过能量最小化和空间结构优化,模板A0A131YR94与长须血蜱NPC2之间的α-碳原子(αC)的成对最小平方根为1.736 Å,冲突评分为1.23。在Ramachandran图谱中,90.51%的残基(118个残基)位于允许的区域,仅1个A13MET残基(0.63%)位于边缘区域附近(表 1)。长须血蜱NPC2的结合腔由5个转角和9个β-折叠形成。结合腔腔壁主要由64个疏水性残基形成,占比为50.46%,包括Val20、Leu30、Tyr36、Val59、Val64、Phe66、Leu94、Tyr100、Pro101和Ile124等。亲水性残基占比为27.42%,也是腔壁的一部分,包括Glu3、Cys25、Thr41、Asp44、Glu65、Cys68、Cys81、Thr94、Cys97、Glu115、Tyr124、Cys127、Cys138、Thr141和Cys150等(图 3A)。
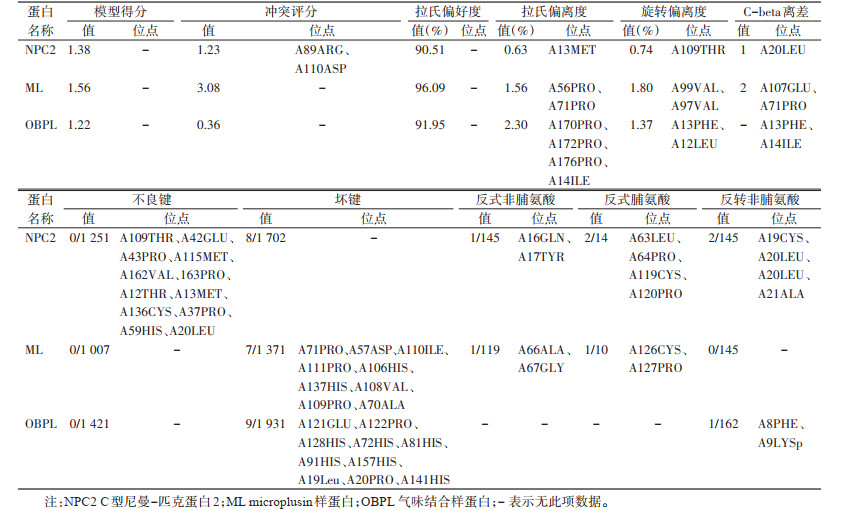
|
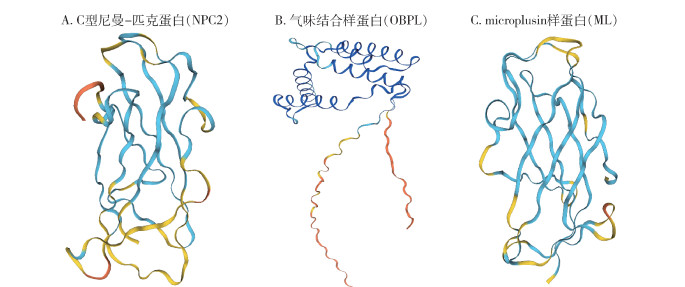
|
| 图 3 长须血蜱3个候选嗅觉相关蛋白的三维结构模型 Figure 3 Three-dimensional structure models of three candidate olfaction-related proteins in Haemaphysalis aponommoides |
| |
长须血蜱OBPL同源结构蛋白5个,包括长角血蜱HAELO(G3BJU6)、蜂蟹螨(Varroa destructor)OBP(7nza,7nyj)等。最后主要基于以下几点选择G3BJU6为基准进行同源建模:(1)依据蛋白结构分类原则,G3BJU6属于蛛形纲气味结合蛋白OBP家族。(2)系统发育结果显示,该序列与同为血蜱属的长角血蜱HAELO G3BJU6蛋白具高度同源性(96.57%),期望值为2×10-21,高于其他基准模型。(3)依据传统的嗅觉蛋白分类方法,该OBPL具有典型的6个Cys残基和5个α-螺旋,同G3BJU6蛋白结构特征类似。(4)长须血蜱OBPL除信号肽外,结构域编码氨基酸是连续的,没有间隙存在。其OBPL蛋白建模结果符合要求(GMQE值为0.80),平均精度pLDDT值为83.19%。通过能量最小化和空间结构优化,模板G3BJU6与长须血蜱OBPL之间的α-碳原子的成对均方根误差(root mean square error,RMSE)为1.856 Å,冲突评分(clashcore)为0.36。在Ramachandran图谱中,91.67%的残基(165个残基)位于允许的区域,2.30%的残基(4个残基,即A14ILE、A170PRO、A172PRO和A176PRO)位于边缘区域附近,显示其整体立体化学质量总体可靠和可接受(表 1)。与蚊虫OBP具有6个α-螺旋不同的是,长须血蜱OBPL的结合腔由5个α-螺旋形成,分别为α1(42~62)、α2(73~90)、α3(62~70)、α4(79~86)和α5(93~103)。长须血蜱OBPL具有C端螺线(η1),N末端没有形成螺旋。结合腔腔壁主要由62个疏水性残基形成,占比为41.83%,包括Thr9、Ile137、Ile143等;亲水性残基占比27.42%也是腔壁的一部分,包括Asp15、Glu37、Asp55、Thr65、Cys70、Try73、Glu79、Asp 91和Cys137等,残基Arg31的氧原子指向空腔中心,形成了空腔的极性表面。见图 3B。
长须血蜱ML蛋白同源结构蛋白11个,包括澳丽花蜱(Amblyomma aureolatum)ML(A0A1E1 WY90)、人NPC2(5kwy、6w5v和2 hka)和牛NPC2(1nep),同理选择A0A1E1 WY90作为同源建模基准,模型GMQE值为0.87,平均精度pLDDT值为91.94%。模板A0A1E1 WY90与长须血蜱ML之间的αC的成对最小RMSE为1.529 Å,冲突评分为3.08。在Ramachandran图谱中,96.09%的残基(144个残基)位于允许的区域,仅1.56%残基(A56PRO和A71PRO 2个残基)位于边缘区域附近(表 1)。长须血蜱ML的结合腔由5个转角和9个β-折叠形成,其中疏水性残基占50.35%,亲水性残基占29.10%。长须血蜱ML蛋白质的结合腔,仅具备Asn39和Asn89氢键结合位点。见图 3C。
2.3 分子对接根据Sybyl-X的Surflex Dock预测,NPC2配基与吲哚碱在12~19个疏水性残基形成的结合口袋中心重叠(图 4A)。与NPC2不同,吲哚碱与结合配体在长须血蜱OBPL口袋的中心重叠,OBPL与吲哚碱结合位点包括Thr9、Ile137和Ile143,Thr65和Cys70与吲哚碱的氧原子之间形成氢键(图 4B)。ML则通过疏水性残基ASN39、ASN89进行结合(图 4C)。
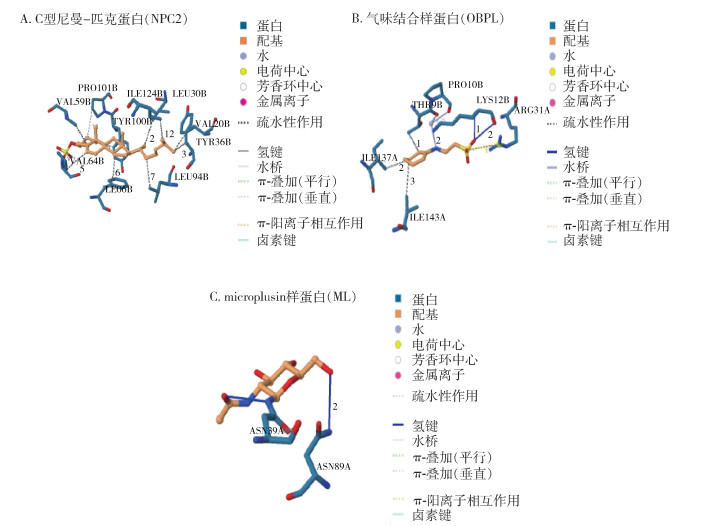
|
| 图 4 长须血蜱3个候选嗅觉相关蛋白与配体的分子对接结果 Figure 4 Molecular docking for three candidate olfaction-related proteins with potential ligands in Haemaphysalis aponommoides |
| |
目前对非昆虫节肢动物的化学感知研究较少[15]。基于这些研究,嗅觉受体(GRs)和离子型受体(IRs)被认为是蜱类化学感知的重要受体。2014年在日本弓背蚁(Camponotus japonicus)中发现了NPC2蛋白[7],这是节肢动物中首次发现的嗅觉相关蛋白,该蛋白在非昆虫节肢动物中能起到信息化学载体的作用,同昆虫中普遍存在的气味结合蛋白(OBP)和CSP功能类似[8]。此后,一些蜱类的嗅觉蛋白研究逐渐被重视。迄今为止,硬蜱属(Ixodes)[4, 9],花蜱属(Amblyoma)[5]、扇头蜱属[4]和钝缘蜱属(Ornithodoros)[16]中已分别发现了多个同源序列。本研究中,3个编码NPC2蛋白、2个编码ML蛋白和1个编码OBPL蛋白的候选基因序列从长须血蜱的转录组数据库中获得,该结果丰富了蜱虫嗅觉基因的相关数据。系统发育分析表明,长须血蜱NPC2、ML和OBPL基因与其他蜱种的同类基因具有一定的同源性,它们的亲缘关系随科、属、种分类阶元的变化又存在一定的特异性,显示出分化的趋势。即使在同一蜱种中,这些基因的表达也存在着一定变异,这与先前的研究结果相似[8]。本研究仅检测了4个候选NPC2基因,其中NPC2-1和NPC2-2同源性较高,分别与血红扇头蜱(R. sanguineus)和森林革蜱(D. silvarum)同源性较高。鉴于蜱类嗅觉相关基因的丰富性和复杂性,未来可能在长须血蜱等重要蜱种中发现更多的嗅觉功能序列。
本研究中,长须血蜱中NPC2和OBPL基因的分子结构和配基结合特点,提示了这些基因参与长须血蜱嗅觉感应的可能性,具体功能还有待进一步的体内结合实验验证。本研究仍有一些明显的局限性。由于需要蜱虫数量较多,参考文献较少,本研究的转录组序列是基于蜱类个体,而不是按器官进行,尤其是第1对足,因此,NPC2、ML和OBPL基因的结果不具备较好的器官针对性。这些基因的定量表达尚需针对不同组织、发育阶段处理进行分析与验证。总之,本研究发现长须血蜱的OBPL和NPC2均具备了参与嗅觉感应的结构特征,为未来探索蜱类高效引诱剂或驱避剂,进而有效控制蜱类和蜱媒病的传播提供了基础数据和技术支撑。
利益冲突 无
| [1] |
Beard CB, Occi J, Bonilla DL, et al. Multistate infestation with the exotic disease-vector tick Haemaphysalis longicornis - United States, August 2017-September 2018[J]. MMWR Morb Mortal Wkly Rep, 2018, 67(47): 1310-1313. DOI:10.15585/mmwr.mm6747a3 |
| [2] |
Sonenshine DE, Kocan KM, de la Fuente J. Tick control: Further thoughts on a research agenda[J]. Trends Parasitol, 2006, 22(12): 550-551. DOI:10.1016/j.pt.2006.09.003 |
| [3] |
Willadsen P. Tick control: Thoughts on a research agenda[J]. Vet Parasitol, 2006, 138(1/2): 161-168. DOI:10.1016/j.vetpar.2006.01.050 |
| [4] |
Josek T, Walden KKO, Allan BF, et al. A foreleg transcriptome for Ixodes scapularis ticks: Candidates for chemoreceptors and binding proteins that might be expressed in the sensory Haller's organ[J]. Ticks Tick Borne Dis, 2018, 9(5): 1317-1327. DOI:10.1016/j.ttbdis.2018.05.013 |
| [5] |
Renthal R, Manghnani L, Bernal S, et al. The chemosensory appendage proteome of Amblyomma americanum (Acari: Ixodidae) reveals putative odorant-binding and other chemoreception-related proteins[J]. Insect Sci, 2017, 24(5): 730-742. DOI:10.1111/1744-7917.12368 |
| [6] |
旷策嫣, 周金林. 蜱的化学感觉系统与驱避剂的研究进展[J]. 中国寄生虫学与寄生虫病杂志, 2022, 40(1): 104-108. Kuang CY, Zhou JL. Research progress on the chemical sensing system and repellents of ticks[J]. Chin J Parasitol Parasit Dis, 2022, 40(1): 104-108. DOI:10.12140/j.issn.1000-7423.2022.01.016 |
| [7] |
Ishida Y, Tsuchiya W, Fujii T, et al. Niemann-Pick type C2 protein mediating chemical communication in the worker ant[J]. Proc Natl Acad Sci USA, 2014, 111(10): 3847-3852. DOI:10.1073/pnas.1323928111 |
| [8] |
Pelosi P, Iovinella I, Felicioli A, et al. Soluble proteins of chemical communication: An overview across arthropods[J]. Front Physiol, 2014, 5: 320. DOI:10.3389/fphys.2014.00320 |
| [9] |
Iovinella I, Ban LP, Song LM, et al. Proteomic analysis of castor bean tick Ixodes ricinus: A focus on chemosensory organs[J]. Insect Biochem Mol Biol, 2016, 78: 58-68. DOI:10.1016/j.ibmb.2016.09.004 |
| [10] |
Zhu J, Guo MB, Ban LP, et al. Niemann-pick C2 proteins: A new function for an old family[J]. Front Physiol, 2018, 9: 52. DOI:10.3389/fphys.2018.00052 |
| [11] |
Xiu CL, Xiao Y, Zhang S, et al. Niemann-pick proteins type C2 are identified as olfactory related genes of Pardosa pseudoannulata by transcriptome and expression profile analysis[J]. Comp Biochem Physiol Part D: Genomics Proteomics, 2019, 29: 320-329. DOI:10.1016/j.cbd.2019.01.004 |
| [12] |
Iovinella I, Mcafee A, Mastrobuoni G, et al. Proteomic analysis of chemosensory organs in the honey bee parasite Varroa destructor: A comprehensive examination of the potential carriers for semiochemicals[J]. J Proteomics, 2018, 181: 131-141. DOI:10.1016/j.jprot.2018.04.009 |
| [13] |
Thambi PJ, Modahl CM, Kini RM. Niemann-pick type C2 proteins in Aedes aegypti: Molecular modelling and prediction of their structure-function relationships[J]. Int J Mol Sci, 2024, 25(3): 1684. DOI:10.3390/ijms25031684 |
| [14] |
崔莹莹, 刘起勇, 张玉栋, 等. 长角血蜱饥饿雌成蜱转录组测序及分析[J]. 中国公共卫生, 2019, 35(7): 876-880. Cui YY, Liu QY, Zhang YD, et al. Sequencing and analysis on transcriptomes of unfed female Haemaphysalis longicornis[J]. Chin J Public Health, 2019, 35(7): 876-880. DOI:10.11847/zgggws1122642 |
| [15] |
Iovinella I, Cappa F, Cini A, et al. Antennal protein profile in honeybees: Caste and task matter more than age[J]. Front Physiol, 2018, 9: 748. DOI:10.3389/fphys.2018.00748 |
| [16] |
Francischetti IMB, Mans BJ, Meng ZJ, et al. An insight into the sialome of the soft tick, Ornithodorus parkeri[J]. Insect Biochem Mol Biol, 2008, 38(1): 1-21. DOI:10.1016/j.ibmb.2007.09.009 |
 2024, Vol. 35
2024, Vol. 35


