扩展功能
文章信息
- 陈敏, 苏超, 石丽媛
- CHEN Min, SU Chao, SHI Li-yuan
- 基于细胞色素C氧化酶亚基Ⅰ基因探讨云南省家鼠鼠疫疫源地黄胸鼠的遗传特性
- Cytochrome c oxidase subunit Ⅰ gene-based genetic characteristics of Rattus tanezumi in domestic rodent plague foci of Yunnan Province
- 中国媒介生物学及控制杂志, 2024, 35(3): 268-273
- Chin J Vector Biol & Control, 2024, 35(3): 268-273
- 10.11853/j.issn.1003.8280.2024.03.002
-
文章历史
- 收稿日期: 2023-11-16
黄胸鼠(Rattus tanezumi)是我国主要家栖鼠种,既往从形态学分类看,我国黄胸鼠有指名亚种和云南亚种2个亚种[1]。2005年我国学者采用世界权威的哺乳动物分类系统(Honacki)将2个亚种统称为黄胸鼠[2-3]。在云南省家鼠鼠疫自然疫源地内,黄胸鼠作为鼠疫主要宿主对形成鼠疫生物群落、维持鼠疫自然疫源性和维护鼠疫生物群落的延续起重要作用[4]。黄胸鼠有高度的适应性,在进化过程中出现了各种各样的能力以适应生存环境,有研究表明以线粒体DNA为基础构建的系统发育树,云贵高原地区表现出明显的单倍体遗传分化,其他地区不明显,从这个角度分析,云南地区的黄胸鼠遗传多样性可能与该地区丰富的物种多样性相关[5-6]。
目前最具吸引力的条形码候选物(即DNA序列)是线粒体细胞色素C氧化酶亚基Ⅰ(cytochrome c oxidase subunitⅠ,COⅠ)基因,长约650 bp。由于COⅠ基因大小和结构较为保守、包含的信息量大、进化速率适中、能够在保证足够变异的同时又很容易被通用引物扩增等特点,已广泛应用于群体遗传学研究[7-8],曾有报道我国黄胸鼠种群从形态上(外形、毛色、头骨、牙齿)具有明显的差别[9]。本研究基于COⅠ基因对云南省不同地理位置和气候环境中分布的黄胸鼠遗传特性进行探讨,旨在为更科学地研判云南省家鼠鼠疫疫源地黄胸鼠的种群来源及扩散,为进一步做好云南省黄胸鼠的种群监测和鼠疫防控提供理论依据。
1 材料与方法 1.1 样本的采集、鉴定及保存2016-2019年从云南省大理白族自治州(大理州)、保山市、德宏傣族景颇族自治州(德宏州)、临沧市、玉溪市、文山壮族苗族自治州(文山州)和西双版纳傣族自治州(西双版纳州)7个地区采用鼠笼捕获的黄胸鼠,经形态学鉴定后,解剖并获取其肝、脾标本,于-80 ℃保存。上述各地市标本数分别为48、39、78、72、60、57和54份。
1.2 基因组DNA提取、PCR扩增及测序使用天根生化科技(北京)有限公司血液/细胞/组织基因组DNA提取试剂盒对所收集的经过鼠疫感染检疫后的标本基因组DNA进行提取,COⅠ扩增使用哺乳动物通用引物MT-R CCTACTCRGCCATTTTACCTATG,MT-F ACTTCTGGGTGTCCAAAGAATCA。扩增体系为25 μl,模板2 μl,正、反向引物各1.5 μl,Mix15 μl,ddH2O10 μl。扩增条件:94 ℃ 5 min;95 ℃1 min,54 ℃ 25 s,72 ℃ 1 min,35个循环;72 ℃ 5 min。实验过程以纯水作为空白对照,扩增产物经琼脂糖凝胶电泳检测后送至北京六合华大基因科技有限公司测序。
1.3 序列分析测序所得序列经Chromas 2.6.6、DNA STARArray star软件进行分析,去除引物序列及测序信号不稳定序列后,进行序列拼接,形成完整序列在美国国立生物技术信息中心(NCBI)进行BLAST同源性比对。用MEGA 11.0.10软件包计算黄胸鼠COⅠ基因的碱基组成及采用邻接(neighbour-joining,NJ)法构建黄胸鼠不同种群单倍型的系统发育树;利用DNASP v5计算单倍型数目、单倍型多样性、核苷酸多样性;使用NETWORK进行单倍型网络图的绘制;使用Arlequin 3.11软件进行分子变异分析(analysis of molecular variance,AMOVA)、基因流(Nm,也称基因迁移)、遗传分化指数(Fst)值的计算。
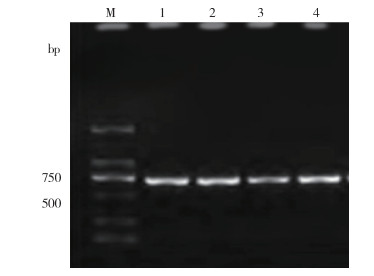
2 结果 2.1 PCR扩增利用扩增引物成功扩增408份黄胸鼠核酸样本COⅠ基因,部分样本扩增结果见图 1。
|
| 注:M表示DNA标记物;1~4表示样本。 图 1 黄胸鼠细胞色素C氧化酶亚基Ⅰ基因扩增结果 Figure 1 Amplification results of cytochrome c oxidase subunit I gene fragments of Rattus tanezumi |
| |
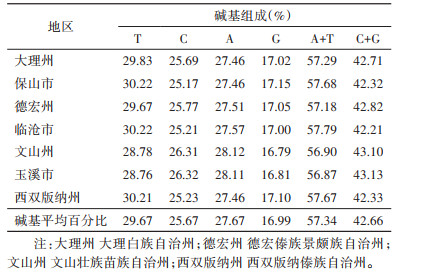
黄胸鼠COⅠ基因序列中T、C、A、G的碱基平均含量分别为29.67%、25.67%、27.67%和16.99%,7个地区黄胸鼠COⅠ基因碱基比例差异不大,A+T含量高于C+G。见表 1。
|
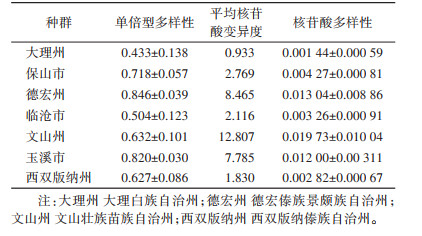
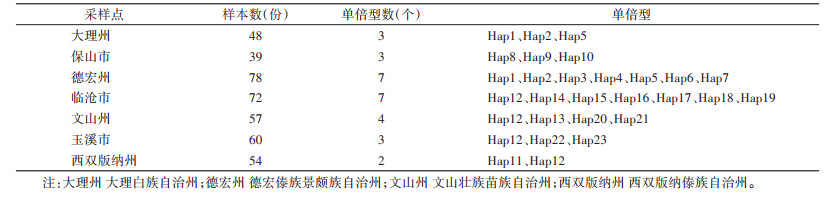
7个种群共定义了23个单倍型,单倍型多样性为0.433~0.846,核苷酸多样性为0.002 82~0.019 73,整体的单倍型多样性由高到低分别为德宏州 > 玉溪市 > 保山市 > 文山州 > 西双版纳州 > 临沧市 > 大理州,核苷酸多样性较高的为文山州、德宏州和玉溪市。单倍型Hap1、Hap2、Hap5为大理州、德宏州种群共有,Hap12为临沧市、文山州、玉溪市和西双版纳州种群共有,其余19个单倍型为各种群特有。见表 2、3。
|
|
对7个调查地区黄胸鼠的COⅠ序列进行聚类分析及单倍型进化网络图分析,不同地理来源的单倍型总共分成3个大支,不同地理种群的单倍型交叉混杂分布。见图 2、3。
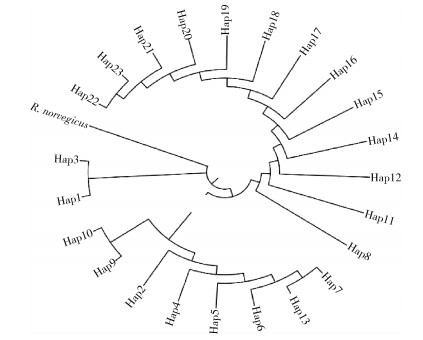
|
| 图 2 基于线粒体细胞色素C氧化酶亚基Ⅰ基因序列构建云南省7个州(市)黄胸鼠种群单倍型邻接系统进化树 Figure 2 Neighbor-joining-based phylogenetic tree of Rattus tanezumi haplotypes based on mitochondrial cytochrome c oxidase subunitⅠgene sequence in seven cities or prefectures of Yunnan Province |
| |
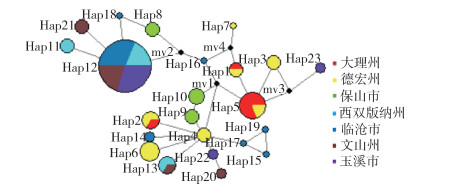
|
| 注:每个实心圆代表1个单倍型,大小与单倍型出现的频率呈正比;每条线代表1次突变;大理州大理白族自治州;德宏州德宏傣族景颇族自治州;文山州文山壮族苗族自治州;西双版纳州西双版纳傣族自治州。 图 3 基于细胞色素C氧化酶亚基Ⅰ基因序列建立的云南省7个州(市)的黄胸鼠单倍型进化网络关系图 Figure 3 Evolutionary network of cytochrome c oxidase subunitⅠgene haplotypes of Rattus tanezumi in seven cities and prefectures of Yunnan Province |
| |
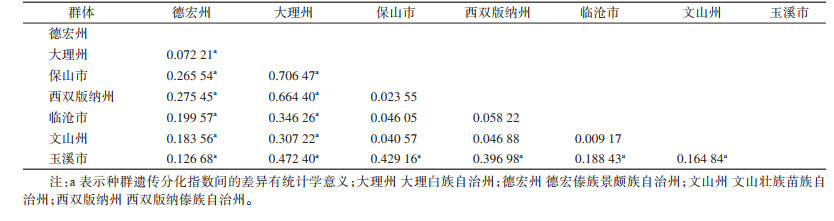
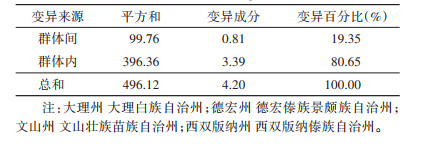
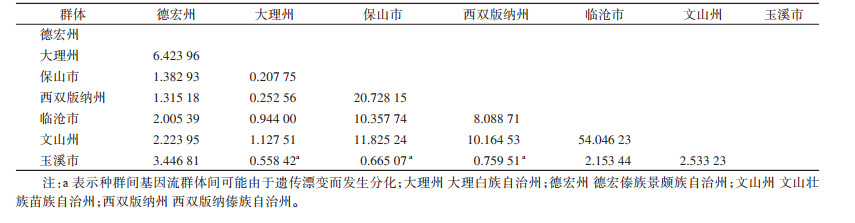
种群间遗传差异分析结果显示,7个地理种群的Fst值为0.009 17~0.706 47,除保山市与西双版纳州、临沧市、文山州,西双版纳州与临沧市、文山州,临沧市与文山州种群间外,其余各地理种群间遗传差异均有统计学意义(均P < 0.05)(表 4)。AMOVA结果表明,来自种群间的遗传变异(19.35%)远低于黄胸鼠种群内的遗传变异(80.65%)(表 5)。7个不同地理种群间基因流范围为0.207 75~54.046 23,见表 6。
|
|
|
本研究获得黄胸鼠COⅠ基因长度为639 bp的同源序列,7个地区黄胸鼠COⅠ基因碱基比例差异不大,A+T含量显著高于C+G,与脊椎动物线粒体DNA的特点相符合[10]。
按照Grant和Bowen[11]提出的标准,单倍型多样性以0.5为临界值,核苷酸多样性以0.005为临界值,二者的值越大,群体的多样性程度越高。本研究中除大理州外其余6个种群均表现出了高单倍型多样性,文山州、德宏州和玉溪市3个种群表现出高核苷酸多样性,单倍型多样性结果与中国部分区域黄胸鼠种群遗传研究中涉及的云南地区种群遗传多样性结果一致[5]。从单倍型系统进化树看出,7个种群的黄胸鼠单倍型出现了3个大的分支且混杂分布。从单倍型分布表和网络分布图分析,7个黄胸鼠种群各自享有较多的单倍型、共享单倍型较少,此结果提示该物种种群遗传多样性普遍较高,且可能蕴藏着较大的进化潜能和丰富的物种资源。从单倍型分布表和进化图看,单倍型Hap4为德宏州独有,其作为中心向其他6个地区种群单倍型发散,推断德宏州Hap4为古老单倍型(由其发出的连线最多,和其他单倍型的联系最为密切)。单倍型Hap12为临沧市、文山州、玉溪市和西双版纳州种群共有且出现频率较高,单倍型Hap5为大理州和德宏州共有,以单倍型Hap12、Hap5为中心发散的单倍型均集中在各自共有单倍型种群,这可能与种群间的地理距离相距较近有关,以地理距离形成了较小的遗传结构。该研究结果与以往针对云南省西南地区4个口岸黄胸鼠种群遗传特性结果类似,本研究中2个遗传多样性指标表明云南省家鼠鼠疫疫源地黄胸鼠种群的群体遗传多样性普遍较高,与云南地区丰富的自然资源及物种多样性相适应[12-13]。
在群体遗传学中衡量群体间遗传分化程度的指标有许多种,较为常见的是Fst[14]。Fst < 0.05表明种群间遗传分化程度较低,0.05~0.15表明种群发生中度遗传分化,Fst > 0.15说明种群间具有较高水平的遗传分化[15]。本研究中7个种群两两之间的Fst均为正值(0.009 17~0.706 47),且两两种群间出现了高、中、低等的遗传分化差异,结合上述州(市)的地理距离、地形地势、生态环境等分析,可能由于地理隔离造成各黄胸鼠种群间出现了不同程度的遗传分化。在群体遗传学上,Nm是指从一个物种的一个种群向另一个种群引入新的遗传物质,从而改变群体“基因库”的组成,通过基因交流向群体中引入新的等位基因,是遗传变异一个非常重要的来源,影响群体遗传多样性,产生新的性状组合。Nm > 1表明群体间的基因流水平较高,群体间遗传分化较小,Nm < 1表明群体间可能由于遗传漂变而发生分化[16]。本研究中除玉溪市与大理州、保山市、西双版纳州种群的Nm < 1,其余种群间均Nm > 1,结合Fst分析,玉溪市与大理州、保山市、西双版纳州种群出现了一定程度的遗传分化,云南省多数黄胸鼠种群间基因交流贫乏,这可能是地理距离隔离所造成。AMOVA分析结果显示,7个黄胸鼠种群间的遗传变异率低于黄胸鼠种群内的遗传变异率,表明云南省黄胸鼠物种基因组稳定。多项研究结果表明遗传距离是评价遗传变异及分化的重要指标,且种群的遗传分化程度会随着地理距离的增加而增加[17-19]。以往对云南省西南地区4个口岸(金水河、磨憨、清水河、姐告)黄胸鼠种群遗传研究结果显示,金水河口岸黄胸鼠与其他3个口岸黄胸鼠亲缘关系较远,说明地理距离对种群遗传分化的影响[13]。由于有研究表明黄胸鼠在云南地区的传播受到地理障碍的限制,不适合做同源相同片段检测分析[20],故本次研究未添加地理距离因素评价种群的遗传变异及分化程度。曾有研究认为较低温度对黄胸鼠的分布区扩大有一定的障碍,黄胸鼠喜热,温度 < 12 ℃不利于其繁殖进而难以成为当地家鼠的优势种群[21-22]。以上7个调查地所处地理位置、海拔形成了不同的地理景观和气候条件,家鼠鼠疫疫源地主要宿主种群数量及分布的遗传生物学规律均存在一定差异。从以上3个遗传分化指标及文献研究综合分析,上述7个黄胸鼠种群出现了部分地理种群间一定程度的遗传分化。
综上所述,本研究基于COⅠ基因分析云南省家鼠鼠疫疫源地黄胸鼠不同种群的遗传特征,结果发现7个黄胸鼠种群表现出丰富的遗传多样性并出现了部分地理种群间的遗传分化。研究为更科学地研判云南省家鼠鼠疫疫源地黄胸鼠的种群来源及扩散、进一步做好云南省黄胸鼠的种群监测和鼠疫防控提供了理论依据。但本次研究仅选择单个线粒体基因的分子标记,难以全面展现真正的群体遗传关系,如进一步分析云南省黄胸鼠的遗传特性,还需要扩大调查范围、持续不间断地开展采样工作及拓展微卫星位点分析等工作。
利益冲突 无
| [1] |
松会武. 黄胸鼠云南亚种研究报告[J]. 贵州农业科学, 1981(6): 21-33. Song HW. The Rattus tanezumi subspecies study in Yunnan[J]. Guizhou Agric Sci, 1981(6): 21-33. |
| [2] |
邓址. 啮齿动物的生态与防治[M]. 北京: 北京师范大学出版社, 1989: 426. Deng Z. Rodent ecology and control[M]. Beijing: Beijing Normal University Publishing Group, 1989: 426. |
| [3] |
Wilson DE, Reeder DM. Mammal species of the world: A taxonomic and geographic reference[M]. 3rd ed. Baltimore: The Johns Hopkins University Press, 2005: 2142.
|
| [4] |
秦长育, 许磊, 张荣祖, 等. 中国鼠疫自然疫源地分型研究V. 鼠疫宿主生物学特征[J]. 中华流行病学杂志, 2012, 33(7): 692-697. Qin CY, Xu L, Zhang RZ, et al. Ecological-geographic landscapes of natural plague foci in China V. Biological characteristics of major natural reservoirs of Yesinia pestis[J]. Chin J Epidemiol, 2012, 33(7): 692-697. DOI:10.3760/cma.j.issn.0254-6450.2012.07.011 |
| [5] |
郭颂. 中国部分区域黄胸鼠种群遗传学研究[D]. 北京: 中国疾病预防控制中心, 2012. Guo S. Study on population genetics of Rattus tanezumi in some regions of China[D]. Beijing: Chinese Center for Disease Control and Prevention, 2012. (in Chinese) |
| [6] |
Lv CC, Zhang LX, Wang RJ, et al. Cryptosporidium spp. in wild, laboratory, and pet rodents in China: Prevalence and molecular characterization[J]. Appl Environ Microbiol, 2009, 75(24): 7692-7699. DOI:10.1128/AEM.01386-09 |
| [7] |
Hebert PDN, Cywinska A, Ball SL, et al. Biological identifications through DNA barcodes[J]. Proc Roy Soc B: Biol Sci, 2003, 270(1512): 313-321. DOI:10.1098/rspb.2002.2218 |
| [8] |
Hebert PDN, Ratnasingham S, deWaard JR. Barcoding animal life: Cytochrome c oxidase subunit 1 divergences among closely related species[J]. Proc Roy Soc B: Biol Sci, 2003, 270 Suppl 1: S96-99. DOI:10.1098/rsbl.2003.0025 |
| [9] |
张荣祖, 杨安峰, 张洁. 云南东南缘兽类动物地理学特征的初步考察[J]. 地理学报, 1958, 24(2): 159-173. Zhang RZ, Yang AF, Zhang J. A preliminary study on the geographical characteristics of fauna in southeastern Yunnan[J]. Acta Geograph Sinica, 1958, 24(2): 159-173. DOI:10.11821/xb195802005 |
| [10] |
肖武汉, 张亚平. 鱼类线粒体DNA的遗传与进化[J]. 水生生物学报, 2000, 24(4): 384-391. Xiao WH, Zhang YP. Genetics and evolution of mitochondrial DNA in fish[J]. Acta Hydrobiol Sin, 2000, 24(4): 384-391. DOI:10.3321/j.issn:1000-3207.2000.04.014 |
| [11] |
Grant WAS, Bowen BW. Shallow population histories in deep evolutionary lineages of marine fishes: Insights from sardines and anchovies and lessons for conservation[J]. J Hered, 1998, 89(5): 415-426. DOI:10.1093/jhered/89.5.415 |
| [12] |
苏军虎. 青藏高原东缘两类典型土著动物种群遗传结构分析[D]. 兰州: 甘肃农业大学, 2014. Su JH. Study on population genetic structure of two kinds of typical native animals in the eastern Qinghai Tibetan Plateau[D]. Lanzhou: Gansu Agricultural University, 2014. (in Chinese) |
| [13] |
刘莹莹, 曹晓梅, 慈颖, 等. 云南省西南地区4个口岸黄胸鼠种群遗传特性分析[J]. 中国媒介生物学及控制杂志, 2020, 31(4): 451-456. Liu YY, Cao XM, Ci Y, et al. Genetic characteristics of Rattus tanezumi populations at four ports of entry in southwest Yunnan Province, China[J]. Chin J Vector Biol Control, 2020, 31(4): 451-456. DOI:10.11853/j.issn.1003.8280.2020.04.015 |
| [14] |
Wright S. The interpretation of population structure by F-statistics with special regard to systems of mating[J]. Evolution, 1965, 19(3): 395-420. DOI:10.1111/j.1558-5646.1965.tb01731.x |
| [15] |
张桂宁, 方弟安. 基于COI基因分析长江下游典型水域翘嘴鲌的遗传多样性[J]. 淡水渔业, 2021, 51(6): 29-35. Zhang GN, Fang DA. Genetic diversity analysis of Culter alburnus Basilewsky in typical waters of the lower Yangtze River based on COI gene[J]. Freshwater Fish, 2021, 51(6): 29-35. DOI:10.3969/j.issn.1000-6907.2021.06.004 |
| [16] |
浦恩念, 高子厚, 李玉琼, 等. 云南部分地区大绒鼠种群遗传多样性与遗传分化研究[J]. 四川动物, 2021, 40(3): 254-260. Pu EN, Gao ZH, Li YQ, et al. Genetic diversity and differentiation of Eothenomys miletus populations in Yunnan Province[J]. Sichuan J Zool, 2021, 40(3): 254-260. DOI:10.11984/j.issn.1000-7083.20200480 |
| [17] |
赵欢, 张伯序, 薛圣伦, 等. 基于COI序列的不同地理种群岩虫遗传多样性研究[J]. 大连海洋大学学报, 2018, 33(1): 33-38. Zhao H, Zhang BX, Xue SL, et al. Genetic diversity of rock worm Marphysa sanguinea from different geographic populations based on mitochondrial COI sequences[J]. J Dalian Ocean Univ, 2018, 33(1): 33-38. DOI:10.16535/j.cnki.dlhyxb.2018.01.006 |
| [18] |
Barker JSF. A global protocol for determining genetic distances among domestic livestock breeds[C]//Proceedings of the 5th world congress on genetics applied to livestock production. Guelph: University of Guelph, 1994, 21: 501-508.
|
| [19] |
Floyd CH, Van Vuren DH, May B. Marmots on great basin mountaintops: Using genetics to test a biogeographic paradigm[J]. Ecology, 2005, 86(8): 2145-2153. DOI:10.1890/04-1227 |
| [20] |
Guo S, Li GC, Liu JL, et al. Dispersal route of the Asian house rat (Rattus tanezumi) on Chinese mainland: Insights from microsatellite and mitochondrial DNA[J]. BMC Genet, 2019, 20(1): 11. DOI:10.1186/s12863-019-0714-3 |
| [21] |
黄文几, 陈延熹, 温业新. 中国啮齿类[M]. 上海: 复旦大学出版社, 1995: 138-141. Huang WJ, Chen YX, Wen YX. Rodents of China [M]. Shanghai: Fudan University Press, 1995: 138-141. (in Chinese) |
| [22] |
吴锡进. 福建中部地区黄胸鼠与褐家鼠繁殖力的初步调查[J]. 中国鼠类防制杂志, 1986, 2(1): 38-40. Wu XJ. Preliminary investigation on the fecundity of Rattus tanezumi and Rattus norvegicus in central Fujian Province[J]. Chin J Rodent Control, 1986, 2(1): 38-40. |
 2024, Vol. 35
2024, Vol. 35


