扩展功能
文章信息
- 刘岳青, 马林源, 马静, 陈开廷, 曹美娜, 王雪薇, 王鹏, 高金亮
- LIU Yue-qing, MA Lin-yuan, MA Jing, CHEN Kai-ting, CAO Mei-na, WANG Xue-wei, WANG Peng, GAO Jin-liang
- 青海血蜱重组蛋白Hq001的结构预测与原核表达
- The recombinant protein, Hq001, in Haemaphysalis qinghaiensis: Structure prediction and prokaryotic expression
- 中国媒介生物学及控制杂志, 2024, 35(2): 138-144
- Chin J Vector Biol & Control, 2024, 35(2): 138-144
- 10.11853/j.issn.1003.8280.2024.02.002
-
文章历史
- 收稿日期: 2023-11-02
2 鄂尔多斯市中心医院分子医学实验室, 内蒙古 鄂尔多斯 017000;
3 内蒙古科技大学包头医学院第一附属医院医学检验科, 内蒙古 包头 014000;
4 内蒙古医科大学鄂尔多斯临床医学院, 内蒙古 鄂尔多斯 017000;
5 河南中医药大学第三附属医院, 河南 郑州 450003
2 Laboratory of Molecular Medicine, Ordos Central Hospital, Ordos, Inner Mongolia 017000, China;
3 Department of Medical Laboratory, The First Affiliated Hospital of Baotou Medical College, Inner Mongolia University of Science & Technology, Baotou, Inner Mongolia 014000, China;
4 Ordos School of Clinical Medicine, Inner Mongolia Medical University, Ordos, Inner Mongolia 017000, China;
5 Third Affiliated Hospital of Henan University of Traditional Chinese Medicine, Zhengzhou, Henan 450003, China
蜱属于节肢动物门蛛形纲蜱螨亚纲寄螨总目蜱总科蜱目,分为软蜱科、硬蜱科和纳蜱科3大科,3个科蜱种在代谢和形态上存在一定差异[1]。青海血蜱(Haemaphysalis qinghaiensis)属于硬蜱科血蜱属,是我国西部地区特有的蜱种,由邓国藩于1978年鉴定,为西部牧区蜱传疾病的主要中间宿主[2-3]。蜱叮咬破损宿主皮肤后,会交替进行唾液输注与血液吸食,同时宿主的凝血级联与炎症反应都会被迅速激发。在此过程中,蜱唾液腺必须释放一系列具有抗凝与抗炎效果的生物活性物质,以保证其持续吸血。这些生物活性因子在医学中具有极大的研究价值。
凝血系统包括血管收缩、血小板聚集和血液凝固。凝血级联中有一系列丝氨酸蛋白酶参与,它们依次相互激活,又在凝血酶原转化为凝血酶时收敛[4]。蜱分泌的唾液中含有脂蛋白、含Kunitz结构域的蛋白、含精氨酸-甘氨酸-天冬氨酸序列的蛋白、金属蛋白酶和防御素等在内的数百种蛋白[5]。含Kunitz型结构域的蛋白酶抑制剂(Kunitz protease inhibitors,KPIs)主要针对宿主凝血系统中的丝氨酸蛋白酶发挥作用,是丝氨酸蛋白酶抑制剂家族的重要成员,许多蜱种也把KPIs作为主要的凝血系统抑制剂[6]。在Kunitz型蛋白家族成员中,牛胰蛋白酶抑制剂(bovine pancreatic trypsin inhibitor,BPTI)是作用机制最为典型的丝氨酸蛋白酶抑制剂。典型的BPTI结构域具有高度保守的三级结构,含有1个或多个抑制结构域。在蜱唾液中KPIs含量较为丰富,承担主要的抗凝血作用[7]。
本实验室在前期研究中从青海血蜱唾液腺cDNA文库中克隆出1个阳性克隆,将该新基因命名为Hq001。经BLASTp比对发现,与来自微小扇头蜱(Rhipicephalus microplus)的典型Kunitz-BPTI型蛋白Boophilin相似。本研究构建了Hq001的原核表达载体并在大肠埃希菌(Escherichia coli)中成功表达了目的蛋白Hq001,利用Hq001所编码蛋白的氨基酸序列进行结构建模与分子动力学(molecular dynamics,MD)模拟,获得了较为可靠的理论模型,并且基于Hq001蛋白理论模型的支持,运用生物信息学工具对Hq001蛋白的功能特性作出评估。
1 材料与方法 1.1 阳性克隆及主要试剂新基因Hq001由鄂尔多斯市中心医院分子医学实验室从青海血蜱cDNA文库中克隆获得并保存。感受态E. coil DH5α、E. coil Rosetta(DE3)(唯地生物技术有限公司);表达载体pET-30a(+)[安诺伦(美国)生物科技有限公司]。
蛋白胨、酵母提取物、丙基硫代-β-D半乳糖苷(isopropylthio-β-D-galactoside,IPTG)、硫酸卡那霉素(Kan+)、10× loading buffer[生工生物工程(上海)股份有限公司];十二烷基硫酸钠聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate-polyacrylamidegel electrophoresis,SDS-PAGE)凝胶制备试剂盒(博士德生物工程有限公司);彩虹180广谱蛋白Marker(11-180KD)[索莱宝(北京)科技有限公司];其他常用试剂均为国产分析纯试剂。
1.2 原核表达载体的构建与表达设计Hq001基因的扩增引物,并引入NdeⅠ和XhoⅠ的限制性内切酶位点。引物序列如下,Hq001F:5'-ATCATATGAATGAGCACGTGGGCA-3';Hq001R:5'-GCCTCGAGCTTTTGAAAATTTTGAAATCT-3'。以Hq001为模板,扩增目的基因片段。PCR条件为:94 ℃预变性3 min;94 ℃ 40 s、56 ℃ 40 s、72 ℃ 40 s,循环25次;72 ℃延伸6 min。用凝胶纯化试剂盒纯化PCR产物,将纯化的PCR产物和表达载体pET-30a用NdeⅠ和XhoⅠ限制性内切酶分别进行双酶切,回收纯化目的片段。将线性化的载体和含有酶切粘性末端的PCR产物用T4DNA连接酶连接,构建原核表达质粒pET30a-Hq001。用所构建的质粒转化E. coil DH5α,获得阳性克隆,并通过酶切和测序验证。
使用重组质粒pET30a-Hq001转化E. coil BL21(DE3)感受态细胞,获得重组菌。将重组菌接种到100 ml LB/Kan培养基中,在37 ℃/220 r/min下培养过夜。将10 ml过夜培养所获种子接种到1 L 2YT/Kan培养液,在相同条件下培养至600 nm处吸光度(A)值达到0.60,然后加入IPTG至终浓度0.50 mmol/L,在25 ℃/220 r/min下再振摇培养4~6 h。随后,将培养物于4 ℃、5 500×g离心20 min,将所得沉淀重悬于0.10 mol/L磷酸盐缓冲液(phosphate-bufferd sodium,PBS)中,pH值为7.8。将重组菌使用JY92-11DN型(宁波新芝物科技有限公司)超声波细胞粉碎机超声破碎30 min,功率为130 w,工作5 s,间隔冷却5 s,全程在冰浴中进行。留取部分超声处理后的细菌裂解液为对照,其余于4 ℃、7 500×g高速离心30 min,分别留取上清与沉淀进行SDS-PAGE分析[8-9]。
1.3 同源建模与序列比对使用Expasy网站中的Translate工具进行翻译[10],网址https://web.expasy.org/translate/。使用SignalP-5.0网站工具对N端信号肽切割位点进行预测[11],网址https://services.healthtech.dtu.dk/services/SignalP-5.0/。将获得的蛋白质序列与美国国立生物技术信息中心(National Center for Biotechnology Information,NCBI)蛋白质数据库与SWISS-Model数据库中其他序列进行比较[12],在Espript 3.0网站上对几种蛋白质序列进行比对[13],网址为https://espript.ibcp.fr/ESPript/cgi-bin/ESPript.cgi[14]。将去除信号肽的蛋白质氨基酸序列进行同源建模,以由AlphaFold程序匹配具有同源性的Kunitz型蛋白质(PDB ID:6 NAN,chain A;2ODY,chain E;1BIK,chain A等)的晶体学模型作为理论Hq001模型的模板,通过本地部署的AlphaFold v2.3软件生成初始理论模型。AlphaFold基于深度神经网络算法,以同源建模的思想与端到端的方式实现蛋白质结构的计算[15]。
1.4 MD模拟将AlphaFold生成的理论模型作为输入,使用格罗宁根化学模拟体系(Groningen machine for chemical simulation,GROMACS)v2023中进行MD模拟,优化理论模型[16]。在MD模拟过程中,采用最陡下降法进行能量最小化以确定能量最低态的理论模型作为最初构象,当迭代达到500 000步或能量梯度 < 1 000 kJ/mol/nm时,能量最小化终止[17]。pH 7.0时带电基团质子化状态的蛋白质结构文件通过H++服务器计算得出[18],服务器相关信息登录http://biophysics.cs.vt.edu/H++网站获得。为保持溶剂盒中模型电荷中性,采用蒙特卡罗离子放置法加入Na+和Cl-离子来中和系统中的电荷。模拟选用力场为AMBER99SB-ILDN[19],水模型为TIP3P。溶质被放入1个立方体盒子中,蛋白质位于盒子中心位置,距离盒子边界的最小距离为10 Å。之后对系统施加恒定温度(300 K)与恒定压力(1 atm),分别对溶剂盒系统施行加温加压各1 ns豫驰,以确定等温等压系综状态。在最终的MD中,首先采用200 ps的带位置约束的位移模拟豫驰系统,然后采用100 ns的无约束位移模拟来优化蛋白质结构,增强结构合理性与稳定性。分别对均方根偏差(root mean square deviation,RMSD)、均方根波动(root mean square fluctuation,RMSF)和总能量变化进行调用分析。使用软件DuIvyTools对分析结果进行绘制,软件开源网址:https://github.com/CharlesHahn/DuIvyTools。
1.5 蛋白优化模型的评价MD模拟结束后,使用PyMOL程序对生成的最终构象进行可视化分析。对于模型的质量评估使用MolProbity[20]、SAVES v6.0[21]和ANOLEA[22]等网站中的软件进行计算。
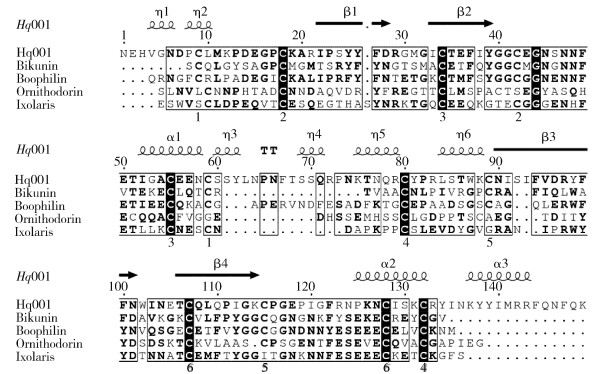
2 结果 2.1 Hq001的序列初步分析新基因Hq001的核苷酸序列长度为773 bp,含有1个483 bp的开放阅读框(open reading frame,ORF),所编码的蛋白质含有169个氨基酸残基,理论相对分子质量为19 500,等电点为8.35。氨基酸序列的GenBank登录号为MN306323。其中N端的19个氨基酸可能为信号肽,序列中含有12个氨基酸为半胱氨酸,可能形成6对二硫键。序列比对发现Hq001所编码的蛋白质与Uniprot中4种具有抗凝血功能的蜱源KPIs序列一致性介于5%~15%,序列相似性介于15%~25%,各序列之间半胱氨酸残基一致性较高,跨群相似性较高,Hq001蛋白可能为KPIs家族成员。见图 1。
|
| 注:α表示α螺旋;β表示β折叠;η表示310螺旋;Bikunin,Uniprot ID为P02760;Boophilin,Uniprot ID为Q8 WPI2;Ornithodorin,Uniprot ID为P56409;Ixolaris,Uniprot ID为Q964Q0。 图 1 Hq001与4种蜱源蛋白酶抑制剂Bikunin、Boophilin、Ornithodorin和Ixolaris的序列比对结果 Figure 1 Sequence alignment between Hq001 and four tick-derived Kunitz protease inhibitors of Bikunin, Boophilin, Ornithodorin, and Ixolaris |
| |
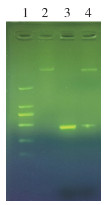
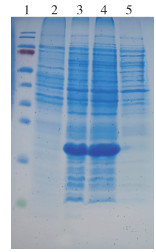
将Hq001的核酸片段克隆入pET-30a表达载体中,构建重组表达质粒pET30a-Hq001。该质粒经酶切和PCR鉴定,发现目的基因片段成功插入表达载体中,基因测序也证实目的基因片段成功克隆入表达载体中,重组表达质粒pET30a-Hq001成功构建(图 2)。SDS-PAGE结果显示,表达菌株经终浓度为0.50 mmol/L的IPTG在25 ℃诱导4 h成功表达了目的蛋白,所表达重组蛋白的相对分子质量约为18 500,与预测的大小一致。将重组菌裂解后发现沉淀中含有大量目的蛋白,而上清中未见目的蛋白的出现,rHq001在E. coil中以包涵体的形式存在。见图 3。
|
| 注:1标准DNA标志物;2 pET30a-Hq001质粒;3 pET30a-Hq001 PCR扩增产物;4 pET30a-Hq001 Nde Ⅰ和Xho Ⅰ酶切产物。 图 2 重组表达质粒pET30a-Hq001的验证 Figure 2 Validation of the recombinant plasmid pET30a-Hq001 |
| |
|
| 注:1标准蛋白标志物;2 BL21株空载;3重组蛋白rHq001总体;4重组蛋白rHq001沉淀部分;5重组蛋白rHq001上清部分。 图 3 rHq001超声破碎后的十二烷基硫酸钠聚丙烯酰胺凝胶电泳分析 Figure 3 Sodium dodecyl sulfate-polyacrylamidegel electrophoresis analysis of rHq001 after ultrasonic fragmentation |
| |
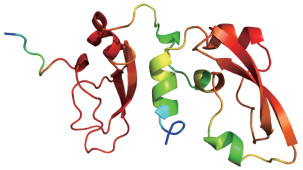
Hq001的每残差置信度(per-residue local distance difference test,pLDDT)评分为82.87,属于较为可信的预测模型,其单个残基的pLDDT评分见图 4。图中单个残基的评分按照光谱颜色变化,红色最好,蓝色较差。可以看到图中蛋白结构包含2个Kunitz结构域,结构域之间以一段铰链区相连。其中结构较为紧凑保守的Kunitz结构域pLDDT评分最高,而N端、C端与铰链连接区域评分均较低。
|
| 图 4 Hq001三级结构预测模型单个残基每残差置信度评分图 Figure 4 Plot of per-residue confidence scores for individual residues of Hq001 tertiary structure prediction model |
| |
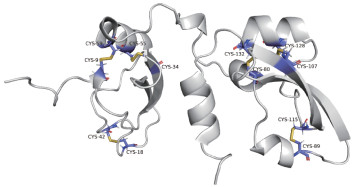
使用PyMOL程序标记出蛋白结构中半胱氨酸位置,蛋白条带结构用白色表示,半胱氨酸sticks结构用紫色表示并添加其氨基酸位点标签(图 5)。观察其三维结构发现其二硫键位置与蜱源凝血途径抑制剂Boophilin[蛋白质结构数据库(protein data bank,PDB)ID:2ODY,chain E]二硫键位置在三维空间中极为相似,并且其半胱氨酸的配对形式也遵循经典的Kunitz结构中以保守的C1-C6、C2-C4、C3-C5形式相结合(图 5),这种二硫键存在形式能够使蛋白质三级结构更加紧凑而稳定。
|
| 图 5 Hq001三级结构预测模型二硫键位置及半胱氨酸位点标注 Figure 5 Disulfide bond position and cysteine site annotation of Hq001 tertiary structure prediction model |
| |
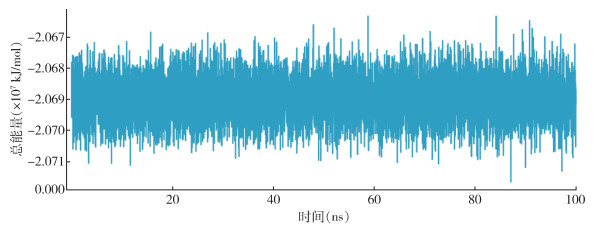
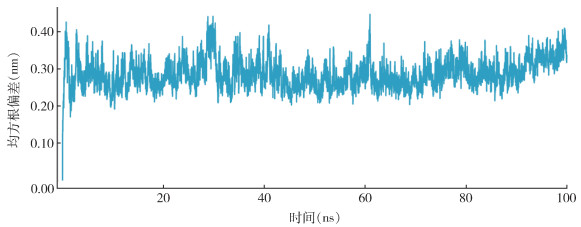
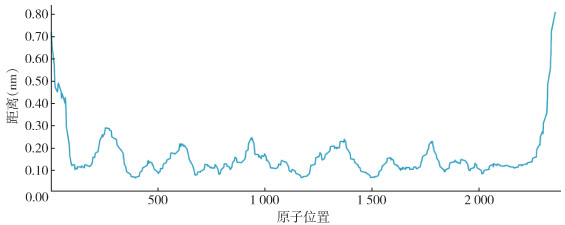
为评估Hq001三维建模的能量与稳定性,并通过优化残基使其结构更加合理,对其执行了100 ns等温等压系综下在水中的MD模拟。结果表明,Hq001整个构象的总能量变化在100 ns的MD模拟过程中相当稳定,波动可以忽略不计(图 6);结构中总α-碳原子的RMSD值在0.24 nm波动(图 7)。图中显示Hq001的RMSD值在MD模拟1 ns左右即达到平衡,并在3 Å左右上下波动。由于二硫键可以稳定Hq001的Kunitz结构域,因此除了N端与C端的灵活区域的原子发生较大位置变化,RMSF在MD模拟过程中变化较小,偏差范围在1~3 Å。这些结果显示Hq001预测模型的结构与其初始结构发生偏离,朝着能量更稳定的空间构象迅速变化(图 8)。根据总能量数据的变化情况,提取能量最低的结构模型(energy=-20 716 640 kJ/mol)作为优化后模型进行评估分析。
|
| 图 6 Hq001在100 ns分子动力学模拟中总能量变化 Figure 6 Change in total energy of Hq001 in 100 ns molecular dynamics simulation |
| |
|
| 图 7 Hq001在100 ns分子动力学模拟中均方根偏差 Figure 7 Root mean square deviation of Hq001 in 100 ns molecular dynamics simulation |
| |
|
| 图 8 Hq001在100 ns分子动力学模拟中均方根波动 Figure 8 Root mean square fluctuation of Hq001 in 100 ns molecular dynamics simulation |
| |
ANOLEA结果显示氨基酸负能态占95.97%。在149个氨基酸当中仅有6个氨基酸为高能态,其中4个分别存在于蛋白质的N末端与C末端。拉氏图是蛋白三维结构评价最为经典的评估手段,用于评估模型的α二面角是否合理。拉氏图评估表明,91.20%的残基位于最有利区域,8.80%位于允许区域,无残基位于慷慨允许区域和不允许区域。上述结果符合拉氏图评价标准。Verfiy3D的评估标准为至少有80%的氨基酸残基的3D-1D评分在0.10以上。通过Verfiy3D程序对Hq001模型进行评价,发现81.21%的残基的3D-1D平均评分在0.10以上。在MolProbity分析中,全原子接触评估得分为2.17,蛋白质几何学评价当中MolProbity得分为1.56,均符合评估体系的可接受范围。
3 讨论在本研究中,针对青海血蜱成虫唾液腺cDNA文库中克隆到的1株新基因Hq001进行研究。Hq001所编码蛋白质的氨基酸序列与多种KPIs具有同源性,含有Kunitz结构域的蛋白质普遍具有抑制1种或多种丝氨酸蛋白酶的活性[7]或作为离子通道阻滞剂发挥作用[23]。
为进一步探究Hq001蛋白质功能,对Hq001进行了原核表达,发现rHq001在E. coil中以包涵体形式成功表达。包涵体的形成通常是因重组蛋白大量表达、细菌胞浆环境减少、真核伴侣缺失和翻译后修饰机制缺失等导致[24]。为获得可溶性蛋白,从改变诱导条件与建立包涵体蛋白纯化方法2个方面进行了尝试。在改变诱导条件方面,先后尝试了降低或不用诱导剂IPTG与逐渐降低诱导温度[25],最终发现在无诱导剂和降低诱导温度的情况下蛋白质表达量显著降低,但仍然以包涵体形式表达。在包涵体纯化方面,先后尝试高浓度尿素与盐酸胍变性,变性蛋白或复性蛋白结合镍柱[26],梯度透析咪唑与添加增溶剂等多种方法[27],但取得可溶性蛋白浓度较低,不足以进行蛋白质功能实验。后续实验中将尝试应用酵母等真核表达系统对Hq001蛋白进行表达,获取可溶性蛋白。
近年来随着神经网络算法技术的不断发展,AlphaFold、RoseTTAFold、Uni-Fold等基于人工智能算法对未知结构进行同源建模的开源软件呈暴发式增长,这大大加速了结构生物学的发展。本研究使用对单体蛋白预测效果较好的AlphaFold对Hq001蛋白进行模型构建,AlphaFold2作为近年来将神经网络模型与同源建模思想相结合的开源软件,能够基于输入的蛋白质序列预测其三级乃至四级结构[15]。虽然这种算法程序极为先进,但仍依赖于蛋白质序列本身的同源性高低。对于与现有解析出的蛋白质结构同源性较低的蛋白质序列来说,此种预测方法仍然较差,但Hq001蛋白与多种KPIs具有共同的保守序列,同源性较高。AlphaFold2使用pLDDT评分来衡量单个残基的置信度,数值范围为0~100。其中pLDDT > 90为高置信度预测模型,pLDDT < 50为低置信度预测模型[28],本研究所构建的Hq001蛋白模型与经典的KPIs表现出极高的相似性。该模型由中段铰链区连接2个保守的Kunitz结构域,其中每个结构域由3对二硫键维持其结构稳定性。根据预测结构不同部位的评分以及其他几种主要模板蛋白的生理功能,推测Hq001蛋白可能作为凝血途径抑制物而发挥作用。Hq001蛋白与Boophilin的保守区域序列与空间构象均极为相似,推测其与Boophilin具有类似的功能。从空间结构上看,二者含有类似的Kunitz结构域,而它们的N端与C端结构域在空间构象上略有不同。Boophilin将N端结构域直接插入到凝血酶的活性位点是为了稳定结合状态,而其C端结构域与蛋白酶的外位点Ⅰ相结合来发挥抑制作用[29-30]。Hq001蛋白也可能以类似的方式发挥其功能。
MD模拟可以模拟蛋白质在一定时间内的结构与动力学变化,对于模拟中蛋白质构象的轨迹进行采样与计算,能够明确蛋白质分子的整体与局部运动情况。RMSD值是较有代表性的参数,一定时间内RMSD值的变化能够反映蛋白质构象的变化程度[31]。Hq001蛋白的RMSD结构表明预测模型构象变化较小,结构较为稳定。此外,Hq001蛋白Kunitz结构域中的残基pLDDT评分较高,而铰链区与N、C末端评分较低,这可能表示保守的Kunitz结构域在进化过程中变化较小。在模拟进行中蛋白空间结构迅速收缩,总的与疏水溶剂的可及面积逐渐降低,这些现象表明蛋白结构逐渐紧凑,向能量更低更为稳定的状态变化。判断模型的可靠性须借由第三方评估软件对模型进行评价,一般认为有3种评价标准结果通过,则认为理论模型是可靠的。经过MD模拟的优化,得到的蛋白质预测模型在几种评价体系中均表现合理,认为此结构可能能够用于结构生物信息学的后续研究。
本研究报道了青海血蜱的1个重组蛋白Hq001的序列信息及其三维空间模型预测评价方法。初步分析表明Hq001蛋白具有典型的双Kunitz结构域,其结构与几种凝血途径相关的丝氨酸蛋白酶抑制剂极为相似。这些信息提示Hq001在凝血途径中可能具有重要的研究价值,可针对此蛋白质进行后续的功能研究,以期发现其在抗凝血方面的更多证据。
利益冲突 无
| [1] |
Gui Z, Wu L, Cai H, et al. Genetic diversity analysis of Dermacentor nuttalli within Inner Mongolia, China[J]. Parasit Vectors, 2021, 14(1): 131-143. DOI:10.1186/s13071-021-04625-5 |
| [2] |
Martins LA, Kotál J, Bensaoud C, et al. Small protease inhibitors in tick saliva and salivary glands and their role in tick-host-pathogen interactions[J]. Biochim Biophys Acta Proteins Proteom, 2020, 1868(2): 140336. DOI:10.1016/j.bbapap.2019.140336 |
| [3] |
陈泽, 刘敬泽. 蜱分类学研究进展[J]. 应用昆虫学报, 2020, 57(5): 1009-1045. Chen Z, Liu JZ. Recent progress in tick taxonomy and a global list of tick species[J]. Chin J Appl Entomol, 2020, 57(5): 1009-1045. DOI:10.7679/j.issn.2095-1353.2020.104 |
| [4] |
Ali A, Zeb I, Alouffi A, et al. Host immune responses to salivary components: A critical facet of tick-host interactions[J]. Front Cell Infect Microbiol, 2022, 12: 809052. DOI:10.3389/fcimb.2022.809052 |
| [5] |
Reck J, Webster A, Dall'Agnol B, et al. Transcriptomic analysis of salivary glands of Ornithodoros brasiliensis Aragão, 1923, the agent of a neotropical tick-toxicosis syndrome in humans[J]. Front Physiol, 2021, 12: 725635. DOI:10.3389/fphys.2021.725635 |
| [6] |
Camillo LDMB, Ferreira GC, Duran AFA, et al. Structural modelling and thermostability of a serine protease inhibitor belonging to the Kunitz-BPTI family from the Rhipicephalus microplus tick[J]. Biochimie, 2021, 181: 226-233. DOI:10.1016/j.biochi.2020.12.014 |
| [7] |
Jmel MA, Voet H, Araújo RN, et al. Tick salivary Kunitz-type inhibitors: Targeting host hemostasis and immunity to mediate successful blood feeding[J]. Int J Mol Sci, 2023, 24(2): 1556. DOI:10.3390/ijms24021556 |
| [8] |
Xu ZM, Yan YJ, Zhang HS, et al. A serpin from the tick Rhipicephalus haemaphysaloides: Involvement in vitellogenesis[J]. Vet Parasitol, 2020, 279: 109064. DOI:10.1016/j.vetpar.2020.109064 |
| [9] |
Chang PP, Li XY, Lin JY, et al. scFv-oligopeptide chaperoning system-assisted on-column refolding and purification of human muscle creatine kinase from inclusion bodies[J]. J Chromatogr B, 2022, 1209: 123410. DOI:10.1016/j.jchromb.2022.123410 |
| [10] |
Duvaud S, Gabella C, Lisacek F, et al. Expasy, the Swiss bioinformatics resource portal, as designed by its users[J]. Nucleic Acids Res, 2021, 49(W1): W216-W227. DOI:10.1093/nar/gkab225 |
| [11] |
Armenteros JJA, Tsirigos KD, Sønderby CK, et al. SignalP 5.0 improves signal peptide predictions using deep neural networks[J]. Nat Biotechnol, 2019, 37(4): 420-423. DOI:10.1038/s41587-019-0036-z |
| [12] |
Bienert S, Waterhouse A, De Beer TAP, et al. The SWISS-MODEL Repository-new features and functionality[J]. Nucleic Acids Res, 2017, 45(D1): D313-D319. DOI:10.1093/nar/gkw1132 |
| [13] |
Robert X, Gouet P. Deciphering key features in protein structures with the new ENDscript server[J]. Nucleic Acids Res, 2014, 42(W1): W320-324. DOI:10.1093/nar/gku316 |
| [14] |
Porter L, Radulović Ž, Kim T, et al. Bioinformatic analyses of male and female Amblyomma americanum tick expressed serine protease inhibitors (serpins)[J]. Ticks Tick Borne Dis, 2015, 6(1): 16-30. DOI:10.1016/j.ttbdis.2014.08.002 |
| [15] |
Jumper J, Evans R, Pritzel A, et al. Highly accurate protein structure prediction with AlphaFold[J]. Nature, 2021, 596(7873): 583-589. DOI:10.1038/s41586-021-03819-2 |
| [16] |
Páll S, Zhmurov A, Bauer P, et al. Heterogeneous parallelization and acceleration of molecular dynamics simulations in GROMACS[J]. J Chem Phys, 2020, 153(13): 134110. DOI:10.1063/5.0018516 |
| [17] |
Bernetti M, Bertazzo M, Masetti M. Data-driven molecular dynamics: A multifaceted challenge[J]. Pharmaceuticals (Basel), 2020, 13(9): 253-279. DOI:10.3390/ph13090253 |
| [18] |
Anandakrishnan R, Aguilar B, Onufriev AV. H++ 3.0: Automating pK prediction and the preparation of biomolecular structures for atomistic molecular modeling and simulations[J]. Nucleic Acids Res, 2012, 40(W1): W537-541. DOI:10.1093/nar/gks375 |
| [19] |
Lindorff-Larsen K, Piana S, Palmo K, et al. Improved side-chain torsion potentials for the Amber ff99SB protein force field[J]. Proteins, 2010, 78(8): 1950-1958. DOI:10.1002/prot.22711 |
| [20] |
Williams CJ, Headd JJ, Moriarty NW, et al. MolProbity: More and better reference data for improved all-atom structure validation[J]. Protein Sci, 2018, 27(1): 293-315. DOI:10.1002/pro.3330 |
| [21] |
Colovos C, Yeates TO. Verification of protein structures: Patterns of nonbonded atomic interactions[J]. Protein Sci, 1993, 2(9): 1511-1519. DOI:10.1002/pro.5560020916 |
| [22] |
Melo F, Feytmans E. Assessing protein structures with a non-local atomic interaction energy[J]. J Mol Biol, 1998, 277(5): 1141-1152. DOI:10.1006/jmbi.1998.1665 |
| [23] |
Zhang GG, Jia HX, Luo L, et al. Molecular and functional characterization of a novel Kunitz-type toxin-like peptide in the giant triton snail Charonia tritonis[J]. Mar Drugs, 2022, 20(11): 686. DOI:10.3390/md20110686 |
| [24] |
Carrió MM, Cubarsi R, Villaverde A. Fine architecture of bacterial inclusion bodies[J]. FEBS Lett, 2000, 471(1): 7-11. DOI:10.1016/s0014-5793(00)01357-0 |
| [25] |
Restrepo-Pineda S, Bando-Campos CG, Valdez-Cruz NA, et al. Recombinant production of ESAT-6 antigen in thermoinducible Escherichia coli: The role of culture scale and temperature on metabolic response, expression of chaperones, and architecture of inclusion bodies[J]. Cell Stress Chaperones, 2019, 24(4): 777-792. DOI:10.1007/s12192-019-01006-x |
| [26] |
Guo XY, Wang RX, Ma RF, et al. Facile purification of active recombinant mouse cytosolic carboxypeptidase 6 from Escherichia coli[J]. Protein Expr Purif, 2022, 197: 106112. DOI:10.1016/j.pep.2022.106112 |
| [27] |
Lipničanová S, Chmelová D, Godány A, et al. Purification of viral neuraminidase from inclusion bodies produced by recombinant Escherichia coli[J]. J Biotechnol, 2020, 316: 27-34. DOI:10.1016/j.jbiotec.2020.04.005 |
| [28] |
Varadi M, Anyango S, Deshpande M, et al. AlphaFold protein structure database: Massively expanding the structural coverage of protein-sequence space with high-accuracy models[J]. Nucleic Acids Res, 2022, 50(D1): D439-D444. DOI:10.1093/nar/gkab1061 |
| [29] |
Soares TS, Watanabe RMO, Tanaka-Azevedo AM, et al. Expression and functional characterization of boophilin, a thrombin inhibitor from Rhipicephalus (Boophilus) microplus midgut[J]. Vet Parasitol, 2012, 187(3/4): 521-528. DOI:10.1016/j.vetpar.2012.01.027 |
| [30] |
Macedo-Ribeiro S, Almeida C, Calisto BM, et al. Isolation, cloning and structural characterisation of boophilin, a multifunctional Kunitz-type proteinase inhibitor from the cattle tick[J]. PLoS One, 2008, 3(2): e1624. DOI:10.1371/journal.pone.0001624 |
| [31] |
Farmer J, Kanwal F, Nikulsin N, et al. Statistical measures to quantify similarity between molecular dynamics simulation trajectories[J]. Entropy (Basel), 2017, 19(12): 646. DOI:10.3390/e19120646 |
 2024, Vol. 35
2024, Vol. 35


