扩展功能
文章信息
- 王金锁, 赵延梅, 魏有文, 周奎章, 李积德, 马英
- WANG Jin-suo, ZHAO Yan-mei, WEI You-wen, ZHOU Kui-zhang, LI Ji-de, MA Ying
- 青海省海东市小型兽类体表寄生蚤种及其鉴定研究
- Species and their identification of ectoparasitic fleas on small mammal in Haidong City, Qinghai Province, China
- 中国媒介生物学及控制杂志, 2023, 34(5): 664-670
- Chin J Vector Biol & Control, 2023, 34(5): 664-670
- 10.11853/j.issn.1003.8280.2023.05.014
-
文章历史
- 收稿日期: 2023-02-02
2 青海省地方病预防控制所鼠疫预防控制科, 青海 西宁 811602
2 Department of Plague Prevention and Control, Qinghai Institute for Endemic Disease Prevention and Control, Xining, Qinghai 811602, China
青海省位于喜马拉雅旱獭(Marmota himalayana)鼠疫自然疫源地的中心地带,为我国鼠疫危害最为严重的地区之一。近年动物间鼠疫连年发生,人间鼠疫呈抬头趋势,严重威胁着当地人民群众的生命安全。海东市因地处我国最大的内陆咸水湖——青海湖东部而得名,介于东经100°41.5′~103°04′,北纬35°25.9′~37°05′。互助土族自治县(互助县)北山国家森林公园和循化撒拉族自治县(循化县)孟达国家级自然保护区均在海东市辖区内,是青海省自然生态体系保存完好的重点区域。它地处祁连山南麓,湟水河、大通河和黄河穿境而过,地貌以河岸阶地和丘陵为主,植被多属干草原型,是青海省的主要农业区。常见动物有草兔(Lepus capensis)、朝鲜姬鼠(Apodemus peninsulae)、间颅鼠兔(Ochotona cansus)、阿拉善黄鼠(Citellus dauricus)、高原鼢鼠(Myospalax aspalax)、黑线姬鼠(A. agrarius)、小家鼠(Mus musculus)、褐家鼠(Rattus norvegicus)、小飞鼠(Pteromys volans)等,代表性的蚤种类有无棘鬃额蚤(Frontopsylla aspiniformis)、红羊新蚤(Neopsylla hongyangensis)、类新蚤(N. compar)、副规新蚤(N. paranoma)、多刺多毛蚤(Hystrichopsylla multidentata)、钩状单蚤(M. hamutus)等。
线粒体DNA具有结构简单、进化速率快和变异低等特点,目前成为有效分子标记被广泛应用于昆虫物种鉴定、物种遗传多样性及系统进化等研究[1]。DNA条形码技术是Hebert等[2]于2004年发展起来的一项利用线粒体细胞色素C氧化酶亚基Ⅰ(cytochrome c oxidase subunit Ⅰ,COⅠ)基因序列发展起来的一项分子生物学分类鉴定技术。Kumar等[3]应用DNA条形码技术对印度的15属63种蚊虫(包括主要疾病媒介在内)进行鉴定,成功鉴定了其中62种,成功率达到98.4%。Weeraratne等[4]对斯里兰卡9个地区的蚊虫进行了DNA条形码鉴定,表明DNA条形码可以识别传统形态学方法无法区分的受损标本。
迄今所知,全世界蚤类已累计发现约2 500余种(亚种),中国境内共有649种(亚种)[5],青海省有169种(亚种)[6]。马英等[7]对海东地区小型兽类做了分子生物学鉴定研究,修正了动物形态学部分鉴定错误,但该地区的寄生蚤种在传统形态学分类上仍存在很大困难,尤其在形态受损和亚种鉴定方面。因此,本文结合鼠疫疫源地调查和监测工作对海东市小型兽类体表寄生蚤的构成、宿主选择和分布开展研究,并对一些常见和重要蚤种提取了DNA条形码数据,作为今后该蚤种分子生物学鉴定的基础资料。
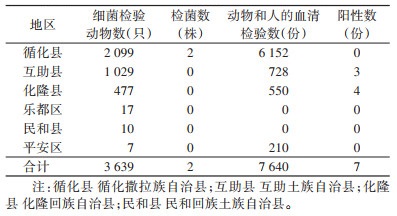
1 材料与方法 1.1 海东市鼠疫监测历史资料收集 1.1.1 细菌学和血清学检测历史资料收集结合海东市鼠疫自然疫源地调查和历年监测工作,整理了该市自判定为鼠疫自然疫源地以来鼠疫菌的检菌数和阳性数。
1.1.2 蚤类历史资料收集通过查阅公开发表的体表寄生蚤相关论文和动物媒介调查报告,收集了海东市蚤类记录。
1.2 常见蚤和疑难种的分类鉴定 1.2.1 实验材料将采自青海省的99份蚤样本用弯钩捡蚤镊轻轻摄出,置于解剖镜下,再用手术刀片将蚤腹部切开几个小切口。用QIAGEN DNA提取试剂盒提取DNA模板,并将DNA模板置于-20 ℃冰箱保存备用。留存蚤体几丁质外骨骼,制作成玻片凭证标本。
1.2.2 蚤种鉴定选择体型完整的蚤体制成玻片标本,依据《中国动物志昆虫纲蚤目》[5]进行外部形态学分类鉴定。
1.2.3 扩增方法PCR反应体系:10×buffer 2.5 μl,正、反向引物各0.5 μl,dNTPs 0.5 μl,模板1~2 μl,Taq DNA聚合酶0.25 μl,ddH2O补齐至25 μl。
扩增条件:94 ℃预变性5 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s,共10个循环;94 ℃变性30 s,50 ℃退火30 s,72 ℃延伸30 s,共30个循环。
选用文献[8]中引物(LepF:ATTCAACCAATCATAAAGATATTGG,LepR:TAAACTTCTGGATGTCCAAAAAATCA)扩增COⅠ目的基因,对无法获得测序结果的样本用蓝白斑筛选法构建克隆载体,用M13通用引物扩增阳性斑点,最后选取电泳目标条带清晰样本送交北京擎科生物科技股份有限公司测序。
1.2.4 COⅠ基因序列分析首先用Chromos软件观察测序峰图质量,拼接结果用Clustal_Ⅹ[9]软件进行序列比对,并结合峰图进行适当的手工调整。同时将测定的序列在美国国立生物技术信息中心(NCBI)上运行局部相似性基本查询工具(BLAST)程序进行序列同源性比对,同源性 > 95%时判定所测序列为研究目标序列。最后运用MEGA 7[10]软件中的Kimura-2-parameter(K2P)[11]模型计算各蚤种间遗传距离。并采用邻接法(neighbor-joining method,NJ法)构建COⅠ基因序列系统发育树,通过1 000次Bootstrap检验分析确定各分支置信度[12]。
2 结果 2.1 海东市鼠疫历史监测数据海东市位于青海省东部,所占面积甚小,多已开垦为耕地,是青海省的主要农业区。自1970年海东市首次于循化县岗察乡检出鼠疫耶尔森菌(鼠疫菌),被判定为鼠疫自然疫源地以来,20世纪80年代对该市进行了鼠疫疫源地调查和监测,均未检出相关阳性材料。1997年互助县五峰乡发生了一起迁入性人间腺鼠疫,2003年化隆县检出4份鼠疫阳性人血清,2012、2013年又在互助县检出3份旱獭鼠疫阳性血清[13],2014-2016年对互助县开展了连续3年的鼠疫监测,未检出阳性材料。该市自判定为鼠疫自然疫源地以来鼠疫菌的检菌数和阳性数等见表 1。
|
海东市调查发现蚤类61种和亚种,隶属于4总科5科28属(表 2),占比超过青海省蚤种总数量的1/3。其中,59种(亚种)属古北界青藏区成分,2种(亚种)属古北界蒙新区成分;古北界和东洋界广布种和兼有种10种,新记录种9种[14];海东市发现6种鼠疫媒介蚤:斧形盖蚤(Callopsylla dolabris)、谢氏山蚤(Oropsylla silantiewi)、细钩盖蚤(C. sparsilis)、直缘双蚤指名亚种(Amphipsylla tuta tuta)、红羊新蚤和人蚤(Pulex irritans)[6]。

|
本文选用海东市历史记录蚤种,尤其是一些传统形态学分类有疑义的蚤种扩增其COⅠ基因序列,共测得4科13属20种99条COⅠ序列,扩增片段有效长度约658 bp,所得序列经在线BLAST程序比对,证实扩增片段确为蚤COⅠ基因片段并提交GenBank。蚤凭证玻片标本保存于青海省地方病预防控制所,相关信息见表 3。
用MEGA 7软件分析序列组成,发现蚤COⅠ基因序列共658个位点,保守位点数234个,变异位点数424个,简约信息位点数392个,自裔位点数32个。A、T、C、G碱基的平均含量分别为26.9%、40.4%、16.9%和15.8%,A+T含量为67.3%,G+C含量为32.7%。
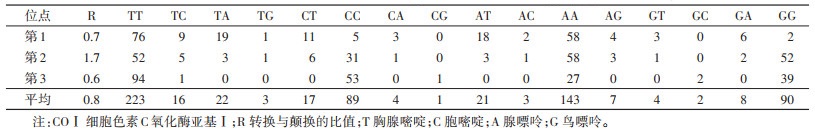
2.3.3 碱基替换使用MEGA 7软件,计算各位点的转换与颠换值及其比值,测得TC平均转换数为16,AG平均转换数为7,TC转换明显大于AG间的转换(表 4)。转换/颠换(R)的平均值为0.8,R < 2,该段序列转换与颠换并未达到饱和,COⅠ基因可靠度高。
|
经MEGA 7软件计算,海东市蚤种内遗传距离为0.2%~2.9%,种间遗传距离为3.3%~22.9%,种间差异是种内差异的12倍,且种间遗传距离最小值(3.3%)明显大于种内遗传距离最大值(2.9%)。
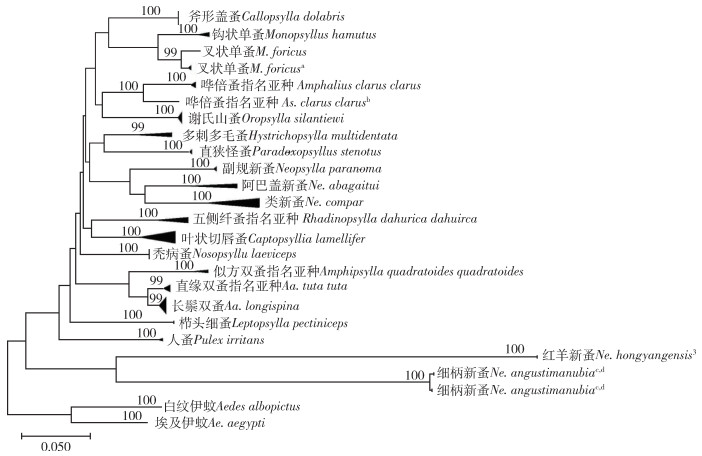
2.3.5 构建NJ系统树基于MEGA 7软件中K2P模型,利用NJ法构建海东市常见寄生蚤COⅠ基因序列的系统发育树(图 1)。发现所有序列在NJ系统发育树中形成了20个高支持率≥99%的分支,3种双蚤聚在1个大的分支,5种新蚤却聚在2个不同的分支。
|
| 注:a提示叉状单蚤存在隐种现象;b倍蚤属雌蚤,形态只鉴定到倍蚤属,结合分子生物学结果复鉴到种;c新蚤属的2个种聚在另一分支;d提示细柄新蚤存在隐种现象。 图 1 基于Kimura-2-parameter模型构建的青海省海东市蚤种邻接法系统发育树 Figure 1 A neighbor-joining phylogenetic tree based on the Kimura 2-parameter model of fleas from Haidong, Qinghai Province, China |
| |
海东市发现和记录蚤共计4总科5科28属61种和亚种,叶状切唇蚤和细钩双蚤属古北界蒙新区的成分,其余59种(亚种)属古北界青藏区的成分,人蚤、缓慢细蚤属广布蚤种,棒形新蚤、无规新蚤、甘肃栉眼蚤、獾副角蚤扇形亚种、鼯鼠盖蚤为两界兼有种,青海长喙蚤、孟达多毛蚤、多刺多毛蚤、喜马狭蚤、细柄新蚤、副规新蚤、短指新北蚤、具钩靴片蚤、宽指青海蚤、半圆茸足蚤、似方双蚤指名亚种、北山盖蚤、刷状同瘴蚤孟达亚种、叉状单蚤、钩状单蚤、新月单蚤为青藏高原特有种。海东蚤类虽在病蚤属、怪蚤属和双蚤属中有个别东洋界物种,但古北界青藏区成分仍居多数,具有黄土高原向青藏高原过渡镶嵌地带物种分布特征。
3.2 修订形态学分类结果海东市蚤种种间遗传距离最小值(3.3%)明显大于种内遗传距离最大值(2.9%),符合DNA条形码种内与种间无重叠的应用标准[15],提示基于COⅠ基因的条形码鉴定技术可作为海东市疑难蚤种鉴定的有力补充手段。本研究中,尽管选择了形态学特征比较明显的非透明蚤标本,但实际过程中仍出现了少量误鉴,尤其雌蚤鉴定错误较多。例如Z19(♀)、Z13(♀)镜检时被鉴定为细钩双蚤,Z049(♀)镜检为青海双蚤,但在分子聚类时却和长鬃双蚤聚为一支,置信度99%。ZF144(♀)镜检时为青海双蚤,分子聚类却和直缘双蚤指名亚种聚为一支,置信度99%。鉴于蚤类外部形态种类鉴别以雄性为主,再结合寄主、地区分布等因素,认为将上述4匹雌蚤分别复鉴为长鬃双蚤和直缘双蚤指名亚种比较合适。另外,编号36_09(♀)因其分类特征不甚明显,形态学鉴定时只鉴定到倍蚤属,聚类结果与哗倍蚤指名亚种聚在高置信度的同一分支,综合考虑了基因序列聚类结果和寄生宿主等因素,将其进一步鉴定为哗倍蚤指名亚种。编号ZF10(♀)镜检时为钩状单蚤,但聚类结果和钩状单蚤并不在同一分支,却和叉状单蚤聚在高置信度的同一支上,这2种蚤雌性外部形态特征差异较小,仅表现为第七腹板后突上翘程度和受精囊头部长短有差异,非透明标本鉴别更加不易,我们结合复鉴凭证标本及分子生物学结果,认为该蚤为叉状单蚤更为准确。综上说明,COⅠ基因和传统形态学分类相结合能更好地解决雌蚤的种类鉴别。
3.3 隐种发现NJ法系统树显示共有20个高支持率分支,说明蚤类应该有20种,这与凭证标本复核结果基本一致,验证了COⅠ基因作为蚤种分子鉴定的实用性。秃病蚤田鼠亚种和秃病蚤指名亚种在NJ法聚类图中聚在置信度99%的同一分支,考虑到这2个亚种差别很小,形态学分类本身也有很多歧义,因此,本研究中把这2个亚种合并到种级阶元,均鉴定为秃病蚤。另外,系统树中细柄新蚤和叉状单蚤又分为2个细小分支,置信度为100%。经复检透明标本后作者认为形态学鉴别无误,提示上述2个蚤种或许存在隐种现象。
3.4 存在问题聚类图中不同属的蚤种均聚在同一分支内,但新蚤属却分别聚在2个分支,第1分支内的副规新蚤、阿巴盖新蚤和类新蚤分支线较短,说明亲缘关系较近,而第2分支内的红羊新蚤和细柄新蚤分支线较长,说明亲缘关系较远。采集信息显示第1分支内的新蚤来自于海东市平安区、互助县、黄南藏族自治州尖扎县、海西蒙古族藏族自治州都兰县,第2分支内的新蚤采自于海南藏族自治州共和县、海北藏族自治州祁连县和果洛藏族自治州久治县。今后需要对上述地区的新蚤开展持续性调查和多基因标记研究,找出分歧结果的原因,相信会有更多新发现和合理解释。
利益冲突 无
| [1] |
赵广宇, 李虎, 杨海林, 等. DNA条形码技术在昆虫学中的应用[J]. 植物保护学报, 2014, 41(2): 129-141. Zhao GY, Li H, Yang HL, et al. Application of DNA barcoding in entomology: A review[J]. Acta Phytophy Sin, 2014, 41(2): 129-141. |
| [2] |
Hebert PDN, Cywinska A, Ball SL, et al. Biological identifications through DNA barcodes[J]. Proc Roy Soc B Biol Sci, 2003, 270(1512): 313-321. DOI:10.1098/rspb.2002.2218 |
| [3] |
Kumar NP, Rajavel AR, Natarajan R, et al. DNA barcodes can distinguish species of Indian mosquitoes (Diptera: Culicidae)[J]. J Med Entomol, 2007, 44(1): 1-7. DOI:10.1093/jmedent/41.5.01 |
| [4] |
Weeraratne TC, Surendran SN, Karunaratne SHPP. DNA barcoding of morphologically characterized mosquitoes belonging to the subfamily Culicinae from Sri Lanka[J]. Parasit Vectors, 2018, 11(1): 266. DOI:10.1186/s13071-018-2810-z |
| [5] |
吴厚永. 中国动物志·昆虫纲·蚤目[M]. 北京: 科学出版社, 2005: 97-133. Wu HY. Fauna Sinica, Insecta: Siphonaptera[M]. Beijing: Science Press, 2005: 97-133. |
| [6] |
王祖郧, 李超. 青海鼠疫[M]. 北京: 人民卫生出版社, 2016: 72-132. Wang ZY, Li C. Plague prevention and control in Qinghai[M]. Beijing: People's Medical Publishing House, 2016: 72-132. |
| [7] |
马英, 李海龙, 鲁亮, 等. DNA条形码技术在青海海东地区小型兽类鉴定中的应用[J]. 生物多样性, 2012, 20(2): 193-198. Ma Y, Li HL, Lu L, et al. Application for identification of small mammals by DNA barcoding in Haidong area, Qinghai province, China[J]. Biodiversity Sci, 2012, 20(2): 193-198. DOI:10.3724/SP.J.1003.2012.06188 |
| [8] |
Hajibabaei M, Janzen DH, Burns JM, et al. DNA barcodes distinguish species of tropical Lepidoptera[J]. Proc Natl Acad Sci USA, 2006, 103(4): 968-971. DOI:10.1073/pnas.0510466103 |
| [9] |
Thompson JD, Gibson TJ, Plewniak F, et al. The Clustal_X windows interface: Flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Res, 1997, 25(24): 4876-4882. DOI:10.1093/nar/25.24.4876 |
| [10] |
Kumar S, Stecher G, Tamura K. MEGA 7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Mol Biol Evol, 2016, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 |
| [11] |
Kimura M. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences[J]. J Mol Evol, 1980, 16(2): 111-120. DOI:10.1007/BF01731581 |
| [12] |
Felsenstein J. Confidence limits on phylogenies: An approach using the bootstrap[J]. Evolution, 1985, 39(4): 783-791. DOI:10.1111/j.1558-5646.1985.tb00420.x |
| [13] |
丛显斌, 刘振才, 李群. 中国鼠疫自然疫源地(1950-2014)[M]. 北京: 人民卫生出版社, 2019: 703-822. Cong XB, Liu ZC, Li Q. Natural foci of plague in China (1950-2014)[M]. Beijing: People's Medical Publishing House, 2019: 703-822. |
| [14] |
王国丽, 蔡理芸. 青海省蚤类新种和新亚种的地理分布[J]. 地方病通报, 1996, 11(4): 44-47. Wang GL, Cai LY. Zoogeographical distribution of the new species and subspecies of fleas in Qinghai province[J]. Endem Dis Bull, 1996, 11(4): 44-47. |
| [15] |
Hebert PDN, Ratnasingham S, De Waard JR. Barcoding animal life: Cytochrome c oxidase subunit 1 divergences among closely related species[J]. Proc Roy Soc B Biol Sci, 2003, 270 Suppl 1: S96-99. DOI:10.1098/rsbl.2003.0025 |
 2023, Vol. 34
2023, Vol. 34



