扩展功能
文章信息
- 章翔, 程蓉蓉, 刘路瑶, 孙萌, 洪宇航, 杨聪山, 柳燕, 殷宗俊, 徐前明
- ZHANG Xiang, CHENG Rong-rong, LIU Lu-yao, SUN Meng, HONG Yu-hang, YANG Cong-shan, LIU Yan, YIN Zong-jun, XU Qian-ming
- 安徽省金寨县大别山地区野生虻Tabanus hypomacros可作为非洲猪瘟病毒机械传播媒介初探
- Tabanus hypomacros: A suspected mechanical transmission vector of African swine fever virus in Dabie Mountain region of Jinzhai county, Anhui province in China
- 中国媒介生物学及控制杂志, 2022, 33(3): 326-330
- Chin J Vector Biol & Control, 2022, 33(3): 326-330
- 10.11853/j.issn.1003.8280.2022.03.002
-
文章历史
- 收稿日期: 2021-09-29
2 安徽医科大学基础医学院, 安徽 合肥 230032;
3 安徽省现代农业生猪产业技术体系, 安徽 合肥 230036
2 School of Basic Medicine, Anhui Medical University, Hefei, Anhui 230032, China;
3 Modern Agricultural Pig Industry Technology System in Anhui Province, Hefei, Anhui 230036, China
非洲猪瘟是一种由非洲猪瘟病毒(ASFV)引起的严重出血性疾病,不同年龄段的猪均易感,发病率和死亡率可高达100%[1-2]。高毒力株的ASFV感染可引起猪高热、食欲不振、皮肤和内脏出血等,甚至于2~10 d内急性死亡。非洲猪瘟传播途径复杂,其病毒媒介包括钝缘蜱属(Ornithodoros)软蜱,以及野猪和家猪等[3],致使病毒很难在猪群中消除。非洲猪瘟传入一个地区后,可引起猪群的高发病率和高死亡率并导致疫情迅速蔓延。另有研究报道,在部分流行地区,猪群急性感染死亡率虽已控制并下降,但亚急性或慢性ASFV感染变得更加频繁[4]。2018年8月3日,辽宁省报告了我国首例非洲猪瘟病例[5]。鉴于ASFV复杂的传播途径以及巨大的社会经济影响,ASFV的流行不仅对中国生猪产业构成巨大威胁,也对全球农业稳定构成威胁[6]。
非洲猪瘟的暴发不仅威胁生物安全性低的家庭养猪场,在生物安全性高的规模化养猪场也经常发生。然而,其确切的传播途径仍不完全清晰。相关研究表明临床亚急性感染、慢性感染或康复的猪有助于非洲猪瘟在流行区持续存在,零星的暴发也可将非洲猪瘟引入无病区[7]。
ASFV是一种虫媒DNA病毒,软蜱科钝缘蜱属是ASFV的天然宿主。ASFV可以在环境中持续数天[3],除了病媒传播和气溶胶传播外,还可通过被污染的衣服和鞋、设备和车辆等媒介传播[7-8]。ASFV在自然宿主和机械媒介之间可直接和间接传播[9],使其在环境中保持活跃的感染循环。ASFV可以在家猪之间传播[10],但野猪向家猪传播的可能性较低,野猪体表发现大量家猪钝缘蜱(O. porcinus)的若虫,证实软蜱是向家猪种群传播ASFV的重要媒介[8]。游走钝缘蜱(O. erraticus)在西班牙首次被确认为ASFV的生物媒介和储存库,该项研究促使研究人员发现了软蜱在非洲是家猪和野猪感染ASFV的来源[11],传播发生在血食期间,受感染的蜱可以长期保存病毒并将其传播给易感宿主。Chen等[12]报道从新疆维吾尔自治区采集的森林革蜱(Dermacentor silvarum)、银盾革蜱(D. niveus)中检测到ASFV特异性的核酸片段,并在进一步饲养的F1代银盾革蜱中检出ASFV核酸,认为革蜱也可以作为ASFV的贮藏宿主。研究表明ASFV也可通过吸血蝇(Stomoxys)叮咬传播,或意外摄入受感染的吸血蝇而传播给猪[13]。
虻(Tabanidae)是多种动物病毒的重要媒介,作为一种吸血媒介也有可能传播病毒[14]。然而,研究者对虻传播ASFV的信息知之甚少。本研究在安徽省金寨县大别山地区收集野生虻,探究安徽省大别山地区虻科食血昆虫在ASFV传播中的潜在作用,为该地区非洲猪瘟防控提供依据。
1 材料与方法 1.1 采样地点野生虻样本采集于安徽省金寨县(北纬31°39′,东经115°29′),该县面积216.5 km2,有15个村庄约45 000名居民,其中有部分家庭以小规模家庭猪场为生。2018年以来,该地区养殖户在委托第三方检测机构开展自检过程中发现零星疑似ASFV核酸检测阳性的猪并迅速开展了定点清除处理。
1.2 样本的采集及处理在居民区和森林交界生态区的3个不同地点用昆虫诱捕器采集昆虫。采获的昆虫被单只放置在含有75%乙醇溶液的2 ml离心管中,置于4 ℃冰箱储存,以供提取核酸样本。
1.3 形态学分类采获的昆虫在体视显微镜下进行形态学观察,利用触角作为分类的关键指标进行鉴定[15]。
1.4 遗传标记分类 1.4.1 DNA提取及细胞色素C氧化酶亚基Ⅰ(COⅠ)基因PCR扩增基于COⅠ基因作为样本昆虫的遗传标记分子,进一步对其鉴定分类。使用DNeasy®血液和组织试剂盒(QIAGEN,德国)从昆虫样品中提取基因组DNA。根据试剂盒说明,每个核酸样品用100 μl洗脱缓冲液中洗脱,并在-20 ℃储存。本研究基于昆虫的COⅠ基因,设计使用的引物序列为COⅠ-F: 5′-ATTCAACCAATCATAAAGATATTGG-3′,COⅠ-R:5′-TAAACTTCTGGATGTCCAAAAAAT-CA-3′[16]。以双翅目昆虫组织核酸样本为阳性对照,ddH2O为阴性对照。PCR反应体系25 μl,包含12.5 μl FastTaq Premix(TOLO生物技术有限公司),5.5 μl ddH2O,正、反向引物各1 μl,以及5 μl DNA模板。PCR扩增反应程序如下:94 ℃预变性1 min;前5个循环包括94 ℃变性30 s,45 ℃退火40 s,72 ℃延伸1 min,接下来30个循环包括94 ℃ 30 s,55 ℃ 40 s,72 ℃ 1 min;最后在72 ℃延伸10 min。
1.4.2 基于COⅠ基因的系统发育分析将纯化的PCR扩增产物送至北京擎科生物科技有限公司进行自动测序。测序所得的核苷酸序列分别用EditSeq 7.1软件进行序列分析,使用MEGA 6.0软件进行分子遗传进化分析。在遗传进化分析过程中选择最大似然法Kimura 2参数模型构建系统发育树,并以蹄斑斑虻(Chrysops dispar)(KC960724)和厩螫蝇(Stomoxys calcitrans)(KM243494)作为外群。
1.5 病毒的分子生物学鉴定 1.5.1 病毒基因组DNA提取及p72基因PCR扩增使用TaKaRa Mini BEST病毒RNA/DNA提取试剂盒Ver.5.0(TAKARA BIO INC,日本)提取病毒基因组DNA。基于世界动物卫生组织和中华人民共和国出入境检验检疫行业标准推荐的p72基因引物:p72-F 5′-ATGGATACCGAGGGAATAGC-3′,p72-R 5′- CTTACCGATGAAAATGATAC-3′[17]进行PCR扩增。以ASFV病毒核酸为阳性对照,ddH2O作阴性对照。PCR反应体系25 μl,包含12.5 μl FastTaq Premix(TOLO生物技术有限公司),5.5 μl ddH2O,正、反向引物各1 μl,以及5 μl DNA模板。基于p72基因PCR扩增反应程序如下:95 ℃ 10 min,35个循环包括94 ℃ 30 s,53 ℃ 30 s,72 ℃ 30 s;最后在72 ℃延长7 min。
1.5.2 基于p72基因的系统发育分析基于p72基因对病毒PCR产物进行测序并进行序列比对,与GenBank公布报道的病毒流行株核苷酸序列选择最大似然法Kimura-2参数模型构建系统发育树。
2 结果 2.1 形态学鉴定触角作为重要的分类学特征。虻的触角分为3个部分:柄节、梗节和鞭节。在虻中,基部的柄节扩大平坦,有或多或少发达的背侧齿,梗节和鞭节都很短。
在研究地点收集的样本昆虫在形态上符合Tabanus分类标准。见图 1。

|
| 图 1 野生虻标本的形态学特征(×40) Figure 1 Morphological characteristics of wild horsefly specimens (×40) |
| |
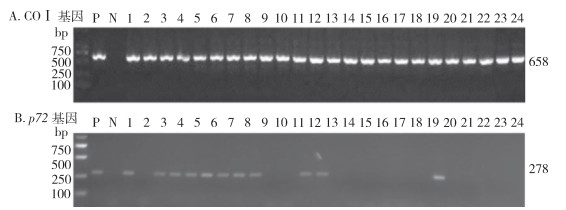
基于COⅠ基因和p72基因的PCR扩增结果见图 2。基于COⅠ基因和p72基因的PCR扩增,分别得到COⅠ基因特异性条带(658 bp)和ASFV特异性条带(278 bp)。
|
| 注:P阳性对照;N阴性对照;1~6为A采样地样本;7~16为B采样地样本;17~24为C采样地样本;COⅠ细胞色素C氧化酶亚基Ⅰ。 图 2 COⅠ基因和p72基因的PCR扩增结果 Figure 2 PCR amplification results of cytochrome c oxidase subunit I gene and p72 gene |
| |
3处采样地虻样本ASFV阳性检出情况不同,其中1处的6只虻中有5只阳性,另2处分别是10只虻中有5只阳性和8只中有1只阳性,ASFV的总阳性检出率为45.83%。
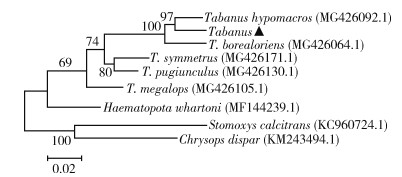
2.3 COⅠ基因的系统发育分析样本昆虫基于COⅠ基因进行PCR扩增并基于COⅠ基因用最大似然法建立其系统发育关系(图 3)。系统发育分析结果显示,本研究中的样本昆虫为T. hypomacros。
|
| 注:▲表示本研究中野生虻细胞色素C氧化酶亚基Ⅰ序列。 图 3 野生虻样本基于COⅠ基因的系统发育分析 Figure 3 Phylogenetic analysis of wild horsefly samples based on cytochrome c oxidase subunitⅠgene |
| |
将从金寨县收集的虻中获得的病毒PCR扩增产物基于p72基因进行系统发育分析,并对PCR产物基于p72基因的C端进行扩增和测序。核苷酸序列分析发现来自不同栖息地的样本昆虫中病毒基于ASFV p72基因的PCR产物基因测序结果几乎相同,该序列信息支持样本为ASFV。见图 4。
|
| 注:▲表示本研究中病毒毒株序列。 图 4 基于14条非洲猪瘟病毒p72基因序列的系统发育分析 Figure 4 Phylogenetic analysis of 14 p72 gene sequences of African swine fever virus |
| |
将样本序列与文献报道的基于p72基因的14株病毒流行株的序列进行比较和遗传进化分析,发现病毒基于p72基因聚成3个不同的进化谱系,每个谱系都局限于一个广泛的地理区域。系统发育关系显示,虻携带的病毒与ASFV中国流行株有很强的相关性。
3 讨论作为一种DNA病毒,ASFV在环境中具有极高的生存能力,其传播途径复杂,确定病毒传播媒介和保藏宿主及了解媒介和宿主之间的接触频率[8],有助于制定预防和缓解非洲猪瘟流行的策略。研究昆虫作为媒介对病毒的传播,可预测受影响地区疫病的流行趋势,同时有助于控制造成病毒传播的媒介因素。
已知虻是一些病原体的重要媒介。生态学研究表明,虻在靠近牧草和森林交错地带最为活跃[18]。然而,在ASFV传播方面,与其他食血性双翅目昆虫相比,虻很少受到关注[19]。在早期的研究中,在吸血蝇吸食含ASFV的血液48 h后,依旧能在吸血蝇体内检测到具有感染性的ASFV存在[20],De Liberato等[14]也证实在螫蝇(S. calcitrans)吸食含ASFV血液12 h后仍可检测到具有感染性的病毒。虻可以储存比螫蝇多5倍的血液[21],提示虻或许可以比螫蝇更持久地携带病毒。
相对于生物传播,机械传播是指病原体在传播过程中并没有在媒介中增殖和发展。决定虻可以机械传播的因素包括媒介虻及宿主的行为特征、病原体和感染程度。虻在血食期间转换宿主的进食习惯,可能是其作为机械载体传播病毒的一个重要因素。在吸食过程中,被吸食动物驱赶可能会导致虻的血食中断,转而在附近的其他动物身上吸食。这种吸食模式有助于ASFV的传播。本研究发现,与自检疑似发生非洲猪瘟的村庄距离不同的3处采样点,样本阳性检出情况与距离村庄远近相关,距离村庄远的采样点阳性样本数量少,这可能与虻的飞行能力有关。在宿主方面,ASFV对家猪的感染性极强。金寨县大别山地区家养黑猪作为ASFV的天然宿主,家猪黑色的毛色性状对虻更有吸引力。此外,病猪在ASFV急性感染阶段表现高热,大多数时间活动减少,使得虻可以在此期间不间断地进食。健康猪在采食过程中也可能会摄入虻,这增强了虻传播病毒的可能性。以上病媒及宿主的相关特性极大地增加了ASFV机械传播的风险。
采样区域地处金寨县大别山腹地,山高林密,加之近年来《野生动物保护法》的严格执行及各项保护措施的落实,当地野猪种群数量扩张迅猛,甚至经常出没于农户田舍周围。野猪作为ASFV的关键宿主,病毒可以在野猪种群中独立循环传播,也可以通过机械传播媒介传播至家猪种群。野猪的活动范围和虻的活动范围高度重叠,一旦野猪携带ASFV,虻的感染极有可能来源于野猪并由此传播至家猪种群。
在大多数地区,虻的活动具有很强的季节性,在本研究地区,虻高频活动发生在夏季,可能与该地区夏季非洲猪瘟流行呈相关性。虻作为ASFV载体是否可以进行生物传播,鉴于软蜱是传播ASFV的重要媒介,已有的研究表明,不同种类的蜱对ASFV有不同的传播能力[22],因此,我们未来的研究将集中于虻对ASFV的生物传播以及虻不同亚种传播ASFV能力是否不同。基于形态学和昆虫线粒体遗传标记COⅠ基因的鉴定方法已被证明可以有效区分许多双翅目物种[23]。
本研究的结果提示,该地区的T. hypomacros可以作为ASFV潜在的机械传播媒介,相对于生物传播,机械传播被认为是一种可以忽略不计的传播途径,但在某些情况下,机械传播可能与生物传播一样有效,其潜在影响难以准确评估或预测。
利益冲突 无
| [1] |
彭志锋, 卢建洲, 蒋增海, 等. 非洲猪瘟病毒疫苗的研究进展[J]. 中国兽医学报, 2020, 40(4): 859-863. Peng ZF, Lu JZ, Jiang ZH, et al. Progress and perspectives for development of African swine fever virus vaccines[J]. Chin J Vet Sci, 2020, 40(4): 859-863. DOI:10.16303/j.cnki.1005-4545.2020.04.33 |
| [2] |
Sánchez-Vizcaíno JM, Mur L, Gomez-Villamandos JC, et al. An update on the epidemiology and pathology of African swine fever[J]. J Comp Pathol, 2015, 152(1): 9-21. DOI:10.1016/j.jcpa.2014.09.003 |
| [3] |
Davies K, Goatley LC, Guinat C, et al. Survival of African swine fever virus in excretions from pigs experimentally infected with the Georgia 2007/1 isolate[J]. Transbound Emerg Dis, 2017, 64(2): 425-431. DOI:10.1111/tbed.12381 |
| [4] |
Fasina FO, Shamaki D, Makinde AA, et al. Surveillance for African swine fever in Nigeria, 2006-2009[J]. Transbound Emerg Dis, 2010, 57(4): 244-253. DOI:10.1111/j.1865-1682.2010.01142.x |
| [5] |
Zhou XT, Li N, Luo YZ, et al. Emergence of African swine fever in China, 2018[J]. Transbound Emerg Dis, 2018, 65(6): 1482-1484. DOI:10.1111/tbed.12989 |
| [6] |
Sánchez-Cordón PJ, Montoya M, Reis AL, et al. African swine fever: A re-emerging viral disease threatening the global pig industry[J]. Vet J, 2018, 233: 41-48. DOI:10.1016/j.tvjl.2017.12.025 |
| [7] |
Mur L, Martínez-López B, Sánchez-Vizcaíno JM. Risk of African swine fever introduction into the European Union through transport-associated routes: Returning trucks and waste from international ships and planes[J]. BMC Vet Res, 2012, 8: 149. DOI:10.1186/1746-6148-8-149 |
| [8] |
Golnar AJ, Martin E, Wormington JD, et al. Reviewing the potential vectors and hosts of African swine fever virus transmission in the United States[J]. Vector Borne Zoonotic Dis, 2019, 19(7): 512-524. DOI:10.1089/vbz.2018.2387 |
| [9] |
Olesen AS, Lohse L, Boklund A, et al. Transmission of African swine fever virus from infected pigs by direct contact and aerosol routes[J]. Vet Microbiol, 2017, 211: 92-102. DOI:10.1016/j.vetmic.2017.10.004 |
| [10] |
Gallardo C, Soler A, Nieto R, et al. Experimental infection of domestic pigs with African swine fever virus Lithuania 2014 genotype Ⅱ field isolate[J]. Transbound Emerg Dis, 2017, 64(1): 300-304. DOI:10.1111/tbed.12346 |
| [11] |
Plowright W, Parker J, Peirce MA. African swine fever virus in ticks (Ornithodoros moubata, Murray) collected from animal burrows in Tanzania[J]. Nature, 1969, 221(5185): 1071-1073. DOI:10.1038/2211071a0 |
| [12] |
Chen Z, Xu XF, Wang YF, et al. DNA segments of African swine fever virus detected for the first time in hard ticks from sheep and bovines[J]. Syst Appl Acarol, 2019, 24(1): 180. DOI:10.11158/saa.24.1.13 |
| [13] |
Olesen AS, Hansen MF, Rasmussen TB, et al. Survival and localization of African swine fever virus in stable flies (Stomoxys calcitrans) after feeding on viremic blood using a membrane feeder[J]. Vet Microbiol, 2018, 222: 25-29. DOI:10.1016/j.vetmic.2018.06.010 |
| [14] |
De Liberato C, Magliano A, Autorino GL, et al. Seasonal succession of tabanid species in equine infectious anaemia endemic areas of Italy[J]. Med Vet Entomol, 2019, 33(3): 431-436. DOI:10.1111/mve.12360 |
| [15] |
Al-Talafha H, Yaakop SB, Ghani IBA. Taxonomic notes and new records of the genus Tabanus Linnaeus 1758 (Diptera: Tabanidae) from Malaysia[J]. AIP Conf Proc, 2016, 1784(1): 060014. DOI:10.1063/1.4966852 |
| [16] |
Hebert PDN, Penton EH, Burns JM, et al. Ten species in one: DNA barcoding reveals cryptic species in the neotropical skipper butterfly Astraptes fulgerator[J]. Proc Natl Acad Sci USA, 2004, 101(41): 14812-14817. DOI:10.1073/pnas.0406166101 |
| [17] |
中华人民共和国国家质量监督检验检疫总局. SN/T 1559-2010非洲猪瘟检疫技术规范[S]. 北京: 中国标准出版社, 2010. General Administration of Quality Supervision, Inspection and Quarantine of the People's Republic of China. SN/T 1559-2010 Protocol of quarantine for African swine fever[S]. Beijing: Standards Press of China, 2010. (in Chinese) |
| [18] |
Baldacchino F, Gardès L, De Stordeur E, et al. Blood-feeding patterns of horse flies in the French Pyrenees[J]. Vet Parasitol, 2014, 199(3/4): 283-288. DOI:10.1016/j.vetpar.2013.10.009 |
| [19] |
Baldacchino F, Desquesnes M, Mihok S, et al. Tabanids: Neglected subjects of research, but important vectors of disease agents![J]. Infect Genet Evol, 2014, 28: 596-615. DOI:10.1016/j.meegid.2014.03.029 |
| [20] |
Mellor PS, Kitching RP, Wilkinson PJ. Mechanical transmission of capripox virus and African swine fever virus by Stomoxys calcitrans[J]. Res Vet Sci, 1987, 43(1): 109-112. DOI:10.1016/S0034-5288(18)30753-7 |
| [21] |
Salem A, Franc M, Jacquiet P, et al. Feeding and breeding aspects of Stomoxys calcitrans (Diptera: Muscidae) under laboratory conditions[J]. Parasite, 2012, 19(4): 309-317. DOI:10.1051/parasite/2012194309 |
| [22] |
De Oliveira RP, Hutet E, Lancelot R, et al. Differential vector competence of Ornithodoros soft ticks for African swine fever virus: What if it involves more than just crossing organic barriers in ticks?[J]. Parasit Vectors, 2020, 13(1): 618. DOI:10.1186/S13071-020-04497-1 |
| [23] |
Cywinska A, Hannan MA, Kevan PG, et al. Evaluation of DNA barcoding and identification of new haplomorphs in Canadian deerflies and horseflies[J]. Med Vet Entomol, 2010, 24(4): 382-410. DOI:10.1111/j.1365-2915.2010.00896.x |
 2022, Vol. 33
2022, Vol. 33


