扩展功能
文章信息
- 范笑忱, 刘璐瑶, 方瑜, 褚凌渺, 冯蕊, 李飞燕, 潘若兮, 王少圣, 孙恩涛
- FAN Xiao-chen, LIU Lu-yao, FANG Yu, CHU Ling-miao, FENG Rui, LI Fei-yan, PAN Ruo-xi, WANG Shao-sheng, SUN En-tao
- 范张食酪螨和腐食酪螨的形态学与分子生物学鉴定
- Morphological and molecular biological identification of Tyrophagus fanetzhangorum and T. putrescentiae
- 中国媒介生物学及控制杂志, 2022, 33(2): 277-280
- Chin J Vector Biol & Control, 2022, 33(2): 277-280
- 10.11853/j.issn.1003.8280.2022.02.021
-
文章历史
- 收稿日期: 2021-11-24
食酪螨属(Tyrophagus)属于蜱螨亚纲(Acari)、真螨总目(Acariformes)、粉螨科(Acaridae)。食酪螨属包括约35种螨类,大多数食酪螨呈世界性分布,存在于自然界和人类的栖息地,是危害储藏物最常见的螨类之一,也是室内过敏原的主要来源之一,可导致人类皮炎和呼吸道过敏[1]。食酪螨形态特征十分接近,难以辨别[2],现食酪螨属部分螨种的分类及形态特征的鉴定存在一定争议[3]。
范张食酪螨(Tyrophagus fanetzhangorum)和腐食酪螨(T. putrescentiae)在外部形态上相似,差异微小,早期国外研究中仅存在两者形态鉴定简单的文字描述和电镜下的特征照片[4-5],未见两者光镜下的显微鉴定照片,对范张食酪螨的表型特征描写也较少,而文字很难描述标本的实际情况,电镜观察对设备要求较高,导致准确鉴定种类十分困难,因此需完善两种近缘螨的形态学鉴定方法,补充光镜照片,使形态学鉴定更便捷准确。目前粉螨种类鉴定主要依据外部形态特征,但对于螨虫的早期形态(卵、幼螨和若螨)、肢体残缺标本及近缘种等较难通过形态鉴定物种[6-7];而应用分子标记鉴定螨类,提高了螨类在系统学和分类鉴定上的可靠性[8],可弥补近缘种形态学鉴定时面临的困难。线粒体细胞色素C氧化酶亚基Ⅰ(COⅠ)基因具有明显的种间差异、较低的种内变异等特性[9],适合研究亲缘关系较近的种间系统发育关系。
本研究对从储藏物分离获得的范张食酪螨和腐食酪螨进行形态学特征分析,并提供光镜下2种螨的整体和局部照片,丰富了其形态学鉴定依据,同时利用COⅠ基因对范张食酪螨和腐食酪螨进行快速准确鉴别。
1 材料与方法 1.1 样本采集、分离和形态鉴定2019年5-7月,从安徽省合肥和铜陵地区久置储藏物中分别采集面粉、大米、稻谷、玉米、饲料、干果等储藏物样本各1~10 g。从样本中挑取单个成螨,进行纯培养。从培养标本中挑取10只以上成螨经蒸馏水清洗,干燥后用封固液固定,制成玻片标本,在高倍镜下进行形态学鉴定[3-5]。其余食酪螨保存在浓度为75%的乙醇溶液中,用于提取基因组DNA。
1.2 DNA提取、引物设计和PCR扩增、测序参照本实验室单只螨基因组DNA的提取方法[10];将成功提取的基因组DNA样本储存于-20 ℃冰箱中备用。扩增引物序列根据参考文献[11]设计:LCO1490 5'-GGTCAACAAATCATAAAGATATTGG-3',HC02198 5'-TAAACTTCAGGGTGACCAAAAAATCA-3'。PCR扩增获得的产物用1%琼脂糖凝胶电泳检测后,送至生工生物工程(上海)股份有限公司测序。
1.3 序列分析将所获序列在美国国立生物技术信息中心网站用BLAST软件进行相似性比对,结合GenBank中食酪螨的基因序列,使用Clustal Ⅹ 1.83软件进行多序列比对,运用MEGA Ⅹ软件进行数据分析,以粗脚粉螨(Acarus siro)作为外群,使用最大似然法构建系统发育树。
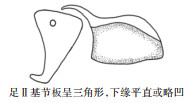
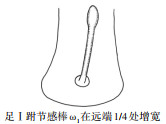
2 结果 2.1 形态学鉴定按照食酪螨属螨类检索表[3-5],根据制作标本的形态学特征鉴定,所采集到的螨类为粉螨科食酪螨属的范张食酪螨与腐食酪螨。2种螨玻片标本在高倍镜下进行观察,发现范张食酪螨足Ⅱ基节板后缘1/3处明显向内凹陷,下缘曲线近似“S”形。腐食酪螨足Ⅱ基节板呈三角形,下缘平直或略凹。此外,范张食酪螨足Ⅰ跗节感棒ω1在远端2/3处明显增宽,而腐食酪螨足Ⅰ跗节感棒ω1在远端1/4处增宽(表 1、图 1)。此外,2种螨的鉴别要点在雌、雄螨中均能观察到。
| 特征部位 | 范张食酪螨 | 腐食酪螨 |
| 足Ⅱ基节 | 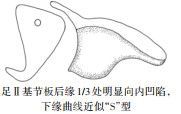
|
|
| 足Ⅰ跗节上的感棒ω1 | 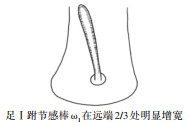
|
|

|
| 注:a范张食酪螨(×200);b腐食酪螨(×200);c范张食酪螨足Ⅰ跗节上感棒ω1(×400);d腐食酪螨足Ⅰ跗节上感棒ω1(×400);e图中箭头所指为范张食酪螨足Ⅱ基节(×400);f图中箭头所指为腐食酪螨足Ⅱ基节(×400)。 图 1 范张食酪螨与腐食酪螨的形态学差异 Figure 1 Morphological differences between Tyrophagus fanetzhangorum and T. putrescentiae |
| |
经PCR扩增后所获2种螨的COⅠ基因序列均为661 bp,经BLAST对比分析,所测得的范张食酪螨COⅠ基因序列与GenBank中收录的范张食酪螨COⅠ基因序列相似性在99.00%以上。同时,所测得的腐食酪螨COⅠ基因序列也与GenBank中收录的腐食酪螨COⅠ基因序列相似性在99.00%以上。
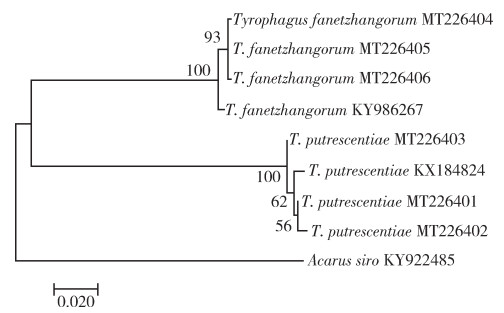
基于GenBank已报道范张食酪螨(登录号:KY986267)、腐食酪螨(登录号:KX184824)和粗脚粉螨(登录号:KY922485)的线粒体COⅠ基因序列,构建最大似然法系统发育树(表 2、图 2);范张食酪螨与腐食酪螨各自聚为一支,支持率达100%,可明显区分,COⅠ在范张食酪螨与腐食酪螨2种间具有一定差异,能用于鉴别这2个近缘种。
| 螨种 | 采样地区 | GenBank登录号 |
| 范张食酪螨 | 中国安徽合肥 | MT226404-MT226406 |
| 西班牙 | KY986267 | |
| 腐食酪螨 | 中国安徽铜陵 | MT226401-MT226403 |
| 中国陕西西安 | KX184824 |
|
| 图 2 基于COⅠ基因的范张食酪螨与腐食酪螨的最大似然法系统发育树 Figure 2 Phylogenetic tree of Tyrophagus fanetzhangorum and T. putrescentiae based on CO Ⅰ gene sequences using maximum likelihood method |
| |
范张食酪螨与腐食酪螨主要分布于储藏食品、面粉和房舍灰尘中,对人体和人类生活环境有害;食酪螨作为粉螨科中常见的螨类,在我国储藏物中分布广泛、数目众多,导致经济和营养价值遭到破坏[12]。准确地鉴定食酪螨有利于科学研究和生物防治的开展。
范张食酪螨与腐食酪螨种间关系近,形态相似,以往的分类和命名中存在争议,因此正确地区分这2种螨是对其进行科学研究的前提。现有文献对腐食酪螨的研究和形态描述较为详尽,但范张食酪螨形态鉴定及分类的研究较少,且缺少光镜下的显微照片,对螨种类的快速鉴别带来不便。本研究通过在高倍镜下对范张食酪螨和腐食酪螨的形态进行观察、比较与拍摄,提供了2种食酪螨外部形态的实物图和线条图,可直观表现出2种螨在足Ⅱ基节的形态、足Ⅰ跗节感棒ω1的形状方面的差异,进而作为2个近缘螨种的鉴别要点,为范张食酪螨提供光镜下更方便和快速的形态学鉴定依据。
单纯依据形态学鉴定螨类时,可能会受发育阶段、标本完整性等因素约束,从而影响结果的准确性。DNA分子标记技术可以克服上述条件的限制,进行螨类的鉴定[13-14],是形态学鉴定的有力补充。郑凌霄等[10]对粉螨科7种样本进行COⅠ基因分析,成功鉴定到种,与形态学鉴定结果一致。江亚杰等[15]验证COⅠ基因片段序列可以对储粮害虫达到种水平的区分,实现有关种类的快速、准确鉴定。本研究选用COⅠ基因作为分子标记对范张食酪螨和腐食酪螨进行鉴定,将所获的食酪螨COⅠ基因片段序列与GenBank中已发表的序列进行BLAST比对,比对结果与形态学鉴定结果一致。使用MEGA Ⅹ构建的最大似然法进化树,显示本实验所得范张食酪螨和腐食酪螨的COⅠ基因序列与自GenBank下载的对应物种序列各自聚为一支,支持率为100%,因此线粒体COⅠ基因作为分子标记能很好地鉴定范张食酪螨与腐食酪螨,且分子生物学鉴定与形态学鉴定结果相一致,从而为形态学研究提供有效的保障。
综上,本研究对范张食酪螨和腐食酪螨的形态鉴别要点进行了比较分析。同时,本研究以COⅠ基因作为分子标记,对2种螨基于线粒体COⅠ基因进行分子生物学鉴定,且与形态学鉴定相互验证,提高螨类近缘种鉴定的准确性,弥补形态学鉴定的不足。
利益冲突 无
| [1] |
Matos-Semedo F, Cruz C, In cio F, et al. House dust mite (HDM) and storage mite (SM) molecular sensitisation profiles and association with clinical outcomes in allergic asthma and rhinitis: Protocol for a systematic review[J]. BMJ Open, 2021, 11(7): e046519. DOI:10.1136/bmjopen-2020-046519 |
| [2] |
Fan QH, Zhang ZQ. Revision of some species of Tyrophagus (Acari: Acaridae) in the oudemans collection[J]. Syst Appl Acarol, 2007, 12(3): 253-280. DOI:10.11158/saa.12.3.11 |
| [3] |
Murillo P, Klimov P, Hubert J, et al. Investigating species boundaries using DNA and morphology in the mite Tyrophagus curvipenis (Acari: Acaridae), an emerging invasive pest, with a molecular phylogeny of the genus Tyrophagus[J]. Exp Appl Acarol, 2018, 75(2): 167-189. DOI:10.1007/s10493-018-0256-9 |
| [4] |
Klimov PB, OConnor BM. Conservation of the name Tyrophagus putrescentiae, a medically and economically important mite species (Acari: Acaridae)[J]. Int J Acarol, 2009, 35(2): 95-114. DOI:10.1080/01647950902902587 |
| [5] |
Fan QH, Zhang ZQ. Tyrophagus (Acari: Astigmata: Acaridae)[M]. Lincoln, New Zealand: Manaaki Whenua Press, 2007: 14-17.
|
| [6] |
Tixier MS, Dennj P, Douin M, et al. Mites of the genus Typhlodromus (Acari: Phytoseiidae) from southern France: Combined morphological and molecular approaches for species identification[J]. Zootaxa, 2019, 4604(2): 242-280. DOI:10.11646/zootaxa.4604.2.2 |
| [7] |
Zhao YE, Zhang WY, Wang RL, et al. Divergent domains of 28S ribosomal RNA gene: DNA barcodes for molecular classification and identification of mites[J]. Parasit Vectors, 2020, 13(1): 251. DOI:10.1186/s13071-020-04124-z |
| [8] |
易忠权, 夏伟, 赵盼雯, 等. DNA标记技术在螨类系统学研究中的应用[J]. 现代生物医学进展, 2017, 17(31): 6196-6200. Yi ZQ, Xia W, Zhao PW, et al. DNA markers in the systematic study of mites[J]. Progr Mod Biomed, 2017, 17(31): 6196-6200. DOI:10.13241/j.cnki.pmb.2017.31.046 |
| [9] |
吴太葆. 基于形态特征和COⅠ基因的粉螨重要类群系统发育研究(蜱螨亚纲: 粉螨亚目)[D]. 南昌: 南昌大学, 2007: 41-45. Wu TB. Studies on phylogenetic relationships among some important groups of acaroid mites based on morphological characters and COⅠ gene (Acari: Acaridida)[D]. Nanchang: Nanchang University, 2007: 41-45. (in Chinese) |
| [10] |
郑凌霄, 尹灿灿, 王逸枭, 等. 基于DNA条形码技术的粉螨种类鉴定研究[J]. 中国媒介生物学及控制杂志, 2019, 30(2): 180-184. Zheng LX, Yin CC, Wang YX, et al. Identification of acaroid mite species based on DNA barcoding[J]. Chin J Vector Biol Control, 2019, 30(2): 180-184. DOI:10.11853/j.issn.1003.8280.2019.02.015 |
| [11] |
Folmer O, Black M, Hoeh W, et al. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit Ⅰ from diverse metazoan invertebrates[J]. Mol Mar Biol Biotechnol, 1994, 3(5): 294-299. |
| [12] |
Cui YB. When mites attack: Domestic mites are not just allergens[J]. Parasit Vectors, 2014, 7: 411. DOI:10.1186/1756-3305-7-411 |
| [13] |
范成. 无损DNA提取技术以及分子鉴定在甲螨中的应用研究[D]. 保定: 河北大学, 2020. Fan C. Study on non-destructive DNA extraction method and molecular identification technology in oribatid mites[D]. Baoding: Hebei University, 2020. (in Chinese) |
| [14] |
王逸枭, 郑凌霄, 管国宇, 等. 居室内检获的羽美绥螨形态和DNA条形码鉴定[J]. 中国媒介生物学及控制杂志, 2018, 29(6): 38-41. Wang YX, Zheng LX, Guan GY, et al. Morphological and DNA barcoding identification of the indoor Ameroseius plumosus[J]. Chin J Vector Biol Control, 2018, 29(6): 38-41. DOI:10.11853/j.issn.1003.8280.2018.06.007 |
| [15] |
江亚杰, 汪中明, 张涛, 等. 基于DNA条形码技术的储粮害虫碎片鉴定研究[J]. 中国粮油学报, 2017, 32(8): 131-135. Jiang YJ, Wang ZM, Zhang T, et al. Molecular identification of stored grain pest fragments by DNA barcoding[J]. J Chin Cereals Oils Assoc, 2017, 32(8): 131-135. DOI:10.3969/j.issn.1003-0174.2017.08.021 |
 2022, Vol. 33
2022, Vol. 33


