扩展功能
文章信息
- 王秋慧, 王浩城, 韩谦, 廖承红
- WANG Qiu-hui, WANG Hao-cheng, HAN Qian, LIAO Cheng-hong
- 埃及伊蚊6个丝氨酸蛋白酶(CLIPs)基因生物信息学分析及功能的初步研究
- Bioinformatics analysis and preliminary functional study of six CLIPs genes in Aedes aegypti
- 中国媒介生物学及控制杂志, 2022, 33(2): 183-190
- Chin J Vector Biol & Control, 2022, 33(2): 183-190
- 10.11853/j.issn.1003.8280.2022.02.004
-
文章历史
- 收稿日期: 2021-12-23
丝氨酸蛋白酶(clip-domain serine proteases,CLIPs)是存在于昆虫和其他节肢动物血淋巴中的非消化性蛋白酶,是昆虫先天免疫反应的关键组成部分。它们由1个或多个氨基端clip结构域、1个连接子序列和1个羧基端具有催化活性的CLIPs结构域组成。相反,一些CLIPs有一个羧基端非催化蛋白酶样结构域,不具有催化作用,它们被称为丝氨酸蛋白酶同源物(clip-domain containing serine protease homologues,CSPHs)。CLIPs基因在昆虫中进化分为5个分支(CLIPA、CLIPB、CLIPC、CLIPD、CLIPE)[1],每个分支在昆虫基因组中都是多基因家族。由CLIPs组成的级联调节激活系统对昆虫中先天免疫反应激活起着调节作用。
CLIPs在昆虫先天免疫反应中具有重要的调节功能,包括细胞因子Spatzle的蛋白水解激活,以形成活性的Toll配体,从而合成抗菌肽,以及黑化反应所需的酚氧化酶原(prophenoloxidase,PPO)的特异性激活。在已确定的免疫途径中,微生物表面分子刺激起始模块化CLIPs的激活,然后激活CLIPC,进而激活CLIPB。然后,活性CLIPB切割并激活效应分子。CLIPA属于CLIPs同源物,不具有蛋白水解活性,但有些可以作为其他CLIPs活性的调节因子。CLIPs广泛存在于昆虫中,并参与了许多重要的生理过程[2]。一旦被激活,它们就会受到血淋巴中的CLIPs抑制剂(serine protease inhibitor,SRPN)的调节[3-6]。
埃及伊蚊(Aedes aegypti)是基孔肯雅热、黄热病和登革热等蚊媒传染病的主要传播媒介,全球成千上万人感染蚊媒传染病,造成极大的经济损失,埃及伊蚊的防控已经成为全球关注的一个问题[7]。目前,在埃及伊蚊中已被发现的82个CLIPs基因中有65个属于CLIPB、CLIPC和CLIPD。CLIPA和CLIPE有17个成员[1]。但是,关于埃及伊蚊CLIPs在先天免疫调节中的功能还知之甚少。本研究中对文献中已发现埃及伊蚊的6个CLIPs进行先天免疫调控功能探索,通过生物信息学的方法进行分析,结合时空表达谱构建,成蚊细菌侵染,研究这6个CLIPs基因的mRNA转录水平变化。为埃及伊蚊部分CLIPs基因功能研究提供基础,有助于更好地理解埃及伊蚊免疫调节机制,进而为病媒生物防控提供潜在靶标。
1 材料与方法 1.1 材料来源埃及伊蚊(Rockfeller株)来自军事科学院军事医学研究院微生物流行病研究所。
1.2 方法 1.2.1 埃及伊蚊6个CLIPs基因生物信息学分析在美国国立生物技术信息中心(NCBI)网站上(https://www.ncbi.nlm.nih.gov/)进行搜索,获取并下载了埃及伊蚊6个CLIPs的mRNA序列和氨基酸序列,这6个CLIPs与其他物种已研究的CLIPs在进化关系上相近,具有一定的研究意义[8]。利用在线工具SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)对埃及伊蚊CLIPs的二级结构进行预测;使用在线软件Clustal Omega(https://www.ebi.ac.uk/Tools/msa/clustalo/)对不同昆虫的CLIPs氨基酸序列进行多序列比对。将全部CLIPs氨基酸序列导入MEGA 7.0软件,经序列比对后,导出为MEGA 7.0软件识别的meg格式,采用邻接法对氨基酸序列构建进化树。其中Bootstrap设为1 000,其余参数采用默认设置,分析不同昆虫CLIPs的进化关系。
1.2.2 埃及伊蚊6个CLIPs基因的时空表达谱构建从埃及伊蚊卵(产卵后3 d)、1~4龄期幼虫、白色和黑色雌蛹、白色和黑色雄蛹(白蛹化蛹后30 min,黑蛹变色后30 min)、雌、雄成虫时期(羽化后3 d)采集样品;从雌性成虫的马氏管、胸部、脂肪体、卵巢和中肠中采集组织样品。每个组织样品大约从30只蚊虫中收集,每个时期样品采集1、2龄幼虫30~50只,其他虫态时期5~10只。采用Trizol法提取埃及伊蚊不同样品的总RNA,用1%的琼脂糖凝胶电泳验证RNA的完整性。每个阶段和组织样本的收集和RNA提取均进行3次重复实验。使用PrimeScript RT reagent Kit(TAKARA,No. RR047A)合成模板cDNA。在LightCycler 480系统中使用SYBR green Master I(Roche)配置反应体系,根据制造商的说明进行,并遵循以下循环条件:95 ℃变性30 s,95 ℃变性5 s,60 ℃变性30 s,进行40个循环。以埃及伊蚊RPS17作为内参基因,每个样品进行3次重复实验。相对基因表达数据采用relative 2-ΔΔct方法[9]进行分析。实验中所使用的引物见表 1。

|
使用革兰阴性菌大肠埃希菌(Escherichia coli,DH5α)和革兰阳性菌金黄色葡萄球菌(Staphylococcus aureus,ATCC43300)进行成蚊侵染。将2种菌分别在37 ℃的Luria-Bertani液体培养基中培养,以600 nm处的吸光度读数监测其生长情况,直至吸光度值达到1左右,离心半径7.7 cm,12 000 r/min,离心3 min收集菌体,观察细菌收集情况。然后将这些颗粒再悬浮在焦炭酸二乙酯(DEPC)处理后的水中,多次离心漂洗,得到最终的大肠埃希菌细胞悬液2.00 mg/ml和金黄色葡萄球菌细胞悬液0.80 mg/ml。将成年雌蚊放入-20 ℃冰箱中进行冷冻麻醉3 min,后放入培养皿中并置于冰上,将上述2种细菌悬液,用油压手动微量注射装置(Eppendorf AG,汉堡,德国)注射至已麻醉成蚊胸腔内,每只约注射1.50 μl,直至成蚊腹部轻微膨胀。每种细菌注射30只雌蚊,同时用DEPC处理后的水作为对照。共进行3次重复实验。收集在细菌感染6、12、24和36 h后存活的成蚊的样本后,进行总RNA提取,合成cDNA,并进行实时荧光定量PCR(real time quantitative PCR,RT-qPCR)检测埃及伊蚊6个CLIPs基因的mRNA表达水平变化。共进行3次重复实验。
1.3 统计学分析使用LightCycler96软件将RT-qPCR导出至Excel 2016软件,采用相对2-ΔΔct方法对相关基因表达数据进行分析。使用GraphPad Prism软件6.04版完成所有统计分析,采用one-way ANOVA检验方法确定对照组和处理组在mRNA表达水平方面的差异。P < 0.05为具有统计学意义。
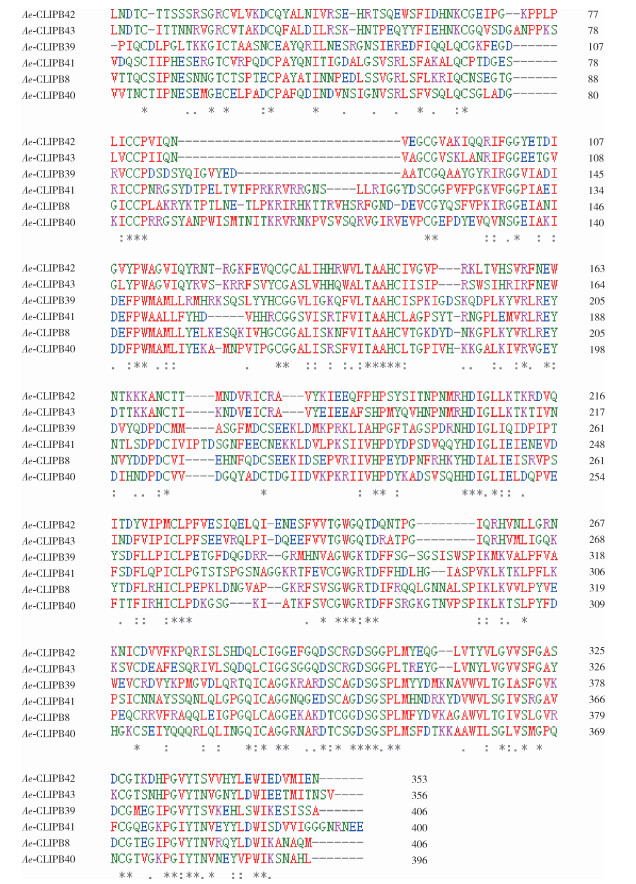
2 结果 2.1 埃及伊蚊6个CLIPs氨基酸序列分析以及二级结构预测在NCBI数据库中搜索并预测了埃及伊蚊CLIPB8、CLIPB39、CLIPB40、CLIPB41、CLIPB42、CLIPB43共6个CLIPs的二级结构。埃及伊蚊6个CLIPs的二级结构主要包括α-螺旋、延伸链以及无规卷曲,同时还存在少量的β-转角(表 2)。其中无规卷曲占比较高,占二级结构组成的53.82%~62.50%,Ae-CLIPB41的无规卷曲比例最高,为62.50%;Ae-CLIPB42的无规卷曲比例最低,为53.82%。每个蛋白均含有α-螺旋,其中Ae-CLIPB39的α-螺旋占17.98%,是6个CLIPs中占比最高的;Ae-CLIPB41的α-螺旋占比最少,仅有14.50%。埃及伊蚊6个CLIPs的二级结构中,β-转角相对较少,其中Ae-CLIPB41的β-转角占比最少,为2.75%,Ae-CLIPB40的β-转角最高,为4.29%。应用在线软件Clustal Omega软件对埃及伊蚊6个CLIPs成员的氨基酸序列进行相似性比对[10],埃及伊蚊6个CLIPs基因家族之间相似性较低,其氨基酸序列的相似性为38.32%(146/381)(图 1),保守区主要集中在羧基端的蛋白酶结构域。

|
|
| 注:使用Clustal Omega对6个丝氨酸蛋白酶进行多序列比对;根据序列相似性≥50%进行比对,其中“*”为高度保守区域(100%相似), “:”为中度保守区域(80%~99%相似),“.”为低度保守区域(50%~80%相似);氨基酸序列的相似性为38.32%(146/381, 图中带有标记的氨基酸个数与氨基酸总数的比值)。 图 1 埃及伊蚊6个丝氨酸蛋白酶保守结构域序列比对 Figure 1 Alignment of conserved domains of six CLIPs in Aedes aegypti |
| |
利用MEGA7软件对埃及伊蚊6个CLIPs基因构建系统进化树(图 2)。这几类昆虫的CLIPs基因位于不同的分支,其中埃及伊蚊6个CLIPs基因分为2个分支,Ae-CLIPB8、Ae-CLIPB39、Ae-CLIPB40和Ae-CLIPB4Ⅰ在同一分支;Ae-CLIPB42和Ae-CLIPB43在同一分支;在前一分支中,Ae-CLIPB8和白纹伊蚊PPAF1的遗传距离最近,同源性最高,二者合并为1个分支,之后与冈比亚按蚊(Anopheles gambiae)CLIPB8合并,最后依次与Ae-CLIPB40、Ae-CLIPB41、Ae-CLIPB39和黑腹果蝇(Drosophila melanogaster)SP5合并为1个大分支;在另一分支中,Ae-CLIPB42和Ae-CLIPB43合并为1个分支,之后依次与An-CLIPB15和An-CLIPB14合并为1个分支。

|
| 注:▲表示本研究中埃及伊蚊6个丝氨酸蛋白酶基因。 图 2 埃及伊蚊6个丝氨酸蛋白酶基因系统进化树 Figure 2 Phylogenetic tree of six CLIPs genes in Aedes aegypti |
| |
埃及伊蚊6个CLIPs基因的表达模式在各发育时期都不同。设定6个CLIPs基因的mRNA表达量在1龄幼虫时期为1,以埃及伊蚊RPS17作为内参基因。6个埃及伊蚊CLIPs基因在埃及伊蚊的各个时期各个组织中均有表达。其中Ae-CLIPB8在幼虫期表达量丰富,在4龄幼虫期表达量最高(F=82.780,P < 0.001),在2龄幼虫表达量次之,均高于其他生长时期;同样的Ae-CLIPB39在4龄幼虫表达量最高(F=82.910,P < 0.001),在雌性成蚊表达量次之,均高于其他生长时期。Ae-CLIPB40在雌性成蚊中表达量最高,在4龄幼虫期表达量次之,高于其他生长时期并且与其他时期差异有统计学意义(F=73.870,P < 0.001)。Ae-CLIPB41在成蚊时期表达量丰富,其中雌性成蚊表达量最高(F=64.790,P < 0.001),在雄性成蚊表达量较高,其余时期表达量均较低;Ae-CLIPB42在4龄幼虫表达量最高(F=87.260,P < 0.001),在黑色雌蛹表达量次之,其余时期表达量都较低;Ae-CLIPB43在雄性成蚊中表达量最高(F=46.230,P < 0.001),在4龄幼虫表达量较高,黑色雄蛹、雌性成蚊次之,其余时期表达量都较低。见图 3。

|
| 注:A~F分别代表埃及伊蚊6个丝氨酸蛋白酶基因在不同发育时期的相对表达水平;图中柱子上只要有相同字母,表示两组间差异无统计学意义(P > 0.05),字母完全不同的两组间差异有统计学意义(P < 0.05)。 图 3 埃及伊蚊6个丝氨酸蛋白酶基因不同发育时期表达水平 Figure 3 Relative expression levels of six CLIPs genes at different developmental stages of Aedes aegypti |
| |
埃及伊蚊6个CLIPs基因在雌性成蚊不同组织的表达模式不同。设定6个CLIPs基因在雌性成蚊马氏管中的表达量为1,以埃及伊蚊RPS17作为内参基因,Ae-CLIPB8在脂肪体表达量最高,其次为胸部,并且与其他时期相比差异有统计学意义(F=87.810,P < 0.001),其余时期表达量都较低;Ae-CLIPB39和Ae-CLIPB42的组织表达模式与Ae-CLIPB8相似,在脂肪体表达量最高,其次为胸部,并且与其他时期相比差异均有统计学意义(F=91.330,P < 0.001;F=135.200,P < 0.001),其余时期表达量都较低;Ae-CLIPB40在胸部表达量最高,并且与其他时期相比差异有统计学意义(F=353.300,P < 0.001),在脂肪体中表达量较高,卵巢次之,其余时期表达量都较低;Ae-CLIPB41和Ae-CLIPB43的组织表达模式相似,在胸部表达量最高,其次为脂肪体,并且与其他时期相比差异均有统计学意义(F=170.800,P < 0.001;F=113.600,P < 0.001),其余时期表达量都较低。见图 4。

|
| 注:A~F分别代表埃及伊蚊6个丝氨酸蛋白酶基因在雌性成蚊不同组织中的相对表达水平;图中柱子上只要有相同字母,表示两组间差异无统计学意义(P > 0.05),字母完全不同的两组间差异有统计学意义(P < 0.05)。 图 4 埃及伊蚊6个丝氨酸蛋白酶基因不同组织表达水平 Figure 4 Relative expression levels of six CLIPs genes in different tissues of Aedes aegypti |
| |
埃及伊蚊6个CLIPs基因在雌性成蚊受到不同种类细菌侵染时的表达模式各有不同。设定6个CLIPs的mRNA表达量在DEPC水处理下为1,以埃及伊蚊RPS17作为内参基因,Ae-CLIPB8和Ae-CLIPB43对于大肠埃希菌和金黄色葡萄球菌的侵染表达量与对照组之间差异无统计学意义(F=1.743,P=0.253;F=3.035,P=0.123);Ae-CLIPB39、Ae-CLIPB40和Ae-CLIPB42三者对于大肠埃希菌的侵染表达量显著增加,差异均有统计学意义(F=26.230,P=0.001;F=259.100,P < 0.001;F=221.900,P < 0.001),但对于金黄色葡萄球菌的侵染表达水平与对照组之间差异无统计学意义(均P > 0.05);Ae-CLIPB41对于金黄色葡萄球菌的侵染表达量显著增加,且差异有统计学意义(F=27.155,P=0.001),但对于大肠埃希菌的侵染表达水平与对照组之间差异无统计学意义(F=4.526,P=0.176)。见图 5。

|
| 注:A~F分别代表埃及伊蚊6个CLIPs在雌性成蚊分别感染大肠埃希菌和金黄色葡萄球菌24 h后的相对表达水平;ns表示差异无统计学意义(P > 0.05);a表示P < 0.01;b表示P < 0.001。 图 5 埃及伊蚊6个丝氨酸蛋白酶基因感染大肠埃希菌和金黄色葡萄球菌后的表达水平 Figure 5 Relative expression levels of six CLIPs genes in the female adult Aedes aegypti after infection with Escherichia coli and Staphylococcus aureus |
| |
本研究采用生物信息学方法分析了埃及伊蚊6个CLIPs的蛋白二级结构特征,比较了氨基酸序列之间的相似性,其保守区主要集中在羧基端的蛋白酶结构域,CLIPs的蛋白酶结构域特征是一个具有催化三元结构域的保守的Tryp_SPc结构域,由His、Asp和Ser氨基酸残基组成[11-12];系统进化树显示,埃及伊蚊6个CLIPs基因与其他物种CLIPs基因之间的进化关系相近,表明CLIPs在节肢动物中的进化相对保守。
CLIPs的细胞外蛋白水解级联涉及多种节肢动物先天免疫反应来应对物理或生物入侵。其特异性片段已经被证明可以快速处理免疫信号,导致局部反应,如马蹄蟹的血淋巴凝结[13],黑腹果蝇抗菌肽合成的激活[14],小龙虾的黑色素化[15-16]。在本研究中,不同的CLIPs基因在埃及伊蚊发育过程表达模式不同,但是这6个CLIPs基因大多数在4龄幼虫和雌性成蚊阶段表达量较为丰富,其原因可能在于4龄幼虫处于埃及伊蚊生长发育阶段形态学发生变化的重要时期,自身需要合成黑色素来保证其蛹期阶段的正常发育,研究发现,在冈比亚按蚊中,目前已确定了几个CLIPs基因参与了疟疾寄生虫的黑化。其中CLIPA2、CLIPA5和CLIPA7抑制黑色素化,这3个CLIPs基因的沉默能够促发寄生虫黑化,敲除CLIPB3,CLIPB4或CLIPB17阻断了蚊虫的黑化[17-18];而雌性成蚊需要吸食血液来满足自身发育,宿主的血液中含有大量细菌,为保证雌虫肠道微生物的平衡,需要自身免疫反应来调节,CLIPs是昆虫先天免疫反应的重要调节因子,有的CLIPs还参与细胞黏附和细菌调理[19]。不同的CLIPs基因在雌性成蚊组织中表达量也具有差异,其在中肠和胸部的表达量相比其他组织较为丰富。雌虫中肠存在大量肠道微生物,雌虫需要不断调节体内肠道微生物的平衡来维持各项生命活动;蚊虫的胸部存在大量血淋巴,未被激活的CLIPs以酶原的形式存在于血淋巴中。这很好地解释了CLIPs基因在中肠和胸部表达量较为丰富的原因。而这些CLIPs基因相对表达较丰富的生长阶段和组织之间的差异也提示CLIPs基因之间形成的级联调节系统是一个复杂的调控网络而并非是单一的级联调控。
在节肢动物中,胞外酶级联反应的中心成分CLIPs调节了Toll通路、免疫缺陷(immune deficiency,IMD)途径、黑化等多种先天免疫反应[20]。研究表明,节肢动物的先天免疫反应主要由2条通路组成,Toll通路和IMD途径[21]。在先前的研究中,埃及伊蚊与果蝇Toll途径基因的同源基因Spz1C、Toll5A、CLIPB5和CLIPB29已经被发现,并被证明介导Toll途径对真菌和革兰阳性菌感染的反应[8, 19];同时,埃及伊蚊CLIPB15和CLIPB22在RNA干扰条件下,显示出对微生物抵抗能力的下降,且对于Toll通路和IMD途径的关键转录因子也有一定的影响[22],这提示部分CLIPB家族成员参与了埃及伊蚊先天免疫的调控。在本研究中,通过对埃及伊蚊成蚊进行革兰阴性菌和革兰阳性菌侵染,发现这6个CLIPs基因在应对革兰阴性菌或革兰阳性菌侵染时有不同的诱导动力学,类似的反应在中华按蚊(An. sinensis)感染沙雷菌时也可发生[23]。同时,并非所有的CLIPs都能对革兰阴性菌以及革兰阳性菌的侵染产生特异性反应,这也说明不同的CLIPs对于细菌的侵染的诱导动力学模式是具有特异性的。Toll通路和IMD途径是细菌、真菌和病毒激活的主要昆虫免疫途径[24],埃及伊蚊在应对不同种类微生物侵染时,CLIPs有着不同的诱导动力学,这意味着在庞大的CLIPs家族中,每个成员所发挥的免疫反应调节功能各有不同。因此,需要进行更加深入的研究去探索每个CLIPs成员能够发挥的功能。
综上,CLIPs已成为许多昆虫免疫反应的重要调节因子。目前,在埃及伊蚊中,CLIPs已进化为A、B、C、D和E共5个亚族,但所有已知或已被预测能够直接调节蚊虫先天免疫反应的CLIPs都属于B亚家族。研究中对埃及伊蚊6个CLIPs的B亚族成员基因进行系统分析,为埃及伊蚊先天免疫反应调控的研究奠定了理论基础。然而,当前还不清楚单一的CLIPs级联是如何在众多生理功能的背景下被调节的。研究在不同的防御反应中导致黑化的调节机制,如伤口愈合或病原体隔离是很有意义的。此外,本研究中部分CLIPs不能够被细菌侵染所诱导,提示CLIPs在埃及伊蚊发育过程中可能具有额外的非免疫功能,就像它们在果蝇胚胎背模式形成或其他有待发现的生理系统中所表现的那样[25]。为进一步提高我们对CLIPs级联反应在埃及伊蚊多种免疫反应中的作用和认识,还需要对其进行更为深入地研究,尤其作为蚊媒传染病的主要传播媒介的埃及伊蚊,其免疫系统与其传播的病原体之间存在复杂的相互作用。
利益冲突 无
| [1] |
Waterhouse RM, Kriventseva EV, Meister S, et al. Evolutionary dynamics of immune-related genes and pathways in disease-vector mosquitoes[J]. Science, 2007, 316(5832): 1738-1743. DOI:10.1126/science.1139862 |
| [2] |
Liu HW, Heng JY, Wang LL, et al. Identification, characterization, and expression analysis of clip-domain serine protease genes in the silkworm, Bombyx mori[J]. Dev Comp Immunol, 2020, 105: 103584. DOI:10.1016/j.dci.2019.103584 |
| [3] |
Gulley MM, Zhang X, Michel K. The roles of serpins in mosquito immunology and physiology[J]. J Insect Physiol, 2013, 59(2): 138-147. DOI:10.1016/j.jinsphys.2012.08.015 |
| [4] |
Gubb D, Sanz-Parra A, Barcena L, et al. Protease inhibitors and proteolytic signalling cascades in insects[J]. Biochimie, 2010, 92(12): 1749-1759. DOI:10.1016/j.biochi.2010.09.004 |
| [5] |
Park JW, Kim CH, Rui J, et al. Beetle immunity[J]. Adv Exp Med Biol, 2010, 708: 163-180. DOI:10.1007/978-1-4419-8059-5_9 |
| [6] |
Jiang HB, Vilcinskas A, Kanost MR. Immunity in lepidopteran insects[J]. Adv Exp Med Biol, 2010, 708: 181-204. DOI:10.1007/978-1-4419-8059-5_10 |
| [7] |
Rajah MM, Pardy RD, Condotta SA, et al. Zika virus: Emergence, phylogenetics, challenges, and opportunities[J]. ACS Infect Dis, 2016, 2(11): 763-772. DOI:10.1021/acsinfecdis.6b00161 |
| [8] |
Zou Z, Shin SW, Alvarez KS, et al. Distinct melanization pathways in the mosquito Aedes aegypti[J]. Immunity, 2010, 32(1): 41-53. DOI:10.1016/j.immuni.2009.11.011 |
| [9] |
Ballester M, Cordr M, CFolch JM. DAG expression: high-throughput gene expression analysis of real-time PCR data using standard curves for relative quantification[J]. PLoS One, 2013, 8(11): e80385. DOI:10.1371/journal.pone.0080385 |
| [10] |
Madeira F, Park YM, Lee J, et al. The EMBL-EBI search and sequence analysis tools APIs in 2019[J]. Nucleic Acids Res, 2019, 47(W1): W636-641. DOI:10.1093/nar/gkz268 |
| [11] |
Di Cera E. Serine proteases[J]. IUBMB Life, 2009, 61(5): 510-515. DOI:10.1002/iub.186 |
| [12] |
Perona JJ, Craik CS. Structural basis of substrate specificity in the serine proteases[J]. Protein Sci, 1995, 4(3): 337-360. DOI:10.1002/pro.5560040301 |
| [13] |
De Gregorio E, Han SJ, Lee WJ, et al. An immune-responsive serpin regulates the melanization cascade in Drosophila[J]. Dev Cell, 2002, 3(4): 581-592. DOI:10.1016/S1534-5807(02)00267-8 |
| [14] |
Ligoxygakis P, Pelte N, Ji CY, et al. A serpin mutant links Toll activation to melanization in the host defence of Drosophila[J]. EMBO J, 2002, 21(23): 6330-6337. DOI:10.1093/emboj/cdf661 |
| [15] |
Dohke K. Studies on prephenoloxidase-activating enzyme from cuticle of the silkworm Bombyx mori: Ⅰ. Activation reaction by the enzyme[J]. Arch Biochem Biophys, 1973, 157(1): 203-209. DOI:10.1016/0003-9861(73)90406-2 |
| [16] |
Dohke K. Studies on prephenoloxidase-activating enzyme from cuticle of the silkworm Bombyx mori: Ⅱ. Purification and characterization of the enzyme[J]. Arch Biochem Biophys, 1973, 157(1): 210-221. DOI:10.1016/0003-9861(73)90407-4 |
| [17] |
Barillas-Mury C. CLIP proteases and Plasmodium melanization in Anopheles gambiae[J]. Trends Parasitol, 2007, 23(7): 297-299. DOI:10.1016/j.pt.2007.05.001 |
| [18] |
Volz J, Osta MA, Kafatos FC, et al. The roles of two clip domain serine proteases in innate immune responses of the malaria vector Anopheles gambiae[J]. J Biol Chem, 2005, 280(48): 40161-40168. DOI:10.1074/jbc.M506191200 |
| [19] |
Wang RG, Lee SY, Cerenius L, et al. Properties of the prophenoloxidase activating enzyme of the freshwater crayfish, Pacifastacus leniusculus[J]. Eur J Biochem, 2001, 268(4): 895-902. DOI:10.1046/j.1432-1327.2001.01945.x |
| [20] |
Lemaitre B, Hoffmann J. The host defense of Drosophila melanogaster[J]. Ann Rev Immunol, 2007, 25: 697-743. DOI:10.1146/annurev.immunol.25.022106.141615 |
| [21] |
Xi ZY, Ramirez JL, Dimopoulos G. The Aedes aegypti toll pathway controls dengue virus infection[J]. PLoS Pathog, 2008, 4(7): e1000098. DOI:10.1371/journal.ppat.1000098 |
| [22] |
Wang HC, Wang QH, Bhowmick B, et al. Functional characterization of two clip domain serine proteases in innate immune responses of Aedes aegypti[J]. Parasit Vectors, 2021, 14(1): 584. DOI:10.1186/s13071-021-05091-9 |
| [23] |
Bai L, Wang LL, Vega-Rodríguez J, et al. A gut symbiotic bacterium Serratia marcescens renders mosquito resistance to Plasmodium infection through activation of mosquito immune responses[J]. Front Microbiol, 2019, 10: 1580. DOI:10.3389/fmicb.2019.01580 |
| [24] |
Sim S, Jupatanakul N, Dimopoulos G. Mosquito immunity against arboviruses[J]. Viruses, 2014, 6(11): 4479-4504. DOI:10.3390/v6114479 |
| [25] |
Castillejo-Lspez C, Häcker U. The serine protease Sp7 is expressed in blood cells and regulates the melanization reaction in Drosophila[J]. Biochem Biophys Res Commun, 2005, 338(2): 1075-1082. DOI:10.1016/j.bbrc.2005.10.042 |
 2022, Vol. 33
2022, Vol. 33


