电子转移过程在自然界和人工应用中具有突出的作用。在生物中电子转移链是光合作用和细胞呼吸作用的重要环节[1-2]。在人工应用中,电子转移现象也具有极其重要的作用,比如催化反应和太阳能的转换[3-8]。在已知的描述电子转移路径的理论当中,最著名的是Marcus在20世纪50年代推导出的电子转移理论[9-12]。自Marcus理论提出以来,在生物电子转移领域已经进行了大量的实验[13]和理论研究[14-17]。特别是Blumberge[18], Hopfield[19]运用Marcus理论对于生物体的电子转移做了系统的工作。电子转移反应速率与耦合矩阵元成二次方关系,而耦合矩阵元与电子转移反应中的电子给体与受体的相互作用密切相关,因此,耦合矩阵元对于电子转移反应的研究具有十分重要的作用。
Cyt c是电子转移蛋白的典型例子。其蛋白内部是由4个吡咯氮原子保持的血红素-Fe。它是一种水溶性碱性蛋白,普遍存在于动物细胞线粒体内膜的外表面,是最保守的蛋白质之一。由于其简单性、稳定性和可用性,Cyt c已成为氧化还原蛋白电子转移的实验和理论研究的主要蛋白之一[20-22]。
纳米技术的快速发展促进了纳米材料在生物传感器、体内成像、催化、抗病原体和药物输送中的应用[23]。在所有金属纳米颗粒中,纳米银颗粒由于具有强大的光学、电学和治疗潜力而引起非常多的关注。特别是在生物医学领域,人们正在深入探索使用纳米银颗粒作为佐剂和纳米粒子的抗菌药物制剂[24]。纳米银颗粒治疗与生物医学应用的快速增长,迫使我们理解纳米银颗粒与蛋白质的相互作用。虽然银纳米颗粒在生物学应用中受到了欢迎和广泛的使用,但是也有文献报道银纳米颗粒的不良反应,这使其成为生物医学应用的一个研究热点[25]。
有文献表明,银团簇可以进入细胞[26]丢失一个电子带一个正电的银离子[27],对富含细胞色素c的线粒体产生生理毒性。而细胞色素c作为生物传递电子的关键蛋白,本身的氧化还原中心就极易得到一个电子或失去一个电子。那么银纳米团簇与细胞色素c之间就很容易发生电子转移反应。
本文主要研究Cyt c分别与带正电的银纳米团簇和不带电银纳米团簇之间的电子转移矩阵元和吸附相互作用,为两者之间的电子转移反应速率的计算提供了基础。
1 材料和方法 1.1 模型搭建使用分辨率为0.19 nm的马心Cyt c的X射线晶体结构(PDB:1HRC.pdb)作为模拟的初始结构。该蛋白包含104个氨基酸残基和1个辅助因子残基(血红素)。血红素的铁离子与1个组氨酸(His18)和1个甲硫氨酸(Met80)配位,血红素环的2个乙烯基通过硫醚键与2个半胱氨酸的硫原子(Cys14和Cys17)共价链接。同时,利用Materials Studio软件生成直径为3 nm的银纳米团簇。
1.2 分子动力学模拟所有分子动力学模拟都使用Namd2.13软件包和Amber03力场。血红素的力场参数及其肽链链接来自Autenrieth等[28]的工作。银纳米团簇的力场参数来自Staelen等[29]的工作。设置两个模拟体系,分别是表面不带电的纳米团簇(P1)与表面带正电的纳米团簇(P2)。每次模拟都会使用最速下降法进行10 000步的最小化模拟,以此消除不良的空间接触。此后使用200 ns的分子动力学模拟松弛蛋白。在模拟过程中,所有氢键受到约束。短距离范德华相互作用的截断距离设置为1.2 nm。采用粒子网格Ewald(PME)方法处理远程静电相互作用。310 K下模拟水溶液中银纳米团簇与Cyt c的吸附作用。积分时间步长2.0 fs。每500步记录1次轨迹文件。分子动力学轨迹可视化用VMD实现[30]。
1.3 电子耦合矩阵元如Marcus[9-12]所述,电子转移与蛋白质内的氧化还原中心、蛋白质的取向和电子转移距离有关。电子耦合矩阵元反映了反映物电子态与产物电子态之间的耦合强度。Balabin等[30]研究隧道通道模型(PTM)计算电子转移中氧化还原中心的耦合强度,该模型将供体和受体之间的介质描述为由相同重复单元组成的桥。本文采用路径通道法计算Cyt c与银纳米团簇之间的耦合矩阵元。Cyt c中的三价Fe作为电子受体,银纳米团簇作为电子给体。连接给体与受体的桥被分解为通过共价键、氢键和贯穿空间跳跃连接的较小亚基。每个连接被分配一个耦合衰减,并且通过结构搜索算法用于计算氧化还原位点的最佳途径。其表达式为
| $ {T_{{\rm{DA}}}} \propto \prod\limits_i {\varepsilon _i^{\rm{C}}} \prod\limits_j {\varepsilon _j^{\rm{H}}} \prod\limits_k {\varepsilon _k^{\rm{S}}}, $ |
其中共价键的衰减因子εC=0.6,来源于Closs和Miller[31]的实验。空间跳跃的衰减因子εS=εC exp[-βS(RS-0.14)],RS代表跳跃距离,单位nm。氢键的衰减因子εH=(εC)2exp[-βS (RH-0.28)],RH为氢键的长度(重原子到重原子之间)。衰减因子βS=17 nm-1后来降低到11 nm-1反映典型的氧化还原辅因子的结合能[32]。
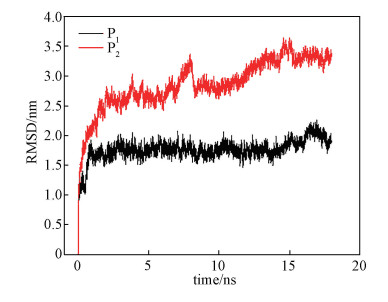
2 结果与讨论 2.1 均方根偏差与均方回转半径为定量地表征Cyt c总的构象变化,计算了骨架原子的均方根偏差(RMSD),如图 1所示。
|
Download:
|
|
P1体系中银纳米团簇体系不带电, P2体系中银纳米团簇体系带正电。 图 1 RMSD随时间的变化曲线 Fig. 1 RMSD curvs of the P1 and P2 systems |
|
RMSD可以表征蛋白质构象的变化。从图 1中可以看出两个系统的RMSD值先增加后稳定在一个值。在带电银纳米团簇的表面RMSD的值最大,为0.33 nm,这表明Cyt c经历了一个较大的结构变化。而在不带电的表面上,Cyt c的RMSD值较小,为0.18 nm。这表明结构变化较小。
同时计算最后3 nm的均方回转半径(Rg)和电子转移耦合矩阵元。通过计算均方回转半径Rg可以反映Cyt c链的构象。对于P1和P2体系,平均Rg值分别为1.33和1.36 nm。又因为在水溶液中的均方回转半径为1.33 nm。这表明带电银纳米团簇会对Cyt c构象产生明显的影响。
2.2 电子转移矩阵元通过分析平衡态最后3 ns的耦合矩阵元,分别得出耦合矩阵元与电子传递路径的统计平均值。数据见表 1。
|
|
表 1 耦合矩阵元的值 Table 1 Value of the coupling matrix elements |
由表 1可知,带正电的P2体系的电子转移距离是不带电的P1体系的电子转移距离的3倍。从表 1中还可得出,P1体系的耦合矩阵元数值比P2体系的数值大3个数量级。说明带电体系对电子耦合矩阵元影响很大,再根据Marcus的电子转移理论,路径越短耦合强度越强,电子转移的速率更可能偏大,更容易发生电子转移。因此这很可能导致P1电子转移速率大于P2的电子转移速率。
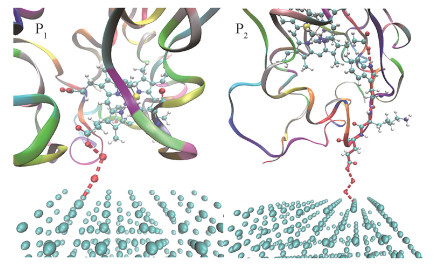
图 2为P1体系与P2体系的电子转移图。其中蛋白质使用Newribbons,氧化还原中心使用球棍模型,银纳米团簇使用CPK,红色柱状通道为PTM构建的电子转移路径通道。从图中可以清晰地看到,P1体系电子转移路径通道要比P2体系的电子转移路径通道短。
|
Download:
|
| 图 2 P1和P2体系的电子转移路径通道 Fig. 2 Electron transfer paths in the P1 and P2 systems | |
运用分子动力学原理,研究Cyt c与银纳米团簇的吸附和电子转移的性质。计算结果表明,带电银纳米团簇对Cyt c有静电相互作用,对Cyt c构象有明显影响,使得电子转移路径较长,电子耦合矩阵元较小,不利于电子转移。不带电的银纳米团簇与Cyt c之间的电子转移路径较短,对构象影响较小,有更大的耦合矩阵元,有利于电子转移的发生。
本文的计算结果是在中国科学院计算机网络信息中心超级计算中心的“元”超级计算机上得到的。
| [1] |
Bauer A, Westkamper F, Grimme S, et al. Catalytic enantioselective reactions driven by photoinduced electron transfer[J]. Nature, 2005, 436(7054): 1139-1140. Doi:10.1038/nature03955 |
| [2] |
Bixon M, Jortner J. Electron transfer: from isolated molecules to biomolecules[C]//Jortner J, Bixon M. Advances in Chemical Physics: Electron Transfer-from Isolated Molecules to Biomolecules, Pt 1. Hoboken: John Wiley & Sons Inc, 1999: pp. 35-202.
|
| [3] |
Chen P Y, Meyer T J. Medium effects on charge transfer in metal complexes[J]. Chemical Reviews, 1998, 98(4): 1439-1477. |
| [4] |
Cleland D M, Irwin G, Wagner P, et al. Linker conjugation effects in rhenium(I) bifunctional hole-transport/emitter molecules[J]. Chemistry-a European Journal, 2009, 15(15): 3682-3690. Doi:10.1002/chem.200802373 |
| [5] |
Collin J P, Laine P, Launay J P, et al. Long-range coupling in a mixed-valence diruthenium complexes containing bis-terpyridine ligands of various lengths as bridges[J]. Journal of the Chemical Society-Chemical Communications, 1993(5): 434-435. Doi:10.1039/C39930000434 |
| [6] |
Devault D. Quantum-mechanical tunnelling in biological-systems[J]. Quarterly Reviews of Biophysics, 1980, 13(4): 387-564. |
| [7] |
Dolg M, Wedig U, Stoll H, et al. Abinitio pseudopotential study of the 1st row transition:metal monoxides and iron monohydride[J]. Journal of Chemical Physics, 1987, 86(4): 2123-2131. Doi:10.1063/1.452110 |
| [8] |
Keita B, Nadjo L. Polyoxometalate-based homogeneous catalysis of electrode reactions:recent achievements[J]. Journal of Molecular Catalysis a-Chemical, 2007, 262(1-2): 190-215. Doi:10.1016/j.molcata.2006.08.066 |
| [9] |
Marcus R A. On the theory of oxidation:reduction reactions involving electron transfer.1[J]. ournal of Chemical Physics, 1956, 24(5): 966-978. |
| [10] |
Marcus R A. On theory of electron-transfer reactions.6. Unified treatment for homogeneous and electrode reactions[J]. Journal of Chemical Physics, 1985, 43(2): 679-701. |
| [11] |
Marcus R A. theory of oxidation-reduction reactions involving electron transfer.4. A statistical-mechanical basis for treating contributions from solvent, ligands, and inert salt[J]. Discussions of the Faraday Society, 1960, 29: 21-31. Doi:10.1039/DF9602900021 |
| [12] |
Marcus R A, Sutin N. electron transfers in chemistry and biology[J]. Biochimica Et Biophysica Acta, 1985, 811(3): 265-322. Doi:10.1016/0304-4173(85)90014-X |
| [13] |
Ishida M, Hwang D, Zhang Z, et al. Beta-functionalized push-pull porphyrin sensitizers in dye-sensitized solar cells:effect of pi-conjugated spacers[J]. Chemsuschem, 2015, 8(17): 2967-2977. Doi:10.1002/cssc.201500085 |
| [14] |
Harihara Pc, Pople J A.. influence of polarization functions on molecular-orbital hydrogenation energies[J]. Theoretica Chimica Acta, 1973, 28(3): 213-222. Doi:10.1007/BF00533485 |
| [15] |
Hush N S. Adiabatic theory of outer sphere electron-transfer reactions in solution[J]. Transactions of the Faraday Society, 1961, 57(4): 557-580. |
| [16] |
Hush N S, Reimers J R. Solvent effects on metal to ligand charge transfer excitations[J]. Coordination Chemistry Reviews, 1998, 177: 37-60. Doi:10.1016/S0010-8545(98)00100-3 |
| [17] |
Jang S J, Newton M D. Theory of torsional non-Condon electron transfer:a generalized spin-boson Hamiltonian and its nonadiabatic limit solution[J]. Journal of Chemical Physics, 2005, 122(2): 024501. Doi:10.1063/1.1828431 |
| [18] |
Blumberger J. Recent advances in the theory and molecular simulation of biological electron transfer reactions[J]. Chemical Reviews, 2015, 115(20): 11191-11238. Doi:10.1021/acs.chemrev.5b00298 |
| [19] |
Hopfield J J. Electron-transfer between biological molecules by thermally activated tunneling[J]. Proceedings of the National Academy of Sciences of the United States of America, 1974, 71(9): 3640-3644. Doi:10.1073/pnas.71.9.3640 |
| [20] |
Baweja L, Balamurugan K, Subramanian V, et al. Effect of graphene oxide on the conformational transitions of amyloid beta peptide:a molecular dynamics simulation study[J]. Journal of Molecular Graphics & Modelling, 2015, 61: 175-185. |
| [21] |
Baweja L, Balamurugan K, Subramanian V, et al. Hydration patterns of graphene-based nanomaterials (gbnms) play a major role in the stability of a helical protein:a molecular dynamics simulation study[J]. Langmuir, 2013, 29(46): 14230-14238. Doi:10.1021/la4033805 |
| [22] |
Bushnell G W, Louie G V, Brayer G D. High-resolution 3-dimensional structure of horse heart cytochrome-c[J]. Journal of Molecular Biology, 1990, 214(2): 585-595. |
| [23] |
Agnihotri S, Mukherji S, Mukherji S. Size-controlled silver nanoparticles synthesized over the range 5-100 nm using the same protocol and their antibacterial efficacy[J]. RSC Advances, 2014, 4(8): 3974-3983. Doi:10.1039/C3RA44507K |
| [24] |
Arakha M, Borah S M, Saleem M, et al. Interfacial assembly at silver nanoparticle enhances the antibacterial efficacy of nisin[J]. Free Radical Biology and Medicine, 2016, 101: 434-445. Doi:10.1016/j.freeradbiomed.2016.11.016 |
| [25] |
Bellamy W, Takase M, Yamauchi K, et al. identification of the bactericidal domain of lactoferrin[J]. Biochimica Et Biophysica Acta, 1992, 1121(1/2): 130-136. |
| [26] |
Costa C, Ronconi J V V, Daufenbach J F, et al. In vitro effects of silver nanoparticles on the mitochondrial respiratory chain[J]. Molecular and Cellular Biochemistry, 2010, 342(1/2): 51-56. |
| [27] |
Foldbjerg R, Dang D A, Autrup H. Cytotoxicity and genotoxicity of silver nanoparticles in the human lung cancer cell line, A549[J]. Archives of Toxicology, 2011, 85(7): 743-750. Doi:10.1007/s00204-010-0545-5 |
| [28] |
Autenrieth F, Tajkhorshid E, Baudry J, et al. Classical force field parameters for the heme prosthetic group of cytochrome c[J]. Journal of Computational Chemistry, 2004, 25(13): 1613-1622. Doi:10.1002/jcc.20079 |
| [29] |
Staelens N, Leherte L, Champagne B, et al. Modeling of structural, energetic, and dynamic properties of few-atom silver clusters embedded in polynucleotide strands by using molecular dynamics[J]. Chemphyschem, 2015, 16(2): 360-369. Doi:10.1002/cphc.201402632 |
| [30] |
Balabin I A, Hu X Q, Beratan D N. Exploring biological electron transfer pathway dynamics with the Pathways Plugin for VMD[J]. Journal of Computational Chemistry, 2012, 33(8): 906-910. Doi:10.1002/jcc.22927 |
| [31] |
Closs G L, Miller J R. Intramolecular long-distance electron:transfer in organic-molecules[J]. Science, 1988, 240(4851): 440-447. Doi:10.1126/science.240.4851.440 |
| [32] |
Tong G S M, Kurnikov I V, Beratan D N. Tunneling energy effects on GC oxidation in DNA[J]. Journal of Physical Chemistry B, 2002, 106(9): 2381-2392. Doi:10.1021/jp013387g |
 2020, Vol. 37
2020, Vol. 37 


