2. 广州大学环境科学与工程学院, 广州 510006
2. School of Environmental Science and Engineering, Guangzhou University, Guangzhou 510006, China
氮是组成生命体的基本元素之一,同时氮循环也是自然界最基本的物质循环过程之一。早期地球上的氮循环由大气反应和缓慢的地质过程控制,在大约27亿年前,微生物代谢活动逐渐进化成为控制与调节地球氮循环的主要驱动力[1]。通过微生物的一系列作用,驱动氮在整个地球系统中循环,维持着整个生态系统中生命的延续和进化。通过长期的探索,科学家们初步建立了微生物氮循环途径,主要包括固氮过程、硝化过程、反硝化过程、氨同化过程以及氨化过程。近年来,由于分子微生物学技术的进步,预测和鉴定了许多参与氮循环过程的新微生物种类。新的微生物及其代谢方式的阐明,使得氮生物地球化学循环理论也在经历不断的修正和完善。细菌的厌氧氨氧化(anaerobic ammonia-oxidation, anammox)、古菌氨氧化(archaea ammonia-oxidation)、硝酸盐异化还原到铵(dissimilatory nitrate reduction to ammonium, DNRA)以及全程氨氧化(complete ammonia-oxidation, comammox)的发现,改变了人们对微生物氮循环过程的传统认知,构建了新的氮循环网络体系[2-5]。本文主要针对氨氧化古菌(ammonia-oxidizing archaea, AOA)在海洋环境中的分布、AOA的氨氧化代谢机制、自养固碳机制及其在海洋生物地球化学循环中的作用等方面的研究进展进行综述,以期对这个领域有更深刻更全面的认识。
1 海洋氨氧化过程概述海洋占据地球表面积的71%,发生在海洋生态系统的氮循环过程是全球氮循环的重要组成部分,对维持海洋乃至全球的生态平衡意义重大。对于海洋氮循环微生物的研究,一直是海洋环境微生物的热点研究领域。由于海洋系统的特殊性,海洋氮循环过程具有明显的空间异质性特征。硝化过程是海洋氮循环的重要环节,连接着氮循环中最还原和最氧化状态的化合物,影响着这些无机氮在海洋系统中的分布。硝化过程包括两个步骤:首先,表层生产力制造的颗粒有机物在向下沉降过程中,不断被微生物矿化分解,释放出氨(NH4+),NH4+在氨氧化微生物(ammonia-oxidizing prokaryote, AOP)的作用下氧化生成亚硝酸盐(NO2-);然后,NO2-在亚硝酸盐氧化细菌(nitrite-oxidizing bacteria, NOB)的作用下氧化生成硝酸盐(NO3-)。氨氧化反应作为硝化过程的第一步,同时也是硝化作用的限速步骤[6]。在海洋水体中,氨氧化过程主要在真光层的底部完成,主要是因为这里光线较弱,氨氧化光抑制被解除,而且有比较充分的底物来源和较适宜的水体溶氧条件。然而,在深海底部也有氨氧化过程的发生,但由于底物及水体溶氧条件的限制,氨氧化速率维持在较低的水平。此外,由于海洋沉积物表层有活跃的有机物矿化作用,提供了充分的NH4+的来源,因此也存在比较活跃的氨氧化作用。早期对于海洋氨氧化过程的研究,基本上是基于氨氧化细菌(ammonia-oxidizing bacteria,AOB)开展的,而AOA的发现,对于海洋硝化过程的认识有了根本性改变,建立了海洋硝化过程的新模式。
2 氨氧化古菌的发现19世纪以前,人们把硝酸盐看作是空气中的氧和氨经土壤催化生成的,是化学作用的产物。直到1890年,Winogradsky[7]利用无机盐培养基成功分离纯化获得第一株具有氨氧化功能的细菌——欧洲亚硝化单胞菌(Nitrosomonas europaea),证实氨氧化过程是由氨氧化微生物催化完成的。在此后的一百多年里,AOB被认为是自然环境中唯一具有氨氧化功能的微生物,对于氨氧化过程的研究也都是围绕AOB开展的。Nitrosomonas europaea全基因组被测序之后,成为一种研究氨氧化微生物的生理生态、系统发育等方面的模式生物[8-10],而编码氨单加氧酶(ammonia mono-oxygenase, AMO)α亚基的amoA基因也从此作为分子标记物用于氨氧化细菌的分子生态学研究[11]。目前的研究结果表明,自然环境中发现的AOB均归属于β-和γ-变形菌纲(Proteobacteria)[12-17]。
然而,在早期对海洋水体氨氧化过程的研究中,存在氨氧化通量与氨氧化微生物活性不匹配的问题,即海洋水体中存在较活跃的氨氧化过程,但海洋水体中具有氨氧化能力的细菌数量却极少。这表明,除AOB外,在海洋水体中可能存在着未知的具有氨氧化活性的微生物。Jordan等[18]针对氮饱和的酸性土壤进行的抑制实验表明,AOB并不是氨氧化作用的主导群体。2004年,Venter等[19]对来自于北大西洋中部的马尾藻海(Sargasso Sea)的海水样品进行环境基因组测序,发现amo基因与古菌基因相关联的现象,预示了古菌在海洋的氨氧化过程中可能发挥重要作用。直到2005年,Schleper等[20-21]的一个偶然发现,为寻找有氧氨氧化能力的古菌提供了重要线索,他们在构建的一个土壤宏基因文库(‘54d9’Genbank entry AJ627422)中的一个43 kb的DNA片断中,发现同时包含1.1b Crenarchaeota的16S rRNA基因和编码氨单加氧酶(AMO)亚基的amoA和amoB基因,推测非嗜热的1.1b Crenarchaeota具有氨氧化作用。2005年,Könneke等[22]在位于西雅图的热带海洋水族馆的水族箱中分离到一株具有氨氧化活性的古菌(其典型菌株被鉴定为Nitrosopumilus maritimus SCM1),并证实SCM1能够以NH4+为唯一氮源和能源进行化能自养生长。进一步的研究表明,海洋中的AOA在全球的碳和氮的循环中可能发挥着重要作用,AOA成为海洋微生物氮循环研究的热点。大量研究结果表明,在海洋环境中,AOA有着广泛的分布,其丰度远高于AOB[23-24],生理学实验也表明AOA比AOB对氨有更高的亲和力,更适合在寡营养条件下生长[25]。这些研究结果都暗示AOA是海洋环境中氨氧化作用的主导群体。
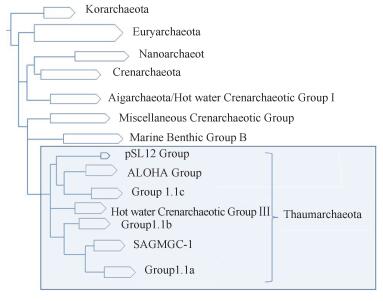
3 氨氧化古菌的多样性及系统进化基于16S rRNA同源性分析,DeLong[26]将从海水中获取的环境样品与可培养的古菌进行比对,并构建系统发育树,将海洋浮游古菌划分为4个分支:Marine Group I,属于泉古菌类群;Marine Group II,III,和IV,属于广古菌类群。进化关系如图 1所示,其中Marine Group I被认为是海洋中分布最广、数量最多的浮游古菌类群,被命名为中温泉古菌(non-thermophilic Crenarchaeota)[27]。此后这类生活在中温环境的泉古菌在土壤、河口、沉积物、海湾等诸多生境中都被发现广泛存在。系统进化分析表明,氨氧化古菌也隶属于中温泉古菌。
但近来,随着更多新的古菌小亚基核糖体RNA(SSU rRNA)和大亚基核糖体RNA(LSU rRNA)序列的获得,研究者们对古菌的系统发育分析表明,中温泉古菌可能代表了一个新的独立的进化分支。Robertson等[28]利用从GenBank下载的712条古菌SSU rRNA基因序列构建的系统发育树,以及Brochier-Armanet等[29]收集的226个古菌和细菌的SSU rRNA和LSU rRNA基因序列进行的同源性分析,都发现在古菌域中的中温泉古菌和嗜热泉古菌形成了2个不同的单系群。紧接着,Brochier-Armanet对包括真核生物、细菌、广古菌、嗜热泉古菌和中温泉古菌在内的53个具有全基因组的物种进行核糖体蛋白(r蛋白)的系统发育分析,并对Cenarcha eumsymbiosum进行比较基因组学分析,建议将中温泉古菌独立为古菌的一个门——奇古菌门(Thaumarchaeota)[29-30]。随后,这一新提出的古菌门迅速得到其他研究证据的支持和认可。目前,奇古菌作为古菌中的一个单独分支,包括属于氨氧化古菌的Group I.1a和I.1b、来自酸性土壤的Group I.1c、来自开放海域的ALOHA Group、来自热泉和热液口的pSL12 Group和HWCGIII (hot water Crenarchaeotic Group III)以及来自地下矿井的SAGMCG-1,它们的系统进化关系如图 1所示。
依靠传统的微生物培养方法和现代的分子生物学手段,越来越多的氨氧化古菌被发现和鉴定(具体见附件表 1)。针对目前这些已被分离或富集到的氨氧化古菌菌株,研究者们进行了基因组学和生理学等多方面研究,对这部分氨氧化古菌的基因组信息和生理学特性都有了一定的了解。但由于氨氧化古菌生长缓慢,代时长,目前来说仍有大部分的氨氧化古菌尚未被人们所认识和发现。Biller等[31]从GenBank中收集来自不同生境的8 296条AOA的amoA基因序列,当以90%的序列相似度作为界定新种的阈值时,得出自然环境中AOA的物种数为315个左右。这就意味着自然界中90%以上的AOA尚未被人类所认识。根据已知的氨氧化古菌16S rRNA和amoA基因的系统发育分析,目前已知的氨氧化古菌可分为4大簇:海洋中的Group I.1a、土壤中的Group I.1b、嗜热的ThAOA和嗜酸的Group I.1a-associated 4个类群。2012年Pester等[32]对NCBI、IMG/M和Camera 3个数据库中收集的12 356条amoA基因序列进行系统发育分析,提出氨氧化古菌可分为5个大簇,利用每个簇中已被分离或富集的氨氧化古菌的名字对该簇命名,包括有:Nitrosopumilus cluster、Nitrososphaera cluster、Nitrosotalea cluster、Nitrosocaldus cluster,以及在当时还未有已知序列的Nitrososphaera sister cluster。
4 AOA的氨氧化代谢途径及其在海洋中的生态分布AOB的氨氧化代谢途径,是NH4+在AMO作用下生成羟胺(NH2OH),然后NH2OH再被羟胺氧化还原酶(hydroxylamine oxidoreductase, HAO)氧化生成NO2-。而目前对于AOA氨氧化过程的具体代谢途径尚不明晰。对于古菌纯培养菌株“Nitrosopumilus marinimus”氨氧化过程的化学计量分析结果表明,古菌氨氧化产生亚硝酸根所消耗的氧和氨的相对量与AOB一致,总反应式为:NH3+1.5O2 →NO2-+H2O+H+[25]。但目前一系列的研究表明,AOA的氨氧化代谢途径不同于AOB。首先,AOA与AOB中编码AMO 3个亚基的基因(amoA、amoB、amoC)的排列顺序存在差异,AOB中AMO基因簇排列较保守,3个亚基的编码基因一般是按照CAB的方式排列[33];而在AOA的基因组中,AMO基因簇不仅存在编码未知功能蛋白的基因的插入片段,且amoA、amoB、amoC在基因簇的组装上也更为多样,不同种属之间也有差别[34]。此外,AOA与AOB中AMO的金属催化中心也不一样,细菌AMO的中心金属元素是铁[35],古菌AMO是以铜作为催化中心的[36]。而通过全基因组分析,在已知的AOA的基因组中并没有发现羟胺氧化还原酶HAO以及细胞色素c的编码基因。综合目前的研究,有3种可能的氨氧化代谢途径:1)氨在AMO作用下氧化生成羟胺,然后羟胺被氧化生成亚硝酸盐,羟胺氧化过程中释放出4个电子,其中2个传递到AMO,用于启动氨的氧化,另外2个进入电子传递链;2)氨同样氧化生成羟胺,但启动这一步的电子来自NO的氧化,羟胺氧化过程释放的4个电子中2个进入电子传递链,2个传递到含铜的亚硝酸盐还原酶(CuNIR)用于亚硝酸盐的还原,亚硝酸盐在CuNIR的作用下还原生成NO,NO再在细胞内部被氧化为亚硝酸盐,释放出2个电子,这2个电子传递到AMO用于氨的氧化;3)氨的氧化并不生成NH2OH,而是生成硝酰基氢化物(HNO),然后HNO被硝酰氧化还原酶(nitroxyl oxidoreductase,NxOR)氧化生成NO2-[36-37]。Martens-Habbena等[25]发现AOA在氨氧化过程中有NO的释放,暗示NO可能是AOA氨氧化过程中给AMO传递电子的氧化还原中介体。Vajrala等[38]通过稳定同位素示踪法发现Nitrosopumilus marinimus的氨氧化过程有NH2OH的生成与消耗,证明AOA氨氧化过程的中间产物可能与AOB一样,都是羟胺。到目前为止,AOA具体的氨氧化代谢途径还无法明确,仍需进一步的研究与探讨。
1992年,Fuhrman等[39]和DeLong[40]分别在不同海域水体中发现,古菌在海洋水体中大量存在,且分布广泛。此后的研究也表明海洋浮游古菌在海洋水体中占据着重要地位,尤其是在真光层以下的深层海水中数量更为丰富,在海洋中层及深层水体中,古菌占据海洋浮游微生物丰度的20%~40%[41-43]。而大部分的海洋浮游古菌都属于中温泉古菌(non-thermophilic Crenarchaeota),即现在的奇古菌门(Thaumarchaeota)。因此可以推断,相对于AOB,AOA在海洋水体中占据着优势地位。
Francis等[44]首先对海水和海洋底泥中AOA的多样性和分布的普遍性做了探索,利用AOA-amoA基因特异性的引物对海水透光层、次氧层、河口和近海底泥等多个地点样品中的AOA-amoA基因进行研究分析,发现在不同的海洋环境中存在不同的AOA群体,而且每个不同的取样点都存在与环境相适应的特异性的AOA,但也存在一部分AOA种类在海洋中具有普遍性的分布。荷兰皇家海洋研究所的Wuchter等[24]在针对北海及北大西洋的研究中,发现北海水体中AOA-amoA基因的丰度与海水中氨浓度的下降以及泉古菌的数量密切相关,而且古菌amoA的丰度比细菌的要高出1~2个数量级,对北大西洋水深1 000 m以上的水体中amoA基因丰度测定结果显示,AOA的丰度也比AOB高1~3个数量级,这表明AOA在海洋硝化作用中可能发挥着重要作用。而宏转录组的分析结果也表明,在不同的海洋环境中,AOA氨氧化代谢都非常活跃[45-47]。Martens-Habbena等[25]通过对比原位测定的海洋氨氧化半饱和常数与实验室测定Nitrosopumilus marinimus SCM1的氨氧化半饱和常数,发现二者在数值上相吻合,暗示AOA主导着海洋大部分的氨氧化反应。综合以上研究可以看出,在大洋及寡营养的开阔海域水体中AOA数量多、多样性高、分布广,是海洋环境中氨氧化作用的主导群体,在海洋氮循环过程中发挥着重要作用。
5 AOA自养固碳的分子机制及其在海洋生态系统中的功能与AOB相比,AOA具有不同的固碳途径。基于氨氧化细菌Nitrosomonas europaea基因组及纯培养水平上的研究表明,氨氧化细菌AOB通过卡尔文循环进行自养固碳[9],1, 5-二磷酸核酮糖羧化酶(RuBisCO)是该过程的关键酶,AOB固定1分子的CO2需消耗3分子的ATP和2分子的NADPH,固碳过程预计占到能量消耗的80%[48]。而针对氨氧化古菌基因组和生理学分析表明,AOA的固碳途径与AOB的完全不同,AOA通过3-羟基丙酸/4-羟基丁酸循环(3-HP/4-HB cycle)固定CO2[36, 49-52](图 2)。相关研究表明,AOA通过3-HP/4-HB循环固定CO2比任何其他自养途径都更为节能,培养实验结果表明,AOA Nitrosopumilus maritimus和AOB Nitrosococcus oceani在氧化等量的氨时,N. maritimus比N.oceani增加的生物量更多[53]。这为AOA在寡营养条件下更具竞争优势提供了解释。氨氧化古菌氨氧化耦联固碳过程如图 2所示,氨氧化古菌通过氨氧化过程从NH4+中获得电子,电子经电子传递链[36]传递,将储存在NH4+中的化学能转换为能量货币ATP,氨氧化古菌经3-HP/4-HB循环,通过消耗ATP固定CO2合成有机物。目前针对氨氧化古菌固碳的研究也得到越来越多的关注,作为编码3-HP/4-HB循环的关键酶乙酰辅酶A羧化酶α亚基的accA(acetyl-CoA carboxylase α-subunit)基因,目前被许多研究作为分子标记来分析环境中自养型的氨氧化古菌[54-59]。

|
Download:
|
| 图 2 氨氧化古菌的氨氧化途径驱动自养固碳[36, 50] Fig. 2 Carbon fixing coupled with ammonia oxidation of ammonia oxidizing archaea[36, 50] | |
由于海洋结构的特殊性,传统观点认为海洋初级生产力过程主要发生在有光合作用进行的海洋真光层(euphotic zone),而海洋内部(真光层以下)由于光限制,其所需能量主要源于上层海洋(真光层)输入的颗粒有机物(particle organic matter, POM),满足海洋内部微生物的能量需求[60]。然而,许多研究表明,深海原核微生物代谢所需碳量(prokaryotic carbon demand, PCD)远超过真光层水体向下输送的POM中所能提供的碳量[61-62],表明在深海内部存在着“新”的途径可提供深海原核微生物生长所需碳源。夏威夷大学的Karl等[63]在针对北太平洋中层水体的研究中,通过分析水体中向下输运的有机碳、有机氮、ATP和RNA的通量,发现在700~900 m的水层中有新颗粒有机碳的生成,指出海洋碳循环过程相比传统认识应该更为复杂,深海内部可能存在“新”的生产力过程,并推测“新”有机碳的生成与上层向下输送的颗粒有机物中生成的NH4+有关。原位培养实验结果表明,在NH4+下降和有NO2-生成的550~750 m的海洋水体中存在着活跃的化能自养过程。利用免疫荧光技术对氨氧化细菌进行计数,在所有深度的沉积物捕获器(sediment trap)中均检测到丰富的AOB,且主要为Nitrosomonas。因此,Karl等[63]提出真光层向海洋内部输送的能量比原来认为的保存在颗粒有机物中的能量要多,氨氧化过程是深海内部生态系统中重要的能量来源途径。Midderlburg[64]对全球海洋的化能自养固碳速率进行估算,得出全球海洋系统化能自养固碳速率为0.77 Pg C·a-1,其中海洋水体占据总化能自养固碳量的52%,为0.4 Pg C·a-1;而沉积物中的微生物化能自养固碳过程主要发生在近海海域,为0.37 Pg C·a-1;并指出氨氧化过程是海洋中化能自养固碳过程能量的主要来源。2006年,Ingalls等[65]通过放射性同位素14C标记海洋浮游古菌的特征膜脂(glycerol dialkyl glycerol tetraethers, GDGTs)对亚热带北太平洋环流中两个不同深度水体中古菌自养固碳能力的研究,推测真光层以下浮游古菌自养固碳占所有古菌碳吸收的83%,并推算海洋浮游古菌的生产力可达到0.6~0.7 Gt C/a,约占到海洋初级生产力(50 Gt C/a)的1%。
随着AOA的发现并受到广泛关注,作为目前在海洋环境中已知的丰度最高的化能自养微生物,AOA被越来越多的研究证明在深海内部的化能自养固碳过程中发挥着重要作用,是深海内部初级生产力的重要贡献者[59, 66-68]。海洋AOA自养固碳过程主要发生在深海水体中。真光层浮游植物通过光合作用产生POM,POM通过物理沉降及混合作用进入到海洋内部,在向下沉降过程中,被微生物逐渐矿化并释放出NH4+,而NH4+在AOA的作用下氧化为NO2-,AOA收获NH4+中储存的化学能固定DIC合成有机碳,NO2-也被进一步氧化成稳定的NO3-储存在深海内部(图 2)。AOA的自养固碳过程提供了深海微型生物食物网的有机碳需求[69],AOA通过化能自养固定生成的部分活性有机碳(labile dissolved organic carbon, LDOC)进入到微型生物碳泵(microbial carbon pump, MCP)[70-71],最终可能转化为惰性溶解有机碳(recalcitrant dissolved organic carbon, RDOC)储存在海洋碳库中,对海洋微型生物碳泵储碳起到重要作用。
6 研究展望近年来,分子生态学作为有力的工具促进了很多新的微生物生态学的认识。分子生态学的方法能够使我们从基因水平了解AOA的多样性及其参与的碳氮转化机制。纯培养AOA以及自然样品的氨氧化率的测定使我们知道不同的AOA有不同的最适生理条件,因此生存在不同的生境中(ecological niches)。多种生理类型的AOA在广泛不同的生态环境中是否都能以不同的菌群结构保证氨氧化活性?AOA的群体是以多大的时间尺度响应环境条件的改变?什么样的环境条件改变最可能会影响环境的AOA氨氧化率以及固碳速率?AOA自养固碳为深海生物圈提供多少能量?生物地球化学和分子生物学的综合方法将会为这些问题的解决提供思路。
| [1] |
Canfield D E, Glazer A N, Falkowski P G. The evolution and future of earth's nitrogen cycle[J]. Science, 2010, 330(6001): 192-196. Doi:10.1126/science.1186120 |
| [2] |
Daims H, Lebedeva E V, Pjevac P, et al. Complete nitrification by Nitrospira bacteria[J]. Nature, 2015, 528(7583): 504-509. Doi:10.1038/nature16461 |
| [3] |
Francis C A, Beman J M, Kuypers M M M. New processes and players in the nitrogen cycle:the microbial ecology of anaerobic and archaeal ammonia oxidation[J]. The ISME Journal, 2007, 1(1): 19-27. Doi:10.1038/ismej.2007.8 |
| [4] |
Klotz M G, Stein L Y. Nitrifier genomics and evolution of the nitrogen cycle[J]. FEMS Microbiology Letters, 2008, 278(2): 146-156. Doi:10.1111/j.1574-6968.2007.00970.x |
| [5] |
Ward B, Capone D, Zehr J. What's new in the nitrogen cycle?[J]. Oceanography, 2007, 20(2): 101-109. Doi:10.5670/oceanog.2007.53 |
| [6] |
Troelstra S R, Wagenaar R, Boer W D. Nitrification in Dutch heathland soils:I. General soil characteristics and nitrification in undisturbed soil cores[J]. Plant and Soil, 1990, 127(2): 179-192. Doi:10.1007/BF00014424 |
| [7] |
Winogradsky S. Recherches sur les organisms de la nitrification[J]. Annales de L Institut Pasteur. Microbiology, 1890, 4: 213-231. |
| [8] |
Bennett K, Sadler N C, Wright A T, et al. Activity-based protein profiling of ammonia monooxygenase in Nitrosomonas europaea[J]. Applied and Environmental Microbiology, 2016, 82(8): 2270-2279. Doi:10.1128/AEM.03556-15 |
| [9] |
Chain P, Lamerdin J, Larimer F, et al. Complete genome sequence of the ammonia-oxidizing bacterium and obligate chemolithoautotroph Nitrosomonas europaea[J]. Journal of Bacteriology, 2003, 185(9): 2759-2773. Doi:10.1128/JB.185.9.2759-2773.2003 |
| [10] |
Kozlowski J A, Price J, Stein L Y. Revision of N2O-producing pathways in the ammonia-oxidizing bacterium Nitrosomonas europaea ATCC 19718[J]. Applied and Environmental Microbiology, 2014, 80(16): 4930-4935. Doi:10.1128/AEM.01061-14 |
| [11] |
Rotthauwe J H, Witzel K P, Liesack W. The ammonia monooxygenase structural gene amoA as a functional marker:molecular fine-scale analysis of natural ammonia-oxidizing populations[J]. Applied and Environmental Microbiology, 1997, 63(12): 4704-4712. Doi:10.1128/AEM.63.12.4704-4712.1997 |
| [12] |
Campbell M A, Chain P S G, Dang H, et al. Nitrosococcus watsonii sp. Nov., a new species of marine obligate ammonia-oxidizing bacteria that is not omnipresent in the world's oceans:calls to validate the names 'Nitrosococcus halophilus' and 'Nitrosomonas mobilis'[J]. FEMS Microbiology Ecology, 2010, 76(1): 39-48. |
| [13] |
Monteiro M, Séneca J, Magalhães C. The history of aerobic ammonia oxidizers:from the first discoveries to today[J]. Journal of Microbiology, 2014, 52(7): 537-547. Doi:10.1007/s12275-014-4114-0 |
| [14] |
Purkhold U, PommereningRöser A, Juretschko S, et al. Phylogeny of all recognized species of ammonia oxidizers based on comparative 16S rRNA and amoA sequence analysis:implications for molecular diversity surveys[J]. Applied and Environmental Microbiology, 2000, 66(12): 5368-5382. Doi:10.1128/AEM.66.12.5368-5382.2000 |
| [15] |
Urakawa H, Garcia J C, Nielsen J L, et al. Nitrosospira lacus sp. Nov., a psychrotolerant, ammonia-oxidizing bacterium from sandy lake sediment[J]. International Journal of Systematic and Evolutionary Microbiology, 2015, 65(1): 242-250. |
| [16] |
Lin W, Kent L C, Hongyue D, et al. D1FHS, the type strain of the ammonia-oxidizing bacterium Nitrosococcus wardiae spec. Nov.:enrichment, isolation, phylogenetic, and growth physiological characterization[J]. Frontiers in Microbiology, 2016, 7(512): 75. |
| [17] |
Ward B B, O'Mullan G D. Worldwide distribution of Nitrosococcus oceani, a marine ammonia-oxidizing γ-proteobacterium, detected by PCR and sequencing of 16S rRNA and amoA genes[J]. Applied and Environmental Microbiology, 2002, 68(8): 4153-4157. Doi:10.1128/AEM.68.8.4153-4157.2002 |
| [18] |
Jordan F L, Cantera J J L, Fenn M E, et al. Autotrophic ammonia-oxidizing bacteria contribute minimally to nitrification in a nitrogen-impacted forested ecosystem[J]. Applied and Environmental Microbiology, 2005, 71(1): 197-206. Doi:10.1128/AEM.71.1.197-206.2005 |
| [19] |
Venter J C, Remington K, Heidelberg J F, et al. Environmental genome shotgun sequencing of the Sargasso Sea[J]. Science, 2004, 304(5667): 66-74. Doi:10.1126/science.1093857 |
| [20] |
Schleper C, Jurgens G, Jonuscheit M. Genomic studies of uncultivated archaea[J]. Nature Reviews Microbiology, 2005, 3(6): 479-488. Doi:10.1038/nrmicro1159 |
| [21] |
Treusch A H, Leininger S, Kletzin A, et al. Novel genes for nitrite reductase and Amo-related proteins indicate a role of uncultivated mesophilic Crenarchaeota in nitrogen cycling[J]. Environmental Microbiology, 2005, 7(12): 1985-1995. Doi:10.1111/j.1462-2920.2005.00906.x |
| [22] |
Könneke M, Bernhard A E, de la Torre, José R, et al. Isolation of an autotrophic ammonia-oxidizing marine archaeon[J]. Nature, 2005, 437(7058): 543-546. Doi:10.1038/nature03911 |
| [23] |
Mincer T J, Church M J, Taylor L T, et al. Quantitative distribution of presumptive archaeal and bacterial nitrifiers in Monterey Bay and the North Pacific Subtropical Gyre[J]. Environmental microbiology, 2007, 9(5): 1162-1175. Doi:10.1111/j.1462-2920.2007.01239.x |
| [24] |
Wuchter C, Abbas B, Coolen M J L, et al. Archaeal nitrification in the ocean[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(33): 12317-12322. Doi:10.1073/pnas.0600756103 |
| [25] |
Martens-Habbena W, Berube P M, Urakawa H, et al. Ammonia oxidation kinetics determine niche separation of nitrifying archaea and bacteria[J]. Nature, 2009, 461(7266): 976-979. Doi:10.1038/nature08465 |
| [26] |
DeLong E F. Oceans of archaea[J]. ASM News, 2003, 69(10): 503-511. |
| [27] |
Delong E F. Everything in moderation:archaea as 'non-extremophiles'[J]. Current Opinion in Genetics & Development, 1998, 8(6): 649-654. |
| [28] |
Robertson C E, Harris J K, Spear J R, et al. Phylogenetic diversity and ecology of environmental archaea[J]. Current Opinion in Microbiology, 2005, 8(6): 638-642. Doi:10.1016/j.mib.2005.10.003 |
| [29] |
Brochier-Armanet C, Boussau B, Gribaldo S, et al. Mesophilic Crenarchaeota:proposal for a third archaeal phylum, the Thaumarchaeota[J]. Nature Reviews Microbiology, 2008, 6(3): 245-252. Doi:10.1038/nrmicro1852 |
| [30] |
张丽梅, 贺纪正. 一个新的古菌类群:奇古菌门(Thaumarchaeota)[J]. 微生物学报, 2012, 52(4): 411-421. |
| [31] |
Biller S J, Mosier A C, Wells G F, et al. Global biodiversity of aquatic ammonia-oxidizing archaea is partitioned by habitat[J]. Frontiers in Microbiology, 2012, 3: 252. |
| [32] |
Pester M, Rattei T, Flechl S, et al. amoA-based consensus phylogeny of ammonia-oxidizing archaea and deep sequencing of amoA genes from soils of four different geographic regions[J]. Environmental Microbiology, 2012, 14(2): 525-539. Doi:10.1111/j.1462-2920.2011.02666.x |
| [33] |
Arp D J, Chain P S, Klotz M G. The impact of genome analyses on our understanding of ammonia-oxidizing bacteria[J]. Annual Review Microbiology, 2007, 61(1): 503. Doi:10.1146/annurev.micro.61.080706.093449 |
| [34] |
Nicol G W, Schleper C. Ammonia-oxidizing Crenarchaeota:important players in the nitrogen cycle?[J]. Trends in Microbiology, 2006, 14(5): 207-212. Doi:10.1016/j.tim.2006.03.004 |
| [35] |
Glass J B, Orphan V J. Trace metal requirements for microbial enzymes involved in the production and consumption of methane and nitrous oxide[J]. Frontiers in Microbiology, 2012, 3: 61. |
| [36] |
Walker C B, de la Torre J R, Klotz M G, et al. Nitrosopumilus maritimus genome reveals unique mechanisms for nitrification and autotrophy in globally distributed marine crenarchaea[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(19): 8818-8823. Doi:10.1073/pnas.0913533107 |
| [37] |
Stahl D A, de la Torre J R. Physiology and diversity of ammonia-oxidizing archaea[J]. Annual Review of Microbiol, 2012, 66(1): 83-101. |
| [38] |
Vajrala N, Martens-Habbena W, Sayavedra-Soto L A, et al. Hydroxylamine as an intermediate in ammonia oxidation by globally abundant marine archaea[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(3): 1006-1011. Doi:10.1073/pnas.1214272110 |
| [39] |
Fuhrman J A, McCallum K, Davis A A. Novel major archaebacterial group from marine plankton[J]. Nature, 1992, 356(6365): 148-149. Doi:10.1038/356148a0 |
| [40] |
Delong E F. Archaea in coastal marine environments[J]. Proceedings of the National Academy of Sciences of the United States of America, 1992, 89(12): 5685-5689. Doi:10.1073/pnas.89.12.5685 |
| [41] |
Karner M B, Delong E F, Karl D M. Archaeal dominance in the mesopelagic zone of the Pacific ocean[J]. Nature, 2001, 409(6819): 507-510. Doi:10.1038/35054051 |
| [42] |
Teira E, Lebaron P, van Aken H, et al. Distribution and activity of bacteria and archaea in the deep water masses of the North Atlantic[J]. Limnology and Oceanography, 2006, 51(5): 2131-2144. Doi:10.4319/lo.2006.51.5.2131 |
| [43] |
Teira E, Reinthaler T, Pernthaler A, et al. Combining catalyzed reporter deposition-fluorescence in situ hybridization and microautoradiography to detect substrate utilization by bacteria and archaea in the deep ocean[J]. Applied and Environmental Microbiology, 2004, 70(7): 4411-4414. Doi:10.1128/AEM.70.7.4411-4414.2004 |
| [44] |
Francis C A, Roberts K J, Beman J M, et al. Ubiquity and diversity of ammonia-oxidizing archaea in water columns and sediments of the ocean[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(41): 14683-14688. Doi:10.1073/pnas.0506625102 |
| [45] |
Baker B J, Lesniewski R A, Dick G J. Genome-enabled transcriptomics reveals archaeal populations that drive nitrification in a deep-sea hydrothermal plume[J]. The ISME Journal, 2012, 6(12): 2269-2279. Doi:10.1038/ismej.2012.64 |
| [46] |
Hollibaugh J T, Gifford S, Sharma S, et al. Metatranscriptomic analysis of ammonia-oxidizing organisms in an estuarine bacterioplankton assemblage[J]. The ISME Journal, 2010, 5(5): 866-878. |
| [47] |
Stewart F J, Ulloa O, DeLong E F. Microbial metatranscriptomics in a permanent marine oxygen minimum zone[J]. Environmental Microbiology, 2012, 14(1): 23-40. Doi:10.1111/j.1462-2920.2010.02400.x |
| [48] |
Kelly D. Bioenergetics of chemolithotrophic bacteria[M]. London: InCompanion to Microbiology, 1978: 363-386.
|
| [49] |
Blainey P C, Mosier A C, Potanina A, et al. Genome of a low-salinity ammonia-oxidizing archaeon determined by single-cell and metagenomic analysis[J]. PLOS ONE, 2011, 6(2): e16626. Doi:10.1371/journal.pone.0016626 |
| [50] |
Hallam S J, Konstantinidis K T, Putuam N, et al. Genomic analysis of the uncultivated marine crenarchaeote cenarchaeum symbiosum[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(48): 18296-18301. Doi:10.1073/pnas.0608549103 |
| [51] |
Spang A, Poehlein A, Offre P, et al. The genome of the ammonia-oxidizing Candidatus nitrososphaera gargensis: insights into metabolic versatility and environmental adaptations[J]. Environmental Microbiology, 2012, 14(12): 3122. Doi:10.1111/j.1462-2920.2012.02893.x |
| [52] |
Tourna M, Stieglmeier M, Spang A, et al. Nitrososphaera viennensis, an ammonia oxidizing archaeon from soil[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(20): 8420-8425. Doi:10.1073/pnas.1013488108 |
| [53] |
Könneke M, Schubert D M, Brown P C, et al. Ammonia-oxidizing archaea use the most energy-efficient aerobic pathway for CO2 fixation[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(22): 8239-8244. Doi:10.1073/pnas.1402028111 |
| [54] |
Bergauer K, Sintes E, van Bleijswijk J, et al. Abundance and distribution of archaeal acetyl-CoA/propionyl-CoA carboxylase genes indicative for putatively chemoautotrophic archaea in the tropical Atlantic's interior[J]. FEMS Microbiology Ecology, 2013, 84(3): 461-473. Doi:10.1111/1574-6941.12073 |
| [55] |
Hu A, Jiao N, Zhang C L. Community structure and function of planktonic Crenarchaeota: changes with depth in the South China Sea[J]. Microbial Ecology, 2011a, 62(3): 549-563. Doi:10.1007/s00248-011-9866-z |
| [56] |
Hu A, Jiao N, Zhang R, et al. Niche partitioning of marine group Crenarchaeota in the euphotic and upper mesopelagic zones of the east china sea[J]. Applied and Environmental Microbiology, 2011b, 77(21): 7469-7478. Doi:10.1128/AEM.00294-11 |
| [57] |
Song Z Q, Wang L, Wang F P, et al. Abundance and diversity of archaeal accA gene in hot springs in Yunnan Province, China[J]. Extremophiles, 2013, 17(5): 871-879. Doi:10.1007/s00792-013-0570-4 |
| [58] |
Yakimov M M, Cono V L, Denaro R. A first insight into the occurrence and expression of functional amoA and accA genes of autotrophic and ammonia-oxidizing bathypelagic Crenarchaeota, of Tyrrhenian Sea[J]. Deep Sea Research Part II:Topical Studies in Oceanography, 2009, 56(11/12): 748-754. |
| [59] |
Yakimov M M, Cono V L, Smedile F, et al. Contribution of crenarchaeal autotrophic ammonia oxidizers to the dark primary production in Tyrrhenian deep waters (Central Mediterranean Sea)[J]. The ISME Journal, 2011, 5(6): 945-961. Doi:10.1038/ismej.2010.197 |
| [60] |
P A del Giorgio, Duarte C M. Respiration in the open ocean[J]. Nature, 2002, 420(6914): 379-384. Doi:10.1038/nature01165 |
| [61] |
Carlson C A, Ducklow H W, Michaels A F. Annual flux of dissolved organic carbon from the euphotic zone in the Northwestern Sargasso Sea[J]. Nature, 1994, 371(6496): 405-408. Doi:10.1038/371405a0 |
| [62] |
Reinthaler T, van Aken H, Veth C, et al. Prokaryotic respiration and production in the meso-and bathypelagic realm of the eastern and western North Atlantic Basin[J]. Limnology and Oceanography, 2006, 51(3): 1262-1273. Doi:10.4319/lo.2006.51.3.1262 |
| [63] |
Karl D M, Knauer G A, Martin J H, et al. Bacterial chemolithotrophy in the ocean is associated with sinking particles[J]. Nature, 1984, 309(5963): 54-56. Doi:10.1038/309054a0 |
| [64] |
Middelburg J J. Chemoautotrophy in the ocean[J]. Geophysical Research Letters, 2011, 38(24): L26404.. |
| [65] |
Ingalls A E, Shah S R, Hansman R L, et al. Quantifying archaeal community autotrophy in the mesopelagic ocean using natural radiocarbon[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(17): 6442-6447. Doi:10.1073/pnas.0510157103 |
| [66] |
Hansman R L, Griffin S, Watson J T, et al. The radiocarbon signature of microorganisms in the mesopelagic ocean[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(16): 6513-6518. Doi:10.1073/pnas.0810871106 |
| [67] |
Herndl G J, Reinthaler T, Teira E, et al. Contribution of archaea to total prokaryotic production in the deep Atlantic ocean[J]. Applied Environmental Microbiology, 2005, 71(5): 2303-2309. Doi:10.1128/AEM.71.5.2303-2309.2005 |
| [68] |
Tetu S G, Breakwell K, Elbourne L D H, et al. Life in the dark:metagenomic evidence that a microbial slime community is driven by inorganic nitrogen metabolism[J]. The ISME Journal, 2013, 7(6): 1227-1236. Doi:10.1038/ismej.2013.14 |
| [69] |
Reinthaler T, van Aken H M, Herndl G J. Major contribution of autotrophy to microbial carbon cycling in the deep North Atlantic Basin[J]. Deep Sea Research Part II:Topical Studies in Oceanography, 2010, 57(16): 1572-1580. Doi:10.1016/j.dsr2.2010.02.023 |
| [70] |
Jiao N, Herndl G J, Hansell D A, et al. Microbial production of recalcitrant dissolved organic matter:long-term carbon storage in the global ocean[J]. Nature Reviews Microbiology, 2010, 8(8): 593-599. Doi:10.1038/nrmicro2386 |
| [71] |
Jiao N, Herndl G J, Hansell D A, et al. The microbial carbon pump and the oceanic recalcitrant dissolved organic matter pool[J]. Nature Reviews Microbiology, 2011, 9(7): 555. |
 2020, Vol. 37
2020, Vol. 37