2. 中国科学院大学化学科学学院, 北京 100049;
3. 广东工业大学材料与能源学院, 广州 510006
2. College of Chemical Sciences, University of Chinese Academy of Sciences, Beijing 100049, China;
3. College of Materials and Energy, Guangdong University of Technology, Guangzhou 510006, China
近年来,生物功能的人工控制因在细胞生物学、药理学和生物纳米技术中的广泛应用而受到关注。在基因表达的人工控制方面,人们通常应用光、热、电场和pH等各种外界刺激来实现。光由于非入侵的方式、时间和空间上的高分辨及无污染等特点在生物应用中有着空前的优势。近年来,光已经被广泛地应用到研究化学生物学、生命科学和医学等各个领域, 尤其是利用光敏感核酸作为新的研究工具调控基因的功能, 极大地促进了基因功能分子学研究的发展,为后续的基因表达网络的构建奠定基础。
近年来,偶氮苯凭借着化学结构稳定、光照后迅速发生异构化等特点,成为光异构化分子的典型代表之一[1-2]。以反式构型(trans)为母体,由于该结构趋于平面结构,因此其热力学性质较其他结构更为稳定,与此同时,在分别施加可见光(>400 nm)以及紫外光(UV < 366 nm)的刺激后,前者能够顺利由顺式构型(cis)变化为trans构型,后者则能够转变为cis构型,其具备的基本特征为稳定性较低[3-5]。受上述特征的影响,偶氮苯类化合物得到广泛应用,在多个领域均发挥出重要作用[6-10]。
将偶氮苯引入到核酸分子中, 从时间和空间上调控生物化学、医学和生理体系方面也引起了科研工作者的广泛兴趣及重点关注。因此,这提供了一条可行性较高的核酸结构改造途径,即插入光响应分子实现核酸结构及其性能的改变,如引入到核糖体、核酸骨架等位置。通过上述方式改造核酸,进而实现对基因转录、核糖核酸酶的有效控制等[11-25]。另外,Tsai等[26]通过光-生物正交配体束缚在时空上控制蛋白质活性的光学切换,利用光快速调节活哺乳动物细胞中蛋白质功能的选择性。Reis等[27]使用可见光实现基于人组蛋白脱乙酰基酶光致变色抑制剂的表观遗传机制的高分辨率,设计一种表观遗传调控转录的化学光学调制探针,可以转化为需要有条件和选择性表观基因组调节的疾病的新治疗策略。Kamiya等[28]使用的光响应性T7启动子和偶氮苯衍生物的化学结构将光开关分子插入T7启动子中的指定位置,在具有可见光的无细胞翻译系统中实现绿色荧光蛋白产生的有效光调节。Bian等[29]设计一种新型可重复使用的环糊精修饰表面,利用环糊精和偶氮苯之间的主客体相互作用实现光交换特定细胞释放。
我们针对本研究进行积极的前期探索,并取得了一定成绩,目前已顺利实现4,4′-二羟甲基偶氮苯的发夹DNA开关的合成。该研究表明,发夹DNA的稳定性会在4,4′-二羟甲基偶氮苯的光异构化的作用下发生变化,在这一过程中需要对反应温度进行控制(Δtm=24 ℃)[30-31],另外,上述操作同样能够影响RNase H酶作用的降解,主要通过将其引入反义核酸两端来实现。此外,还研究了DNA引物延伸相关的聚合酶链反应的光调控。在这一过程中,需要重点关注一个因素,即稳定双链是否能够形成。因此,需要插入保护链,形成稳定的发夹结构,确保不发生引物延伸现象。通过紫外光照处理,可以脱去DNA模板的保护链,为后续引物结合操作创造条件,确保引物延伸能够顺利实现[32]。通过上述实验操作保证实验结果的准确性与可重复性。
本文提出对生命活动过程中核酸转录进行光调控,通过对DNA双链的非模板链进行偶氮苯修饰,研究T7 RNA聚合酶催化下其指导的RNA转录反应的光调控可行性。参考常规含T7启动子DNA模板研究RNA的转录,将偶氮苯引入非模板链的启动子区域末端,然后分别连接5、7、9、11 nt的4种长度保护链。根据我们以前的研究,反式的偶氮苯使得非模板链形成发夹结构,而紫外光照后的顺式偶氮苯打破非模板的发夹结构。我们希望通过顺反异构化改变偶氮苯周围的DNA双链体的局部结构,影响DNA与酶之间的相互作用,从而光调节RNA的转录。此外,还通过改变在启动子末端的保护链的长度详细研究光调节效率,结果发现较短长度修饰的偶氮苯保护链启动子可以实现明确的光调节,进一步解释了DNA模板双链体的解离或双链体的局部结构影响光调节的机制。
1 实验部分 1.1 材料与试剂选用分析纯试剂药品。实验所用常规药品均购自于北京化工厂,主要有:石油醚(PE)、三乙胺(TEA)、无水硫酸钠(Na2SO4)、乙酸乙酯(EA)、氯化钠(NaCl)、200~300目硅胶、氢氧化钠(NaOH);自芜湖华仁科技有限公司购入二氯甲烷(DCM)、乙腈(C2H3N)、四氢呋喃(THF)等,主要用于无水反应,以及标准脱氧核苷亚磷酰胺单体用于DNA合成;自生工科技有限公司购入转录试剂、引物等分子试剂;自Thermo Fisher购入SYBR gold核酸染色剂;自宝如亿科技有限公司购入T7 RNA聚合酶。
1.2 主要仪器与设备主要应用到的仪器与设备有:1260高效液相色谱系统(安捷伦)、400 M核磁共振仪(AVANCE)、ChemiDoc XRS高灵敏化学发光成像系统、DNA合成仪(AppliedBiosystems)、UV1800紫外分光光度计(岛津)。
1.3 偶氮苯衍生物修饰DNA的固相合成与纯化首先向合成柱中装入Universal-CPG,装入量为35 mg左右,即1 μmol,主要以CPG载体为依据。然后从5′到3′编辑目标寡聚核苷酸序列,主要依据为雅磷酰胺单体的装载位置规律。将DNA核苷亚磷酰胺单体由3′端向5′端向固相CPG的连接,均采取常规合成方法,具体参照相关操作手册,在这一过程中使用的设备为ABI 394DNA/RNA固相合成仪。最后,想要顺利得到目标序列的固相,需要实现在CPG的寡聚核酸上亚磷酰胺单体的偶联,每个单体均需按照特定的顺序完成偶联。具体参照如下步骤:首先利用2 mL的离心管盛装固相CPG,向其注入1 mL浓氨水;其次为氨解过程,该过程温度需控制在50 ℃以内,顺利实现固相切除;然后进行离心处理,为HPLC分离纯化提供条件,离心力为1 000~13 000 r·min-1。本实验液相纯化DNA采用安捷伦反相C18制备柱,参照之前发表的液相色谱条件进行分离纯化,最后取得目标溶液做脱DMT、沉降除盐处理,最后进行ESI-MS质谱鉴定,应用的DNA为400 pmol。
1.4 偶氮苯修饰的DNA光异构化首先利用1×PBS溶解T7NC到T7NC3序列,配置溶液浓度为2.5 μmol/L;其次为退火处理,时间为5 min,温度为95 ℃;然后进行紫外照射,容器为石英比色皿,照射条件为365 nm, 7 mW/cm2。按照20 s的时间间隔分别测定吸光度,共计测量时间为2 min。再采取白光灯照射,照射条件为>400 nm,11 W。紫外照射为反式到顺式,白光灯照射为顺式到反式,吸光度测量时间间隔均为20 s,分析软件选用Origin 8.0。
1.5 修饰后的模板指导的转录将模板链和非模板链(2:1)均匀混合,与预配好的10×RNA Thermopol反应溶液混合,溶液规格为2 μL;添加NTP,溶液规格为1 μL;进行退火处理,时间为5 min,温度为95 ℃。退火结束后需要自然冷却,设置两组实验,变量为紫外照射和不光照,前者的处理时间为5 min,为了实现后续的充分结合,进行10 min的孵育操作,孵育条件为无光照。完成上述处理后向体系内加入50 U/μL T7 RNA聚合酶,在此基础上进行涡旋振荡处理,转录产物的获得,需经历2 h的孵育过程,温度保持在37 ℃。将预配好的上样缓冲液加入体系内,二者为1:1的体积比,充分混匀后继续孵育,孵育时间为5 min,孵育温度为90 ℃。预配好20%变性聚丙烯酰胺凝胶(内含7 mol/L尿素),样品量为10 mL。接下来选择1×TBE缓冲液进行聚丙烯凝胶电泳操作。跑完电泳后用SYBR gold染色,并进行成像分析。
2 结果与讨论 2.1 偶氮苯修饰转录模板的设计,合成及结构鉴定在本研究中,合成了偶氮苯修饰的非模板链(non-template)及模板链(template),前期操作主要以常规T7模板转录序列为参考,目的在于深入分析偶氮苯修饰所起到的作用。在模板中引入T7启动子序列和转录序列,主要目的在于满足T7 RNA聚合酶高依赖性的要求。值得一提的是,T7启动子序列具备两个功能区,RNAP识别区(-11~-7位点)和解旋区(-4~-1位点),即结合位点以及解螺旋位点。具体参照图 1。

|
Download:
|
| 图 1 转录所用DNA模板和偶氮苯修饰的非模板DNA序列 Fig. 1 DNA template for transcription and non-template DNA sequence modified with azobenzene | |
将偶氮苯修饰在非模板链上,在T7 Promoter区通过偶氮苯引入保护短链形成发夹结构,而紫外光照后的偶氮苯转为顺式,非模板的发夹结构稳定性降低,将影响DNA模板与T7 RNA聚合酶之间的相互作用,从而调节RNA的转录活性。考虑到保护链的长度可能影响其转录活性,因而末端保护链设计长度选择了如下4个规格,分别为5、7、9、11 nt,分别对应于T7NC、T7NC1、T7NC2和T7NC3,见图 1。
按照前期研究建立的方法合成偶氮苯衍生物[32],产物为4,4′-二羟甲基偶氮苯,其两端羟基分别进行DMT保护反应以及合成偶氮苯亚磷酰胺单体,从而可进一步连接在核酸链中。利用ABI 394 DNA合成仪进行偶氮苯修饰DNA合成,分别进行切除并脱保护以及HPLC分离操作。鉴定结果具体参照表 1。
|
|
表 1 偶氮苯修饰的非模板DNA的ESI-MS鉴定 Table 1 ESI-MS identification of azobenzene modified non-template DNA |
将偶氮苯修饰的DNA(T7NC、T7NC1、T7NC2和T7NC3)利用1×PBS溶解,溶液浓度为2.5 μmol/L,其次为退火处理,时间为5 min,温度为95 ℃。没有光照时,偶氮苯-核酸的共轭体T7NC中偶氮苯呈现热稳定的反式形式,其紫外光谱在335 nm(π-π*)处呈现肩峰。分别进行紫外灯照射处理以及白光光照处理,得到图 2(a)和2(b)所示的峰图。由图可以观察到,增色效应较为显著的波长为260 nm。由于DNA链长度增加,偶氮苯浓度相对减少,导致其紫外特征峰偏低,因而在335 nm处吸光度普遍偏低,而在430 nm(n-π*)处几乎不可见。另外,还可以观测到在上述两个光照条件下,T7NC1、T7NC2和T7NC3均有类似峰变化趋势,具体分别见图 2(c)、2(d),图 2(e)、2(f)和图 2(g)、2(h)。可知,偶氮苯被修饰到寡核苷酸中,能起到光异构化的作用。此外,偶氮苯修饰的DNA通过365 nm的紫外光照120 s即可达到主要含有顺式结构的光稳态,而可见光照120 s它们将从顺式返回原始的反式结构。

|
Download:
|
| (a)和(b),(c)和(d),(e)和(f),(g)和(h)分别対应于T7NC、T7NC1、T7NC2和T7NC3紫外可见光照下紫外光谱的变化。 图 2 偶氮苯修饰的DNA在不同时间的紫外和可见光照下紫外光谱变化 Fig. 2 Changes of UV spectra of azobenzene-modified DNA under irradiations of different durations with UV and visible light | |
T7 RNA聚合酶能够催化rNTP的5′-磷酸基与下一个rNTP的3′-OH之间磷酸二酯键的建立,如此一来,便顺利实现了新生RNA链的延伸。接下来,进一步探讨T7 RNA聚合酶催化下偶氮苯修饰非模板DNA调控转录的可行性,分别设置5、7、9、11 nt长度的保护链,通过上述保护链发挥调控作用。T7启动子包括两个功能区域,其中,-11~-7是T7 RNA聚合酶的结合区域(图 1)。在酶结合区域的配对过程中,T7NC1、T7NC2和T7NC3这3个序列的保护链均发挥了重要作用。
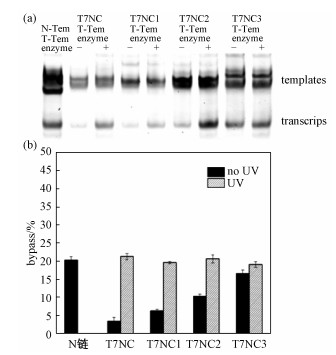
图 3是模板链和非模板链(template,T7NC,T7NC1,T7NC2和T7NC3)1:1混合后T7 RNA聚合酶催化15 min的结果。图 3(a),1通道是正常的非模板链和模板链1:1混合后,在T7 RNA聚合酶催化下RNA转录的结果。2、4、6、8通道是T7NC、T7NC1、T7NC2和T7NC3与模板链混合后没有紫外光照时,在T7 RNA聚合酶催化下的结果,而3、5、7、9通道是T7NC、T7NC1、T7NC2和T7NC3与模板链混合后紫外光照后酶催化下的结果。显然,对于含T7NC的模板DNA几乎没有发生RNA转录(图 3(a),2通道),而光照后,有明显的转录产物生成(图 3(a),3通道)。通过定量分析(图 3(b)),发现RNA转录产物从光照前3.4%增加到光照后21.4%,大约有6.25倍的变化。同样,对于含有T7NC1、T7NC2和T7NC3的DNA模板,紫外光照后其指导的RNA转录产物也有所增加(图 3(a),5通道对4通道,7通道对6通道,9通道对8通道),增加的效率分别为4、3、1.25倍。因此,非模板链上通过偶氮苯连接5个碱基的发夹结构与模板链组合指导的RNA转录具有最佳的光控效果。这是因为光照前反式偶氮苯使得非模板链上保护链与其结合力很强,阻碍了非模板链与模板链的结合,从而影响T7 RNA聚合酶与模板DNA的结合,所以影响了RNA转录活性。而紫外光照后,形成的顺式偶氮苯使得非模板链上发夹结构稳定性减弱,非模板链利于与模板链结合,有利于T7 RNA聚合酶的结合,从而恢复RNA转录活性。值得注意的是,随着非模板链上保护链的长度增加(T7NC、T7NC1、T7NC2和T7NC3),光照前后RNA转录产物增加的程度减小,这可能由于非模板上保护链的长度越长,往往对应着更高的发夹结构稳定性,且该稳定性不会在光照条件下改变,也就不会引起T7 RNA聚合酶与DNA模板结合的改变。与Liu等[33]报道的模板侧链引入偶氮苯单元控制RNA转录活性比较,本文通过单一偶氮苯修饰在T7启动子末端,紫外光照引发更有效地调控RNA转录活性,而他们通过多个偶氮苯修饰提高调控效率,但也将不可避免地需要长时间的紫外光照。
|
Download:
|
| 图 3 偶氮苯修饰非模板链(N-Tem)和模板形成的双链在T7 RNA聚合酶催化15 min后RNA转录的变性胶分析 Fig. 3 Denatured gel analysis of azo-modified non-template strands (N-Tem) and template-derived double strands after 15 min of T7 RNA polymerase catalyzed RNA transcription | |
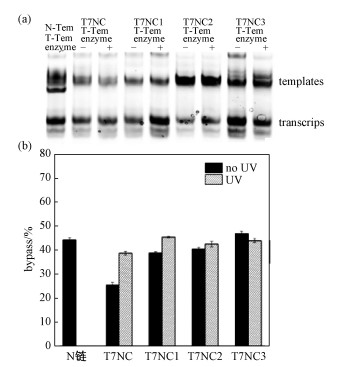
对于偶氮苯修饰的模板与正常的转录模板的转录过程,随时间变化进行分析。发现,随着时间的延长,RNA的转录产物也将增多。如图 4,反应时间为30 min时,含T7NC序列的DNA模板对RNA转录仍然表现出一定的光控制能力,光照前后差异达到1.56倍。含T7NC1序列的DNA模板对RNA转录在光照后增加为1.15倍。而对于含T7NC2和T7NC3序列的DNA模板,转录产物光照前后几乎无明显差异。
|
Download:
|
| 图 4 偶氮苯修饰非模板链(N-Tem)和模板形成的双链在T7 RNA聚合酶催化30 min后RNA转录的变性胶分析 Fig. 4 Denatured gel analysis of azo-modified non-template strands (N-Tem) and template-derived double strands after 30 min of T7 RNA polymerase catalyzed RNA transcription | |
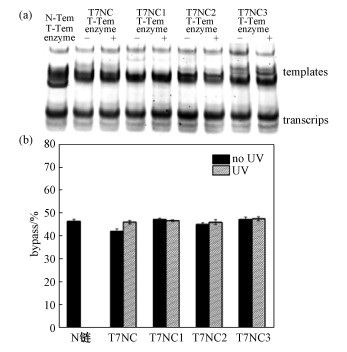
当酶催化反应时间延长为45 min时,会出现图 5所示的结果,即转录产率大致相同。紫外光照前后转录效果没有明显变化,可能是随着时间的推移,偶氮苯异构化会导致模板DNA双螺旋结构被破坏,在此结构打开的过程中顺利实现了模板的插入。如此一来,启动子序列会被释放,进而转向连续延伸模式。转录活动能够在上述条件下正常进行,主要原因在于模板配对的氢键作用不会受到偶氮苯保护链结构的决定性影响。
|
Download:
|
| 图 5 偶氮苯修饰非模板链(N-Tem)和模板形成的双链在T7 RNA聚合酶催化45 min后RNA转录的变性胶分析 Fig. 5 Denatured gel analysis of azo-modified non-template strands (N-Tem) and template-derived double strands after 45 min of T7 RNA polymerase catalyzed RNA transcription | |
对与偶氮苯修饰的DNA模板指导的RNA转录的时间变化研究表明,RNA转录活性光调控效果主要归结于催化效率的调控,这也与文献[30-34]报道的结果相一致。
3 结论在本研究中,主要针对偶氮苯单元修饰在DNA模板上对RNA转录的光控制行为进行系统而深入的探讨,首先进行保护链的筛选,即5、7、9、11个碱基,然后与DNA模板相连接,进行两个不同光照条件的对比,探究其转录效率的变化趋势。研究显示,光控转录效果最好的一组模板为含T7NC序列DNA模板。但是随着时间的延长,光控效果明显减弱,说明T7 RNA聚合酶的转录机理限制时间上的调控。
| [1] |
Beharry A A, Woolley G A. Azobenzene photoswitches for biomolecules[J]. Chemical Society Reviews, 2011, 40(8): 4422-4437. |
| [2] |
Szymanski W, Beierle J M, Kistemaker H A, et al. Reversible photocontrol of biological systems by the incorporation of molecular photoswitches[J]. Chemical Reviews, 2013, 113(8): 6114-6178. |
| [3] |
Kumar G S, Neckers D C. Photochemistry of azobenzene -containing polymers[J]. Chemical Reviews, 1989, 89(8): 1915-1925. |
| [4] |
Liu Z F, Hashimoto K, Fujishima A. Photoelectro -chemical information-storage using an azobenzene derivative[J]. Nature, 1990, 347(6294): 658-660. |
| [5] |
Zimmerman G, Chow L Y, Paik U J. The photochemical isomerization of azobenzene[J]. Journal of the American Chemical Society, 1958, 80(14): 3528-3531. |
| [6] |
Yan Y, Chen J I, Ginger D S. Photoswitchable oligonucleotide-modified gold nanoparticles:controlling hybridization stringency with photon dose[J]. Nano Letter, 2012, 12(5): 2530-2536. |
| [7] |
Yang Y, Endo M, Hidaka K, et al. Photo-controllable DNA origami nanostructures assembling into predesigned multiorientational patterns[J]. Journal of the American Chemical Society, 2012, 134(51): 20645-20653. |
| [8] |
Lohmann F, Ackermann D, Famulok M. Reversible light switch for macrocycle mobility in a DNA rotaxane[J]. Journal of the American Chemical Society, 2012, 134(29): 11884-11887. |
| [9] |
Sahoo J P, Sudhir Kumar. Medicinal interest of AZO-based organic compounds:a review[J]. Asian Journal of Pharmaceutical and Clinical Research, 2016, 9(1): 33-39. |
| [10] |
Jones M W, Mantovani G, Blindauer C A, et al. Direct peptide bioconjugation/PEGylation at tyrosine with linear and branched polymeric diazonium salts[J]. Journal of the American Chemical Society, 2012, 134(17): 7406-7413. |
| [11] |
Yan Y, Wang X, Chen J I, et al. Photoisomerization quantum yield of azobenzene-modified DNA depends on local sequence[J]. Journal of the American Chemical Society, 2013, 135(22): 8382-8387. |
| [12] |
Nakasone Y, Ooi H, Kamiya Y, et al. Dynamics of inter-DNA chain interaction of photoresponsive DNA[J]. Journal of the American Chemical Society, 2016, 138(29): 9001-9004. |
| [13] |
Kingsland A, Samai S, Yan Y, et al. Local density fluctuations predict photoisomerization quantum yield of azobenzene-modified DNA[J]. Journal of Physical Chemistry Letters, 2016, 7(15): 3027-3031. |
| [14] |
Chaulk S G, MacMillan A M. Caged RNA:photo-control of a ribozyme reaction[J]. Nucleic Acids Res, 1998, 26(13): 3173-3178. |
| [15] |
Liang X, Asanuma H, Komiyama M. Photoregulation of DNA triplex formation by azobenzene[J]. Nucleic Acids Research, 2002, 124(9): 1877-1883. |
| [16] |
Liu M, Asanuma H, Komiyama M. Azobenzene-tethered T7 promoter for efficient photoregulation of transcription[J]. Journal of the American Chemical Society, 2006, 128(3): 1009-1015. |
| [17] |
Matsunaga D, Asanuma H, Komiyama M. Photoregulation of RNA digestion by RNase H with azobenzene-tethered DNA[J]. Journal of the American Chemical Society, 2004, 126: 11452-11453. |
| [18] |
Wang X, Huang J, Zhou Y, et al. Conformational switching of G-quadruplex DNA by photoregulation[J]. Angewandte Chemie International Edition, 2010, 49(31): 5305-5309. |
| [19] |
Beharry A A, Sadovski O, Woolley G A. Azobenzene photoswitching without ultraviolet light[J]. Journal of the American Chemical Society, 2011, 133(49): 19684-19687. |
| [20] |
Dong M, Babalhavaeji A, Samanta S, et al. Red-shifting azobenzene photoswitches for in vivo use[J]. Accounts of Chemical Research, 2015, 48(10): 2662-2670. |
| [21] |
Nishioka H, Liang X, Asanuma H. Effect of the ortho modification of azobenzene on the photoregulatory efficiency of DNA hybridization and the thermal stability of its cis form[J]. Chemistry, 2010, 16(7): 2054-2062. |
| [22] |
Poloni C, Szymanski W, Hou L, et al. A fast, visible-light-sensitive azobenzene for bioorthogonal ligation[J]. Chemistry, 2014, 20(4): 946-951. |
| [23] |
Bock L C, Griffin L C, Latham J A, et al. Selection of single-stranded-dna molecules that bind and inhibit human thrombin[J]. Nature, 1992, 355(6360): 564-566. |
| [24] |
Riccardi C, Russo Krauss I, Musumeci D, et al. Fluorescent thrombin binding aptamer-tagged nanoparticles for an efficient and reversible control of thrombin activity[J]. ACS Applied Materials & Interfaces, 2017, 9(41): 35574-35587. |
| [25] |
Thevarpadam J, Bessi I, Binas O, et al. Photo-responsive formation of an intermolecular minimal G-quadruplex motif[J]. Angewandte Chemie International Edition, 2016, 55(8): 2738-2742. |
| [26] |
Tsai Y H, Essig S, James J R, et al. Selective, rapid and optically switchable regulation of protein function in live mammalian cells[J]. Nature Chemistry, 2015, 7(7): 554-561. |
| [27] |
Reis S A, Ghosh B, Hendricks J A, et al. Light-controlled modulation of gene expression by chemical optoepigenetic probes[J]. Nature Chemistry Biololgy, 2016, 12(5): 317-323. |
| [28] |
Kamiya Y, Takagi T, Ooi H, et al. Synthetic gene involving azobenzene-tethered T7 promoter for the photocontrol of gene expression by visible light[J]. ACS Synthetic Biology, 2015, 4(4): 365-370. |
| [29] |
Bian Q, Wang W, Wang S, et al. Light-triggered specific cancer cell release from cyclodextrin/azobenzene and aptamer-modified substrate[J]. ACS Applied Materials & Interfaces, 2016, acsami.6b09734. |
| [30] |
Wu L, Wu Y, Jin H, et al. Photoswitching properties of hairpin ODNs with azobenzene derivatives at the loop position[J]. Med Chem Comm, 2015, 6: 461-468. |
| [31] |
孔德佳, 莫蒙武, 季禾茗, 等. 偶氮苯作用的核酸开关在生物体内的可逆光调控[J]. 中国科学:化学, 2018, 48(7): 698-711. |
| [32] |
季禾茗, 孔德佳, 莫蒙武, 等. 偶氮苯修饰的DNA对引物延伸的光调控[J]. 中国科学院大学学报, 2018, 36(1): 470-479. |
| [33] |
Liu M, Asanuma H, Komiyama M. Azobenzene-tethered T7 promoter for efficient photoregulation of transcription[J]. Journal of the American Chemical Society, 2006, 128(3): 1009-1015. |
| [34] |
Liang X, Wakuda R, Fujioka K, et al. Photoregulation of DNA transcription by using photoresponsive T7 promoters and clarification of its mechanism[J]. The FEBS Journal, 2010, 277(6): 1551-1561. |
 2020, Vol. 37
2020, Vol. 37 


