2. 中国科学院大学化学科学学院, 北京 100049;
3. 江西科技师范大学药学院, 南昌 330013
2. College of Chemical Sciences, University of Chinese Academy of Sciences, Beijing 100049, China;
3. College of Pharmacy, Jiangxi Science and Technology Normal University, Nanchang 330013, China
人工操纵特定的生命过程或基因功能一直是生命科学的前沿研究方向[1-3]。以往的报道是利用小分子抑制剂来抑制特定酶的活性从而调控生命过程,即在生命体中引入外源小分子,但外源小分子在一进入生命体后就会立即开始发挥作用,因此很难在时间和空间上实现选择性调控[4]。近些年来,科研工作者曾尝试多种方法以实现对生命过程的多维度调控,例如改变温度、改变pH、改变电场、施加磁场、给予光照等。与传统的外界刺激相比,光作为刺激手段具有独特的优势[5]。首先光是一种清洁的、非入侵式的外界刺激,已广泛应用于生物医学领域的光学照明和荧光成像。其次,光可以精确地从时间和空间上进行宏观调控,在光敏物质抵达特定位置后,通过特定波长的光照控制药物的释放。最后,光控释放的效率较高,特定波长的光可激发某些功能基团并发生各种光化学反应(化学键的断裂和异构化等)。
使用光反应变色分子调控生物过程具有极好的发展潜力,在化学生物学、药物研究和医学具有深远的意义。我们和其他研究组曾使用光笼的化合物修饰的核酸,实现生物体内基因表达的光控制[6-9]。但是这种方法只能单次地增加或降低核酸的活性,不能可逆地控制核酸的功能。偶氮苯是光异构化分子的典型代表[10-11],化学结构稳定,光照后异构化迅速。母体的反式构型(trans)几乎是平面结构且热力学相对稳定,紫外光(UV < 366 nm)照后形成稳定性较低的具有弯曲构象的顺式形态(cis),而在可见光(>400 nm)光照刺激下又可实现cis构象向trans构象的转变[12-14]。基于这一特性,偶氮苯类化合物近年来被广泛地应用在各种材料领域,例如,光控DNA自组装[15-16]、光制动纳米机械[17]、纳米药物输送材料[18]等。
近年来,随着偶氮类化合物光控DNA动力学的基础研究得到迅速的发展[19-21],进一步增加了人们使用这一工具调控功能生物大分子(核酸和蛋白质)的信心[22-23]。这种光响应分子通常被引入到核酸不同的结构位置上,例如,磷酸骨架、碱基和核糖体部分等,改变核酸的结构和结合性能,从而将其应用于光开关RNA沉默、基因转录的可逆控制、适配体的可逆识别、核糖核酸酶的可逆控制等[24-28]。
新颖的或改进的偶氮苯衍生物的设计对于核酸的化学修饰也是许多化学工作者争相研究的热点[29-30]。在前期研究中,我们已经设计合成4,4′-二羟甲基偶氮苯的发夹DNA开关,发现4,4′-二羟甲基偶氮苯的光异构化使得发夹DNA的稳定性发生巨大改变(ΔTm=24 ℃)[31-32],并将其置于反义核酸的两端形成哑铃型核酸,成功地光控制靶向RNA在RNase H酶作用的降解[33]。从时间和空间上光控制复制[34-36]、转录[37-38]和翻译[3, 33, 39-40]过程使得核酸工具分子的发展越来越引起人们的关注。这里,我们研究DNA引物延伸相关的聚合酶链反应的光调控。聚合酶链反应用于特定DNA序列的试管分离和指数扩增,引物和模板能否形成适当稳定的双链是反应的关键因素。如示意图 1,我们在DNA模板与引物的结合区通过4,4′-二羟甲基偶氮苯的连接加入一段保护链,由于反式的偶氮苯与邻近的碱基对之间堆积作用,DNA模板自身形成稳定的发夹结构而使得其与引物不能够互补结合,从而引物延伸不能发生。反之,通过紫外光照偶氮苯分子从反式转变为顺式,这种异构化作用所产生的扭转力使得DNA模板的保护链离去,从而DNA模板可以结合引物,在聚合酶的作用下能够使得引物延伸进行。对比骨架楔入型偶氮苯的设计[34],通过偶氮苯共价键连接模板的保护链具有可逆性,并且单一位点的修饰不影响正常的生理活动。考虑到发夹DNA的模板与引物和茎短链之间存在结合的竞争,我们使用25 mer长度的DNA模板和不同长度的保护链组成的发夹DNA,通过筛选发夹DNA的长度分别为5、6、7和8个碱基长度的保护链,以及长度为12,15和17 mer的引物,发现能够高效调控引物延伸的发夹DNA和引物组合,为从时间和空间上动力学控制生理条件下生物活性分子提供非常有用的工具,也将为分子水平上研究基因功能、基因表达网络以及疾病的发生和发展提供一种新的策略和研究手段。
|
Download:
|
| 图 1 偶氮苯亚磷酰胺单体的合成 Fig. 1 Synthetic procedure of azobenzene phosphoramidite | |
实验所用的药品是分析纯。所有溶液用超纯水配制。对硝基苯甲醇,4,4′-二甲氧基三苯基氯甲烷,四氮唑,双(二异丙基氨基)(2-氰基乙氧基)膦均购自偶合公司;锌粉,冰醋酸,四氢呋喃购自Macklin公司;色谱级乙腈购自阿拉丁公司;4种脱氧核苷购自芜湖华仁科技有限公司。引物订购自生工公司;DNA聚合酶订购自NEB。
1.2 主要仪器设备仪器使用AVANCE的Ⅲ型400 M核磁共振仪;Applied Biosystems的ABI 394型DNA合成仪;安捷伦1260高效液相色谱系统;岛津UV1800紫外分光光度计和BIO-RAD的ChemiDoc XRS高灵敏化学发光成像系统。
1.3 偶氮苯衍生物的合成 1.3.1 合成4,4′-二羟甲基偶氮苯参考前期的工作[31]。称取6.0 g(26 mmol)对硝基苯甲醇,配制并加入70 mL(5.7 mol/L)的氢氧化钠水溶液,升温至100 ℃,再缓慢投入7.0 g(100 mmol)锌粉,回流搅拌1 h,过滤得到橙色滤渣。再用甲醇溶解滤渣,在空气下加热至回流反应8 h。最后用旋转蒸发仪除去甲醇,得到3.4 g橙色固体,收率为56.6%。
1.3.2 合成4-羟甲基-4′-羟甲基-(4,4′-二甲氧基三苯甲基)-偶氮苯称取4,4′-二羟甲基偶氮苯1.5 g(6.5 mmol)溶解在40 mL无水四氢呋喃中,分3批次,按每次投料间隔3 h的方法共加入4,4′-二甲氧基三苯基氯甲烷(DMT-Cl)2.1 g(6.5 mmol),反应全程通氮气保护,TLC板监测进程。最后得到原料,DMT一取代,DMT二取代的混合粗产品。湿法上样过柱层析分离,最后得到4-羟甲基-4′羟甲基-(4,4′-二甲氧基三苯甲基)-偶氮苯2.0 g,收率为55.5%。
1.3.3 合成4-羟甲基-(β-氰乙基-N, N′-二异丙基亚磷酰胺)-4′-羟甲基-(4,4′-二甲氧基三苯甲基)-偶氮苯称取上一步得到的产物0.160 g(0.290 mmol),加入四氮唑0.021 g(0.30 mmol),干燥过夜。抽真空后氮气保护,加入磷试剂0.120 g,最后用针筒打入2 mL重蒸后的乙腈使产物的浓度为0.15 mmol/mL左右,随后冰浴下搅拌反应30 min,TLC板监测,反应完成后,用0.22 μm有机系滤膜过滤,待下一步DNA合成使用。
1.4 偶氮苯衍生物修饰DNA的固相合成用DNA固相合成仪按照常规合成的方法经过脱DMT、ETT活化、偶联、盖帽,氧化的步骤将DNA核苷亚磷酰胺单体由3’端向5’端连接到固相。其中普通DNA单体的偶联时间为120 s,偶氮苯衍生物单体的偶联时间增加到600 s。直到所有的亚磷酰胺单体依次偶联到CPG的寡聚核酸上,即可得到目标序列的固相。
1.5 纯化偶氮苯衍生物修饰的DNA将得到的固相加入1 mL的浓氨水,50 ℃下氨解8 h后离心,取上清液浓缩后过滤,HPLC提纯制备。液相色谱条件:紫外检测波长260 nm,流动性A为TEAA,B为乙腈;梯度洗脱程序:0~40 min,0~40% B;40~45 min,40%~100% B;45~50 min,100% B;50~55 min,100%~0 B;55~60 min,0 B;流速1.0 mL/min;柱温:40 ℃。最后取得目标峰溶液加乙酸做脱DMT处理,浓缩旋干后加25 μL NaCl溶液(3 mol/L),涡旋溶解,最后加入1 mL无水乙醇溶液。置于-20 ℃冰箱过夜,然后离心(1 000~13 000 rpm),固体保存标记,上清液移至另外的EP管中,取400 pmol DNA的量做ESI-MS质谱鉴定。
1.6 偶氮苯修饰DNA的光异构化将C1到C4序列溶解在1×PBS中配成2.5 μmol/L的溶液,退火后转移至石英比色皿先用紫外灯(365 nm, 7mW/cm2, 反式到顺式)照射,用岛津UV 1800紫外分光光度计每间隔20 s测一次吸光度,120 s后再用白光灯(>400 nm,11 W,顺式到反式)照射,每间隔20 s测一次吸光度,记录并用软件Origin 8.0作图分析。
1.7 Tm值测定将样品溶解在1×PBS样品溶解标准缓冲液中退火后进行实验,测定梯度温度下的260 nm紫外吸收(Beckman DU 800 Nucleic Acid/Protein Analyzer程序升温1 ℃/min)得到熔解曲线,根据dA/dT作图得知Tm值。同样,我们将样品紫外光照光照10 min(365 nm,11 mW/cm2)再测定熔解温度曲线。记录并用软件Origin 8.0作图分析。
1.8 修饰后的DNA引导引物延伸将引物和模板等比例的溶解于2 μL 10×Thermopol反应溶液(20 mmol/L Tris-HCl, 10 mmol/L KCl, 10 mmol/L (NH4)2SO4, 2 mmol/L MgSO4, pH 8.8)中,再加入过量的dNTP,95 ℃下高温退火5 min,降温至室温后,进行平行实验,一组不光照,一组用紫外灯(365 nm)照射5 min,然后在黑暗条件下分别孵育10 min使引物和模板链充分结合。最后均加入1 U的DNA聚合酶37 ℃下分别孵育1 h。终反应体积为20 μL。通过20%的变性聚丙烯酰胺凝胶在150 V电压条件下电泳2 h,最后应用化学发光凝胶成像系统进行表征,条带分析。
2 结果与讨论 2.1 偶氮苯修饰DNA的合成参照我们以前的报道[33],以对硝基苯甲醇为原料,在浓NaOH水溶液中,Zn作催化剂合成4,4′-二羟甲基偶氮苯。偶氮苯一端羟基与4,4′-二甲氧基三苯基氯甲烷(DMT-Cl)反应进行保护,另一端羟基与2-氰乙基-N,N,N′,N′-四异丙基亚磷酰二胺(磷试剂)反应合成偶氮苯的亚磷酰胺单体(图 1)。值得注意的是最后一步与磷试剂反应,偶氮苯衍生物的投料比例较少有利于DNA的偶联,否则可能剩余部分的磷试剂首先与固相上裸露的羟基反应,从而导致偶氮苯偶联效率下降。这里,直接以重蒸的乙腈为溶剂,4,4′-二羟甲基偶氮苯:四氮唑:磷试剂的投料比例为1:0.8:0.8,通过薄层层析硅胶色谱监控偶氮苯原料不再减少,反应30 min以上,使得磷试剂反应完全,然后将反应液过滤,待用于DNA偶联。
偶氮苯修饰DNA合成使用ABI 394 DNA合成仪按照常规的亚磷酰胺固相合成的方法进行。在偶联偶氮苯的亚磷酰胺单体时,将DNA核苷亚磷酰胺单体偶联时间从通常的120 s增加到1 200 s,偶联效率几乎与标准的DNA亚磷酰胺单体相当。取下装有固载相的柱子,再用浓氨水将寡核苷酸从固相上切除并脱保护,最后进行HPLC分离。制备提纯后的样品进行ESI-MS质谱鉴定,结果如表 1,其实际测得的分子量和理论值相符。
|
|
表 1 偶氮苯修饰的DNA的ESI-MS鉴定 Table 1 ESI-MS spectra of azobenzene linked DNA |
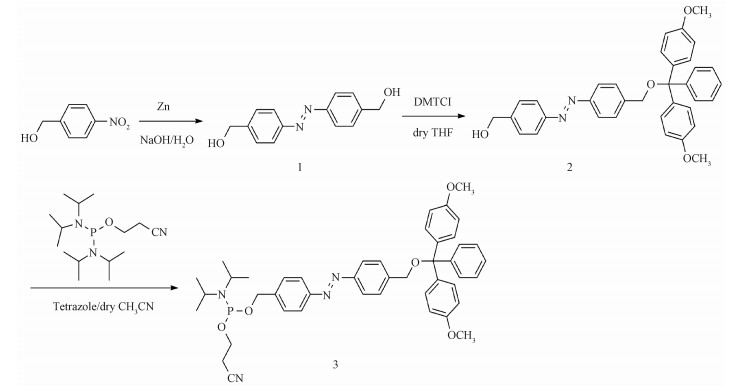
将偶氮苯修饰的DNA(C1、C2、C3和C4)溶解在1×PBS中配成2.5 μmol/L的溶液,95 ℃下退火形成稳定的结构。反式是偶氮苯的热稳定形式,其紫外光谱在335 nm (π-π*)处呈现明显的肩峰。对于C1溶液,通过365 nm紫外灯照,可以明显观察到335 nm处的峰强随着光照时间增加而降低,直到照射80 s,此肩峰基本消失,而430 nm(n-π*)处的峰强略有升高(图 2(a))。此外,再用白光光照,335 nm处的峰强随着光照时间增加而增强,430 nm处峰强略有下降(图 2(b))。同样,C2、C3和C4的溶液通过紫外和可见光照均呈现335 nm和430 nm处峰强的类似变化(图 2(c)、2(d),图 2(e)、2(f)和图 2(g)、2(h))。说明偶氮苯被修饰到设计的寡核苷酸中,并且能起到光异构化的作用。此外,260 nm是核酸的特征吸收峰,在DNA变性时吸光度升高表现出增色效应,说明偶氮苯光异构化可能影响DNA双链的结构,将进一步通过进行热力学稳定性的研究加以佐证。
|
Download:
|
| 图 2 偶氮苯修饰的DNA在不同时间的紫外和可见光照下紫外光谱的变化 Fig. 2 UV/Vis absorbance spectra of the C1, C2, C3, and C4 forms of azobenzene linked DNA before and after light illumination | |
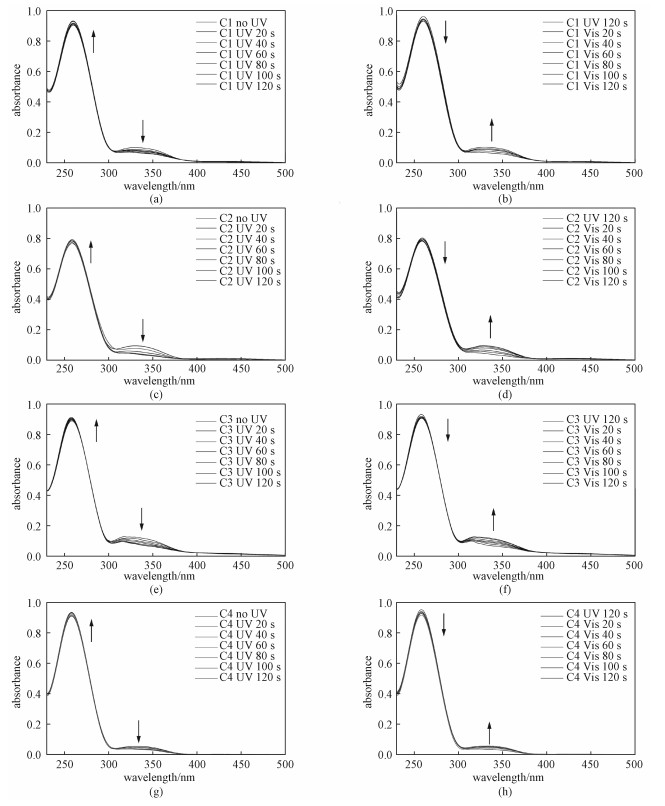
我们前期研究[29, 31]发现,当4,4′-羟甲基偶氮苯代替4、5和6个碱基对的发夹DNA的环状结构,偶氮苯的光异构化能够使得发夹DNA的稳定性发生巨大改变(ΔTm=24 ℃)。这里进一步研究不改变发夹DNA的环状结构上4,4′-羟甲基偶氮苯,仅仅增加一边茎碱基数目,偶氮苯的光异构化是否仍然可能开关发夹DNA的稳定性。图 3展示紫外光照前后C1、C2、C3和C4的熔点曲线,很明显发现C1和C2光照前后的熔链曲线有着较大差异(图 3(a)和3(b)),熔点温度分别从光照前60.2和67.7 ℃,光照后降低到49.8和53.5 ℃,它们熔点温度的变化分别为10.4和14.2 ℃(表 2)。这主要因为偶氮苯的异构化影响其与邻位碱基之间堆积作用以及邻位碱基对的氢键作用,导致寡聚核酸的稳定性降低。尤其是C2,具有6个碱基对,紫外光照后的稳定性降低(ΔTm=14.2 ℃)最多。然而,随着茎碱基数目的增多,偶氮修饰的DNA通过紫外光照后的稳定性降低的程度变小,C3的ΔTm仅是6.9 ℃而茎碱基数目增加到8个碱基对(C4)时光照后的Tm值几乎没有变化。这可能是偶氮苯两端互补配对的序列太长(≥8 bp),偶氮苯异构化所产生的扭转力不足以影响双链的结合。
|
Download:
|
| 图 3 偶氮苯修饰的DNA光照前后的熔点曲线 Fig. 3 Typical melting curves of the C1, C2, C3, and C4 forms of azobenzene linked DNA before and after UV irradiation | |
|
|
表 2 偶氮苯修饰的DNA光照前后的熔点温度 Table 2 Tm comparison of azobenzene linked DNA before and after UV irradiation |
引物延伸的基本原则之一是引物与模板序列要紧密互补结合,因而偶氮苯修饰的DNA作为模板能否和引物形成适当稳定的双链是引物延伸的关键因素。在偶氮苯修饰的DNA中较长的茎碱基链原则上与引物互补结合,但是短链与长链形成的茎碱基对的稳定性可能影响长茎碱基链与引物的结合,而偶氮苯异构化可能影响茎碱基对的稳定性。因此,考虑到发夹DNA的长链茎与引物和短链茎之间存在结合的竞争,我们通过筛选发夹DNA的短链长度分别为5、6、7和8个碱基长度的C1、C2、C3和C4,以及长度为12 mer(Pri.12),15 mer(Pri.15)和17 mer(Pri.17)的引物,评价偶氮苯修饰的DNA与引物结合的光调控。
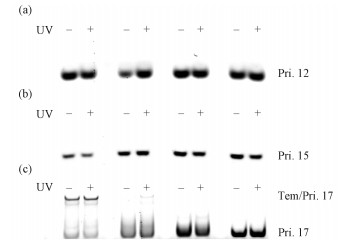
图 4(a)显示Pri.12和模板序列光照前后都没有很好的结合,可能因为本身引物链就比较短,模板链自身能形成双链结构的部分较引物长,即使是偶氮苯在顺式形态下,影响模板的双链结构,但C1、C2、C3,C4模板单链部分和引物能互补配对的长度为7、6、5和4 bp,仍然是不稳定结合,所以在凝胶电泳上无法看到Pri.12和模板的结合。当Pri.15和C1、C2、C3,C4模板单链部分能互补配对的长度增加为10、9、8和7 bp,引物依旧无法很好的参与模板链自身形成的双链结构进行竞争。图 4(b)显示Pri.15和C1只有微弱的结合,而其和C2、C3、C4都不稳定结合。不同的是当Pri.17和C1、C2、C3,C4模板单链部分能互补配对的长度增加为12、11、10和9 bp时,C1和Pri.17可以形成较为稳定的双链结构,这意味引物和模板长链部分互补配对的长度为12 bp时,引物和模板能够有相对稳定的结合,而光照前后这种结合力的变化不是很明显。正如所期望的,C2和Pri.17孵育后在光照前几乎没有结合,而在光照后它们有明显的结合条带,结合部分增加7%左右。由于保护链的增长,C3、C4和Pri.17在光照前后变化也不太明显。
|
Download:
|
| 图 4 偶氮苯修饰的DNA和引物结合的光调控 Fig. 4 Photoregulation of the binding of azobenzene linked DNA with the primer | |
反式是偶氮苯的热稳定形式,起到稳定模板链自身双链结构的作用,所以引物链不能很好与模板链紧密结合,降低了引物的延伸产率。当受外界的紫外光刺激时,偶氮苯能在比较短的时间发生异构化转变为顺式结构,破坏模板链自身双链的碱基堆叠与配对作用,导致双链不稳定,结合力降低。同时引物与模板链不仅存在着配对关系,还会与模板的茎短链存在着竞争配对,从而彻底打开模板自身的双链结构,引物和模板紧密结合,进行DNA的复制。虽然影响引物延伸的因素有很多,但是我们主要从DNA模板、不同长度的引物和不同的酶这三方面来研究并筛选光控引物延伸最佳效率的条件。
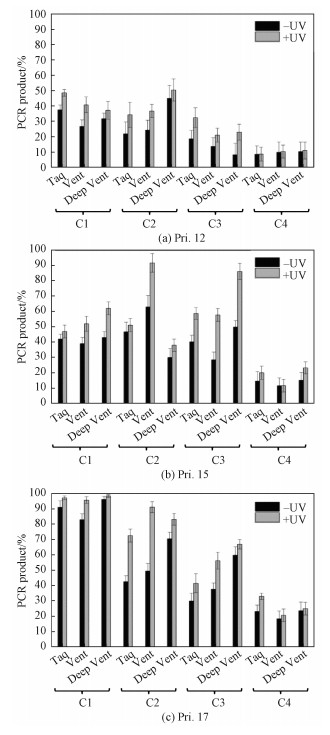
由图 5可见,胶图中最下方的条带为起始引物条带,最上面的条带为延伸全长产物,中间的片段为反应过程中未完全延伸的产物。在以Pri.12作为引物时,其与C1、C2、C3和C4配对区的碱基对分别为7、6、5和4 bp。由图 5(a)和6(a)可知,C1作为模板,紫外光照前,在Taq、Vent、Deep Vent酶的作用下引物延伸产率分别为37.5%,26.7%和31.7%,而紫外光照后,引物延伸产率存在不同程度增加(48.6%,40.7%和37.2%)。与C1相比,C2和C3,模板链的保护短链分别从5个碱基增加到6和7个碱基,与引物配对的碱基对也相应从7 bp减少到6和5 bp,引物延伸的效率通常有所下降。且它们自身经紫外光照后,引物延伸产率比光照前也存在不同程度增加。这是因为随着模板链自身的双链长度增加,配对区长度逐渐减短,结合力变弱,引物在竞争位置逐渐处在劣势,大大降低了模板和引物的结合,所以延伸的产率逐渐减少。C1、C2和C3光照前后指导的引物延伸都有5%~15%的差异,正如我们所预期的,偶氮苯的顺式形态影响了模板自身双链的稳定性,通过配对区的结合,引物“挤走”竞争区的干扰碱基从而自身参与模板链的结合,从而增加延伸效率。而C4光照前后引物的延伸几乎都不明显,这也与光照前后Tm值无明显变化的结果相一致,偶氮苯开关不足以使得12 mer引物对8 bp长度的茎碱基对有所影响。
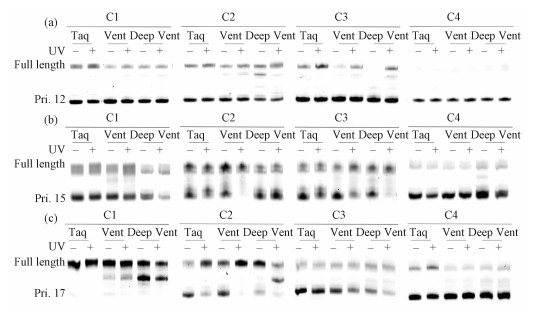
|
Download:
|
| 图 5 偶氮苯修饰的DNA指导引物延伸 Fig. 5 Photomodulaton of the DNA primer extension using azobenzene linked DNA template | |
|
Download:
|
| 图 6 偶氮苯修饰的DNA指导引物延伸的定量分析 Fig. 6 Quantitative analysis for primer extension | |
在以Pri.15作为引物时,其与C1、C2、C3和C4配对区的碱基对增加为10、9、8和7 bp,因而较Pri.12表现出更强的结合力,提高了“挤走”竞争区模板端位短链的可能性。图 5(b)和6(b)显示,Pri.15作为引物表现出比Pri.12更多量的聚合物链反应产物。此外,光照前后引物延伸效率差异比较明显,其中C3,在Vent酶作用下引物延伸效率仅为28.4%,而紫外光照后引物延伸效率达到57.8%,光照前后的PCR产率增加超过1倍。在Deep Vent酶的作用下C3光照前后指导的PCR产率也增加73%。C2在Vent作用下光照后指导的PCR产率很高(91.8%),但光照前指导PCR反应产率也较高(63.1%),因而光照前后的产率也表现出差异,仅为28.7%。对于C4,不管是光照前还是光照后,其指导的引物延伸产率都比较低,偶氮苯开关仍不足以使得15 mer引物对8 bp长度的茎碱基对有所影响。
在以Pri.17作为引物时,其与C1、C2、C3和C4配对区的碱基对增加为12、11、10和9 bp,因而较Pri.12和Pri.15表现出更强的结合力,“挤走”竞争区模板端位短链的可能性更大。图 5(c)和图 6(c)均显示,C1光照前引物延伸的效率已经很大(83.2%以上),紫外光照后引物延伸的效率96.0%以上,由于光照前的背景较高,所以光照前后PCR效率没有明显的变化。说明偶氮苯开关调控的影响不大,原因是配对区的双链较长,即使是竞争区引物不能和模板链紧密结合,配对区也能够指导引物继续延伸。然而,C2,由于配对区的碱基对增加,紫外光照前降低了引物延伸效率的背景,Vent催化下延伸产率仅为49.6%,紫外光照射后的延伸产率达到91.4%,增加了84%。C3的调控比C2的调控略有不足,而C4受到偶氮苯自身调控能力和配对区结合力的限制延伸结果也不够理想。
此外,使用同一种DNA模板,在不同酶的作用下,引物延伸的调控效果只有略微的区别,其中Vent酶表现略好的调控效果。更多的影响调控效果的是DNA模板和引物的组合,这是由于发夹DNA的模板与短保护链和引物之间存在结合的竞争。我们发现,具有7个保护碱基的C3对Pri.15及6个碱基短链的C2对Pri.17具有普遍较好的光调控延伸效果。其中C3,在Vent酶作用下紫外光照前后引物延伸效率增加超过1倍。而C2,由于引物长度增加,尽管紫外光照前增加了引物延伸的背景,但Vent催化下紫外光照射后的延伸产率达到91.4%,也增加了84%,这也与DNA与引物的结合实验中光照前后具有不同的结合力相一致。
2.6 偶氮苯修饰的DNA指导引物延伸的可逆研究为评价偶氮苯引入到核酸分子中对引物延伸的可逆调控,我们选择C2序列与Pri.15在Vent酶作用下,通过紫外光和可见光交替照射来研究引物延伸的可逆行为。图 7(a)表示溶液退火后37 ℃下共孵育58 min,18和38 min分别用UV光和可见光照射2 min,每隔9 min取等量溶液电泳表征(光照时间除外)。线性结果如图 7(b),前18 min反应速率线性拟合为1.22,UV光照射后反应加快,斜率为2.17,增加1.7倍,可见光照后反应速率又下降到较低水平,仅为0.4。证明偶氮苯修饰的模板能够可逆的控制引物延伸速率。
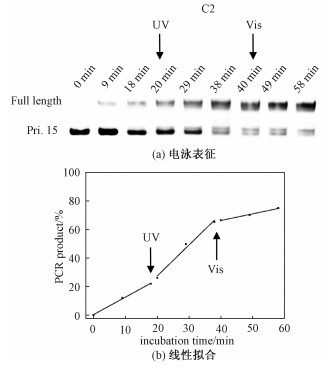
|
Download:
|
| 图 7 偶氮苯修饰的DNA(C2)指导引物延伸的可逆光调控 Fig. 7 Reversible photoregulation of primer extension using C2 with UV and visible light | |
本文研究偶氮苯单元修饰在DNA模板上对引物延伸的光控制行为,筛选5、6、7和8个碱基的保护链通过4,4′-二羟甲基偶氮苯连接的25 mer DNA模板,在紫外光照前后调控的引物延伸效率。结果表明具有7个保护碱基的C3对Pri.15及6个碱基短链的C2对Pri.17具有普遍较好的光调控延伸效果,并且因聚合酶的改变而对引物的调控效果只有略微不同。因而我们断定偶氮苯修饰的DNA模板对引物延伸的光控制行为的主要影响是因为偶氮苯单元修饰模板所形成的竞争区和引物与模板形成的配对区之间的相互竞争。
| [1] |
Wang R W, Jin C, Zhu X Y, et al. Artificial base zT as functional "element" for constructing photoresponsive DNA nanomolecules[J]. Journal of the American Chemical Society, 2017, 139(27): 9104-9107. DOI:10.1021/jacs.7b02865 |
| [2] |
Gorbea C, Mosbruger T, Cazalla D. A viral Sm-class RNA base-pairs with mRNAs and recruits microRNAs to inhibit apoptosis[J]. Nature, 2017, 550(7675): 275-279. DOI:10.1038/nature24034 |
| [3] |
Gomez-Santacana X, Pittolo S, Rovira X, et al. Illuminating phenylazopyridines to photoswitch metabotropic glutamate receptors:from the flask to the animals[J]. ACS Central Science, 2017, 3(1): 81-91. DOI:10.1021/acscentsci.6b00353 |
| [4] |
Adedeji A O, Singh K, Calcaterra N E, et al. Severe acute respiratory syndrome coronavirus replication inhibitor that interferes with the nucleic acid unwinding of the viral helicase[J]. Antimicrobial Agents and Chemotherapy, 2012, 56(9): 4718-4728. DOI:10.1128/AAC.00957-12 |
| [5] |
Velema W A, Szymanski W, Feringa B L. Photopharmacology:beyond proof of principle[J]. Journal of the American Chemical Society, 2014, 136(6): 2178-2191. |
| [6] |
Wu L, Pei F, Zhang J H, et al. Synthesis of site-specifically phosphate-caged siRNAs and evaluation of their RNAi activity and stability[J]. Chemistry-A European Journal, 2014, 20(38): 12114-12122. DOI:10.1002/chem.v20.38 |
| [7] |
Hemphill J, Govan J, Uprety R, et al. Site-specific promoter caging enables optochemical gene activation in cells and animals[J]. Journal of the American Chemical Society, 2014, 136(19): 7152-7158. DOI:10.1021/ja500327g |
| [8] |
Wu L, Wang Y, Wu J Z, et al. Caged circular antisense oligonucleotides for photomodulation of RNA digestion and gene expression in cells[J]. Nucleic Acids Research, 2013, 41(1): 677-686. DOI:10.1093/nar/gks996 |
| [9] |
Wang Y, Wu L, Wang P F, et al. Manipulation of gene expression in zebrafish using caged circular morpholino oligomers[J]. Nucleic Acids Research, 2012, 40(21): 11155-11162. DOI:10.1093/nar/gks840 |
| [10] |
Szymanski W, Beierle J M, Kistemaker H A, et al. Reversible photocontrol of biological systems by the incorporation of molecular photoswitches[J]. Chemical Reviews, 2013, 113(8): 6114-6178. DOI:10.1021/cr300179f |
| [11] |
Beharry A A, Woolley G A. Azobenzene photoswitches for biomolecules[J]. Chemical Society Reviews, 2011, 40(8): 4422-4437. |
| [12] |
Liu Z F, Hashimoto K, Fujishima A. Photoelectrochemical information-storage using an azobenzene derivative[J]. Nature, 1990, 347(6294): 658-660. DOI:10.1038/347658a0 |
| [13] |
Kumar G S, Neckers D C. Photochemistry of azobenzene-containing polymers[J]. Chemical Reviews, 1989, 89(8): 1915-1925. DOI:10.1021/cr00098a012 |
| [14] |
Zimmerman G, Chow L Y, Paik U J. The photochemical isomerization of azobenzene[J]. Journal of the American Chemical Society, 1958, 80(14): 3528-3531. DOI:10.1021/ja01547a010 |
| [15] |
Yang Y Y, Endo M, Hidaka K, et al. Photo-controllable DNA origami nanostructures assembling into predesigned multiorientational patterns[J]. Journal of the American Chemical Society, 2012, 134(51): 20645-20653. DOI:10.1021/ja307785r |
| [16] |
Yan Y Q, Chen J I, Ginger D S. Photoswitchable oligonucleotide-modified gold nanoparticles:controlling hybridization stringency with photon dose[J]. Nano Letters, 2012, 12(5): 2530-2536. DOI:10.1021/nl300739n |
| [17] |
Lohmann F, Ackermann D, Famulok M. Reversible light switch for macrocycle mobility in a DNA rotaxane[J]. Journal of the American Chemical Society, 2012, 134(29): 11884-11887. DOI:10.1021/ja3042096 |
| [18] |
Tunitskaya V L, Kochetkov S N. Structural-functional analysis of bacteriophage T7 RNA polymerase[J]. Biochemistry-Moscow, 2002, 67(10): 1124-1135. DOI:10.1023/A:1020911223250 |
| [19] |
Nakasone Y, Ooi H, Kamiya Y, et al. Dynamics of inter-DNA chain interaction of photoresponsive dNA[J]. Journal of the American Chemical Society, 2016, 138(29): 9001-9004. DOI:10.1021/jacs.6b02525 |
| [20] |
Kingsland A, Samai S, Yan Y Q, et al. Local density fluctuations predict photoisomerization quantum yield of azobenzene-modified DNA[J]. The Journal of Physical Chemistry Letters, 2016, 7(15): 3027-3031. DOI:10.1021/acs.jpclett.6b00956 |
| [21] |
Yan Y Q, Wang X, Chen J L, et al. Photoisomerization quantum yield of azobenzene-modified DNA depends on local sequence[J]. Journal of the American Chemical Society, 2013, 135(22): 8382-8387. DOI:10.1021/ja403249u |
| [22] |
Lubbe A S, Szymanski W, Feringa B L. Recent developments in reversible photoregulation of oligonucleotide structure and function[J]. Chemical Society Reviews, 2017, 46(4): 1052-1079. DOI:10.1039/C6CS00461J |
| [23] |
Chen H R, Zhang H Y, Pan J, et al. Dynamic and progressive control of DNA origami conformation by modulating DNA helicity with chemical adducts[J]. ACS Nano, 2017, 10(5): 4989-4996. |
| [24] |
Liu M Z, Asanuma H, Komiyama M. Azobenzene-tethered T7 promoter for efficient photoregulation of transcription[J]. Journal of the American Chemical Society, 2006, 128(3): 1009-1015. DOI:10.1021/ja055983k |
| [25] |
Wang X L, Huang J, Zhou Y Y, et al. Conformational switching of G-quadruplex DNA by photoregulation[J]. Angewandte Chemie-International Edition, 2010, 49(31): 5305-5309. DOI:10.1002/anie.v49:31 |
| [26] |
Matsunaga D, Asanuma H, Komiyama M. Photoregulation of RNA digestion by RNase H with azobenzene-tethered DNA[J]. Journal of the American Chemical Society, 2004, 126(37): 11452-11453. DOI:10.1021/ja0471976 |
| [27] |
Liang X G, Asanuma H, Komiyama M. Photoregulation of DNA triplex formation by azobenzene[J]. Journal of the American Chemical Society, 2002, 124(9): 1877-1883. DOI:10.1021/ja011988f |
| [28] |
Chaulk S G, MacMillan A M. Caged RNA:photo-control of a ribozyme reaction[J]. Nucleic Acids Research, 1998, 26(13): 3173-3178. DOI:10.1093/nar/26.13.3173 |
| [29] |
Dong M X, Babalhavaeji A, Samanta S, et al. Red-shifting azobenzene photoswitches for in vivo use[J]. Accounts of Chemical Research, 2015, 48(10): 2662-2670. DOI:10.1021/acs.accounts.5b00270 |
| [30] |
Nishioka H, Liang X G, Asanuma H. Effect of the ortho modification of azobenzene on the photoregulatory efficiency of DNA hybridization and the thermal stability of its cis form[J]. Chemistry-A European Journal, 2010, 16(7): 2054-2062. DOI:10.1002/chem.v16:7 |
| [31] |
Wu L, Wu Y, Jin H W, et al. Photoswitching properties of hairpin ODNs with azobenzene derivatives at the loop position[J]. MedChemCommun, 2015, 6(3): 461-468. DOI:10.1039/C4MD00378K |
| [32] |
Wu L, Koumoto K, Sugimoto N. Reversible stability switching of a hairpin DNA via a photo-responsive linker unit[J]. Chem Commun, 2009, 14(14): 1915-1917. |
| [33] |
Wu L, He Y J, Tang X J. Photoregulating RNA digestion using azobenzene linked dumbbell antisense oligodeoxynucleotides[J]. Bioconjugate Chemistry, 2015, 26(6): 1070-1079. DOI:10.1021/acs.bioconjchem.5b00125 |
| [34] |
Young D D, Edwards W F, Lusic H, et al. Light-triggered polymerase chain reaction[J]. Chemical Communications, 2008(4): 462-464. DOI:10.1039/B715152G |
| [35] |
Wang Q, Yi L, Liu L L, et al. A thermostable azo-linker for reversible photoregulation of DNA replication[J]. Tetrahedron Letters, 2008, 49(34): 5087-5089. DOI:10.1016/j.tetlet.2008.06.027 |
| [36] |
Yamazawa A, Liang X G, Asanuma H, et al. Photoregulation of the DNA polymerase reaction by oligonucleotides bearing an azobenzene[J]. Angewandte Chemie-International Edition, 2000, 39(13): 2356-2357. DOI:10.1002/(ISSN)1521-3773 |
| [37] |
Kamiya Y, Takagi T, Ooi H, et al. Synthetic gene involving azobenzene-tethered T7 promoter for the photocontrol of gene expression by visible light[J]. ACS Synthetic Biology, 2015, 4(4): 365-370. DOI:10.1021/sb5001092 |
| [38] |
Asanuma H, Tamaru D, Yamazawa A, et al. Photoregulation of the transcription reaction of T7 RNA polymerase by tethering an azobenzene to the promoter[J]. Chembiochem, 2002, 3(8): 786-789. DOI:10.1002/1439-7633(20020802)3:8<786::AID-CBIC786>3.0.CO;2-P |
| [39] |
Wang X Y, Liang X G. Azobenzene-modified antisense oligonucleotides for site-specific cleavage of RNA with photocontrollable property[J]. RSC Advances, 2016, 6(96): 93398-93402. DOI:10.1039/C6RA20954H |
| [40] |
Tian T, Song Y Y, Wang J Q, et al. Small-molecule-triggered and light-controlled reversible regulation of enzymatic activity[J]. Journal of the American Chemical Society, 2016, 138(3): 955-961. DOI:10.1021/jacs.5b11532 |
 2019, Vol. 36
2019, Vol. 36 






