在药物研发过程中,许多潜在的药物由于跨膜性质不佳而被淘汰,因此,药物跨膜能力是药物开发过程中必须考虑的重要因素[1-3]。细胞穿膜肽是一类序列长度小于30个氨基酸的短肽,可以穿过细胞膜而不对细胞造成损害,因此可以作为载体将药物、核酸和蛋白质等生物活性分子转运到细胞中[4-9],从而使活性分子更好地发挥作用。近年来,细胞穿膜肽作为潜在的药物递送载体已经引起人们的广泛关注,探究细胞穿膜肽的作用机理对细胞穿膜肽的临床应用研究具有重要意义。
S4(13)是从螺旋抗菌肽Dermaseptin S4中截取出来的一段细胞穿膜肽(图S1),它由13个氨基酸残基(ALWKTLLKKVLKA)构成,其中的Lys氨基酸残基使S4(13)整体带正电。研究表明S4(13)经过核定位信号肽修饰之后,可以参与肿瘤细胞中siRNA的递送过程和实现基因沉默,同时具有对细胞毒性低的优点[9-14]。已有的实验数据证明S4(13)能够进入细胞[13],它的穿膜特点决定了它的衍生物具有递送核酸的作用,但是,关于S4(13)穿膜的具体机理尚不清楚[15]。目前的研究中只有S4(13)修饰后的产物S4(13)-PV穿膜的相关报道,即Mano等通过抑制内吞途径的实验,发现S4(13)-PV进入细胞的过程并没有受到影响,证明了S4(13)-PV的穿膜过程普遍是非内吞的作用机制[16],并且他们用实验说明了S4(13)-PV是以直接易位的方式进入细胞膜,细胞膜的组分会对穿膜过程产生影响[16-17]。Padari等[18]通过圆二色谱法发现S4(13)-PV会组装成特定大小和形状的聚集体。虽然现有实验支持S4(13)直接易位的作用机制,但是目前还不清楚S4(13)是以单分子形式还是以聚集体的形式穿膜。
本文使用分子动力学模拟[19-20]的方法研究细胞穿膜肽S4(13)与质膜的相互作用,用1-棕榈酰基-2-油酰基磷脂酰胆碱(POPC)和1-棕榈酰基-2-油酰基磷脂酰甘油(POPG)2种磷脂分子搭建的双分子层分别代表真核生物质膜和原核生物质膜,对单个和多个S4(13)分子在单纯水相和水膜两相的环境下分别进行常规分子动力学模拟,通过伞形采样模拟单个多肽分子在膜上的吸附和扩散以及多个多肽分子在水溶液中的聚集过程,并寻找多肽和质膜作用过程中涉及的关键残基,以此为穿膜肽跨生物膜运输活性分子的研究提供理论参考,帮助设计更理想的细胞穿膜肽。
1 研究方法 1.1 构建分子动力学模拟体系本项工作用到的细胞穿膜肽S4(13)的结构(图S1)来自于穿膜肽数据库CPPsite2.0[21-23],该结构具有两亲性,疏水性氨基酸和亲水性氨基酸分别分布在螺旋两侧;分别以POPC和POPG构造质膜模型,以模拟真核和原核生物细胞膜[24]。磷脂分子POPC和POPG的结构从PubChem数据库[25]中获取。使用Gaussian 09[26]选择HF/6-31 G*方法和基组建立磷脂分子的GAFF力场[27]参数,步骤包括:首先对磷脂分子进行结构优化,然后利用Amber 18[28]中的antechamber程序计算每个原子的RESP静电荷[29-30],最后利用parmchk2程序补充相关的力场参数。完成磷脂分子力场参数的构建之后,用Packmol[31]和Amber 18软件的tleap程序分别搭建如表 1所示8个体系的起始结构,根据Packmol盒子的大小和磷脂分子以及水分子的密度确定每个体系中磷脂分子和水分子的个数。分别用S1~S8表示本文中8个常规分子动力学模拟的体系(S1~S3体系用于分析单个多肽的性质,S4~S8用于分析多个多肽的性质)。S4~S6体系中,5个多肽分子在空间上分布较远,S7体系中5个多肽分子分布较紧密,S8体系中4个多肽分子来自于S7体系平衡后的聚集体结构。
|
|
表 1 常规分子动力学模拟的体系 Table 1 System list of classical MD simulations |
所有的动力学模拟和分析均使用Amber 18软件包进行。S4(13)采用Amber ff14SB力场[32],磷脂分子采用GAFF力场,所有的水分子都采用TIP3P水[33]。用tleap程序为其添加抗衡离子以及周期边界盒子,生成动力学模拟所需要的拓扑文件和坐标文件,最终进行相应时长的分子动力学模拟。每个体系进行分子动力学模拟之前,首先经历3步优化的过程除去体系中的不合理原子接触和碰撞,释放初始结构中不合理因素带来的张力,达到势能面较平稳的状态:1) 约束多肽,有质膜的体系同时约束磷脂分子,优化除此之外的水分子和抗衡离子;2) 约束多肽的骨架原子,有质膜的体系对磷脂分子的头尾原子进行约束,对多肽侧链以及其他组分进行优化;3) 不加任何约束进行优化。这3步优化都是先采用最陡下降法,后采用共轭梯度的方法进行优化。之后,每个体系在NPT系综条件(压力设置为100 kPa)下经历7步升温过程,将体系从0 K均匀地加热到310 K,无质膜的体系经历500 ps的升温过程,有质膜的体系经历1 ns,直至体系密度达到平稳。对每个平稳后的体系在NVT系综条件下进行如表 1所示相应时长的分子动力学模拟,模拟步长设置为2 fs,轨迹每隔1 ps写入一次,用Berendsen thermostat恒温热浴方法控制温度维持稳定在310 K附近,用SHAKE算法[34]约束含氢共价键的伸缩振动,将长程相互作用的截断值设置为1.0 nm,使用particle mesh Ewald (PME)方法[35]处理分子模拟中的长程静电相互作用。
1.3 伞形采样分子动力学模拟为说明单个S4(13)分子在不同质膜上的吸附和扩散行为,应用伞形采样方法将S4(13)分子分别从POPC和POPG质膜中心沿着垂直于质膜平面的z轴方向拉到距离质膜中心6 nm的位置处,定义质膜中心和多肽分子质心的距离作为反应坐标。设置伞形采样窗口数为61,每个采样窗口的宽度设为0.1 nm,施加外力的力常数大小设置为1 046 kJ/(mol·nm2),每个采样窗口模拟的时间为10 ns。对每个采样窗口,分子动力学模拟的步长都设置为2 fs,每隔2 ps写入一次轨迹,在310 K温度条件下进行模拟。另外,为说明多个S4(13)分子在水相中发生聚集的趋势,对S8体系中聚集体的稳定结构,施加外力将其中一个S4(13)分子从其他3个S4(13)分子的质心位置沿着远离聚集体的方向拉到距离质心3 nm的位置,定义被拉的S4(13)分子的质心和另外3个S4(13)分子的质心的距离作为反应坐标,设置伞形采样的窗口数为26,其他模拟条件与S4(13)在质膜上所进行的伞形采样模拟条件一致。最后,使用加权柱状图分析方法通过Wham程序[36-37]计算这3组伞形采样动力学模拟过程中的自由能,绘制平均力势(potential of mean force, PMF)曲线。
1.4 分子动力学轨迹分析各个体系的动力学轨迹分析都由Amber 18软件包中的cpptraj程序[38]完成。包括多肽骨架原子均方根偏差(root mean square deviation, RMSD)的计算、氢键的统计(采用cpptraj默认的氢键标准)、二级结构的统计、回转半径(radius of gyration,Radgyr)的计算、质量密度的计算、相关系数的计算、接触强度的计算以及特定帧结构的提取。
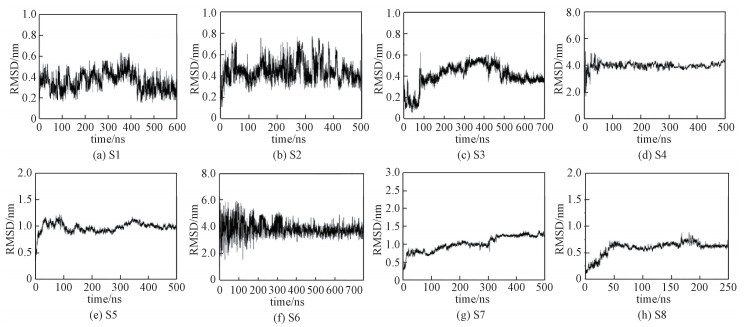
2 结果与讨论 2.1 轨迹分析判断体系的稳定性各个体系升温过程中的密度变化(图S2)反映了密度在升温过程中都已达到平衡。S4(13)骨架原子均方根偏差(RMSD)的计算结果(图S3)表明每个体系在动力学轨迹最后150 ns都已经达到稳定和平衡。为使每个体系有足够的采样,每个体系进行一定长时间的分子动力学模拟后,均选用最后150 ns轨迹进行各种性质的统计与分析。RMSD数据还提示含有单个S4(13)分子的体系S1~S3的RMSD较小,含有多个S4(13)分子的体系S4~S7的RMSD较大且相互存在差异:S5和S7体系的RMSD值稳定在1 nm左右,说明S4(13)可能维持结合状态,受到比较稳定的相互作用;而S4和S6体系的RMSD稳定在4 nm左右,可能是因为体系中有灵活性比较大的多肽分子。
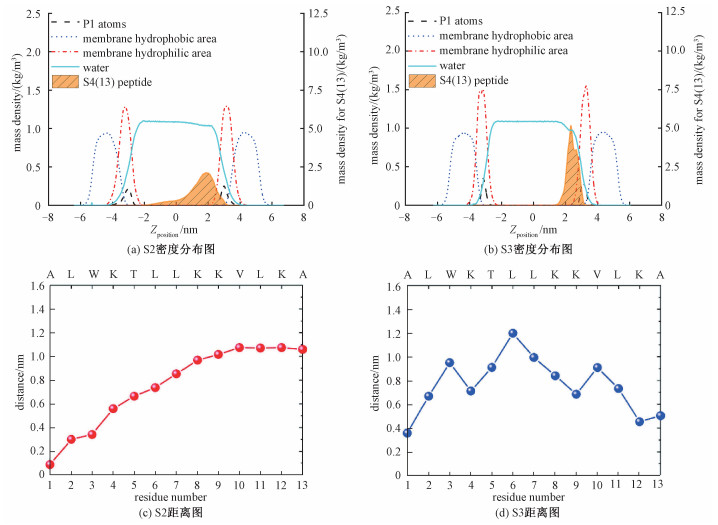
2.2 单个S4(13)分子与不同质膜相互作用对比统计最后150 ns动力学轨迹中,S2和S3体系中磷脂亲水端、磷脂疏水端、磷原子、水分子和S4(13)分子在垂直于质膜双层平面法线方向的质量密度分布和不同残基的Cα原子与磷原子平面的距离(图 1),用来描述多肽整体和各个残基在多肽结合到质膜表面时相对于质膜的位置。密度分布图(图 1(a)、1(b))中,为方便观察,将质膜的重心位置定义为Zposition=0,考虑到S4(13)的密度远小于其他组分,其密度使用右侧坐标轴,比例尺为其他组分密度的1/5。S4(13)的密度分布越靠近质膜,说明肽与质膜的作用越强;残基的Cα原子和磷原子平面的距离越小,提示该残基越靠近质膜,说明这个残基参与肽与质膜相互作用的可能性更大。结果表明S4(13)和POPG膜有更强的相互作用,这归因于POPG膜体系的磷脂分子带负电,可以和带正电的S4(13)分子形成较强的静电相互作用。
|
Download:
|
| 图 1 沿z轴方向体系S2、S3各组分的密度分布图和各个残基的Cα原子距离磷原子平面的距离图 Fig. 1 The mass density distribution map of the components and the distance between Cα atoms of each residue and averageplane of phosphorus atoms along z direction in systems S2 and S3 | |
通常2层磷脂亲水头部间的距离可以用于描述质膜的厚度,密度分布图表明S4(13)分子对POPG膜和POPC膜厚度的影响可以忽略不计。随着模拟的进行,2个体系S4(13)分子的质量密度(图(1(a)、1(b)),橙色区域)都集中分布在水相和质膜亲水端区域内,说明S4(13)分子与2种质膜都有作用。但是,S2体系的S4(13)分子在垂直于POPC膜平面方向上密度分布更广,S3体系的S4(13)分子在垂直于POPG膜平面方向上密度分布更集中,说明S4(13)分子在POPC膜体系中有较大的运动自由度,在POPG膜中运动受到限制。
图 1(c)表明S4(13)在POPC膜中只有多肽链N-端的3个氨基酸残基Ala-1、Leu-2和Trp-3靠近了磷原子平面,与质膜的亲水端形成相互作用;图 1(d)表明在POPG膜体系中,S4(13)的两端和质膜都有作用,其中Ala-1、Lys-12和Ala-13是距离磷原子平面最近的3个残基;S4(13)中间部分的Lys-4和Lys-9残基也和质膜有作用。综上,与POPC膜有作用的主要是疏水性氨基酸残基,与POPG膜有作用的主要是多肽链头尾部分的氨基酸残基加上中间部分的带电残基,这说明静电相互作用对于多肽和细菌质膜的作用有重要意义。
为研究S4(13)穿过质膜在膜中的扩散过程,利用增强采样的方法对这一过程进行模拟。因此,采用伞形采样的方法模拟S4(13)分子从质膜中心扩散到水相中的过程,从而对比S4(13)在2种质膜上吸附和扩散的难易程度。
对伞形采样的数据应用加权直方图分析方法计算平均力势(图 2),PMF曲线表示S4(13)的自由能沿着质膜中心到水相距离变化(定义质膜中心的位置为0)。图中结果表明单分子S4(13)吸附到POPG膜表面上比吸附到POPC膜表面上更容易, 其自由能呈现下降趋势,表明S4(13)吸附到POPG膜表面是自发的过程。在POPC膜体系中,多肽吸附到膜上首先自由能需升高5.19 kJ/mol。而对于多肽从膜中心向水相扩散的过程:在POPC体系中,多肽质心在z轴方向0.425 nm位置处和膜的结合最稳定,在POPG体系中,在z轴方向0.192 nm位置处和膜的结合最稳定。在POPC膜体系中从质膜中心扩散到水相的势垒为181.45 kJ/mol;而对于POPG膜体系,多肽从质膜中心扩散到水相的势垒为136.28 kJ/mol,该结果表明多肽S4(13)不容易以单分子的形式穿过细胞膜。

|
Download:
|
| 图 2 POPC和POPG膜体系中S4(13)从质膜中心沿z轴方向向水层扩散的平均力势曲线 Fig. 2 PMF profiles for the peptide S4(13) to move from membrane center to aqueous solution along z direction in POPC and POPG systems | |
回转半径可以表征体系聚集的程度,如图 3所示,黑线是回转半径随时间的变化,用红线拟合变化趋势。在S4、S6和S7体系中,根据回转半径曲线的趋势可以看出聚集现象的发生。而对于S5体系,不容易发生聚集现象。这表明在POPC膜和水溶液中,S4(13)容易发生聚集,在POPG膜上由于和质膜形成强的相互作用,S4(13)的运动受到限制不容易发生聚集。

|
Download:
|
| 图 3 各个体系S4(13)骨架原子的回转半径随模拟时间变化曲线 Fig. 3 The radius of gyration changes for backbone atoms of S4(13) in each system with simulation time | |
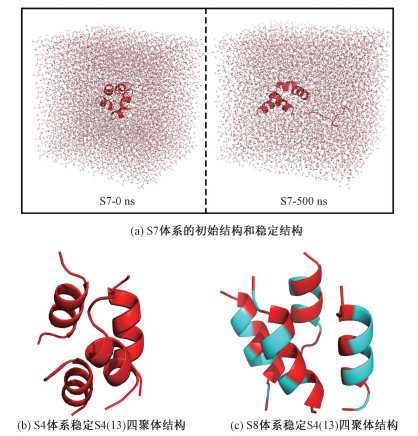
图 4中4个体系的初始结构和模拟后的稳定结构对比单个S4(13)与多个S4(13)分子与不同质膜的作用,每个体系的S4(13)都用红色的cartoon模型表示,磷原子显示为橙色小球模型,水分子和磷脂分子都显示为线状模型。为便于观察,隐藏抗衡离子。S2和S3体系采用侧视图,S4和S5体系采用俯视图,多肽的起始位置都被放置在距离膜表面相同距离的位置处。由图结果可知,平衡后的4个体系中的构象都发生了很大的改变:S2和S3体系的结果和2.2小节的分析一致,多肽S4(13)分子的两端都被锚定在POPG膜上,而在POPC膜上只有一端被固定。S4体系的5个S4(13)分子在POPC膜上发生了聚集,倾向于形成四聚体结构(图S4(b));S5体系的5个S4(13)分子和膜有较强的静电相互作用,这限制了分子的运动,导致S4(13)分子在POPG膜上不能发生聚集。S7水相体系的起始和平衡后的结构和S8体系稳定后的S4(13)结构也证明多个S4(13)分子在水相中容易发生聚集形成四聚体(图S4(a)、S4(c))。此外,游离在聚集体之外的单个S4(13),因为没有形成聚集体结构失去了螺旋结构。S6体系由于水相中S4(13)分子运动较灵活且间隔较远,没有形成四聚体,但是有形成四聚体结构的趋势。通过观察S7和S8体系的结果,也证明S4(13)在水相中有形成四聚体的倾向。

|
Download:
|
| 图 4 S2~S5体系模拟前的初始结构和模拟后的稳定结构 Fig. 4 The initial structures before simulations and the equilibrated structures after simulations for systems S2-S5 | |
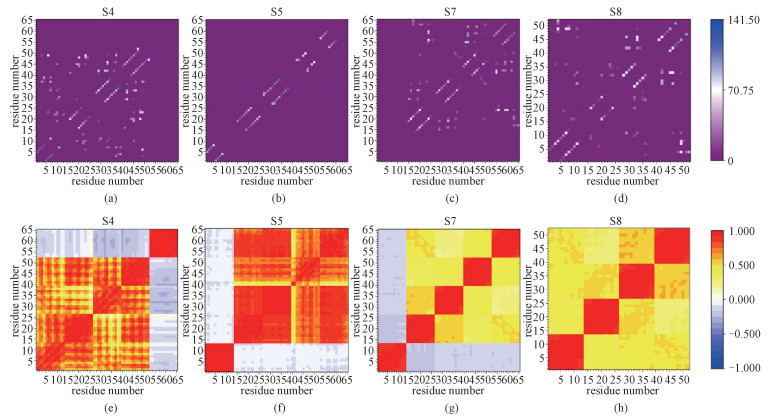
对S4、S5、S7和S8体系中S4(13)进行残基间接触强度和轨迹相关系数的分析(图 5),S4和S5接触强度的结果和图 4一致,结合S7和S8体系的结果,说明S4(13)在POPG膜上不聚集,在POPC膜和水溶液环境下才会倾向于形成四聚体。S4、S7和S8体系相关系数的结果(图 5(e)、5(g)、5(h))与接触强度的结果(图 5(a)、5(c)、5(d))一致,图中4个S4(13)分子的相关性很强,S5体系由于S4(13)和质膜表面有较强的结合作用,因此表现出运动的一致性,所以会存在4个分子运动相关性很强的情况(图 5(f)),这从另一方面说明了多个S4(13)分子和POPG膜的稳定结合作用。
|
Download:
|
| 图 5 S4、S5、S7和S8体系S4(13)残基间的接触热图(a~d)和相关系数热图(e~h) Fig. 5 The contact intensity maps (a-d) and correlation maps (e-h) for residues of S4(13) for systems S4, S5, S7, and S8 | |
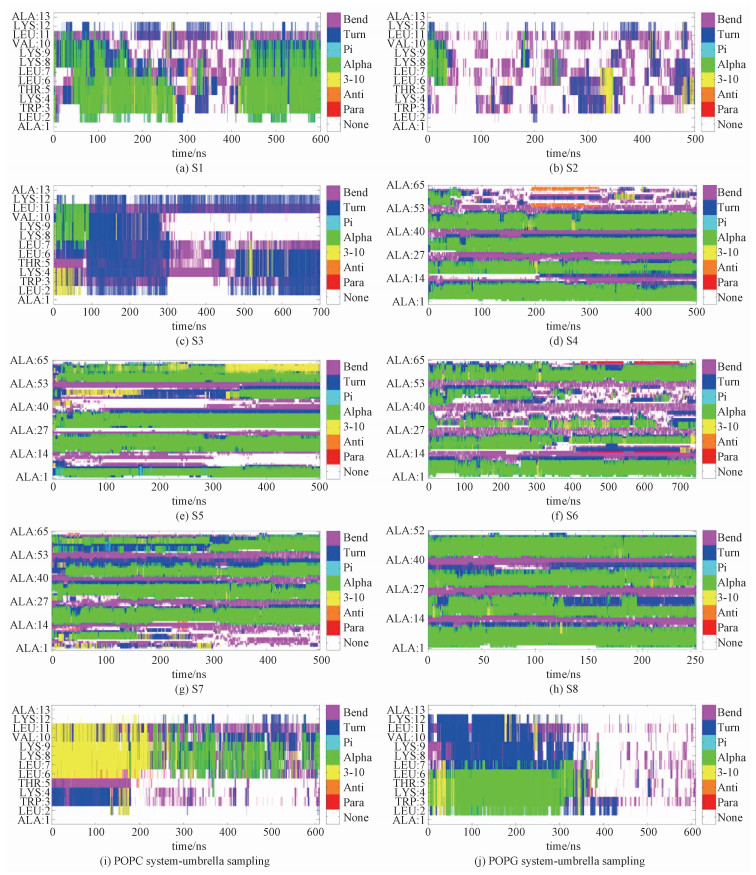
文献表明,S4(13)可以形成螺旋结构,S4(13)在穿膜过程中伴随着构象的变化,这种结构变化与S4(13)穿膜机理可能有联系。常规分子动力学模拟的结果表明单个S4(13)分子和质膜相互作用时,螺旋结构被破坏,氢键分析结果也表明分子内氢键占有率极低。对各体系进行二级结构随模拟时间变化的分析(图S6),表 2中显示的是S1~S8体系每个S4(13)分子二级结构的时间占比,S4~S8体系中S4(13)分子的二级结构随时间的变化显示,聚集体的α-螺旋比例较高,提示形成S4(13)分子聚集体有利于维持α-螺旋结构。对单个S4(13)分子在POPC和POPG膜上的伞形采样的轨迹进行二级结构分析,结果表明单个S4(13)分子嵌入到膜内区域时倾向于维持螺旋结构,在水相倾向于形成无规卷曲的结构,因此膜相疏水环境比水相环境更有利于单个多肽分子维持螺旋结构,这对聚集体与质膜的作用也具有很好的参考意义。
|
|
表 2 各个体系模拟过程中S4(13)各种二级结构的占比(体系有多个多肽的标记为Pep1~5) Table 2 The proportion of second structure for S4(13) in each system during the simulation time(The systems with multiple peptides are listed as Pep1-5) |
氢键是维持超分子结构稳定的重要相互作用。在S4(13)与质膜结合初期,分子内氢键是维持螺旋结构的关键,分子间氢键则对多肽与质膜的结合发挥重要作用。统计S2和S3体系中S4(13)与质膜作用的氢键,取出占有率靠前的氢键(表 3、表 4),对于POPC的体系,主要是S4(13)分子的Ala-1和Trp-3残基与质膜形成氢键,因此只是肽链的一端与质膜有较强的相互作用。对于POPG体系,Lys-9、Lys-12、Lys-4和Ala-1残基参与形成氢键。从形成氢键的数量上对比,也进一步证明了POPG和S4(13)分子的结合更强。综上,S2和S3体系的氢键分析表明:静电相互作用对于多肽与细菌质膜的结合发挥了重要作用,带负电的POPG质膜更容易和带正电的多肽S4(13)形成氢键;不带电的POPC质膜更倾向于和S4(13)的疏水残基作用。这和前面单个S4(13)分子与不同质膜作用的结果一致,也和S2和S3体系的结构图一致。
|
|
表 3 S2体系中S4(13)和POPC膜形成的氢键 Table 3 The hydrogen bonds formed between S4(13) and POPC membrane for S2 system |
|
|
表 4 S3体系中S4(13)和POPG膜形成的氢键 Table 4 The hydrogen bonds formed between S4(13) and POPG membrane for S3 system |
对比单个S4(13)分子结合到质膜表面的构象变化,分析伞形采样体系S4(13)与质膜的分子间氢键相互作用(表S1、S2)和S4(13)分子内氢键相互作用(图S5)。POPC体系形成的分子间氢键比POPG体系形成的分子间氢键更稳定一些,这与S4(13)结合在质膜表面的情况不同,S4(13)在质膜内形成了更多与POPC质膜的氢键作用,可能是膜内的疏水环境导致。同样,POPC和POPG膜体系的分子内氢键的数量图(图S5)显示在质膜内的数量要大于水相环境中的数量,因此在膜内更容易维持螺旋结构,在水相螺旋结构被破坏。在POPC膜内S4(13)的分子内氢键的数量更多。这些说明S4(13)在POPC膜内更稳定,这个结论和PMF曲线(图 2)给出的结论一致。
对于维系多个S4(13)体系聚集体存在相互作用的情况,S8体系聚集体之间的接触强度(表S3)和分子间氢键分析(表S4)表明,聚集体之间的结合依赖于疏水残基间的疏水相互作用。接触强度可以看作一定距离内所有作用力的集合,提取0.5 nm以内的接触强度大于50的相互作用残基,表中显示疏水性残基对S4(13)分子聚集起着关键作用。S4和S7体系内S4(13)分子间的接触强度(表S5、表S6)进一步证明了疏水残基对于形成聚集体的重要性。
2.6 多个S4(13)分子在水相的聚集为证明S4(13)分子能自发在水溶液中形成聚集体,进行了伞形采样动力学模拟,得到平均力势的结果如图 6(结构图表示反应坐标为1.0和2.0 nm处的S4(13)结构,分别对应于单个S4(13)分子和其他3个S4(13)分子结合最稳定的状态和完全远离聚集体结构的状态,其中疏水性氨基酸残基显示为红色,亲水性氨基酸残基显示为天蓝色)。从图中可知单个多肽从聚集体中解离需跨越的势垒约为14.60 kJ/mol,这表明单个多肽在水相中形成聚集体是有利的,即S4(13)的聚集现象是普遍存在的。

|
Download:
|
| 图 6 水溶液体系伞形采样模拟的平均力势(PMF)曲线 Fig. 6 The PMF plots of umbrella sampling simulation for water system | |
本文利用分子动力学模拟的手段,分析单个S4(13)和多个S4(13)在真核生物质膜(POPC)和细菌质膜(POPG)中发生的相互作用,模拟结果表明:POPG膜对S4(13)分子具有较强的吸引作用,可以约束S4(13)两端的Ala-1和Ala-13残基,并且与S4(13)多肽链中间部分的带电残基Lys-4、Lys-9和Lys-12有重要的相互作用,将多肽锚定在质膜表面;而POPC膜对S4(13)的吸引作用较弱,只能吸引S4(13)分子一端的Ala-1、Leu-2和Trp-3残基。S4(13)和质膜的氢键分析表明静电相互作用有利于多肽在质膜界面的稳定,而疏水相互作用有利于多肽在质膜内部的稳定。S4(13)在POPC和POPG质膜上的伞形采样模拟表明单个S4(13)分子都可以和质膜发生作用,被吸附到POPG膜上更容易,但都不容易穿过质膜。多个S4(13)分子与不同质膜的模拟结果表明S4(13)分子在水溶液和POPC膜上容易以四聚体的形式聚集,所以S4(13)分子更倾向于以聚集体的形式穿过生物膜。伞形采样模拟的结果也表明在水相中容易发生S4(13)分子的聚集。分析残基间的相互作用表明S4(13)聚集主要依赖于疏水性氨基酸残基。另外,单个S4(13)分子在与质膜作用的初始阶段螺旋结构会被破坏,但在膜内能维持螺旋结构的稳定。当S4(13)分子以聚集体形式存在时,螺旋结构能较稳定地存在。综上,该研究结果为探究细胞穿膜肽和质膜的作用提供了理论依据,这对于拓展细胞穿膜肽在临床上的应用有重要意义。
附录|
|
表 S1 POPC伞形采样体系多肽和质膜形成的氢键 TableS1 The hydrogen bonds between peptide and membrane in POPC umbrella sampling system |
|
|
表 S2 POPG伞形采样体系多肽和质膜形成的氢键 TableS2 The hydrogen bonds between peptide and membrane in POPG umbrella sampling system |
|
|
表 S3 S8体系中接触强度大于50的氨基酸残基对 TableS3 The contact residue-pairs with contact intensity larger than 50 in system S8 |
|
|
表 S4 S8体系聚集体之间的氢键 TableS4 The hydrogen bonds within S4(13) tetramer in system S8 |
|
|
表 S5 S4体系中接触强度大于50的氨基酸残基对 TableS5 The contact residue-pairs with contact intensity larger than 50 in system S4 |
|
|
表 S6 S7体系中接触强度大于50的氨基酸残基对 TableS6 The contact residue-pairs with contact intensity larger than 50 in system S7 |

|
Download:
|
| 图 S1 S4(13)的三维结构图(cartoon模型) Fig. S1 3D structure of S4(13) (cartoon model) | |

|
Download:
|
| 图 S2 各个体系升温过程中的密度变化图 Fig. S2 The density plots of each system during the heating process | |
|
Download:
|
| 图 S3 各个体系S4(13)骨架原子的RMSD随时间变化曲线 Fig. S3 The RMSD plots for backbone atoms of S4(13) in each system as a function of time | |
|
Download:
|
| 图 S4 S7体系的初始结构、稳定结构和S4、S8体系中S4(13)稳定四聚体结构 Fig. S4 The initial and equilibrated conformers of system S7 and the equilibrated tetramer structures of S4(13) in systems S4 and S8 | |

|
Download:
|
| 图 S5 伞形采样体系分子内氢键的数量随时间的变化 Fig. S5 The number of intramolecular hydrogen bonds of umbrella sampling systems with time | |
|
Download:
|
| 图 S6 各体系S4(13)二级结构随模拟时间的变化 Fig. S6 The secondary structure changes of S4(13) in each system during the simulation time | |
| [1] |
Lipinski C A, Lombardo F, Dominy B W, et al. Experimental and computational approaches to estimate solubility and permeability in drug discovery and development settings[J]. Advanced Drug Delivery Reviews, 1997, 23(1-3): 3-25. Doi:10.1016/S0169-409X(96)00423-1 |
| [2] |
涂展春, 欧阳钟灿. 生物膜弹性的几何理论[J]. 中国科学院研究生院学报, 2008, 25(6): 849-854. |
| [3] |
秦飞, 姚鑫. 环糊精固载量不同的壳聚糖药物载体合成及性能[J]. 中国科学院大学学报, 2015, 32(1): 51-56. |
| [4] |
Dissanayake S, Denny W A, Gamage S, et al. Recent developments in anticancer drug delivery using cell penetrating and tumor targeting peptides[J]. Journal of Controlled Release, 2017, 250: 62-76. Doi:10.1016/j.jconrel.2017.02.006 |
| [5] |
Borrelli A, Tornesello A L, Tornesello M L, et al. Cell penetrating peptides as molecular carriers for anti-cancer agents[J]. Molecules, 2018, 23(2): 295. Doi:10.3390/molecules23020295 |
| [6] |
Taylor R E, Zahid M. Cell penetrating peptides, novel vectors for gene therapy[J]. Pharmaceutics, 2020, 12(3): 225. Doi:10.3390/pharmaceutics12030225 |
| [7] |
Wang F H, Wang Y, Zhang X, et al. Recent progress of cellpenetrating peptides as new carriers for intracellular cargo delivery[J]. Journal of Controlled Release, 2014, 174: 126-136. Doi:10.1016/j.jconrel.2013.11.020 |
| [8] |
何小林, 王大成. 中国马氏钳蝎神经毒素BmK M1和M4的晶体结构研究[J]. 中国科学院研究生院学报, 2001, 18(1): 97-100. Doi:10.3969/j.issn.1002-1175.2001.01.014 |
| [9] |
Singh T, Murthy A S N, Yang H J, et al. Versatility of cell-penetrating peptides for intracellular delivery of siRNA[J]. Drug Delivery, 2018, 25(1): 1996-2006. Doi:10.1080/10717544.2018.1543366 |
| [10] |
Cardoso A M S, Trabulo S, Cardoso A L, et al. S4(13)-PV cell-penetrating peptide induces physical and morphological changes in membrane-mimetic lipid systems and cell membranes: Implications for cell internalization[J]. Biochimica et Biophysica Acta(BBA)-Biomembranes, 2012, 1818(3): 877-888. Doi:10.1016/j.bbamem.2011.12.022 |
| [11] |
Morais C M, Cardoso A M, Aguiar L, et al. Lauroylated histidine-enriched S413-PV peptide as an efficient gene silencing mediator in cancer cells[J]. Pharmaceutical Research, 2020, 37(10): 188. Doi:10.1007/s11095-020-02904-x |
| [12] |
Morais C M, Cardoso A M, Cunha P P, et al. Acylation of the S413-PV cell-penetrating peptide as a means of enhancing its capacity to mediate nucleic acid delivery: relevance of peptide/lipid interactions[J]. Biochimica et Biophysica Acta(BBA)-Biomembranes, 2018, 1860(12): 2619-2634. Doi:10.1016/j.bbamem.2018.10.002 |
| [13] |
Hariton-Gazal E, Feder R, Mor A, et al. Targeting of nonkaryophilic cell-permeable peptides into the nuclei of intact cells by covalently attached nuclear localization signals[J]. Biochemistry, 2002, 41(29): 9208-9214. Doi:10.1021/bi0201466 |
| [14] |
Trabulo S, Mano M, Faneca H, et al. S413-PV cell penetrating peptide and cationic liposomes act synergistically to mediate intracellular delivery of plasmid DNA[J]. The Journal of Gene Medicine, 2008, 10(11): 1210-1222. Doi:10.1002/jgm.1247 |
| [15] |
Mano M, Teodósio C, Paiva A, et al. On the mechanisms of the internalization of S413-PV cell-penetrating peptide[J]. The Biochemical Journal, 2005, 390(Pt2): 603-612. |
| [16] |
Mano M, Henriques A, Paiva A, et al. Cellular uptake of S413-PV peptide occurs upon conformational changes induced by peptide-membrane interactions[J]. Biochimica et Biophysica Acta(BBA)-Biomembranes, 2006, 1758(3): 336-346. Doi:10.1016/j.bbamem.2006.01.014 |
| [17] |
Mano M, Henriques A, Paiva A, et al. Interaction of S413-PV cell penetrating peptide with model membranes: relevance to peptide translocation across biological membranes[J]. Journal of Peptide Science, 2007, 13(5): 301-313. Doi:10.1002/psc.842 |
| [18] |
Padari K, Koppel K, Lorents A, et al. S413-PV cell-penetrating peptide forms nanoparticle-like structures to gain entry into cells[J]. Bioconjugate Chemistry, 2010, 21(4): 774-783. Doi:10.1021/bc900577e |
| [19] |
Xu Y C, Jiang H L. Molecular dynamics simulations studies on the structure-function relationship of protein targets related to Alzheimer's disease[J]. Journal of the Graduate School of the Chinese Academy of Sciences, 2009, 26(2): 280-287. |
| [20] |
方磊, 计明娟. PTP1B选择性抑制剂的分子动力学模拟及结合自由能计算[J]. 中国科学院研究生院学报, 2009, 26(1): 58-64. |
| [21] |
Gautam A, Singh H, Tyagi A, et al. CPPsite: a curated database of cell penetrating peptides[J]. Database: the Journal of Biological Databases and Curation, 2012, 2012: bas015. |
| [22] |
Kardani K, Bolhassani A. Cppsite 2.0:an available database of experimentally validated cell-penetrating peptides predicting their secondary and tertiary structures[J]. Journal of Molecular Biology, 2020, 433(11): 166703. |
| [23] |
Agrawal P, Bhalla S, Usmani S S, et al. CPPsite 2.0:a repository of experimentally validated cell-penetrating peptides[J]. Nucleic Acids Research, 2016, 44(D1): D1098-D1103. Doi:10.1093/nar/gkv1266 |
| [24] |
Bagheri M, Keller S, Dathe M. Interaction of W-substituted analogs of cyclo-RRRWFW with bacterial lipopolysaccharides: the role of the aromatic cluster in antimicrobial activity[J]. Antimicrobial Agents and Chemotherapy, 2011, 55(2): 788-797. Doi:10.1128/AAC.01098-10 |
| [25] |
Kim S, Chen J, Cheng T J, et al. PubChem 2019 update: improved access to chemical data[J]. Nucleic Acids Research, 2019, 47(D1): D1102-D1109. Doi:10.1093/nar/gky1033 |
| [26] |
Frisch M J, Trucks G W, Schlegel H B, et al. Gaussian 09, Revision A. 01[CP]. Gaussian, Inc, Wallingford CT, 2009.
|
| [27] |
Wang J M, Wolf R M, Caldwell J W, et al. Development and testing of a general amber force field[J]. Journal of Computational Chemistry, 2004, 25(9): 1157-1174. Doi:10.1002/jcc.20035 |
| [28] |
Case D A, Ben-Shalom I Y, Brozell S R, et al. AMBER 2018[CP]. University of California, San Francisco, 2018.
|
| [29] |
Sousa da Silva A W, Vranken W F. ACPYPE-AnteChamber PYthon parser interface[J]. BMC Research Notes, 2012, 5(1): 367. Doi:10.1186/1756-0500-5-367 |
| [30] |
Wang J M, Cieplak P, Kollman P A. How well does a restrained electrostatic potential (RESP) model perform in calculating conformational energies of organic and biological molecules?[J]. Journal of Computational Chemistry, 2000, 21(12): 1049-1074. Doi:10.1002/1096-987X(200009)21:12<1049::AID-JCC3>3.0.CO;2-F |
| [31] |
Martínez L, Andrade R, Birgin E G, et al. PACKMOL: a package for building initial configurations for molecular dynamics simulations[J]. Journal of Computational Chemistry, 2009, 30(13): 2157-2164. Doi:10.1002/jcc.21224 |
| [32] |
Maier J A, Martinez C, Kasavajhala K, et al. ff14SB: improving the accuracy of protein side chain and backbone parameters from ff99SB[J]. Journal of Chemical Theory and Computation, 2015, 11(8): 3696-3713. Doi:10.1021/acs.jctc.5b00255 |
| [33] |
Jorgensen W L, Chandrasekhar J, Madura J D, et al. Comparison of simple potential functions for simulating liquid water[J]. The Journal of Chemical Physics, 1983, 79(2): 926-935. Doi:10.1063/1.445869 |
| [34] |
Ryckaert J P, Ciccotti G, Berendsen H J C. Numerical integration of the Cartesian equations of motion of a system with constraints: molecular dynamics of n-alkanes[J]. Journal of Computational Physics, 1977, 23(3): 327-341. Doi:10.1016/0021-9991(77)90098-5 |
| [35] |
Darden T, York D, Pedersen L. Particle mesh Ewald: an N·log(N) method for Ewald sums in large systems[J]. The Journal of Chemical Physics, 1993, 98(12): 10089-10092. Doi:10.1063/1.464397 |
| [36] |
Kumar S, Rosenberg J M, Bouzida D, et al. The weighted histogram analysis method for free-energy calculations on biomolecules. I. The method[J]. Journal of Computational Chemistry, 1992, 13(8): 1011-1021. Doi:10.1002/jcc.540130812 |
| [37] |
Souaille M, Roux B. Extension to the weighted histogram analysis method: combining umbrella sampling with free energy calculations[J]. Computer Physics Communications, 2001, 135(1): 40-57. Doi:10.1016/S0010-4655(00)00215-0 |
| [38] |
Roe D R, Cheatham T E. PTRAJ and CPPTRAJ: software for processing and analysis of molecular dynamics trajectory data[J]. Journal of Chemical Theory and Computation, 2013, 9(7): 3084-3095. Doi:10.1021/ct400341p |
 2021, Vol. 38
2021, Vol. 38 


