弗林蛋白(Furin)与众多疾病的发生密切相关,比如乳腺癌、胰腺癌、炭疽病等,有效抑制其活性显得至关重要。1989年Fuller等[1]对哺乳动物的fur基因[2]的序列进行分析,发现fur基因编码的蛋白与酵母Kexin有很高的同源性,并将其命名为Furin。1990年,Furin被确认是一种成对的碱性氨基酸内切蛋白酶,随后大量Furin的同源物陆续被发现,这些同源物统称为前体蛋白转化酶,它们都能切割前体蛋白进而将其激活,其中Furin是第一个被研究且研究最清楚的前体蛋白转化酶[3]。
Furin作为前体蛋白转换酶,最开始也是从没有活性的前体蛋白(Furin前体)中得到的。Furin前体由前肽和Furin共2部分组成[4],对其进行2次自我剪切,将前肽与Furin分离,生成具有生物活性的分子。第1次剪切是在内质网[5-6]中迅速完成的,前肽被切割,但没有与Furin彻底分离,而是附着在Furin上,进而抑制Furin发挥作用。第2次剪切是在高尔基体中缓慢发生,此时环境的pH发生改变,导致前肽中对pH敏感的序列被切割,使其与Furin完全分离,Furin没有了自身前肽的抑制而具有了生物活性,进而开始切割其他前体蛋白并将其激活来发挥作用[7]。
Furin切割的氨基酸序列有一定规律,(P1)Arg-X-X-Arg↓(P4)(↓为切割位点)是Furin能识别的最短序列[8],其中X表示任意氨基酸,但Furin更容易识别P2位置为碱性的Lys或Arg的氨基酸序列[9]。Furin可以切割生长因子、基质金属蛋白酶和病毒的糖蛋白等蛋白[10],这些前体蛋白与很多疾病相关,这也使得Furin在各种的疾病中被研究,并取得了一定进展。
Furin可切割多种癌症相关的前体蛋白,已被证实的有:膜型基质金属蛋白酶1(membrane type-1 matrix metalloproteinase, MT1-MMP)、血管内皮生长因子C(vascular endothelial growth factor C, VEGF-C)、血小板衍生生长因子(platelet derived growth factor, PDGF)、胰岛素生长因子1型受体(insulin-like growth factor 1, IGF-1R)、转化生长因子β(transforming growth factor-β, TGFβ)、去整合素基质金属蛋白酶(a disintegrin and metalloprotease, ADAM)[11]等。Furin通过切割这些前体蛋白,使其发挥生物活性,导致正常细胞癌化[12]。有研究证实Furin参与了头颈部鳞状细胞癌及和乳腺癌的癌症进程[11],在对乳腺癌的研究中发现,Furin通过对抑癌基因(phosphatase and tensin homolog deleted on chromosome ten, PTEN)产生抑制效应,降低它的活性,从而增强乳腺癌细胞的表达[13]。也有研究表明,Furin可促进胰腺癌细胞的上皮-间质转化(epithelial to mesenchy-mal transition, EMT)[14],抑制Furin的表达是治疗胰腺癌的一种有效方法。此外Furin还参与与卵巢癌相关的前体蛋白的表达[15]。Furin介导炭疽毒素进入细胞[16],当毒素与细胞膜表面的蛋白受体结合后,Furin被激活,将结合体切割,毒性蛋白被释放入细胞液中,使人中毒。
Furin与众多疾病相关,寻找抑制Furin活性的药物十分必要。目前,人们设计了许多Furin的抑制剂,这些抑制剂可分为小分子抑制剂和基于蛋白的大分子抑制剂。其中最具吸引力的是小分子化合物,因为它们可以通过合成路线获得,具有持久的代谢和蛋白水解稳定性等特点。小分子抑制剂又可细分为肽类抑制剂、非肽类抑制剂和肽的类似物抑制剂[17]。
1) 肽类抑制剂
Furin是从Furin前体中得到的,Furin前体包含前肽和Furin这2部分,前肽的存在会抑制Furin发挥作用。研究发现,前肽中一些特定的氨基酸序列会对Furin起到抑制作用,如果P1-P2位是双碱性氨基酸会起到很大的抑制效应[18]。另外在多肽中引入一对二硫键,首尾相连形成环肽,通过这种方法可以形成很多新的抑制剂。例如,利用这一方法使大麦丝氨基酸蛋白酶抑制剂-2转变为十八环肽抑制剂-2,这大大提高了抑制效率[19]。
2) 肽的类似物抑制剂
将Furin作用的底物的肽键(-CO-NH-)进行改造,把能被剪切的肽键换成不能被剪切的甲基酮键(-COCH2-),即换成Arg-X-X-Arg↓-(COCH2)-X,Arg-X的肽键替换为(-COCH2-),改造后的肽类似物对Furin的抑制性很强,抑制常数为3.4nmol/L[20]。
3) 非肽类抑制剂
非肽类抑制剂有对细胞的穿透力强、专一性高和不易被降解等优点,目前大多数是对于此类抑制剂的研究。抑制活性最好的非肽类抑制剂是琥珀酰(andrographolide, SEA)[21],另外一些含有吡啶和喹啉环的化合物也对Furin有很高的抑制性[22]。
鉴于以上研究,我们基于Furin受体进行虚拟筛选,通过分子对接,分子动力学模拟,最终筛选出最具潜能的Furin小分子抑制剂。
1 材料和方法 1.1 模型搭建本文所使用的Furin蛋白(PDB ID 5JXH)的X射线晶体结构来源于蛋白质数据库RCSB PDB,分辨率为2.0Å(1Å=0.1nm)。该蛋白质包含473个残基,活性中心已经确定,由12个残基组成活性口袋,分别是ASP153、HIS194、ALA252、SER253、TRP254、GLY255、PRO256、GLU257、ASP258、ASN295、THR367和SER368[23]。
1.2 分子动力学模拟用Amber18的antechamber工具创建配体小分子的力场参数。通过加入Na+和Cl-使模拟体系电荷为0。然后将每个体系浸入厚度为1.2nm的TIP3P水分子周期边界盒子中。采用PME方法处理静电相互作用。每个体系经历3步优化过程。首先采用最陡下降法,然后采用共轭梯度法,最后经历7步升温过程,将体系温度从0K升到310K[24]。每个体系模拟100ns,用RMSD评估和检查体系的动力学稳定性。对于共价结合的氢原子,采用Shake算法。使用Berendsen算法保持温度恒定。使用Amber18中的CPPTRAJ模块对轨迹进行分析。
1.3 结合自由自由能计算有2种方法可以估计对接复合物的结合自由能:分子力学-广义Born表面积法(MMGBSA)和分子力学泊松-Boltzmann表面积法(MMPBSA)。2种方法在计算体系的非成键的相互作用能的贡献都是采用的分子力学的方法。在溶液效应中,非极性部分对整个体系的自由能的影响都是通过计算分子表面积而得到[25]。不同的是,在计算溶液效应中,计算极性部分对整个体系的自由能的贡献时,MMPBSA是通过解Possion-Boltzmanne(PB)方程的方法得到,MMGBSA通过广义Born模型得到。2种方法得到的自由能都是相对的,所以我们采用MMGBSA方法,分析分子运动轨迹,得到受体、配体和复合物的各项能量和总能量,并通过以下方程估计系统的净能量:
| $ \Delta G_{\text {Binding }}=\Delta G_{\text {Complex }}-\Delta G_{\text {Receptor }}-\Delta G_{\text {Ligand }}. $ |
上述每一项都包含几种键合能与非键合能的计算,如范德华相互作用能、静电相互作用能、极性对溶剂化能的贡献等。
2 结果与讨论 2.1 构建药效团模型基于Furin受体的关键残基以及残基和抑制剂分子间的相互作用构建药效团。我们构建的Furin药效团模型,是基于2个关键残基GLY255、GLU257和氢键相互作用构建的3个药效团。其中,以GLY255为氢键供体和氢键受体构建了2个药效团,以GLU257为氢键受体构建了1个药效团。
2.2 基于药效团模型的虚拟筛选利用SYBYL进行第1次筛选,筛选的分子库为ZINC数据库,筛选的小分子数目为9800个,第1次筛选的原则基于类药五原则(Lipinski rules)和pan assay interference compounds(PAINS)。第1次筛选后,剩余小分子数目9000个。基于Furin药效团模型进行第2次筛选,利用SYBYL中的部分匹配功能,筛选满足药效团的小分子。经过筛选,最后选中22个小分子。
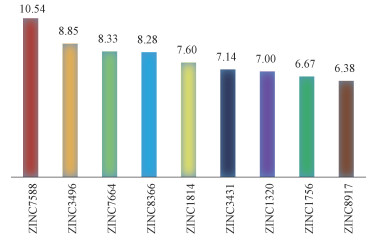
2.3 分子对接利用SYBYL将选中的22个小分子与Furin受体进行对接,根据能量匹配和位置匹配,进行打分排序,选取打分值高于6分的小分子展示,如图 1所示。筛选出打分排名靠前的4个小分子,从图 1可以看出,分别为ZINC7588、ZINC3496、ZINC7664和ZINC8366,4个小分子和受体Furin对接后的复合物结构分别为F7588、F3496、F7664和F8366。
|
Download:
|
| 图 1 小分子与Furin的对接得分 Fig. 1 Docking score of small molecule and Furin | |
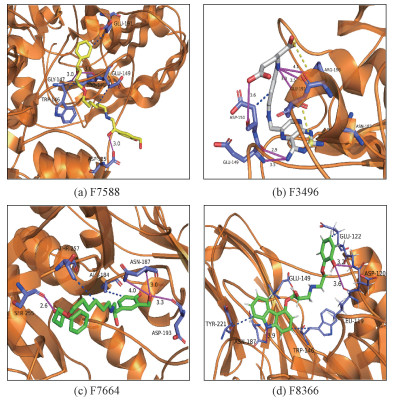
为了更清楚地认识ZINC7588、ZINC3496、ZINC7664、ZINC8366与Furin之间的相互作用,对4个小分子与Furin的结合模式进行分析,如图 2所示。图中显示了小分子与Furin之间的非键相互作用和关键残基,不同颜色的线分别表示氢键、疏水相互作用和盐桥。
|
Download:
|
| 粉实线表示氢键, 蓝虚线表示疏水相互作用, 黄虚线表示盐桥 图 2 F7588、F3496、F7664、F8366的结合模式图 Fig. 2 Binding mode of F7588, F3496, F7664, and F8366 | |
从图 2(a)可以看出ZINC7588与Furin的6个残基发生了相互作用,其中Asp125、Gly147和Pro148与Furin形成氢键,键长均为3.0Å;Trp146、Glu149和Glu191与Furin之间有疏水相互作用。从图 2(b)可以看出ZINC3496与Furin的5个残基发生相互作用,形成7个氢键,其中Glu149和Arg190分别与Furin形成2个氢键,键长分别为3.5、2.9、4.1、3.5Å;Asp150、Asn187和Glu191分别与Furin形成1个氢键,键长分别为3.6、2.9和3.0Å;Asp150与Furin之间有疏水相互作用;Arg190和Glu191与Furin形成盐桥。从图 2(c)可以看出ZINC7664与Furin的5个残基发生相互作用,其中Asn187与Furin形成2个氢键,键长分别为3.0和4.0Å;Asp193和Ser255分别与Furin形成1个氢键,键长分别为3.3和3.6Å;Ala184和Thr257与Furin之间有疏水相互作用。从图 2(d)可以看出ZINC8366与Furin的7个残基发生相互作用,其中Leu119、Asp120和Asn187与Furin形成氢键,键长分别为3.6、3.2和3.9Å。Glu122、Trp146、Glu149和Tyr221与Furin之间有疏水相互作用。
2.4 均方根偏差对接后的4个复合物结构F7588、F3496、F7664、F8366为初始构象,利用Amber 18软件进行100ns分子动力学模拟,得到4组轨迹,并对4组轨迹进行数据整理和分析。首先,对轨迹分析计算得到均方根偏差(RMSD)值随时间变化的图像,如图 3所示。

|
Download:
|
| 图 3 100ns分子动力学中F7588、F3496、F7664和F8366的RMSD Fig. 3 RMSD profile of F7588, F3496, F7664, and F8366 during 100ns MD simulation | |
图 3表明,4组轨迹均达到平衡,其中F7588、F7664和F8366的平均RMSD值稳定在2.0Å左右,F3496的平均RMSD值稳定在2.25Å左右,均在可接受范围内,表明4个小分子配体都可以紧密地结合在Furin受体的活性口袋中。
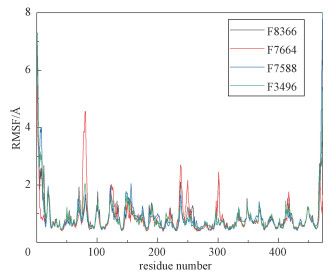
2.5 均方根扰动均方根扰动(RMSF)可以表示分子中各个原子的自由程度。对4组轨迹进行RMSF分析表明,Furin关键残基的RMSF值没有大幅度的波动,如图 4所示。
|
Download:
|
| 图 4 100ns分子动力学中F7588、F3496、F7664和F8366的RMSF图 Fig. 4 RMSF profile of F7588, F3496, F7664, and F8366 during 100ns MD simulation | |
对活性口袋中的关键残基的RMSF值进行分析,从图 4可以看出,小分子与Furin作用的关键残基的RMSF值处于一个相对较低的水平,平均值大约为0.8Å,表明由于Furin受体与小分子配体结合而使关键残基失去了灵活性,结合很稳定。
2.6 MMGBSA结合自由能计算通过计算复合物的结合自由能,评估Furin受体和小分子对接后的复合物的结合稳定性。对4个复合物的分子动力学模拟进行MMGBSA计算,得到4个复合物的各项能量平均值,如表 1所示。
|
|
表 1 F7588、F3496、F7664和F8366的结合自由能 Table 1 Binding free energies of F7588, F3496, F7664, and F8366 |
表 1列出了在MMGBSA中体系的结合自由能,结合自由能越低,表明结合稳定性越强。可以看出,在各项能量贡献中,复合物之间的相互作用主要是范德华相互作用能和静电相互作用能贡献,其中F7664的范德华相互作用能最低,为-47.47kcal/mol,F3496的静电相互作用能最低,为-98.60kcal/mol。极性溶剂化能不利于受体-抑制剂的结合,F3496的极性溶剂化能最高,为112.29kcal/mol。MMGBSA预测的复合物的净结合能由低到高分别是F7664、F3496、F8366和F7588。所以综合对比,小分子ZINC7664更容易与Furin受体结合,并且结合更稳定。
3 结论本文通过虚拟筛选、分子对接和分子动力学模拟的方法,进行基于Furin受体的虚拟筛选,以寻找Furin的潜在抑制剂。利用Sybyl基于Furin受体药效团模型进行虚拟筛选和分子对接,从ZINC数据库中筛选出4个小分子ZINC7588、ZINC3496、ZINC7664和ZINC8366,对4组复合物进行分子动力学模拟表明,它们在65ns达到平衡,保持了结构的稳定性,RMSD值稳定在2.0Å左右。在RMSF中可以看到,关键残基的RMSF保持较低水平,在0.8Å左右,也可以说明小分子与Furin受体结合稳定。对4组复合物进行MMGBSA分析对比,发现ZINC7664的结合自由能最低,为-39.28kcal/mol。综上所知,小分子ZINC7664与Furin受体的结合最稳定,具有较好的应用前景,可用于设计有效的抑制Furin发挥作用的药物,为治疗与Furin相关的疾病提供可能。
本文的计算结果是中国科学院计算机网络信息中心超级计算中心“元”上计算得到。
| [1] |
Fuller R S, Brake A J, Thorner J. Intracellular targeting and structural conservation of a prohormone-processing endoprotease[J]. Science, 1989, 246(4929): 482-486. Doi:10.1126/science.2683070 |
| [2] |
Roebroek A J M, Schalken J A, Bussemakers M J G, et al. Characterization of human c-Fes/fps reveals a new transcription unit (fur) in the immediately upstream region of the proto-oncogene[J]. Molecular Biology Reports, 1986, 11(2): 117-125. Doi:10.1007/BF00364823 |
| [3] |
Nakayama K. Furin: a mammalian subtilisin/Kex2p-like endoprotease involved in processing of a wide variety of precursor proteins[J]. The Biochemical Journal, 1997, 327(Pt 3): 625-635. |
| [4] |
Osadchuk T V, Shybyryn O V, Kibirev V K. Chemical structure and properties of low-molecular furin inhibitors[J]. Ukrainian Biochemical Journal, 2016, 88(6): 5-25. Doi:10.15407/ubj88.06.005 |
| [5] |
Gawlik K, Shiryaev S A, Zhu W, et al. Autocatalytic activation of the furin zymogen requires removal of the emerging enzyme's N-terminus from the active site[J]. PLoS One, 2009, 4(4): e5031. Doi:10.1371/journal.pone.0005031 |
| [6] |
Thomas G. Furin at the cutting edge: from protein traffic to embryogenesis and disease[J]. Nat Rev Mol Cell Biol, 2002, 3(10): 753-766. Doi:10.1038/nrm934 |
| [7] |
Salvas A, Benjannet S, Reudelhuber T L, et al. Evidence for proprotein convertase activity in the endoplasmic reticulum/early Golgi[J]. FEBS Letters, 2005, 579(25): 5621-5625. Doi:10.1016/j.febslet.2005.09.029 |
| [8] |
张友义, 刘伟娜, 赵慧娜, 等. Furin的生物学功能[J]. 生命的化学, 2019, 39(2): 353-359. |
| [9] |
Henrich S, Cameron A, Bourenkov G P, et al. The crystal structure of the proprotein processing proteinase furin explains its stringent specificity[J]. Nature Structural Biology, 2003, 10(7): 520-526. Doi:10.1038/nsb941 |
| [10] |
Rockwell N C, Thorner J W. The kindest cuts of all: crystal structures of Kex2 and furin reveal secrets of precursor processing[J]. Trends in Biochemical Sciences, 2004, 29(2): 80-87. Doi:10.1016/j.tibs.2003.12.006 |
| [11] |
Jaaks P, Bernasconi M. The proprotein convertase furin in tumour progression[J]. International Journal of Cancer, 2017, 141(4): 654-663. Doi:10.1002/ijc.30714 |
| [12] |
王涛, 赵晶, 杨安钢. 弗林蛋白酶: 一种广泛参与前体蛋白切割的内切蛋白酶[J]. 医学分子生物学杂志, 2006, 3(3): 202-205. Doi:10.3870/j.issn.1672-8009.2006.03.009 |
| [13] |
Li Y F, Chu J H, Li J, et al. Cancer/testis antigen-Plac1 promotes invasion and metastasis of breast cancer through Furin/NICD/PTEN signaling pathway[J]. Molecular Oncology, 2018, 12(8): 1233-1248. Doi:10.1002/1878-0261.12311 |
| [14] |
Zhang Y, Zhou M, Wei H, et al. Furin promotes epithelial-mesenchymal transition in pancreatic cancer cells via Hippo-YAP pathway[J]. International Journal of Oncology, 2017, 50(4): 1352-1362. Doi:10.3892/ijo.2017.3896 |
| [15] |
Longuespée R, Couture F, Levesque C, et al. Implications of proprotein convertases in ovarian cancer cell proliferation and tumor progression: insights for PACE4 as a therapeutic target[J]. Translational Oncology, 2014, 7(3): 410-419. Doi:10.1016/j.tranon.2014.04.008 |
| [16] |
Sergeeva O A, van der Goot F G. Anthrax toxin requires ZDHHC5-mediated palmitoylation of its surface-processing host enzymes[J]. PNAS, 2019, 116(4): 1279-1288. Doi:10.1073/pnas.1812588116 |
| [17] |
韩锦铂. 前体蛋白加工酶furin的天然抑制剂及相互作用蛋白研究[D]. 上海: 中国科学院研究生院(上海生命科学研究院), 2007.
|
| [18] |
Basak A, Lazure C. Synthetic peptides derived from the prosegments of proprotein convertase 1/3 and furin are potent inhibitors of both enzymes[J]. The Biochemical Journal, 2003, 373(Pt 1): 231-239. |
| [19] |
Villemure M, Fournier A, Gauthier D, et al. Barley serine proteinase inhibitor 2-derived cyclic peptides as potent and selective inhibitors of convertases PC1/3 and furin[J]. Biochemistry, 2003, 42(32): 9659-9668. Doi:10.1021/bi034418w |
| [20] |
Jean F, Boudreault A, Basak A, et al. Fluorescent peptidyl substrates as an aid in studying the substrate specificity of human prohormone convertase PC1 and human furin and designing a potent irreversible inhibitor[J]. Journal of Biological Chemistry, 1995, 270(33): 19225-19231. Doi:10.1074/jbc.270.33.19225 |
| [21] |
Basak A, Cooper S, Roberge A G, et al. Inhibition of proprotein convertases-1, -7 and furin by diterpines of Andrographis paniculata and their succinoyl esters[J]. The Biochemical Journal, 1999, 338(Pt 1): 107-113. |
| [22] |
Podsiadlo P, Komiyama T, Fuller R S, et al. Furin inhibition by compounds of copper and zinc[J]. Journal of Biological Chemistry, 2004, 279(35): 36219-36227. Doi:10.1074/jbc.M400338200 |
| [23] |
Dahms S O, Arciniega M, Steinmetzer T, et al. Structure of the unliganded form of the proprotein convertase furin suggests activation by a substrate-induced mechanism[J]. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(40): 11196-11201. |
| [24] |
方磊, 计明娟. PTP1B选择性抑制剂的分子动力学模拟及结合自由能计算[J]. 中国科学院研究生院学报, 2009, 26(1): 58-64. |
| [25] |
侯廷军, 朱丽荔, 陈丽蓉, 等. EGFR和4-苯胺喹唑啉类抑制剂之间相互作用模式的研究[J]. 化学学报, 2002, 60(6): 1023-1028. |
 2023, Vol. 40
2023, Vol. 40 


