2. 国家儿童医学中心 复旦大学附属儿科医院临床检验中心细菌室, 上海 201102
2. Laboratory of Microbiology of Center for Clinical Laboratory, Children's Hospital of Fudan University, National Children's Medical Center, Shanghai 201102, China
近年来,耐碳青霉烯类肠杆菌目细菌(carba-penem-resistant Enterobacterales, CRE),特别是耐碳青霉烯类肺炎克雷伯菌(carbapenem-resistant Klebsiella pneumoniae, CRKP)引起的感染在全球范围内呈上升趋势,对人类健康构成重大威胁[1]。CRKP存在区域差异[2],不同国家的多中心研究[3]发现,同一中心内儿童患者和成人患者的细菌克隆存在不同,不同区域儿童和成人患者也存在不同。尽管每年国内外均有相关的流行病学监测报告[4-5],但重点关注的是成年人群,而不是针对儿童。中国细菌耐药监测网(CHINET)监测数据显示,肺炎克雷伯菌(Klebsiella pneumoniae, KP)对亚胺培南和美罗培南的耐药率分别从2005年的3.0%、2.9%上升至2018年的25.5%、26.3%,2019—2021年的耐药率呈下降趋势,但其检出率仍超过23%[6]。根据中国儿童细菌耐药监测组(ISPED)的监测结果,儿童分离的KP对美罗培南的耐药率由2016年的13.4%上升至2020年的23%,并且在不同年龄组中新生儿患者CRE感染比例最高[7]。2021年全国细菌耐药监测网(CARSS)数据[8]表明,儿童CRKP检出率高于成人,应加强对儿童患者的关注。
KP对碳青霉烯类抗生素的耐药机制主要是产碳青霉烯酶,如Ambler A类β-内酰胺酶blaKPC,B类金属内酰胺酶blaNDM和D类β-内酰胺酶blaOXA-48[9]。CRKP在不同年龄患儿中表现出不同的分子特征[10],据报道B类碳青霉烯酶blaNDM-1主要存在于新生儿,而A类碳青霉烯酶blaKPC-2主要存在于非新生儿和成人患者中。另外,携带blaKPC-2的肺炎克雷伯菌多位点序列分型(multilocus sequence typing, MLST)的优势序列型为ST 11型,但同时也存在于其他序列型中,如ST 15型[11]。而携带blaNDM-1基因的KPN优势序列存在于不同的ST分型序列中[12-13]。对于儿童患者CRKP主要的耐药机制,blaNDM-1和blaKPC-2的临床及分子流行病学比较的相关数据目前尚未有报道。本研究对儿童患者2017—2020年分离CRKP菌株中碳青霉烯酶基因blaNDM-1和blaKPC-2阳性的菌株进行分析,对blaNDM-1和blaKPC-2阳性菌株的临床和分子流行病学特征进行比较。
1 资料与方法 1.1 资料来源选取某儿童医院儿童住院患者2017—2020年临床分离的CRKP。根据指南[14]推荐,CRKP定义为对包括亚胺培南、美罗培南和厄他培南中任何一种药物耐药的临床分离株。纳入标准:研究期间的非重复患者(年龄<18岁)临床分离的CRKP菌株,将每例患者首次入院后分离到的第一株CRKP菌株纳入研究。
1.2 调查内容查阅菌株来源患者的病历资料,获得患者的基本临床特征,包括人口统计学资料(年龄、性别等)、标本来源、科室、住院时间及预后等。
1.3 细菌培养、鉴定及药敏试验采用常规细菌培养方法对临床微生物标本进行细菌培养,细菌鉴定采用MALDI-TOF biotyper细菌质谱鉴定仪(德国布鲁克公司)进行鉴定。采用VITEK 2 Compact对菌株进行仪器法药敏试验。仪器法药敏试验采用AST GN13抗菌药物敏感性试验卡片(法国梅里埃公司)检测菌株对亚胺培南和厄他培南的最低抑菌浓度(MIC),其中对仪器法检出的厄他培南耐药菌株进一步采取纸片扩散法进行复核,测定菌株对美罗培南的抑菌圈直径。采用标准菌株大肠埃希菌ATCC 25922,大肠埃希菌ATCC 35218(产酶株)作为仪器法药物敏感性试验质控菌株,采用标准菌株大肠埃希菌ATCC 25922作为纸片扩散法药物敏感性试验质控菌株。
1.4 碳青霉烯酶基因检测通过聚合酶链式反应(PCR)检测碳青霉烯酶耐药基因(A组碳青霉烯酶blaKPC;B组碳青霉烯酶blaNDM),引物参考文献[10]的引物。PCR扩增子采用Sanger测序,所有DNA序列与Pubmed数据库中可采用的参考菌株进行BLAST检索比较。
1.5 多位点序列分型(MLST)参考法国巴斯德研究所MLST网站(https://bigsdb.pasteur.fr/klebsiella.html)公布的肺炎克雷伯菌MLST方法,对7个管家基因rpoB、gapA、mdh、pgi、phoE、infB和tonB的片段进行PCR扩增和序列测定,测序结果与上述网站的数据库进行比对,获得各基因位点的编号和MLST型别。利用BioNumerics软件绘制CRKP的最小生成树。
1.6 统计学方法应用统计软件SPSS 21.0进行统计分析。非正态分布计量资料采用中位数(四分位数)[M(P25,P75)]表示,两组间比较用Mann-Whitney U检验;率的比较采用Fisher确切概率法。以P≤0.05为差异有统计学意义。
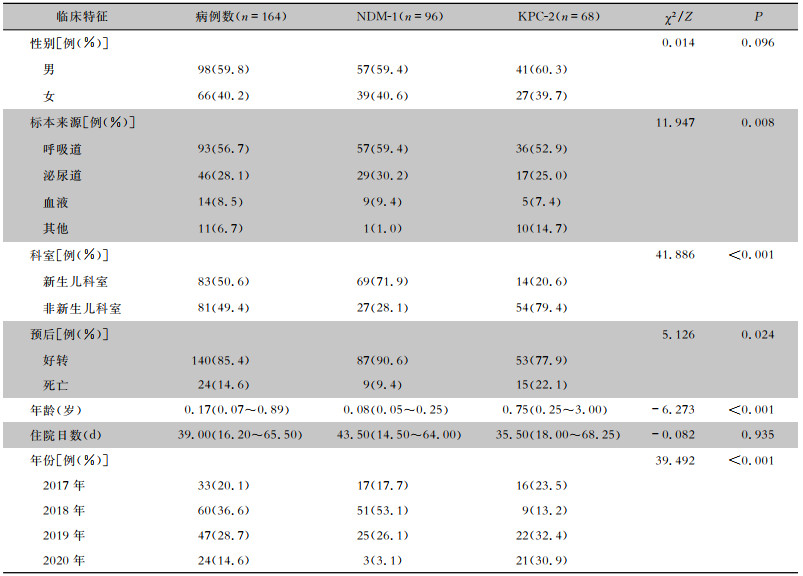
2 结果 2.1 产NDM-1和产KPC-2患者的临床特点比较2017—2020年,共收集164株符合条件的CRKP,来源患者中男性98株,女性66株;标本来源主要为呼吸道,其次是泌尿道和血液,其他标本来源11株,包括创口分泌物和脑脊液各3株,引流液2株,脓液、胸腔积液和眼分泌物各1株。菌株主要分布在新生儿重症监护病房(NICU)和新生儿科,其次是儿科重症监护病房(PICU)、内科和外科;140例患儿最后好转出院,死亡24例,病死率14.6%;所有菌株均携带碳青霉烯耐药基因,其中96株携带blaNDM-1,68株携带blaKPC-2。
2017—2019年主要为产NDM-1菌株,2019年后主要为产KPC-2菌株。对其临床特点进行比较,携带blaNDM-1和blaKPC-2的CRKP菌株在标本来源、科室分布、预后以及患者年龄比较,差异均有统计学意义(均P<0.05)。产KPC-2的患者病死率较产NDM-1的患者高(22.1% VS 9.4%),差异有统计学意义(P<0.05)。见表 1。
| 表 1 产NDM-1和产KPC-2患者的临床特征比较 Table 1 Comparison in clinical characteristics between patients with NDM-1-procucing and KPC-2-producing CRKP strains |
|
共发现31种不同的亚型,其中ST 11最为流行(31.10%,51株),其次是ST 17(23.78%,39株),ST 278(10.98%,18株),ST 1883(3.66%,6株),ST 2735(3.05%,5株),ST 846(2.44%,4株),ST 1640(2.44%,4株),其他亚型检出率均较低,不足2%。从时间分布来看,2017年以ST 11(27.27%,9株)和ST 278(21.21%,7株)为主,2018年以ST 17(56.67%,34株)和ST 278(18.33%,11株)为主,而2019和2020年以ST 11(31.91%,15株;79.17%,19株)为主,见图 1。
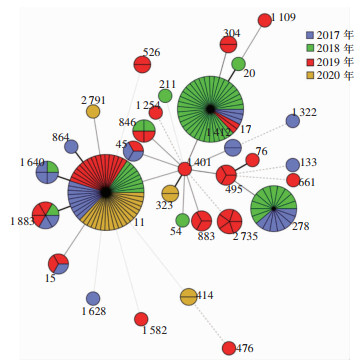 |
| 图 1 2017—2020年CRKP菌株MLST分型数据的BioNumerics分析 Figure 1 BioNumerics analysis on MLST of CRKP strains, 2017-2020 |
产NDM-1和产KPC-2的CRKP菌株MLST分型也存在一定差异,产NDM-1的CRKP菌株以ST 17(40.63%,39株)和ST 278(18.75%,18株)为主,而产KPC-2的CRKP菌株以ST 11(73.53%,50株)为主,见图 2。
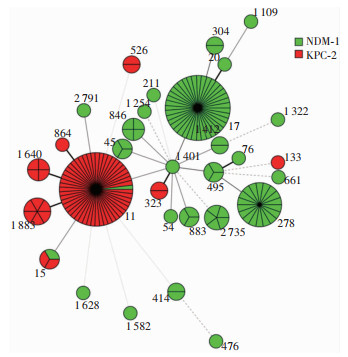 |
| 图 2 NDM-1和KPC-2 CRKP菌株MLST分型数据的BioNumerics分析 Figure 2 BioNumerics analysis on MLST of NDM-1-procu-cing and KPC-2-producing CRKP strains |
CRKP对第一、二、三代头孢菌素以及酶抑制剂氨苄西林/舒巴坦、哌拉西林/他唑巴坦、头孢哌酮/舒巴坦的耐药率均达90%以上。对碳青霉烯类药物美罗培南、厄他培南和亚胺培南的耐药率也在90%以上,2020年均达到100%。未发现对多黏菌素B和替加环素耐药的菌株。
CRKP对头孢吡肟、阿米卡星、庆大霉素、复方磺胺甲口恶唑(SMZ)和呋喃妥因的耐药率呈波动上升趋势,在2018年下降,随后又逐渐上升。对氨曲南的耐药率呈缓慢上升趋势,对磷霉素的耐药率呈缓慢下降趋势,见图 3。
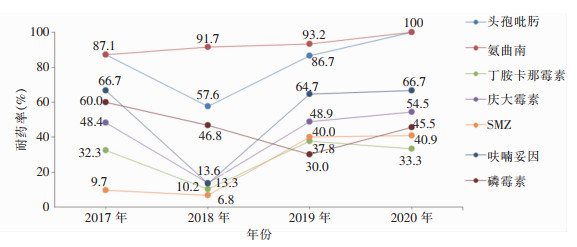 |
| 图 3 2017—2020年CRKP对常用抗菌药物的耐药率 Figure 3 Resistance rates of CRKP strains to commonly used antimicrobial agents, 2017-2020 |
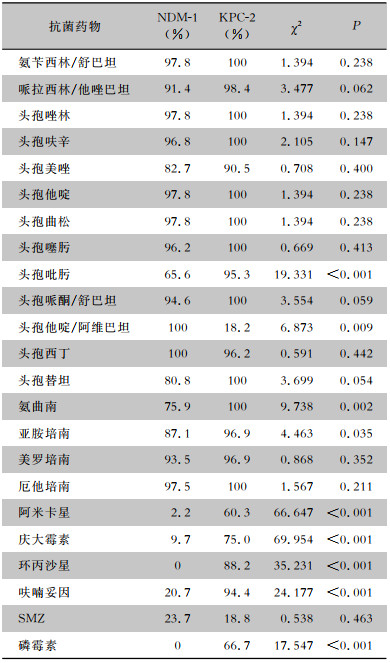
产NDM-1与产KPC-2的CRKP分离株耐药率亦存在差异。产KPC-2的CRKP分离株对头孢吡肟、氨曲南、亚胺培南、阿米卡星、庆大霉素、环丙沙星、呋喃妥因、磷霉素的耐药率均高于产NDM-1的CRKP分离株,差异均有统计学意义(均P<0.05)。产NDM-1的CRKP分离株对头孢他啶/阿维巴坦的耐药率高于产KPC-2的CRKP分离株,差异有统计学意义(P<0.05),见表 2。
| 表 2 产NDM-1和产KPC-2的CRKP菌株对常用抗菌药物的耐药率比较 Table 2 Comparison in resistance rates between NDM-1-procucing and KPC-2-producing CRKP strains to commonly used antimicrobial agents |
|
研究CRKP感染的流行病学对制定临床治疗方案和评价不同治疗策略的效果具有重要作用。目前关于儿童患者感染不同耐药基因的CRKP临床及分子流行病学比较的相关数据较少。本研究对2017—2020年某儿童医院分离的产NDM-1和产KPC-2的CRKP菌株临床特征、分子分型及其耐药性特点进行分析比较,结果显示产NDM-1和产KPC-2的CRKP菌株在科室分布、年龄和患者预后上存在差异,分子分型亦存在不同,且产KPC-2的CRKP菌株较产NDM-1的CRKP菌株表现出更加严峻的耐药特征,更高的病死率。
在科室分布和年龄上,产NDM-1和产KPC-2的CRKP存在差异。国内研究[15]指出,不同于成人患者主要产KPC型碳青霉烯酶,儿童分离的CRE菌株主要是产NDM-1型金属酶。在碳青霉烯酶基因NDM变异中,NDM-1被认为是最常见的类型[16],可在肺炎克雷伯菌多种克隆群中流行播散。先前对CRKP分子流行病学研究[10, 17-18]也表明,NDM-1是新生儿分离株对碳青霉烯类耐药的主要耐药机制。本文研究结果亦显示产NDM-1的CRKP主要分布在小年龄的新生儿,产KPC-2的CRKP以大年龄的非新生儿患者居多。需要关注的是,该院儿童CRKP患者其耐药基因出现由携带NDM-1到KPC-2转变的趋势。blaKPC-2是最常见的A类β-内酰胺酶,在大年龄儿童及成人患者中报道较多,且有许多医院感染暴发的报道[19-20]。与其他碳青霉烯酶基因相比,blaKPC-2表现出更强的传播能力和更高的毒力[21-22]。
分子分型方面,产NDM-1和产KPC-2的CRKP亦存在差异。164株CRKP分离株中发现31种ST型别,其中ST 11、ST 17、ST 278、ST 1883和ST 2735是CRKP的主要ST型别。有研究[23-24]表明ST 11为产KPC酶KP的主要克隆型,本研究中ST 11占31.10%,产NDM-1的CRKP菌株的MLST型以ST 17型和ST 278型为主,产KPC-2的CRKP菌株的MLST型主要是ST 11型。值得注意的是,该院儿童患者CRKP其ST型别一直在发生动态改变。对于中国的非新生儿和成人患者,ST 11一直是主要的ST类型,并没有发生显著变化[10, 19]。研究[25]报道,携带KPC的ST 11 KPN分离株的流行通常与质粒或转座子介导的水平基因转移有关,因此,应特别关注中国儿童患者中ST 11-KPC-KPN高风险克隆株的潜在传播。
此外,产NDM-1和产KPC-2的CRKP药敏结果及感染患者的预后也显示出较大差异。携带碳青霉烯酶KPC-2和NDM-1的CRKP菌株表现出不同的耐药特征,此与其他儿童研究[26]结果相似。CRKP均为多重耐药菌,其中对第一、二、三代头孢菌素以及酶抑制剂等耐药率均达90%以上,耐药形式严峻。与NDM-1相比,KPC-2表现出更严重的耐药性,对头孢吡肟、氨曲南、亚胺培南、阿米卡星、庆大霉素、呋喃妥因、磷霉素具有更高的耐药性,且预后较差,应引起临床和感控的重视。
本研究对儿童患者首次分离到的产KPC-2和NDM-1的CRKP进行临床及分子流行病学的比较,发现了产KPC-2和NDM-1的CRKP其临床和分子分型的差异,但是本研究也存在一定缺陷。首先,本研究为单中心研究,其结果是否与其他地区一致,值得后期多中心的研究进一步比较。另外,仅分析2017—2021年4年的数据,时间较短,后续将继续收集菌株进行动态监测。此外,产KPC-2和NDM-1的CRKP临床及分子流行病学差异的存在可能与传播、聚集性感染有关,但是需要进一步同源性监测进行证明。最后,作为重要的治疗药物[27],本研究对头孢他啶/阿维巴坦、多黏菌素B和替加环素的药物敏感性数据有限。
综上所述,产KPC-2和NDM-1的CRKP表现出不同的临床和分子流行病学特征,产KPC-2型CRKP表现出更严重的耐药性,且患者预后较差,给医院感染防控带来更严峻的挑战。
利益冲突:所有作者均声明不存在利益冲突。
| [1] |
Zhang D, Hu SS, Sun JY, et al. Antibiotic consumption versus the prevalence of carbapenem-resistant Gram-negative bacteria at a tertiary hospital in China from 2011 to 2017[J]. J Infect Public Health, 2019, 12(2): 195-199. DOI:10.1016/j.jiph.2018.10.003 |
| [2] |
Wyres K, Holt K. Regional differences in carbapenem-resis-tant Klebsiella pneumoniae[J]. Lancet Infect Dis, 2022, 22(3): 309-310. DOI:10.1016/S1473-3099(21)00425-4 |
| [3] |
Castagnola E, Tatarelli P, Mesini A, et al. Epidemiology of carbapenemase-producing Enterobacteriaceae in a pediatric hospital in a country with high endemicity[J]. J Infect Public Health, 2019, 12(2): 270-274. DOI:10.1016/j.jiph.2018.11.003 |
| [4] |
Hu FP, Zhu DM, Wang F, et al. Current status and trends of antibacterial resistance in China[J]. Clin Infect Dis, 2018, 67(Suppl 2): S128-S134. |
| [5] |
Veeraraghavan B, Walia K. Antimicrobial susceptibility profile & resistance mechanisms of global antimicrobial resistance surveillance system (GLASS) priority pathogens from India[J]. Indian J Med Res, 2019, 149(2): 87-96. DOI:10.4103/ijmr.IJMR_214_18 |
| [6] |
胡付品, 郭燕, 朱德妹, 等. 2021年CHINET中国细菌耐药监测[J]. 中国感染与化疗杂志, 2022, 22(5): 521-530. Hu FP, Guo Y, Zhu DM, et al. CHINET surveillance of antimicrobial resistance among the bacterial isolates in 2021[J]. Chinese Journal of Infection and Chemotherapy, 2022, 22(5): 521-530. |
| [7] |
Fu P, Xu HM, Jing CM, et al. Bacterial epidemiology and antimicrobial resistance profiles in children reported by the ISPED program in China, 2016 to 2020[J]. Microbiol Spectr, 2021, 9(3): e0028321. DOI:10.1128/Spectrum.00283-21 |
| [8] |
全国细菌耐药监测网. 2021年全国细菌耐药监测报告[J]. 中华检验医学杂志, 2023, 46(6): 566-581. China Antimicrobial Resistance Surveillance System. 2021 national antimicrobial resistance surveillance report[J]. Chinese Journal of Laboratory Medicine, 2023, 46(6): 566-581. DOI:10.3760/cma.j.cn114452-20230119-00040 |
| [9] |
Lutgring JD. Carbapenem-resistant Enterobacteriaceae: an emerging bacterial threat[J]. Semin Diagn Pathol, 2019, 36(3): 182-186. DOI:10.1053/j.semdp.2019.04.011 |
| [10] |
Yin LJ, He LY, Miao J, et al. Actively surveillance and appropriate patients placements' contact isolation dramatically decreased carbapenem-resistant Enterobacteriaceae infection and colonization in pediatric patients in China[J]. J Hosp Infect, 2020, 105(3): 486-494. DOI:10.1016/j.jhin.2020.03.031 |
| [11] |
肖士林, 陈东科, 秦天, 等. 携带blaKPC-2型耐药基因肺炎克雷伯菌的流行克隆研究[J]. 中华医院感染学杂志, 2020, 30(1): 1-5. Xiao SL, Chen DK, Qin T, et al. Study of epidemic clone of Klebsiella pneumoniae carrying blaKPC-2 gene[J]. Chinese Journal of Nosocomiology, 2020, 30(1): 1-5. |
| [12] |
王群, 叶梅毅, 王芳, 等. 携带blaNDM-1耐药基因肺炎克雷伯菌的耐药性及分子流行特征研究[J]. 现代预防医学, 2020, 47(13): 2426-2430. Wang Q, Ye MY, Wang F, et al. Drug resistance and molecular epidemiological characteristics of Klebsiella pneumoniae carrying blaNDM-1 resistance gene[J]. Modern Preventive Medicine, 2020, 47(13): 2426-2430. |
| [13] |
Yin D, Zhang L, Wang A, et al. Clinical and molecular epidemiologic characteristics of carbapenem-resistant Klebsiella pneumoniae infection/colonization among neonates in China[J]. J Hosp Infect, 2018, 100(1): 21-28. DOI:10.1016/j.jhin.2018.05.005 |
| [14] |
NCCLS news. M100-S11, performance standards for antimicrobial susceptibility testing[J]. Clin Microbiol Newsl, 2001, 23(6): 49. |
| [15] |
胡付品, 郭燕, 朱德妹, 等. 2018年CHINET中国细菌耐药性监测[J]. 中国感染与化疗杂志, 2020, 20(1): 1-10. Hu FP, Guo Y, Zhu DM, et al. CHINET surveillance of bacterial resistance in China: 2018 report[J]. Chinese Journal of Infection and Chemotherapy, 2020, 20(1): 1-10. |
| [16] |
Ahmad N, Khalid S, Ali SM, et al. Occurrence of blaNDM variants among Enterobacteriaceae from a neonatal intensive care unit in a northern India hospital[J]. Front Microbiol, 2018, 9: 407. DOI:10.3389/fmicb.2018.00407 |
| [17] |
Yin L, He L, Miao J, et al. Carbapenem-resistant Entero-bacterales colonization and subsequent infection in a neonatal intensive care unit in Shanghai, China[J]. Infect Prev Pract, 2021, 3(3): 100147. DOI:10.1016/j.infpip.2021.100147 |
| [18] |
Qin SS, Fu Y, Zhang QJ, et al. High incidence and endemic spread of NDM-1-positive Enterobacteriaceae in Henan Pro-vince, China[J]. Antimicrob Agents Chemother, 2014, 58(8): 4275-4282. DOI:10.1128/AAC.02813-13 |
| [19] |
李青, 油丽萍, 陈爱文, 等. 携带blaKPC-2基因的肺炎克雷伯菌ST 789型引起医院感染暴发[J]. 中华医院感染学杂志, 2016, 26(2): 251-253, 262. Li Q, You LP, Chen AW, et al. Outbreak of nosocomial infections due to Klebsiella pneumoniae ST 789 isolates harboring blaKPC-2 gene[J]. Chinese Journal of Nosocomiology, 2016, 26(2): 251-253, 262. |
| [20] |
张会, 赵辉, 董丹丹, 等. 携带blaKPC-2基因肺炎克雷伯菌引起儿童医院感染暴发的研究[J]. 中国实验诊断学, 2019, 23(1): 136-139. Zhang H, Zhao H, Dong DD, et al. Nosocomial outbreak of KPC-2-producing ST 11 Klebsiella pneumoniae in a children's hospital in China[J]. Chinese Journal of Laboratory Diagnosis, 2019, 23(1): 136-139. DOI:10.3969/j.issn.1007-4287.2019.01.055 |
| [21] |
Deshpande LM, Rhomberg PR, Sader HS, et al. Emergence of serine carbapenemases (KPC and SME) among clinical strains of Enterobacteriaceae isolated in the United States me-dical centers: report from the MYSTIC program (1999-2005)[J]. Diagn Microbiol Infect Dis, 2006, 56(4): 367-372. DOI:10.1016/j.diagmicrobio.2006.07.004 |
| [22] |
Nordmann P, Cuzon G, Naas T. The real threat of Klebsiella pneumoniae carbapenemase-producing bacteria[J]. Lancet Infect Dis, 2009, 9(4): 228-236. DOI:10.1016/S1473-3099(09)70054-4 |
| [23] |
王建友, 周阿旺, 陈丹. 碳青霉烯酶在碳青霉烯类耐药肺炎克雷伯杆菌的分布及分子流行病学[J]. 中国微生态学杂志, 2015, 27(5): 517-520, 526. Wang JY, Zhou AW, Chen D. The distribution and molecular epidemiology of carbapenemase in carbapenem-resistant Klebsiella pneumoniae[J]. Chinese Journal of Microecology, 2015, 27(5): 517-520, 526. |
| [24] |
王步补, 阮战伟, 项小婵, 等. 耐亚胺培南肺炎克雷伯菌分子流行病学及耐药传播机制研究[J]. 全科医学临床与教育, 2014, 12(1): 18-22. Wang BB, Ruan ZW, Xiang XC, et al. Molecular epidemiology and drug-resistance transfer mechanism of imipenem resis-tant Klebsiella pneumonia[J]. Clinical Education of General Practice, 2014, 12(1): 18-22. |
| [25] |
Tang Y, Fu P, Zhou Y, et al. Absence of the type I-E CRISPR-Cas system in Klebsiella pneumoniae clonal complex 258 is associated with dissemination of IncF epidemic resistance plasmids in this clonal complex[J]. J Antimicrob Chemother, 2020, 75(4): 890-895. DOI:10.1093/jac/dkz538 |
| [26] |
Liao WJ, Liu Y, Zhang W. Virulence evolution, molecular mechanisms of resistance and prevalence of ST 11 carbapenem-resistant Klebsiella pneumoniae in China: a review over the last 10 years[J]. J Glob Antimicrob Resist, 2020, 23: 174-180. DOI:10.1016/j.jgar.2020.09.004 |
| [27] |
Doi Y. Treatment options for carbapenem-resistant Gram-nega-tive bacterial infections[J]. Clin Infect Dis, 2019, 69(Suppl 7): S565-S575. |



