金黄色葡萄球菌(Staphylococcus aureus, SA)是一种在自然界广泛存在的病原菌,在临床上可以引起多种感染性疾病,如皮肤软组织感染、血流感染和中枢神经系统感染等[1]。近年来,因全球范围内广谱抗菌药物的过度使用,SA的耐药率呈现不断上升趋势,除了耐药基因的表达外,还有一个重要的原因是生物膜的形成。生物膜是细菌在生长过程中黏附于生物或非生物表面后,被细菌自身分泌的胞外物质包裹而形成的细菌聚集膜样物,可以保护细菌免受宿主免疫系统、抗菌药物及物理机械压力的影响[2]。SA生物膜相关感染往往会造成治疗失败以及复发性感染,不仅导致疾病的发病率和病死率上升,还会给全球经济造成巨大的损失[3]。目前研究[4]表明, 抗菌药物在亚抑菌浓度(sub-MIC)下会对SA生物膜的形成产生影响,且不同抗菌药物影响细菌生物膜形成的作用机制不同。因此,本综述将对SA生物膜的形成过程及基因调控、不同抗菌药物在sub-MIC下对SA生物膜形成的影响及作用机制进行概述,以期为临床有效控制及治疗SA生物膜相关感染提供一定依据。
1 sub-MIC的定义及意义最低抑菌浓度(MIC)是抑制细菌生长的最低药物浓度,敏感菌株在抗菌药物浓度高于MIC时被抑制,而耐药菌株则需要更高的抗菌药物浓度才能被抑制,其上限被称为防突变浓度(MPC),两者之间的浓度集合被称为突变选择窗口(MSW)。MSW理论认为,耐药突变菌株的选择性富集只发生在MSW内,当抗菌药物浓度低于MIC时,虽然治疗无效但也不会导致耐药菌的富集[5]。随着研究人员对细菌耐药机制的深入研究,进一步提出了sub-MIC和最小选择浓度(MSC)的概念,补充了MSW理论。研究人员将野生型敏感菌和同源的耐药菌按1 ∶1的比例混合,当抗菌药物浓度较低时,因耐药菌存在适应性代价,故敏感菌具有生长优势;当抗菌药物浓度增加时,耐药菌逐渐克服适应性代价,两者生长速度相同、适应性相同的药物浓度即为MSC,当抗菌药物浓度高于MSC但仍低于MIC时就可以选择性富集耐药突变菌株[6-7]。见图 1。因此,sub-MIC是指低于MIC但仍对细菌具有选择作用的抗菌药物浓度,由于药物在体内浓度分布不同和药物自身不良反应的影响,以及临床长期应用抗菌药物导致菌群MIC值升高,细菌不可避免会处于sub-MIC环境[8]。当菌群处于sub-MIC环境时,不仅可以富集预先存在的耐药突变菌株,还可以通过增加适应性进化频率来产生更高水平的耐药突变,最终导致药物治疗失败[9]。
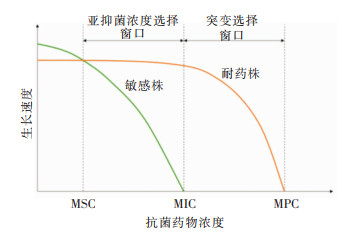 |
| 图 1 细菌在sub-MIC选择窗口和传统突变选择窗口中的生长[9] |
生物膜是细菌附着于表面后被自身分泌的胞外基质(ECM)包裹形成的结构性群落,ECM主要由胞外多糖(EPS)、胞外DNA(eDNA)、蛋白质、脂类和其他生物分子组成[10]。目前关于生物膜的研究主要以体外模型为主,尽管参与生物膜形成的分子成分因细菌种类不同而异,但广泛认可的生物膜生长模型包括三个连续阶段:黏附、聚集/成熟、分离/扩散[11]。随着研究的深入,Moormeier等[12]借助微流控活细胞分析系统和延时显微镜实时观察SA生物膜的发展过程,并提出了五阶段生物膜生长模型,即黏附、聚集、外流、成熟、扩散。见图 2。接下来将对五个阶段及相关基因调控进行概述。
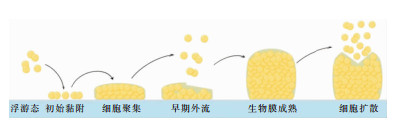 |
| 图 2 SA生物膜生长模型 |
生物膜形成的第一步是浮游的SA黏附到生物或非生物表面。SA与生物表面之间主要通过一组N端含有信号肽序列、C端含有“LPXTG”模体(motif)的表面蛋白即细胞壁锚定蛋白进行黏附,其中一类与ECM成分结合的蛋白分子称为识别黏附基质分子的微生物表面成分(MSCRAMMs)[13]。MSCRAMMs主要包括凝集因子A(ClfA)和凝集因子B(ClfB)、纤维蛋白结合蛋白A(FnBPA)和纤维蛋白结合蛋白B(FnBPB)、丝氨酸-天冬氨酸重复序列蛋白(Sdr)、骨唾液酸结合蛋白(Bbp)、胶原黏附素(Cna)等[14]。这些蛋白由其对应的基因编码,可以促进细菌与生物表面进行特异性黏附。而SA与非生物表面之间则是通过静电作用和疏水作用实现非特异性黏附[15]。除此之外,SA自身分泌的自溶素(atlA)亦有助于细菌在聚苯乙烯表面黏附[16]。
2.2 细胞聚集完成初始黏附后的细菌在存在流体剪切力的情况下很容易脱离表面,因此,SA会产生多种因子帮助细胞聚集从而增加生物膜的稳定性。首先,SA在黏附过程中产生的一些细胞外蛋白可以在最初附着后即发挥促进细胞聚集的作用,如FnBPA、FnBPB、ClfB和SdrC[17]。其次,多糖胞间黏附素(PIA)已被证明可以在SA生物膜形成的第二阶段即细胞聚集中发挥重要作用[18]。PIA的合成主要受ica基因的调控,ica由四个功能基因(icaA、icaD、icaB、icaC)和一个调节基因(icaR)组成,其表达容易受环境条件(如高温、高渗透压、抗菌药物等)的影响,因此,只有特定菌株或在特定条件下SA可以依赖PIA实现细胞聚集[19]。此外,研究[20]表明,SA可以通过烯醇化酶和甘油三醛-3-磷酸脱氢酶(GAPDH)在低pH值环境下使细胞自我裂解释放eDNA和细胞质蛋白,从而促进细胞聚集。
2.3 早期外流过去认为,SA与其他细菌一样有着相似的生物膜形成过程[21],但研究人员通过延时显微镜对SA生物膜形成过程观察后发现,在聚集阶段开始后约6 h,细胞出现了明显的释放,即早期外流阶段[12]。此阶段主要由SA自身分泌的核酸酶(nuc)介导,nuc是一种热稳定的核酸内切酶,也是SA的一种重要毒力因子,可以降解生物膜的主要成分eDNA引起细胞早期外流[22]。SA nuc突变体缺少早期外流阶段,同时也无法观察到生物膜成熟所需的微菌落结构的形成[12]。因此,SA独特的早期外流阶段便于生物膜进行结构重建,进而形成更有利于生物膜发展的结构形态。
2.4 生物膜成熟无论何种细菌,其生物膜成熟的关键都在于微菌落结构的形成[23],SA亦不例外。通过延时显微镜观察SA生物膜形成发现,在生物膜成熟过程中,不断有细菌脱离黏附,残留在基底层的细胞则生成小簇菌落结构,即微菌落[13]。这些微菌落不断解离和生成,使得生物膜高度结构化,以便于细菌进行营养交换与废物排除[24]。在此过程中,SA主要通过一类具有表面活性剂性质的α-螺旋多肽两性分子,即酚溶性调节蛋白(PSMs)实现生物膜成熟。PSMs可以破坏生物膜的非共价作用,使得生物膜结构得到修饰和重塑进而逐渐趋于成熟,而PSMs的表达则受Agr系统的严格调控[25]。
2.5 细胞扩散生物膜积累成熟后,生物膜内的细胞可以通过扩散重新恢复到浮游状态,因此细胞扩散既是生物膜生长和发展过程的最后阶段,也是另一个生长周期的开始[26]。此阶段主要受机械力、表面活性剂PSMs以及降解生物膜基质分子酶(如蛋白酶等)的影响,因此细胞扩散很大程度上亦受Agr系统调控[27]。在Agr系统中,首先由启动子P2、P3分别启动RNA II和RNA III的转录,然后再由RNA II编码AgrA、AgrB、AgrC和AgrD蛋白,其中AgrD编码自诱导肽前体(pre-AIP),在AgrB的作用下成熟为自诱导肽(AIP),当细胞外AIP浓度达阈值时,Agr系统被激活,AgrC结合AIP后使AgrA磷酸化,从而再反作用于P2、P3启动子以及其他几个转录靶点,调控相关细胞外蛋白酶的表达而促进生物膜扩散[28]。同时,AgrA还可以直接调控PSMs的表达,从而有助于生物膜的扩散[29]。
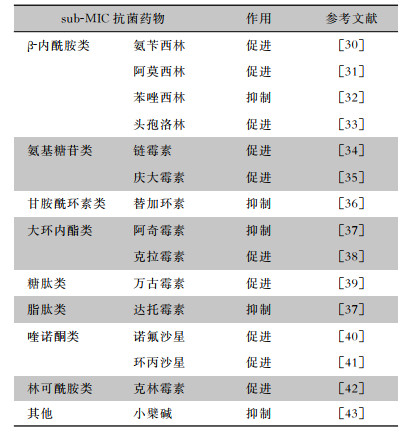
3 sub-MIC抗菌药物影响SA生物膜形成的作用机制sub-MIC抗菌药物既可以促进生物膜形成,也可以抑制生物膜形成。现已充分证实不同抗菌药物在sub-MIC下影响SA生物膜形成[30-43],本文针对常见抗菌药物对SA生物膜的影响进行概述。见表 1。由于生物膜形成过程中涉及众多基因及其复杂的调控机制,至今还无法将常用抗菌药物与SA生物膜形成的作用靶点一一对应。因此,以下部分总结了目前sub-MIC抗菌药物影响SA生物膜形成的作用机制。见图 3。
| 表 1 sub-MIC抗菌药物对SA生物膜的作用 |
|
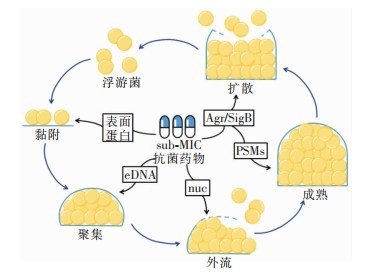 |
| 图 3 sub-MIC抗菌药物影响SA生物膜形成的作用机制 |
初始黏附是生物膜形成的早期关键步骤。SA可以通过细胞表面蛋白与表面相互作用,也可以通过静电作用和疏水作用与表面黏附。已有多项研究观察到SA在sub-MIC抗菌药物作用下,其表面蛋白含量(包括黏附相关蛋白)会发生变化。通过对氨苄西林诱导和非氨苄西林诱导的生物膜进行转录组学分析发现,SA在sub-MIC氨苄西林作用下,一些编码表面蛋白基因以及黏附相关基因的表达会上调,有助于细菌形成生物膜[30]。同时,研究[34, 40]表明,sub-MIC链霉素或诺氟沙星可以通过改变SA表面电荷和疏水性增加细菌与表面的初始黏附,促进生物膜的形成。
3.2 释放eDNA细菌生物膜主要由EPS、eDNA、蛋白质、脂类和其他生物分子组成[10]。在过去的生物膜研究中,EPS被认为是主要和最重要的组成部分,然而随着研究的不断深入,人们发现eDNA在生物膜形成中发挥着重要作用[44]。由于生理、生长速率或细胞周期的不同,大多数细菌在抗菌药物敏感性上会表现出一定的异质性,即一些细胞在抗菌药物浓度低于MIC时就会死亡[45-46]。因此,sub-MIC抗菌药物影响生物膜形成的另一种潜在机制可能是细胞死亡后eDNA的释放。sub-MIC阿莫西林可以诱导SA eDNA的释放而增加生物膜的形成[31]。eDNA除了可以直接释放到细胞外空间促进生物膜形成外,还可以封闭在膜囊泡内参与生物膜的形成[47], 如sub-MIC万古霉素刺激SA生物膜形成的潜在机制是膜囊泡分泌的增加[39]。
3.3 调控Agr系统群体感应系统(QS系统)是细菌细胞间的一种通讯系统,控制着许多基因的表达以响应群体密度[48],而Agr系统是SA QS系统的重要部分[49]。Agr系统在生物膜形成过程中起着至关重要的调控作用,因此,sub-MIC抗菌药物可以通过直接影响Agr系统而影响SA生物膜的形成,也可以通过影响Agr系统调节下游基因的表达而影响生物膜的形成。例如,sub-MIC环丙沙星促进SA生物膜形成的作用机制是sub-MIC环丙沙星结合了AgrC受体,从而影响下游agrA、icaA及icaR基因的表达[41]。sub-MIC莫匹罗星则通过上调cidA的表达刺激SA生物膜的形成,故cidA的表达受到群体感应Agr系统的正向调节[50]。最近研究[51]表明,SA在sub-MIC左氧氟沙星和万古霉素作用下可能引起Agr系统发生自发基因突变,刺激群体欺骗效应,导致生物膜形成的增加。
3.4 改变nuc活性nuc是SA分泌的一种胞外核酸内切酶,主要由nuc1基因编码,其不仅可以降解eDNA引起细胞早期外流从而促进生物膜的形成[22],还可以切断中性粒细胞胞外陷阱(NET)的主干DNA从而实现免疫逃逸,避免NET对生物膜产生杀伤作用[52]。因此,sub-MIC抗菌药物可以通过影响nuc活性从而影响SA生物膜的形成,如多西环素在sub-MIC下可抑制nuc活性,从而抑制SA生物膜的形成[53]。
3.5 触发应激反应为了在不同的环境条件下生长,SA可以利用高效的信号转导系统调整细胞生理机能,从而响应环境刺激,这种信号转导系统的一个关键转录因子是σB(SigB),其主要参与细菌的应激反应[54]。因此,sub-MIC抗菌药物可以通过影响σB而影响应激反应的发生,从而调节SA生物膜的形成。如sub-MIC克林霉素可以替代σB,从而触发细菌的应激反应,并上调生物膜相关基因如atlA、agrA、psm、fnbA和fnbB的表达,使得SA生物膜的形成增加[42]。
4 小结sub-MIC抗菌药物会影响SA的生物膜结构和基因表达,也会对细菌的毒力表达和耐药性产生不同程度的影响。目前关于sub-MIC抗菌药物作用下生物膜的相关研究大部分为体外研究,且样本量较小,因而难免造成抽样误差的增加,使得研究结果的可靠性下降。因此,不同抗菌药物在sub-MIC状态下对SA生物膜的影响还存在争议。同时,正如本综述中所提到的,sub-MIC抗菌药物影响SA生物膜是一个涉及多基因的复杂过程,虽然与之相关的研究很多,但大多数未阐明具体的作用机制。因此,对于sub-MIC抗菌药物对SA生物膜形成的影响及相关机制还需要更多的研究进一步证明。
利益冲突:所有作者均声明不存在利益冲突。
| [1] |
Guo YL, Song GH, Sun ML, et al. Prevalence and therapies of antibiotic-resistance in Staphylococcus aureus[J]. Front Cell Infect Microbiol, 2020, 10: 107. DOI:10.3389/fcimb.2020.00107 |
| [2] |
Flemming HC, Wingender J, Szewzyk U, et al. Biofilms: an emergent form of bacterial life[J]. Nat Rev Microbiol, 2016, 14(9): 563-575. DOI:10.1038/nrmicro.2016.94 |
| [3] |
Idrees M, Sawant S, Karodia N, et al. Staphylococcus aureus biofilm: morphology, genetics, pathogenesis and treatment strategies[J]. Int J Environ Res Public Health, 2021, 18(14): 7602. DOI:10.3390/ijerph18147602 |
| [4] |
李婧, 温海楠, 王晓辉, 等. 亚抑菌浓度抗菌药物对细菌生物膜影响的研究进展[J]. 临床检验杂志, 2021, 39(5): 386-389. Li J, Wen HN, Wang XH, et al. Research progress on the effects of sub-inhibitory concentration of antibiotics on bacte-rial biofilm[J]. Chinese Journal of Clinical Laboratory Science, 2021, 39(5): 386-389. |
| [5] |
Drlica K. The mutant selection window and antimicrobial resistance[J]. J Antimicrob Chemother, 2003, 52(1): 11-17. DOI:10.1093/jac/dkg269 |
| [6] |
Sandegren L. Selection of antibiotic resistance at very low antibiotic concentrations[J]. Ups J Med Sci, 2014, 119(2): 103-107. DOI:10.3109/03009734.2014.904457 |
| [7] |
Liu AN, Fong A, Becket E, et al. Selective advantage of resistant strains at trace levels of antibiotics: a simple and ultrasensitive color test for detection of antibiotics and genotoxic agents[J]. Antimicrob Agents Chemother, 2011, 55(3): 1204-1210. DOI:10.1128/AAC.01182-10 |
| [8] |
王方舟, 倪文涛, 崔俊昌. 抗菌药物亚抑菌浓度及其临床意义[J]. 中国感染与化疗杂志, 2017, 17(5): 597-601. Wang FZ, Ni WT, Cui JC. Sub-inhibitory concentration of antimicrobial agents and its clinical implications[J]. Chinese Journal of Infection and Chemotherapy, 2017, 17(5): 597-601. |
| [9] |
Li J, Xie SY, Ahmed S, et al. Antimicrobial activity and resistance: influencing factors[J]. Front Pharmacol, 2017, 8: 364. DOI:10.3389/fphar.2017.00364 |
| [10] |
Karygianni L, Ren Z, Koo H, et al. Biofilm matrixome: extracellular components in structured microbial communities[J]. Trends Microbiol, 2020, 28(8): 668-681. DOI:10.1016/j.tim.2020.03.016 |
| [11] |
Kostakioti M, Hadjifrangiskou M, Hultgren SJ. Bacterial biofilms: development, dispersal, and therapeutic strategies in the dawn of the postantibiotic era[J]. Cold Spring Harb Perspect Med, 2013, 3(4): a010306. |
| [12] |
Moormeier DE, Bose JL, Horswill AR, et al. Temporal and stochastic control of Staphylococcus aureus biofilm development[J]. mBio, 2014, 5(5): e01341-14. |
| [13] |
Moormeier DE, Bayles KW. Staphylococcus aureus biofilm: a complex developmental organism[J]. Mol Microbiol, 2017, 104(3): 365-376. DOI:10.1111/mmi.13634 |
| [14] |
Foster TJ. The MSCRAMM family of cell-wall-anchored surface proteins of Gram-positive cocci[J]. Trends Microbiol, 2019, 27(11): 927-941. DOI:10.1016/j.tim.2019.06.007 |
| [15] |
Spengler C, Nolle F, Thewes N, et al. Using knock-out mutants to investigate the adhesion of Staphylococcus aureus to abiotic surfaces[J]. Int J Mol Sci, 2021, 22(21): 11952. DOI:10.3390/ijms222111952 |
| [16] |
Leonard AC, Goncheva MI, Gilbert SE, et al. Autolysin-mediated peptidoglycan hydrolysis is required for the surface display of Staphylococcus aureus cell wall-anchored proteins[J]. Proc Natl Acad Sci U S A, 2023, 120(12): e2301414120. DOI:10.1073/pnas.2301414120 |
| [17] |
Speziale P, Pietrocola G, Foster TJ, et al. Protein-based biofilm matrices in Staphylococci[J]. Front Cell Infect Micro-biol, 2014, 4: 171. |
| [18] |
McCarthy H, Rudkin JK, Black NS, et al. Methicillin resis-tance and the biofilm phenotype in Staphylococcus aureus[J]. Front Cell Infect Microbiol, 2015, 5: 1. |
| [19] |
Nguyen HTT, Nguyen TH, Otto M. The staphylococcal exo-polysaccharide PIA-biosynthesis and role in biofilm formation, colonization, and infection[J]. Comput Struct Biotechnol J, 2020, 18: 3324-3334. DOI:10.1016/j.csbj.2020.10.027 |
| [20] |
Foulston L, Elsholz AKW, DeFrancesco AS, et al. The extracellular matrix of Staphylococcus aureus biofilms comprises cytoplasmic proteins that associate with the cell surface in response to decreasing pH[J]. mBio, 2014, 5(5): e01667-14. |
| [21] |
Periasamy S, Joo HS, Duong AC, et al. How Staphylococcus aureus biofilms develop their characteristic structure[J]. Proc Natl Acad Sci U S A, 2012, 109(4): 1281-1286. DOI:10.1073/pnas.1115006109 |
| [22] |
Schilcher K, Horswill AR. Staphylococcal biofilm development: structure, regulation, and treatment strategies[J]. Microbiol Mol Biol Rev, 2020, 84(3): e00026-19. |
| [23] |
Jamal M, Ahmad W, Andleeb S, et al. Bacterial biofilm and associated infections[J]. J Chin Med Assoc, 2018, 81(1): 7-11. DOI:10.1016/j.jcma.2017.07.012 |
| [24] |
Peng Q, Tang XH, Dong WY, et al. A review of biofilm formation of Staphylococcus aureus and its regulation mechanism[J]. Antibiotics (Basel), 2022, 12(1): 12. DOI:10.3390/antibiotics12010012 |
| [25] |
Zaman M, Andreasen M. Cross-talk between individual phenol-soluble modulins in Staphylococcus aureus biofilm enables rapid and efficient amyloid formation[J]. Elife, 2020, 9: e59776. DOI:10.7554/eLife.59776 |
| [26] |
Lister JL, Horswill AR. Staphylococcus aureus biofilms: recent developments in biofilm dispersal[J]. Front Cell Infect Microbiol, 2014, 4: 178. |
| [27] |
Le KY, Dastgheyb S, Ho TV, et al. Molecular determinants of staphylococcal biofilm dispersal and structuring[J]. Front Cell Infect Microbiol, 2014, 4: 167. |
| [28] |
Tan L, Li SR, Jiang B, et al. Therapeutic targeting of the Staphylococcus aureus accessory gene regulator (agr) system[J]. Front Microbiol, 2018, 9: 55. DOI:10.3389/fmicb.2018.00055 |
| [29] |
Le KY, Villaruz AE, Zheng Y, et al. Role of phenol-soluble modulins in Staphylococcus epidermidis biofilm formation and infection of indwelling medical devices[J]. J Mol Biol, 2019, 431(16): 3015-3027. DOI:10.1016/j.jmb.2019.03.030 |
| [30] |
Liu JY, Yang L, Hou YC, et al. Transcriptomics study on Staphylococcus aureus biofilm under low concentration of ampicillin[J]. Front Microbiol, 2018, 9: 2413. DOI:10.3389/fmicb.2018.02413 |
| [31] |
Mlynek KD, Callahan MT, Shimkevitch AV, et al. Effects of low-dose amoxicillin on Staphylococcus aureus USA300 biofilms[J]. Antimicrob Agents Chemother, 2016, 60(5): 2639-2651. DOI:10.1128/AAC.02070-15 |
| [32] |
Jo A, Ahn J. Phenotypic and genotypic characterisation of multiple antibiotic-resistant Staphylococcus aureus exposed to subinhibitory levels of oxacillin and levofloxacin[J]. BMC Microbiol, 2016, 16(1): 170. DOI:10.1186/s12866-016-0791-7 |
| [33] |
Lázaro-Díez M, Remuzgo-Martínez S, Rodríguez-Mirones C, et al. Effects of subinhibitory concentrations of ceftaroline on methicillin-resistant Staphylococcus aureus (MRSA) biofilms[J]. PLoS One, 2016, 11(1): e0147569. DOI:10.1371/journal.pone.0147569 |
| [34] |
Kumar A, Ting YP. Streptomycin favors biofilm formation by altering cell surface properties[J]. Appl Microbiol Biotechnol, 2016, 100(20): 8843-8853. DOI:10.1007/s00253-016-7793-0 |
| [35] |
Mitchell G, Brouillette E, Séguin DL, et al. A role for sigma factor B in the emergence of Staphylococcus aureus small-colony variants and elevated biofilm production resulting from an exposure to aminoglycosides[J]. Microb Pathog, 2010, 48(1): 18-27. DOI:10.1016/j.micpath.2009.10.003 |
| [36] |
袁春妹, 张能华, 傅跃青, 等. 替加环素对耐甲氧西林金黄色葡萄球菌生物膜的清除作用[J]. 浙江临床医学, 2021, 23(12): 1717-1720. Yuan CM, Zhang NH, Fu YQ, et al. The clearing effect of tigecycline on methicillin-resistant Staphylococcus aureus biofilm[J]. Zhejiang Clinical Medical Journal, 2021, 23(12): 1717-1720. |
| [37] |
Zhou Y, Liu MJ, Liao XY, et al. New attempts to inhibit methicillin-resistant Staphylococcus aureus biofilm? a combination of daptomycin and azithromycin[J]. Infect Drug Resist, 2023, 16: 7029-7040. DOI:10.2147/IDR.S433439 |
| [38] |
Ferrer MD, Rodriguez JC, álvarez L, et al. Effect of antibio-tics on biofilm inhibition and induction measured by real-time cell analysis[J]. J Appl Microbiol, 2017, 122(3): 640-650. DOI:10.1111/jam.13368 |
| [39] |
He XL, Yuan FL, Lu F, et al. Vancomycin-induced biofilm formation by methicillin-resistant Staphylococcus aureus is associated with the secretion of membrane vesicles[J]. Microb Pathog, 2017, 110: 225-231. DOI:10.1016/j.micpath.2017.07.004 |
| [40] |
Kumar A, Ting YP. Effect of sub-inhibitory antibacterial stress on bacterial surface properties and biofilm formation[J]. Colloids Surf B Biointerfaces, 2013, 111: 747-754. DOI:10.1016/j.colsurfb.2013.07.011 |
| [41] |
朱娟, 李颖佳. 组氨酸激酶AgrC在亚抑菌浓度环丙沙星促进金黄色葡萄球菌生物膜形成中的作用机制[J]. 中华检验医学杂志, 2022, 45(2): 151-156. Zhu J, Li YJ. Role of histidine kinase AgrC in Staphylococcus aureus biofilm enhancing effects by sub-minimal inhibitory concentration ciprofloxacin[J]. Chinese Journal of Laboratory Medicine, 2022, 45(2): 151-156. |
| [42] |
Schilcher K, Andreoni F, Dengler Haunreiter V, et al. Modulation of Staphylococcus aureus biofilm matrix by subinhibitory concentrations of clindamycin[J]. Antimicrob Agents Chemother, 2016, 60(10): 5957-5967. DOI:10.1128/AAC.00463-16 |
| [43] |
Zhang CF, Li ZL, Pan Q, et al. Berberine at sub-inhibitory concentration inhibits biofilm dispersal in Staphylococcus aureus[J]. Microbiology (Reading), 2022, 168(9): 001243. |
| [44] |
Okshevsky M, Meyer RL. The role of extracellular DNA in the establishment, maintenance and perpetuation of bacterial biofilms[J]. Crit Rev Microbiol, 2015, 41(3): 341-352. DOI:10.3109/1040841X.2013.841639 |
| [45] |
Davis KM, Isberg RR. Defining heterogeneity within bacterial populations via single cell approaches[J]. Bioessays, 2016, 38(8): 782-790. DOI:10.1002/bies.201500121 |
| [46] |
Wen XS, Gehring R, Stallbaumer A, et al. Limitations of MIC as sole metric of pharmacodynamic response across the range of antimicrobial susceptibilities within a single bacterial species[J]. Sci Rep, 2016, 6: 37907. DOI:10.1038/srep37907 |
| [47] |
Campoccia D, Montanaro L, Arciola CR. Tracing the origins of extracellular DNA in bacterial biofilms: story of death and predation to community benefit[J]. Biofouling, 2021, 37(9-10): 1022-1039. DOI:10.1080/08927014.2021.2002987 |
| [48] |
Whiteley M, Diggle SP, Greenberg EP. Progress in and promise of bacterial quorum sensing research[J]. Nature, 2017, 551(7680): 313-320. DOI:10.1038/nature24624 |
| [49] |
Salam AM, Quave CL. Targeting Virulence in Staphylococcus aureus by chemical inhibition of the accessory gene regulator system in vivo[J]. mSphere, 2018, 3(1): e00500-17. |
| [50] |
Jin Y, Guo YJ, Zhan Q, et al. Subinhibitory concentrations of mupirocin stimulate Staphylococcus aureus biofilm formation by upregulating cidA[J]. Antimicrob Agents Chemother, 2020, 64(3): e01912-19. |
| [51] |
He L, Lv HY, Wang YN, et al. Antibiotic treatment can exa-cerbate biofilm-associated infection by promoting quorum cheater development[J]. NPJ Biofilms Microbiomes, 2023, 9(1): 26. DOI:10.1038/s41522-023-00394-4 |
| [52] |
Bhattacharya M, Berends ETM, Zheng XH, et al. Leukocidins and the nuclease nuc prevent neutrophil-mediated killing of Staphylococcus aureus biofilms[J]. Infect Immun, 2020, 88(10): e00372-20. |
| [53] |
Rosman CWK, van der Mei HC, Sjollema J. Influence of sub-inhibitory concentrations of antimicrobials on micrococcal nuclease and biofilm formation in Staphylococcus aureus[J]. Sci Rep, 2021, 11(1): 13241. DOI:10.1038/s41598-021-92619-9 |
| [54] |
Valle J, Echeverz M, Lasa I. σB inhibits poly-N-acetylglucosamine exopolysaccharide synthesis and biofilm formation in Staphylococcus aureus[J]. J Bacteriol, 2019, 201(11): e00098-19. |



