2. 中国辐射防护研究院
2. China Institute for Radiation Protection
氡(Rn)及其子体是存在于环境空气中的天然辐射源,占人类接受天然本底照射总剂量的一半以上[1]。氡及其子体被国际癌症研究机构(IARC)归为Ⅰ类致癌因素,成为位居吸烟之后的第二大致肺癌因素[2]。然而目前尚缺乏用于氡致肺癌早期筛查的分子标志物。circRNA是一类在人体组织及细胞中广泛表达,具有组织、时间及疾病特异性的内源非编码RNA。其异常表达及调控对疾病的发生、发展具有重要作用[3]。circRNA独特的共价闭环结构使其免受外切酶的影响而表达稳定[4],因此有望成为潜在的肿瘤诊断标志物及治疗靶点。
本研究围绕氡致肺癌的关键危害因素α粒子及其作用靶点支气管上皮细胞,利用基因芯片技术研究α粒子分次累积照射诱发BEAS-2B 细胞circRNA的差异表达,筛选α照射致细胞损伤潜在标志分子,为氡致肺癌的发生机制及早期筛查分子标志物的研究提供实验数据。
1 材料与方法 1.1 细胞系、主要试剂及仪器人支气管上皮细胞由中国疾病预防控制中心辐射防护与核安全医学所苟巧老师惠赠。BEGM培养基(瑞士Lonza/Clonetics公司);荧光标记试剂盒、基因表达洗涤试剂盒、基因表达杂交试剂盒及8 × 60K基因芯片(美国 Agilent Technologies公司);TRIzol(RNA抽提试剂)(美国Sigma公司);RNA纯化试剂盒(德国Qiagen公司);NanoDrop2000超微量分光光度计(美国Thermo公司)。
1.2 辐射源利用苏州大学的α粒子照射装置,装置由α粒子源(241Am)、源支架和盖玻片盘等构成,剂量率为0.138 Gy/min[5]。
1.3 细胞培养与照射在含100 U/ml青霉素和100 U/ml链霉素的BEGM培养基中,于37℃、5%CO2条件下培养人支气管上皮细胞BEAS-2B。取对数生长期的BEAS-2B细胞接种于带无菌圆形盖玻片的六孔板中,调整细胞数量为3 × 104个/孔~5 × 104个/孔。次日,将接种有细胞的盖玻片从六孔板中取出,置于α粒子照射装置的样品槽中接受照射。本次研究采用分次照射的方式,每次照射剂量为0.1 Gy。细胞照射后继续传代培养,传代培养10代后再次进行照射,累计照射4次,传代培养40代。以未照射且培养相同代数的细胞为对照组。期间获得的细胞照射模型命名为2B-0.1Gy(n)-m,其中,n代表照射次数,m代表传代次数,如2B-0.1Gy(2)-20细胞模型为细胞经过α粒子累计照射2次后,累计传代20代的细胞模型。最终获得细胞模型包括2B-0.1Gy(1)-10、2B-0.1Gy(2)-20、2B-0.1Gy(3)-30、2B-0.1Gy(4)-40。最终选取2B-0.1Gy(2)-20和2B-0.1Gy(4)-40及其对照组细胞用于circRNA表达的检测分析。
1.4 BEAS-2B细胞总RNA的分离提取及纯化分别取2B-0.1Gy(2)-20、2B-0.1Gy(4)-40细胞及相应的对照组细胞,1 500 r/min(r = 76 mm)离心10 min收集细胞沉淀,利用TRIzol试剂分别提取各实验组细胞总RNA,并利用QIAGEN RNeasy® Kit对RNA进行纯化。
1.5 cDNA与cRNA的合成cDNA与cRNA的合成采用荧光标记试剂盒完成。取0.2 µg RNA于0.2 ml离心管中,加入2 μl阳性内参混合液(Spike Mix)及0.8 μl T7启动子引物(T7 Promoter Primer),65 ℃ 10 min,冰浴5 min以合成cDNA第一链;然后,加入7 μl cDNA第二链反应液(Master Mix)后进行PCR扩增获得双链DNA,PCR反应条件为40 ℃ 2 h;70 ℃ 15 min。最后,在上述体系中加入6 µl反转录反应液,40 ℃孵育2 h,合成荧光标记cRNA并纯化。
1.6 基因芯片杂交基因芯片杂交采用基因表达杂交试剂盒完成。取1.65 µg纯化后的cRNA于55 µl cRNA反应体系中,60 ℃ 30 min进行片段化,加入55 µl杂交反应试剂(GE Hyb)。随后,将含有cRNA样本的混合液加入到芯片中,65 ℃ 10 r/min滚动杂交17 h。
1.7 芯片扫描及差异表达circRNA的筛选将洗涤后的基因芯片于Agilent扫描仪中扫描,分辨率为5 μm,扫描仪自动以100%和10%光电倍增信号强度(PMT)各扫描一次,Agilent软件对两次扫描结果自动合并。采用Feature Extraction10.7.1.1软件(美国Agilent Technologies公司)处理原始图像提取原始数据。利用Genespring12.5软件(美国Agilent Technologies公司)进行quantile标准化和后续处理。标准化后的数据进行过滤,在用于比较的每组样本中至少有1组100%标记为Detected的探针留下进行后续分析。利用倍数变化值进行差异基因筛选,筛选的标准为上调或者下调倍数变化值 ≥ 2.0。在上述获得的差异表达circRNA基础上,对实验组2B-0.1Gy(2)-20及2B-0.1Gy(4)-40差异表达circRNA进行比对分析,筛选共同差异表达circRNA。
1.8 候选差异表达circRNA的功能预测分析circRNA相互作用miRNA预测及功能分析:利用CircInteractome在线分析工具[6]进行候选差异表达circRNA 相互作用miRNA的预测;进一步利用DIANA-miRPath v3.0在线分析工具[7]对候选circRNA相互作用miRNA进行KEGG分析,通过miRNA的通路分析来间接预测候选circRNA的生物学功能。
circRNA母源基因及相互作用蛋白预测与功能分析:利用CircInteractome在线分析工具进行差异表达circRNA母源基因分析及相互作用靶蛋白预测;进一步利用在线数据库Kaplan Meier plotter[8]对circRNA母源基因及circRNA相互作用靶蛋白表达基因进行生存分析,间接评估候选差异表达circRNA的生物学功能;利用Metascape在线分析软件[9]构建circRNA母源基因及circRNA结合蛋白的蛋白质相互作用网络,预测候选circRNA可能的相互作用网络。
2 结果 2.1 基因芯片分析结果通过对累计照射2次,传代20代的细胞模型2B-0.1Gy(2)-20及累计照射4次,传代40代的细胞模型2B-0.1Gy(4)-40进行circRNA芯片检测分析。结果显示,两种细胞模型均呈现circRNA的差异表达。其中,2B-0.1Gy(2)-20细胞模型与对照组相比,筛选获得差异表达circRNA 357个,上调表达175个,下调表达182个;2B-0.1Gy(4)-40细胞模型与对照组相比,筛选获得差异表达circRNA 451个,上调表达145个,下调表达306个。对两个细胞模型差异表达circRNA进行比对分析,获得共同差异表达circRNA 13个,其中表达趋势一致的有6个,分别为hsa_circ_0040439、hsa_circ_0046249、hsa_circ_0006589、hsa_circ_0090531、hsa_circ_0030355、hsa_circ_0079321。本研究将两实验组共同差异表达,且表达趋势一致的6个circRNA作为候选circRNA。表1列出两个实验组共同差异表达circRNA。
|
|
表 1 2B-0.1Gy(2)-20及2B-0.1Gy(4)-40两种细胞模型共同差异表达circRNA |
利用CircInteractome在线分析工具对表1中的6个表达趋势一致的候选circRNA进行相互作用miRNA预测分析,结果显示,与6个circRNA相互作用miRNA达195个,其中,hsa_circ_0040439相互作用miRNA 75个;hsa_circ_0046249相互作用miRNA 42个;hsa_circ_0006589相互作用miRNA 8个;hsa_circ_0090531相互作用miRNA 57个;hsa_circ_0030355相互作用miRNA 47个;hsa_circ_0079321相互作用miRNA 9个。
进一步利用mirPath v.3在线分析工具对候选circRNA相互作用miRNA进行KEGG分析,结果显示,6个候选circRNA相互作用miRNA均参与了一些生物学信号通路。其中,hsa_circ_0040439相互作用miRNA富集KEGG 通路31个,显著性富集居前5的通路为:脂肪酸生物合成、病毒致癌、癌症中的蛋白多糖、Hippo信号通路及赖氨酸降解通路;hsa_circ_0046249相互作用miRNA富集KEGG 通路44个,显著性富集居前5的通路为:脂肪酸生物合成、朊病毒病、细胞周期、病毒致癌及内质网中的蛋白质加工;hsa_circ_0006589相互作用miRNA富集KEGG 通路5个,分别为:脂肪酸生物合成、脂肪酸代谢、甾体生物合成、粘连及甲状腺激素信号通路;hsa_circ_0090531相互作用miRNA富集KEGG 通路52个,显著性富集居前5的通路为:癌症中的蛋白多糖、朊病毒病、脂肪酸生物合成、病毒致癌及Hippo信号通路;hsa_circ_0030355相互作用miRNA富集KEGG 通路44个,显著性富集居前5的通路为:朊病毒病、脂肪酸生物合成、粘连、Hippo信号通路及肾细胞癌;hsa_circ_0079321相互作用miRNA富集KEGG 通路8个,显著性富集居前5的通路为:癌症中的microRNA、脑胶质瘤、缬氨酸、亮氨酸和异亮氨酸生物合成、背腹轴形成及癌症的中枢碳代谢。
对6个circRNA涉及的195个相互作用miRNA进行KEGG通路的聚类分析,根据mirPath v.3所含数据库中信息,其中55个miRNA参与KEGG通路,构建KEGG通路热图1。结合上述各circRNA参与KEGG通路,由热图可知候选circRNA相互作用miRNA主要参与脂肪酸生物合成、赖氨酸降解、脂肪酸代谢、ECM-受体相互作用、Hippo信号通路、癌症中的蛋白多糖、粘连及朊病毒病KEGG通路。
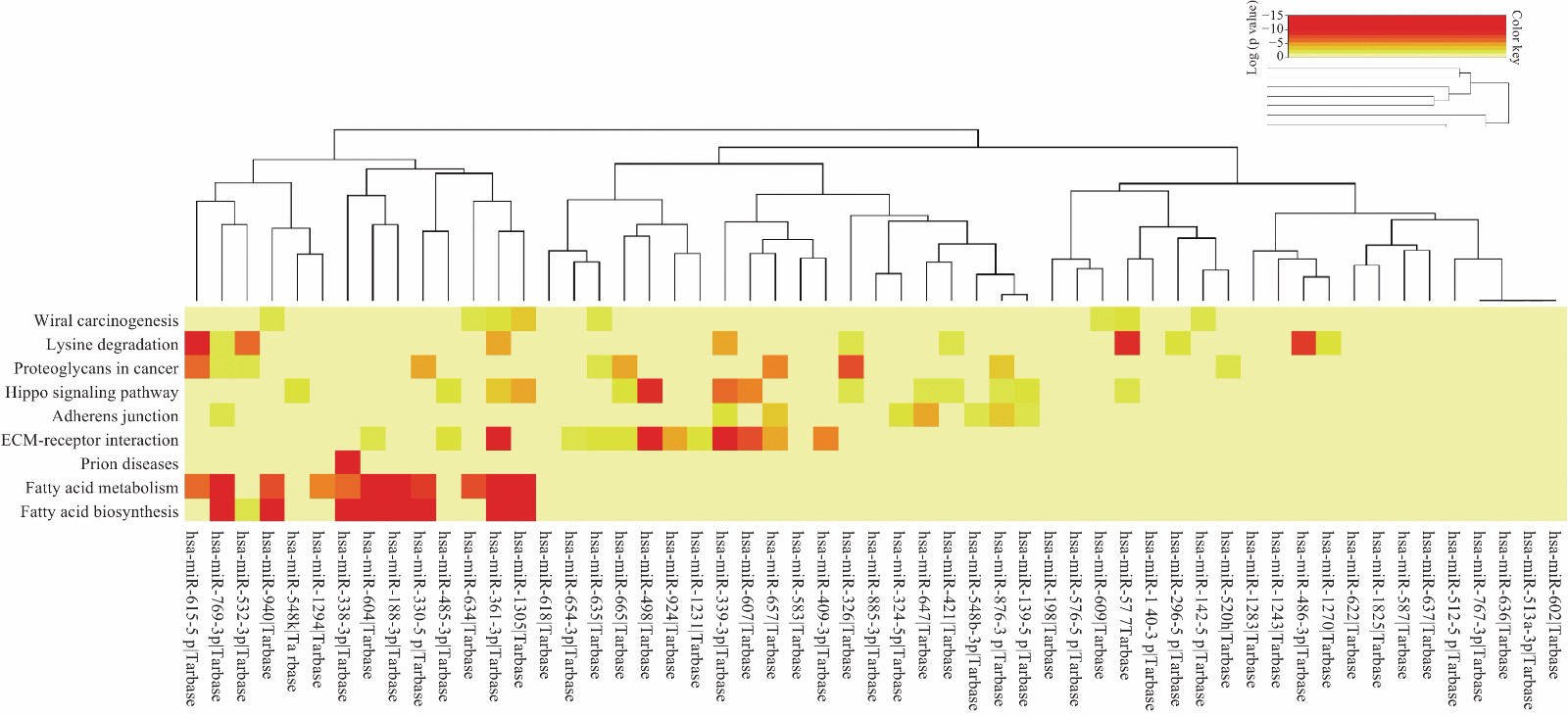
|
图 1 候选circRNA相互作用miRNA KEGG通路聚类图 |
利用CircInteractome在线分析工具对表1中的13个共同差异表达circRNA进行母源基因及circRNA结合蛋白的预测分析,结果如表3。利用在线数据库Kaplan Meier plotter对肺癌中6个候选circRNA母源基因及circRNA结合蛋白表达基因进行生存分析,结果显示,6个母源基因中有2个与患者总生存期(OS)显著相关,分别为P4HB(HR = 1.23,95%CI:1.09~1.44,P< 0.05)、SLC35D2(HR = 0.8,95%CI:0.7~0.9,P < 0.05);6个circRNA结合蛋白中有5个与患者总生存期显著相关,分别为EIF4A3( HR = 1.53,95%CI:1.35~1.74,P < 0.05)、AGO2( HR = 1.5,95%CI:1.27~1.77,P < 0.05)、FMRP( HR = 0.83,95%CI:0.73~0.94,P < 0.05)、FUS( HR = 0.73,95%CI:0.64~0.82,P < 0.05)、HuR( HR = 1.3,95%CI:1.15~1.48,P < 0.05)。上述与总生存期(OS)显著相关的母源基因及circRNA结合蛋白覆盖了筛选出的6个表达趋势一致的circRNA,揭示6个候选circRNA可能具有潜在的致癌性。部分基因生存曲线见 图2、3。进一步通过Metascape在线分析软件对候选circRNA母源基因及circRNA结合蛋白进行蛋白质相互作用分析,构建相互作用网络图,结果如图4。14个基因相关蛋白通过共同参与的生物学过程形成2个蛋白相互作用模块子结构,其中,AGO1、AGO2、AGO3、IGF2BP1、IGF2BP2、IGF2BP3、EIF4A3、HNRNPC、U2AF2、PTBP1、FUS 11个蛋白通过mRNA分解代谢过程、RNA分解代谢过程及翻译的负调节形成MCODE1模块;TAF15、DGCR8、EWSR1 3个蛋白通过Large Drosha complex形成MCODE2模块,如图4B。14个基因相关蛋白通过两个功能模块与其他相关蛋白相互作用形成相互交织更大的相互作用网络图,如图4A。上述相互作用揭示,差异表达circRNA通过与特异或共同RNA结合蛋白相互作用,进而参与相同的生物学过程。
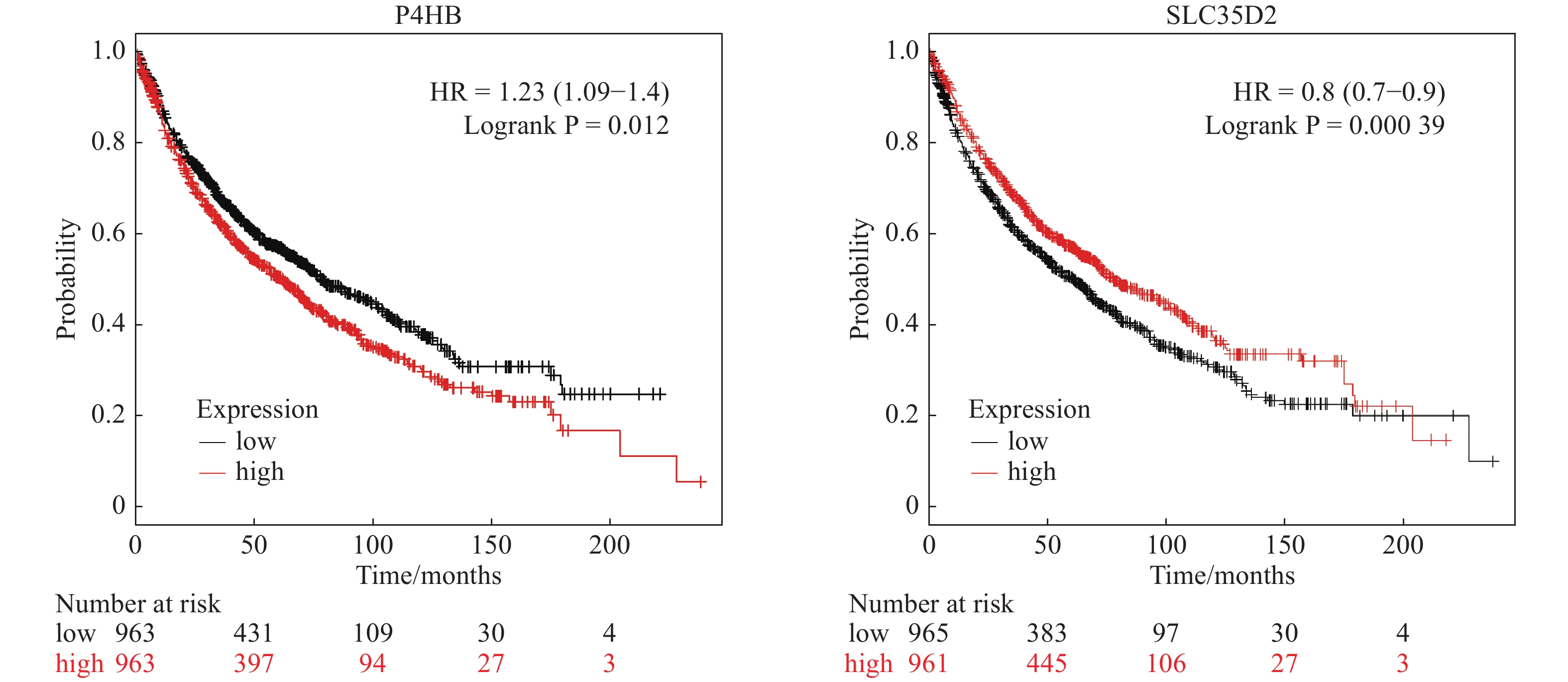
|
图 2 候选circRNA母源基因生存分析 |
|
|
表 2 候选circRNA的母源基因及circRNA结合靶蛋白预测分析结果 |
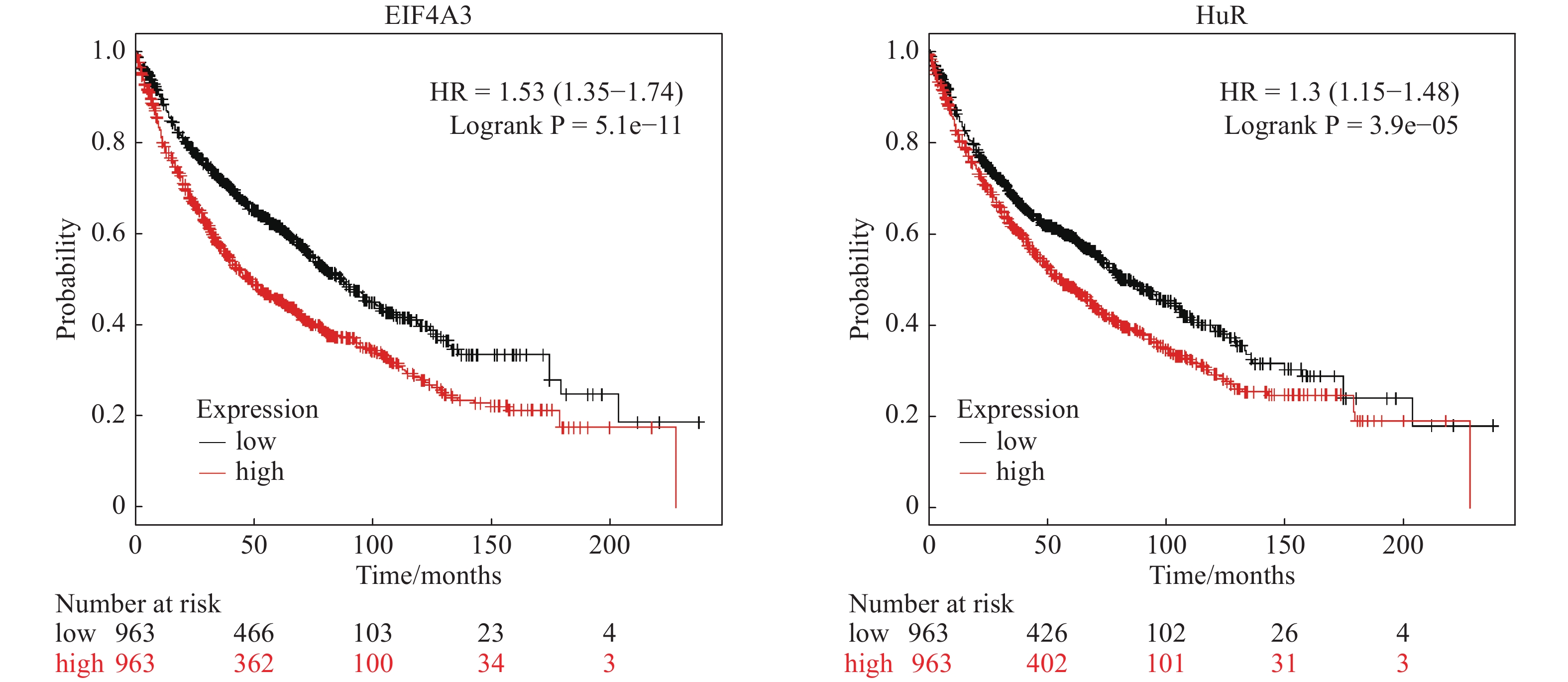
|
图 3 候选circRNA部分结合蛋白表达基因生存分析 |
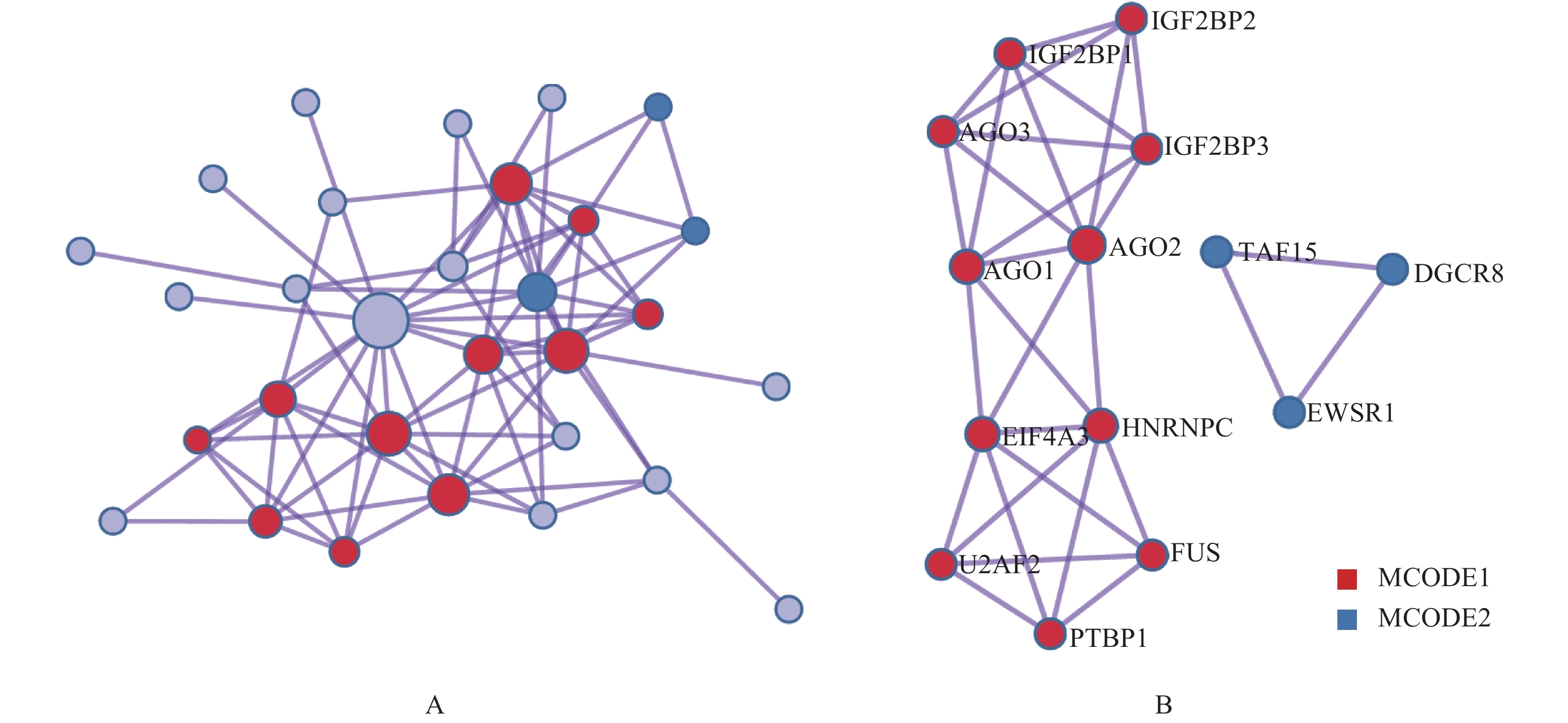
|
图 4 候选circRNA母源基因及circRNA结合蛋白相互作用图 |
基于1988年以来氡暴露矿工队列研究,国际癌症研究机构(IARC)将氡列为肺癌致癌因素。氡衰变后可产生一系列的氡子体,氡及其子体对健康的主要危害在于其发射的高能量α 粒子,吸入体内的氡及子体衰变产生的α 粒子作用于支气管上皮细胞,进而诱发细胞损伤及肺癌的发生[10]。在2013年我国新调整发布的《职业病分类和目录》[11]中,高氡暴露所致肺癌是唯一被明确指出的一种放射性肿瘤类型。铀矿工是主要的高氡暴露人群,铀矿工的个人剂量在核燃料循环领域居于首位,定期的职业健康体检是健康监护的主要手段,以达到疾病的早发现、早治疗。然而目前缺少氡致肺癌早期筛查分子标志物。
circRNA于1976年首次被发现后[12],由于其普遍性、保守性、组织细胞特异性和稳定性等自身特征,使其在临床样本的检测中可能优于线性RNA而具有更高的诊断价值[3],同时成为肿瘤领域的研究热点。本研究围绕氡暴露危害的主要危害因素及关键作用靶点,利用α粒子源照射人支气管上皮细胞,构建细胞损伤模型,通过基因芯片筛选差异表达circRNA,希望寻找α粒子致细胞损伤早期分子靶标。为了模拟氡对人体的长期危害,本研究利用α粒子,通过分次照射的方法构建细胞模型,以期望利用不同照射次数和传代次数的细胞模型来反映α辐射引起细胞损伤阶段及损伤程度的差异。具体而言,细胞经1次照射后,传代培养10代再进行下一次照射,累计照射4次,共传代培养40代,每次照射剂量为0.1Gy。最终获得不同照射剂量和不同培养代数的细胞模型。对累计照射2次,传代20代的细胞模型2B-0.1Gy(2)-20及累计照射4次,传代40代的细胞模型2B-0.1Gy(4)-40进行circRNA芯片检测分析,通过比较两个模型差异表达circRNA,获得两个模型均差异表达且趋势一致的circRNA 6个。
竞争性内源RNA(ceRNA)假说认为,mRNA、假基因、长链非编码RNA(LncRNA)及环状RNA(circRNA)通过竞争性结合miRNA,进而调控基因的表达。circRNA可作为miRNA“海绵”,与miRNA反应元件竞争性结合相应的miRNA,进而调控靶基因的表达[13]。因此,我们假设,在α粒子照射致支气管上皮细胞损伤过程中,circRNA作为miRNA的“海绵”参与细胞损伤的生物学调节过程。CircInteractome在线分析显示,6个表达趋势一致的ciRNA作用的靶miRNA达195个。这些miRNA涉及KEGG通路分析揭示,hsa_circ_0040439等6个候选circRNA可能作为miRNA“海绵”,通过脂肪酸生物合成、脂肪酸代谢、癌症中的蛋白多糖、病毒致癌等通路参与α粒子照射致支气管上皮细胞损伤的生物学过程。
circRNA可与RNA结合蛋白(RBP)结合,如AGO蛋白、RNA聚合酶II等,充当“RBP海绵”,调节RBP功能,影响基因的表达[14]。本研究预测显示,候选circRNA能与EIF4A3、AGO1、AGO2、AGO3、FUS、HuR、IGF2BP1、IGF2BP2、IGF2BP3等蛋白结合,推测其在RNA或RBP复合物的组装中发挥重要作用。此外,候选circRNA相互作用蛋白的生存分析及文献检索显示,部分蛋白的差异表达与肺癌患者总生存期显著相关,且与癌症的发生发展密切相关,如HuR[15]、AGO1[16]、AGO2[15-16]、IGF2BP3[17]、DGCR8[18]。因此,我们推测,差异表达circRNA通过与RBP的结合,将这些蛋白因子分类并传递到特定的位置,进而改变剪接模式或mRNA稳定性,参与α粒子照射致支气管上皮细胞损伤中基因表达的调控,甚至恶性转化的进程。
总之,本研究通过基因芯片技术证实α粒子照射可诱导人支气管上皮细胞circRNA的改变,初步筛选得到hsa_circ_0040439、hsa_circ_0039264、hsa_circ_0046249等差异表达circRNA,这些circRNA可能在α粒子辐射损伤中发挥作用,但其具体作用机制尚需进一步研究证实。
| [1] |
王茂枝, 潘自强, 刘森林, 等. 北京新镇地区室外氡子体水平调查[J]. 中国辐射卫生, 2018, 27(5): 430-431. DOI:10.13491/j.issn.1004-714x.2018.05.002 |
| [2] |
王欣, 陈英民, 王垒, 等. 山东省矿井下工作人员职业性照射初步评价[J]. 中国辐射卫生, 2018, 27(3): 273-275. DOI:10.13491/j.issn.1004-714x.2018.03.022 |
| [3] |
冯笑, 金玉, 龚莉莎, 等. 环状RNA: 潜在的肿瘤诊断生物标记物[J]. 国际检验医学杂志, 2018, 39(19): 2423-2428. DOI:10.3969/j.issn.1673-4130.2018.19.023 |
| [4] |
Jeck W R, Sorrentino J A, Wang K, et al. Circular RNAs are abundant, conserved, and associated with ALU repeats[J]. RNA, 2013, 19(2): 141-157. DOI:10.1261/rna.035667.112 |
| [5] |
嵇卫星, 田文倩, 尹晓明, 等. 一种用于细胞实验的新型α粒子照射装置的建造和验证[J]. 辐射研究与辐射工艺学报, 2015, 33(2): 29-35. |
| [6] |
Dudekula D B, Panda A C, Grammatikakis I, et al. CircInteractome: a web tool for exploring circular RNAs and their interacting proteins and microRNAs[J]. RNA Biol, 2016, 13(1): 34-42. DOI:10.1080/15476286.2015.1128065 |
| [7] |
Vlachos I S, Zagganas K, Paraskevopoulou M D, et al. DIANA-miRPath v3.0: deciphering microRNA function with experimental support[J]. Nucleic Acids Res, 2015, 43(W1): W460-W466. DOI:10.1093/nar/gkv403 |
| [8] |
Nagy Á, Lánczky A, Menyhárt O, et al. Validation of miRNA prognostic power in hepatocellular carcinoma using expression data of independent datasets[J]. Sci Rep, 2018, 8(1): 9227. DOI:10.1038/s41598-018-27521-y |
| [9] |
Zhou Y Y, Zhou B, Pache L, et al. Metascape provides a biologist-oriented resource for the analysis of systems-level datasets[J]. Nat Commun, 2019, 10(1): 1523. DOI:10.1038/s41467-019-09234-6 |
| [10] |
王伟鹏, 孙静, 陈玉之, 等. 线粒体DNA减少在氡及其子体致人支气管上皮细胞凋亡中的作用[J]. 辐射研究与辐射工艺学报, 2014, 32(1): 29-33. |
| [11] |
国卫疾控.《职业病分类和目录》〔2013〕48号[S].2013.
|
| [12] |
Sanger H L, Klotz G, Riesner D, et al. Viroids are single-stranded covalently closed circular RNA molecules existing as highly base-paired rod-like structures[J]. Proc Natl Acad Sci USA, 1976, 73(11): 3852-3856. DOI:10.1073/pnas.73.11.3852 |
| [13] |
徐新宇, 张鑫, 孙轶华. 竞争性内源RNA调控网络在心血管疾病中的研究进展[J]. 临床检验杂志, 2019, 37(6): 430-433. |
| [14] |
Sun H D, Xu Z P, Sun Z Q, et al. Down-regulation of circPVRL3 promotes the proliferation and migration of gastric cancer cells[J]. Sci Rep, 2018, 8(1): 10111. DOI:10.1038/s41598-018-27837-9 |
| [15] |
Chen Y J, Yang F, Fang E H, et al. Circular RNA circAGO2 drives cancer progression through facilitating HuR-repressed functions of AGO2-miRNA complexes[J]. Cell Death Differ, 2019, 26(7): 1346-1364. DOI:10.1038/s41418-018-0220-6 |
| [16] |
Wang M, Zhang L, Liu Z Y, et al. AGO1 may influence the prognosis of hepatocellular carcinoma through TGF-β pathway[J]. Cell Death Dis, 2018, 9(3): 324. DOI:10.1038/s41419-018-0338-y |
| [17] |
Lochhead P, Imamura Y, Morikawa T, et al. Insulin-like growth factor 2 messenger RNA binding protein 3(IGF2BP3) is a marker of unfavourable prognosis in colorectal cancer[J]. Eur J Cancer, 2012, 48(18): 3405-3413. DOI:10.1016/j.ejca.2012.06.021 |
| [18] |
Guo Y Q, Tian P, Yang C H, et al. Silencing the double-stranded RNA binding protein DGCR8 inhibits ovarian cancer cell proliferation, migration, and invasion[J]. Pharm Res, 2015, 32(3): 769-778. DOI:10.1007/s11095-013-1219-9 |




