2. 青海师范大学, 青海省青藏高原药用动植物资源重点实验室, 青海 西宁 810008;
3. 青海师范大学高原科学与可持续发展研究院, 青海 西宁 810016
2. Key Laboratory of Medicinal Animal and Plant Resources of the Qinghai-Tibet Plateau in Qinghai Province, Qinghai Normal University, Xining 810008, China;
3. Academy of Plateau Science and Sustainability, Qinghai Normal University, Xining 810016, China
叶绿体是植物一种重要的半自主性细胞器, 拥有独立的基因组[1]。研究表明, 高等植物叶绿体基因组长度一般介于120~180 kb, 通常为典型的环状四分体结构, 包括1个大单拷贝区(large single copy region, LSC)、1个小单拷贝区(small single copy region, SSC) 和2个反向重复区(inverted repeats region a/b, IRa/IRb)[2, 3]。叶绿体基因组编码110~130个基因, 具有合成蛋白质、淀粉、色素等多种功能[4, 5]。被子植物中, 叶绿体基因组(cpDNA) 多为母系遗传, 仅少数为双亲或父系遗传, 因具有高度保守、结构稳定、进化速率慢、分子质量小等优点, 被广泛应用于物种界定、系统进化、遗传多样性、群体遗传结构及物种形成等领域, 取得理想的研究成果[6, 7]。譬如, Jansen等[8]基于葡萄(Vitis vinifera) 和其他26种被子植物叶绿体基因组序列构建系统发育树, 从全新的角度阐明了葡萄在被子植物中的系统发育关系; 梁凤萍等[9]基于27种菊科植物叶绿体基因组序列进行系统性研究, 为菊科植物的分类界定和系统进化提供了更准确的证据。
葵花大蓟(Cirsium souliei) 是菊科(Compositae)、管状花亚科(Carduoideae)、蓟属(Cirsium) 的一种多年生药用草本植物, 别名“聚头蓟”, 主要分布于青海、甘肃、四川和西藏等地, 生于海拔1 930~4 800 m的山坡、路旁、林缘、荒地、河滩地、田间、水旁潮湿地[10, 11]。葵花大蓟全草入药, 具有凉血止血、散瘀消肿等功效, 主治吐血、鼻出血、尿血、子宫出血、崩漏、黄疸、疮痈等症状[10, 12]。目前, 国内外学者对葵花大蓟的耐逆特性和化学成分进行了研究, 发现葵花大蓟具有较好的温度耐受性, 并从中分离鉴定出多种化合物[13-16]。然而, 有关葵花大蓟叶绿体基因组结构特征、系统位置及亲缘关系的研究至今未见报道。据此, 本研究采用高通量测序技术对葵花大蓟的叶绿体基因组进行测序和数据分析, 阐明叶绿体基因组的结构特征; 在此基础上, 通过对葵花大蓟及其45种近缘种系统发育树的构建, 明晰其系统位置及亲缘关系, 以期为葵花大蓟遗传多样性、群体遗传结构、系统进化及物种形成机制提供理论依据。
材料与方法实验材料 新鲜无病虫害的葵花大蓟叶片采自青海省海北藏族自治区祁连县默勒镇(N37.72898°, E100.6297°, alt. 3 471.12 m), 采集后迅速放于变色硅胶中干燥保存。凭证标本存放于中国科学院西北高原生物研究所青藏高原生物标本馆(HNWP)。
基因组DNA提取和测序 采用改良的CTAB法提取基因组总DNA[17], 然后利用1.0%琼脂糖凝胶电泳检测DNA的完整性, Nanodrop检测DNA的纯度(OD 260/280比值) 以及Qubit 2.0荧光定量仪对样品DNA浓度定量; DNA样品检测合格后, 使用Covaris超声破碎仪随机破碎合格的DNA样品, 接着对测序片段进行末端修复、3′端加A尾、加测序接头和纯化; 通过PCR扩增构建DNA文库, 质检合格后利用Illumina NovaSeq6000高通量测序平台(北京诺禾致源科技有限公司) 进行测序。
叶绿体基因组组装和注释 原始序列(raw reads) 经Fastap软件[18]过滤、去除低质量序列, 获得待分析的高质量序列(clean reads)。以蓟(Cirsium japonicum; Genbank登录号: NC_053767.1) 叶绿体基因组序列为参考, 运用NOVOPlasty软件[19]对葵花大蓟叶绿体基因组序列进行组装, 设置为默认参数; 然后, 以葵花大蓟近缘种林蓟(Cirsium schantarense; Genbank登录号: MN871982) 和丝路蓟(Cirsium arvense; Genbank登录号: NC_036965) 为参考物种, 利用在线工具GeSeq (https://chlorobox.mpimp-golm.mpg.de/geseq.html)[20]和Plastid Genome Annotator (PGA) 软件[21]分别对葵花大蓟的叶绿体基因组进行注释, 并将两种注释结果对比矫正, 去除错误及冗余注释; 接着, 采用在线工具Chloroplot (https://irscope.shinyapps.io/Chloroplot/)[22]绘制葵花大蓟叶绿体基因组图谱; 最后, 将注释好的葵花大蓟叶绿体基因组序列提交至NCBI数据库(序列登录号ON720162)。
密码子偏好性和SSR位点分析 利用Codon W1.4.2软件[23]检测葵花大蓟叶绿体基因组中大于200 bp蛋白编码基因的密码子使用频率和相对密码子使用频率(RSCU); 使用在线程序MISA (https://webblast.ipk-gatersleben.de/misa/index.php?action=1)[24]对葵花大蓟叶绿体基因组序列进行SSR位点分析, 其中单核苷酸、二核苷酸、三核苷酸、四核苷酸、五核苷酸和六核苷酸的重复数分别设置为8、5、3、3、3和3。
叶绿体基因组比较分析 运用在线软件IRscope (https://irscope.shinyapps.io/irapp/)[25]对葵花大蓟及蓟属8种植物的叶绿体基因组进行IR区边界比较分析; 以丝毛飞廉(Carduus crispus; Genbank登录号: NC_053726.1) 为参考基因组, 利用在线软件mVista (http://genome.lbl.gov/vista/mvista/submit.html)[26]中的Shuffle-LAGAN模型对上述9种植物的叶绿体基因组作比较分析。
系统发育分析 采用MAFFT v7软件[27], 对从GenBank数据库中下载的菊科管状花亚科45种植物及葵花大蓟的叶绿体基因组序列进行比对, 以菊科舌状花亚科(Cichorioideae) 中苣买菜(Sonchus arvensi) 为外类群, 运用IQ-TREE[28]软件构建46种管状花亚科植物的最大似然(maximum likelihood, ML) 树, bootstrap值设置为5 000, 最佳模型设置为TVM+F+R2。
结果与分析 1 叶绿体基因组结构、分类、功能及特征葵花大蓟叶绿体基因组全长152 470 bp, 由一个长度为83 485 bp的LSC、一个长度为18 623 bp的SSC和一对长度为25 181 bp的反向重复区(inverted repeat regions, IRs) 构成的环状四分体结构(图 1)。葵花大蓟叶绿体基因组总GC含量为37.7%, 其中SSC区、LSC区和IRs区的GC含量分别为35.8%、31.3%和43.1%。葵花大蓟叶绿体基因组共编码134个基因, 包括蛋白编码基因89个、tRNA基因37个、rRNA基因8个, 其中17个基因存在两个拷贝(表 1)。此外, 葵花大蓟叶绿体基因组中共发现13个基因含有内含子, 其中7个基因含有1个内含子, 6个基因含有2个内含子(表 1)。
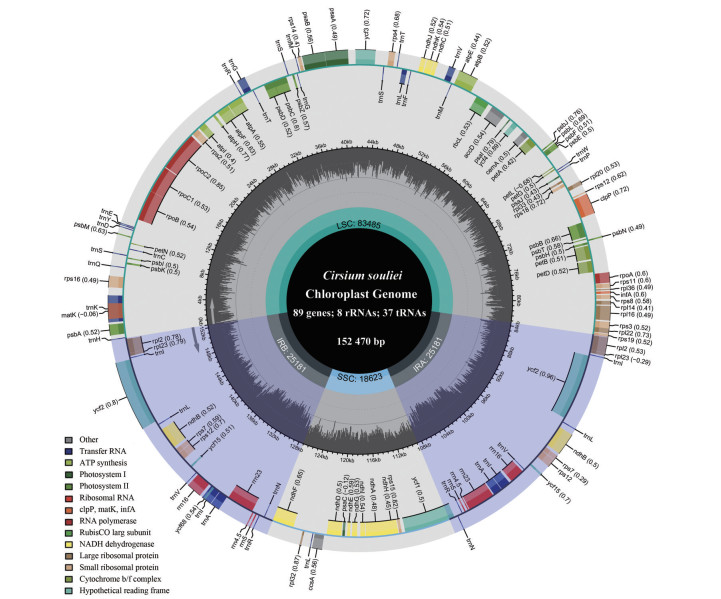
|
Figure 1 Circularized map of the chloroplast genome of C. souliei |
| Table 1 Gene annotation of the chloroplast genome of C. souliei. a, bPresent an intron and two introns in protein-coding genes, respectively; cPresents duplicated genes |
本研究从葵花大蓟叶绿体基因组中, 获得71条大于200 bp的蛋白编码序列(coding DNA sequence, CDS), 用于密码子偏好性分析。研究结果表明, 葵花大蓟叶绿体基因组71条CDS序列共编码25 473个密码子。其中, 编码亮氨酸(Leu) 的密码子占比最多, 为10.51%; 编码半胱氨酸(Cys) 的密码子占比最少, 为1.10% (表 2)。同时, 还检测到30个相对同义密码子使用度(RSCU) 大于1的密码子, 除UUG外其余密码子都以A/U结尾, 且编码精氨酸(Arg) 的密码子AGA的RSCU值最高, 为1.88 (表 2和图 2)。
| Table 2 Relative synonymous codon usage of each amino acid in C. souliei. *Represents termination codon |
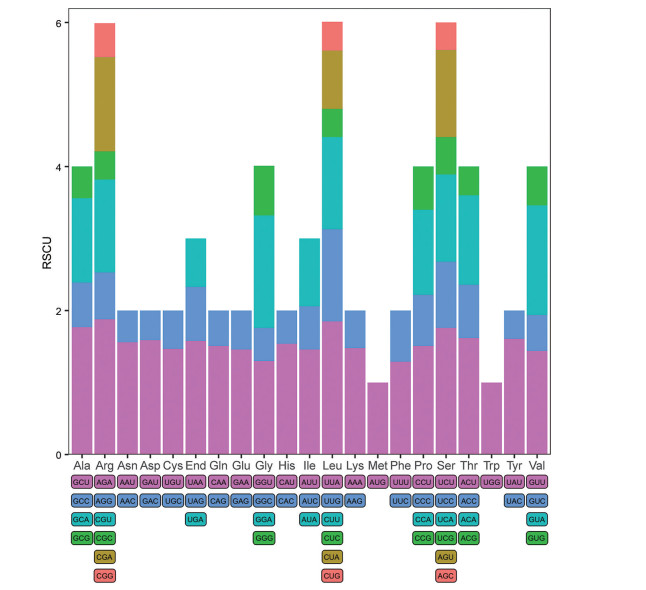
|
Figure 2 RSCU analysis of each amino acid in C. souliei |
葵花大蓟叶绿体基因组共检测到188个SSR位点, 其中单核苷酸重复有108个, 包括A、C和T 3种重复类型; 二核苷酸重复有4个, 仅有AT和TA两种类型; 三核苷酸重复有67个, 包括AAC、AAG、AAT、ACC、ACG、AGA、AGT、ATA等29种类型; 四核苷酸重复有9个, 包括AAAT、AATC、ATAA、GATT、GTTT、TATT和TTTC 7种类型(图 3A)。此外, 葵花大蓟叶绿体基因组中大部分SSR位点位于蛋白编码序列(CDS), 占比约为47%, 其次在基因间隔区(intergenic spacer, IGS) 的SSR位点约为45%, 而位于内含子(intron) 中的SSR位点占比最小, 仅为8% (图 3B)。
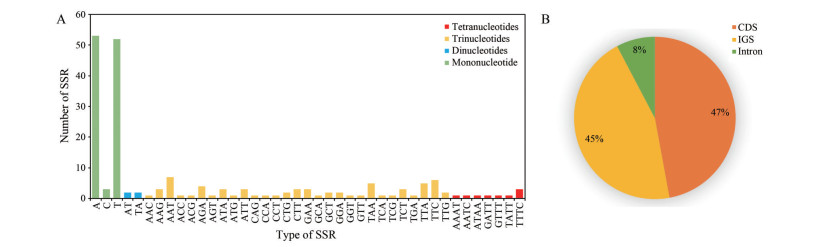
|
Figure 3 SSR types and distributions in the chloroplast genome of C. souliei. A: Types and numbers of SSR in the chloroplast genome of C. souliei; B: Distribution of SSRs in the coding DNA sequence (CDS), intergenic spacer (IGS) and intron regions |
叶绿体基因组IR区扩张和收缩分析表明, 蓟属所有参试物种在LSC/IR和SSC/IR边界区具有相似的基因种类、排列和长度, 但在这些边界区也存在明显的扩张和收缩(图 4)。就LSC/IRb边界区而言, 葵花大蓟、丝路蓟、绵毛蓟(C. eriophorum)、刺儿菜(C. setosum)、蓟和C. nipponicum中rps19基因均向IRb区扩张了60 bp; IRb/SSC边界区, 除葵花大蓟和蓟的ycf1基因缺失外, 其余蓟属物种的ycf1基因均向IRb区扩张了6~10 bp; 同样, SSC/IRa边界区, 蓟属所有参试物种的ycf1基因均向IRa区扩张了566~571 bp (图 4)。此外, 叶绿体基因组全序列比对分析显示, 蓟属所有参试物种的叶绿体基因组高度保守, 未检测到大量的基因缺失和变异(图 5), 但也有微量的基因变异存在, 如在petN-psbM、trnR-UCU-trnfM-CAU、petD-rpoA、ndhB-ycf15、trnL-UAG-ndhF、ycf1等区域; 并且, 参试物种叶绿体基因组中LSC区和SSC区的遗传变异显著高于IR区(图 5)。
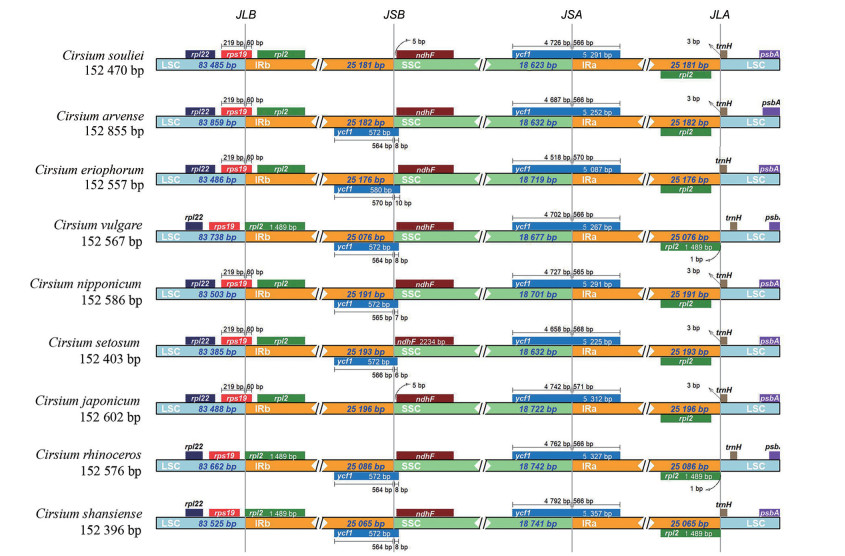
|
Figure 4 Boundary analysis of IR regions in chloroplast genomes from nine Cirsium species |
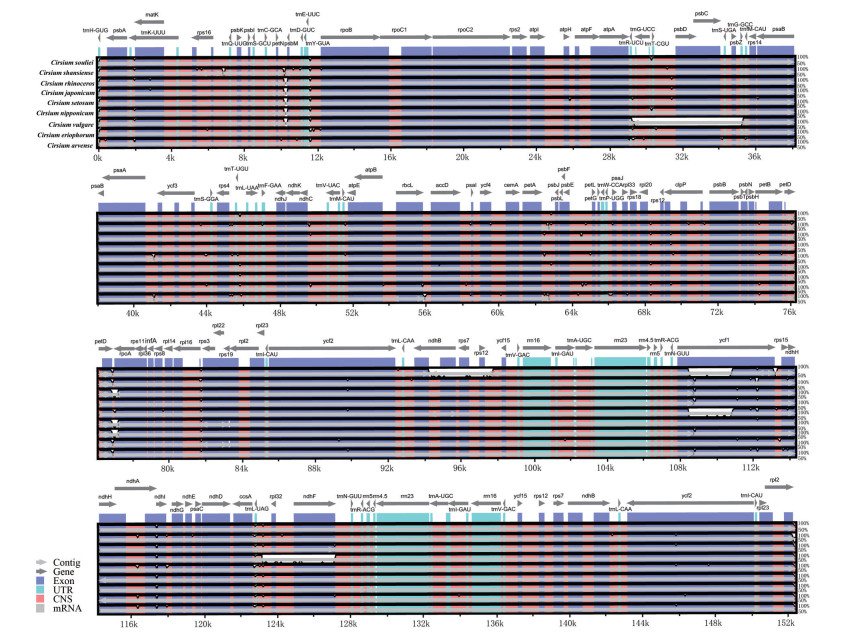
|
Figure 5 Full sequence alignment of chloroplast genomes of nine Cirsium species. The top arrows show transcription direction; purple color indicates the exons of protein-coding genes (exon); cyan color indicates tRNAs and rRNAs; pink-orange color shows conserved non-coding sequence (CNS); The X-axis represents the positions in the cp genome while Y-axis represents percentage identity within 50%-100% |
以苣买菜为外类群, 基于管状花亚科46种植物叶绿体基因组构建的最大似然(ML) 树结果显示, 管状花亚科46种植物聚为Clade 1和Clade 2两大分支, 其中Clade 1由飞廉亚族(Carduinae) 和矢车菊亚族(Centaureinae) 的物种构成, Clade 2由刺苞亚族(Carlininae) 和旋覆花亚族(Inulinae) 的物种组成(图 6)。在Clade 1中, 葵花大蓟与翼蓟为姊妹类群, 构成一个单系分支, 支持率(bootstrap) 高达100%, 表明两者亲缘关系较近; 并且, 其与飞廉属(Carduus) 丝毛飞廉和C. tenuiflorus构成的单系分支互为姊妹类群, 支持率为100% (图 6)。此外, 牛口刺(C. shansiense) 和丝路蓟构成的单系分支与刺儿菜互为姊妹类群, 支持率为100%; 蓟和C. rhinoceros为姊妹种, 构成一个单系分支, 支持率也为100%; 然而, 蓟属的绵毛蓟与C. nipponicum构成的单系分支与Saussurea tanguensis也为姊妹类群, 但这些物种与蓟属其他物种亲缘关系较远(图 6)。由此看来蓟属参试物种并非是一个单系类群。
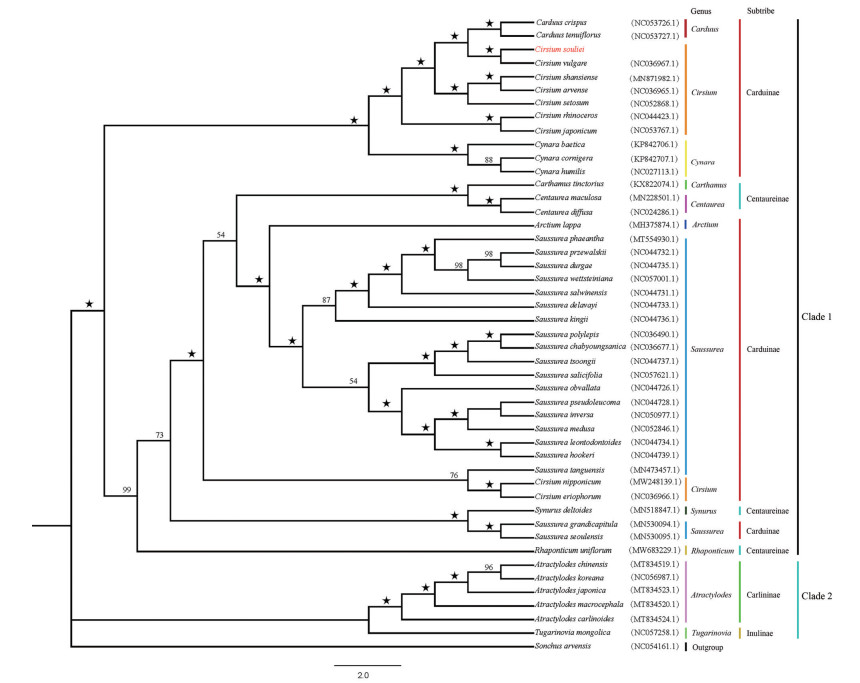
|
Figure 6 Phylogenetic tree of 46 species in Carduoideae based on the chloroplast genomes using maximum likelihood (ML). "★" Represent fully supported nodes |
研究表明, 陆生植物叶绿体基因组大小常介于120~180 kb之间, 基因结构、组成和排列顺序较为保守[3]。葵花大蓟叶绿体基因组长度为152 470 bp、GC含量为37.7%、共编码134个基因, 这与先前报道的蓟属其他植物的叶绿体基因组结构特征基本类似[29-31], 因此葵花大蓟的叶绿体基因组结构相对保守, 这进一步验证了先前研究结论的可靠性和合理性[3]。
IR、LSC和SSC区扩张和收缩是植物进化中的常见现象, 也是造成叶绿体基因组长度差异与结构变异的主要原因[32, 33]。譬如, 苏玥等[34]通过对乳苣(Mulgedium tataricum) 及其他5个菊科莴苣族(Lactuceae) 植物叶绿体基因组LSC/IR和SSC/IR边界区扩张和收缩的研究, 发现莴苣族6个物种的叶绿体基因组序列整体高度相似, 但rps19、rpl2、ycf1、trnN、ndhF、trnH和rpl23等基因在LSC/IR和SSC/IR边界区的位置和大小存在稍许差异。本研究结果也同样表明, 葵花大蓟及蓟属其他8种植物叶绿体基因组的LSC/IR和SSC/IR边界基因排列和类型高度相似, 仅有少量差异, 如葵花大蓟和蓟的ycf1基因仅存在IRb/SSC边界区的丢失和SSC/IRa边界区向IRa区的扩张; 同时, 葵花大蓟、丝路蓟、绵毛蓟、刺儿菜、蓟和C. nipponicum的rps19基因也存在向IRb区扩张的现象。此外, 叶绿体基因组序列比对结果显示, 蓟属所有参试物种叶绿体基因组中LSC和SSC区的遗传变异显著高于IR区, 尤其存在高变异区域, 如petN-psbM、trnR-UCU-trnfM-CAU、petD-rpoA、ndhB-ycf15、trnL-UAG-ndhF和ycf1, 其往往会发生快速的核苷酸替换, 因而有望从这些区域中开发用于蓟属种间鉴定和系统发育分析的分子标记[35]。
叶绿体简单序列重复(SSR) 拷贝数变异是一种重要的分子标记, 因其具有比核基因与线粒体SSR更大的分类距离, 被广泛应用于植物群体遗传多样性和系统发育研究[36-38]。通过对葵花大蓟叶绿体基因组的分析, 本研究共检测到188个SSR位点, 其主要位于CDS区; 并且, 单核苷酸A/T碱基在SSR位点中出现频率最高, 这与先前报道的菊科其他植物的叶绿体基因组类似, 如乳苣、茵陈蒿(Artemisia capillaris) 和银叶蒿(Artemisia argyrophylla) 等[34, 39]。然而, 葵花大蓟SSR位点的三核苷酸和四核苷酸重复类型与已报道的菊科其他植物却有较大差异, 如葵花大蓟检测到的三核苷酸和四核苷酸类型分别有29种和7种, 远高于菊科其他物种[39-42], 说明葵花大蓟与菊科其他植物叶绿体基因组SSR重复类型差异较大, 必将为葵花大蓟SSR引物开发和系统发育研究提供较好的基础。
研究表明, 自然界存在的20种氨基酸中, 除甲硫氨酸(Met) 和色氨酸(Trp) 由唯一密码子编码外, 其他氨基酸均对应2~6个同义密码子, 由于同义密码子在机体内的使用频率存在差异, 从而形成植物密码子的使用偏性, 即密码子偏好性[43]。密码子偏好性是生物体进化的重要特征, 其对基因功能、物种进化等研究具有重要意义[44]。本研究密码子偏好性分析表明, 亮氨酸(Leu) 是葵花大蓟叶绿体基因组中占比最高的氨基酸, 并且相对同义密码子使用度(RSCU) 大于1的30个密码子通常以A/U结尾, 这与先前发表的菊科植物基本相似[41, 45], 认为这可能是由于叶绿体基因组富含大量A、U碱基, 从而导致不同植物的密码子偏好性不同, 但植物间亲缘关系越近则往往具有相似的密码子使用模式, 表明植物密码子偏好性在进化关系上较为保守[46, 47]。
探究菊科植物的系统发育关系现已成为当前系统与进化植物学研究的热点领域[48, 49]。纵观国内外研究进展, 发现菊科植物系统发育的研究主要集中于科内属间与一些属内[9, 34, 50, 51], 而关于蓟属种内系统发育分析的报道较少[29-31], 尤其有关葵花大蓟系统发育关系的研究目前尚属空白。本研究基于菊科管状花亚科46种植物的叶绿体基因组数据, 通过对其系统发育的分析发现葵花大蓟与翼蓟为姊妹类群, 靴带支持率高达100%, 表明两者具有较近的亲缘关系。
作者贡献: 郑长远负责实验数据分析和论文初稿撰写; 苏旭和刘玉萍负责实验设计和论文修改; 刘涛、王亚男、苏丹丹、张雨和秦娜参与野外采样和数据分析。全体作者都阅读并同意最终文本。
利益冲突: 无相关利益冲突。
| [1] |
Corriveau JL, Coleman AW. Rapid screening method to detect potential biparental inheritance of plastid DNA and results for over 200 angiosperm species[J]. Am J Bot, 1988, 75: 1443-1458. |
| [2] |
Raman G, Park KT, Kim JH, et al. Characteristics of the completed chloroplast genome sequence of Xanthium spinosum: comparative analyses, identification of mutational hotspots and phylogenetic implications[J]. BMC genomics, 2020, 22: 855. |
| [3] |
Xu WB, Xia BS, Li XW. The complete chloroplast genome sequences of five pinnate-leaved Primula species and phylogenetic analyses[J]. Sci Rep, 2020, 10: 20782. DOI:10.1038/s41598-020-77661-3 |
| [4] |
Palmer JD. Comparative organization of chloroplast genomes[J]. Annu Rev Genet, 1985, 19: 325-354. DOI:10.1146/annurev.ge.19.120185.001545 |
| [5] |
Rodríguez-Ezpeleta N, Brinkmann H, Burey SC, et al. Monophyly of primary photosynthetic eukaryotes: green plants, red algae, and glaucophytes[J]. Curr Biol, 2005, 15: 1325-1330. DOI:10.1016/j.cub.2005.06.040 |
| [6] |
Zhang YJ, Li DZ. Advances in phylogenomics based on complete chloroplast genomes[J]. Plant Diver and Resour (植物分类与资源学报), 2011, 33: 365-375. |
| [7] |
Du XY, Zeng T, Feng Q, et al. The complete chloroplast genome sequence of yellow mustard (Sinapis alba L.) and its phylogenetic relationship to other Brassicaceae species[J]. Gene, 2020, 731: 144340. DOI:10.1016/j.gene.2020.144340 |
| [8] |
Jansen RK, Kaittanis C, Saski C, et al. Phylogenetic analyses of Vitis (Vitaceae) based on complete chloroplast genome sequences: effects of taxon sampling and phylogenetic methods on resolving relationships among rosids[J]. BMC Evol Biol, 2006, 6: 32. DOI:10.1186/1471-2148-6-32 |
| [9] |
Liang FP, Wen XN, Gao HY, et al. Analysis of chloroplast genomes features of Asteraceae species[J]. Genom Appl Biol (基因组学与应用生物学), 2018, 37: 5437-5447. |
| [10] |
Chen YL, Shi Z. Flora Reipublicae Popularis Sinicae: Vol 78 (中国植物志第78卷)[M]. Beijing: Science Press, 1987: 86.
|
| [11] |
Liu SW. Flora Qinghaiica (青海植物志)[M]. Xining: Qinghai People's Publishing House, 1996: 439.
|
| [12] |
Northwest Institute of Plateau Biolog, CAS. Economic Flora of Qinghai (青海经济植物志)[M]. Xining: Qinghai People's Publishing House, 1987: 611.
|
| [13] |
Yang D, Luo Q, Cao ML, et al. Temperature stress on physiological indices of Compositae weeds and their adaptive significance[J]. J Jishou Univ (吉首大学学报), 2006, 27: 75-79. |
| [14] |
He ZW, Lv ZP, Wu WS, et al. Lipophilic chemical constituents from Cirsium souliei[J]. Acta Bot Bor-Occid Sin (西北植物学报), 2007, 27: 1884-1887. |
| [15] |
Lv MY, Ge H, Liu SJ, et al. Studies on the chemical constituents of Cirsium souliei (Franch.) Mattf.[J]. Northwest Pharm J (西北药学杂志), 2013, 28: 118-121. |
| [16] |
He XF, He ZW, Jin XJ, et al. Caryolane-type sesquiterpenes from Cirsium souliei[J]. Phytochem Lett, 2014, 10: 80-85. DOI:10.1016/j.phytol.2014.08.003 |
| [17] |
Doyle JJ, Doyle JL. A rapid DNA isolation procedure for small quantities of fresh leaf tissue[J]. Phytochem Bull, 1987, 19: 11-15. |
| [18] |
Chen SF, Zhou YQ, Chen YR, et al. Fastp: an ultra-fast all-in-one FASTQ preprocessor[J]. Bioinformatics, 2018, 34: i884-i890. DOI:10.1093/bioinformatics/bty560 |
| [19] |
Dierckxsens N, Mardulyn P, Smits G. NOVOPlasty: de novo assembly of organelle genomes from whole genome data[J]. Nucleic Acids Res, 2017, 45: e18. DOI:10.1093/nar/gkw1060 |
| [20] |
Tillich M, Lehwark P, Pellizzer T, et al. GeSeq - versatile and accurate annotation of organelle genomes[J]. Nucleic Acids Res, 2017, 45: W6-W11. DOI:10.1093/nar/gkx391 |
| [21] |
Qu XJ, Moore MJ, Li DZ, et al. PGA: a software package for rapid, accurate, and flexible batch annotation of plastomes[J]. Plant Methods, 2019, 15: 50. DOI:10.1186/s13007-019-0435-7 |
| [22] |
Zheng SY, Poczai P, Hyvönen J, et al. Chloroplot: an online program for the versatile plotting of organelle genomes[J]. Front Genet, 2020, 11: 576124. DOI:10.3389/fgene.2020.576124 |
| [23] |
Sharp PM, Li WH. The codon adaptation index - a measure of directional synonymous codon usage bias, and its potential applications[J]. Nucleic Acids Res, 1987, 15: 1281-1295. DOI:10.1093/nar/15.3.1281 |
| [24] |
Beier S, Thiel T, Münc T, et al. Misa-web: a web server for microsatellite prediction[J]. Bioinformatics, 2017, 33: 2583-2585. DOI:10.1093/bioinformatics/btx198 |
| [25] |
Amiryousefi A, Hyvönen J, Poczai P. IRscope: an online program to visualize the junction sites of chloroplast genomes[J]. Bioinformatics, 2018, 34: 3030-3031. DOI:10.1093/bioinformatics/bty220 |
| [26] |
Frazer KA, Pachter L, Poliakov A, et al. VISTA: computational tools for comparative genomics[J]. Nucleic Acids Res, 2004, 32: W273-W279. DOI:10.1093/nar/gkh458 |
| [27] |
Katoh K, Misawa K, Kuma K, et al. MAFFT: a novel method for rapid multiple sequence alignment based on fast Fourier transform[J]. Nucleic Acids Res, 2002, 30: 3059-3066. DOI:10.1093/nar/gkf436 |
| [28] |
Nguyen LT, Schmidt HA, von Haeseler A, et al. IQ-TREE: a fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies[J]. Mol Biol Evol, 2015, 32: 268-274. DOI:10.1093/molbev/msu300 |
| [29] |
Nam SJ, Kim JM, Kim YS, et al. The complete chloroplast genome of Korean endemic species, Cirsium rhinoceros (H. Lév. & Vaniot) Nakai (Asteraceae)[J]. Mitochondrial DNA B, 2019, 4: 2351-2352. DOI:10.1080/23802359.2019.1627940 |
| [30] |
Xie BX, Jiang CY, Zhu ZY, et al. The complete chloroplast genome of Cirsium setosum and its phylogenetic analysis[J]. Mitochondrial DNA B, 2019, 4: 3756-3757. DOI:10.1080/23802359.2019.1681921 |
| [31] |
Xu JS, Dang H, Li HZ. The complete chloroplast genome of Cirsium shansiense and its phylogenetic analysis[J]. Mitochondrial DNA B, 2020, 5: 1134-1135. DOI:10.1080/23802359.2020.1721032 |
| [32] |
Kim KJ, Lee HL. Complete chloroplast genome sequences from Korean ginseng (Panax schinseng Nees) and comparative analysis of sequence evolution among 17 vascular plants[J]. DNA Res, 2004, 11: 247-261. DOI:10.1093/dnares/11.4.247 |
| [33] |
Xue S, Shi T, Luo WJ, et al. Comparative analysis of the complete chloroplast genome among Prunus mume, P. armeniaca, and P. salicina[J]. Hort Res, 2019, 6: 89. DOI:10.1038/s41438-019-0171-1 |
| [34] |
Su Y, Liu JJ, Wan B, et al. Chloroplast genome structure characteristic and phylogenetic analysis of Mulgedium tataricum[J]. J Agric Sci Technol (中国农业科技导报), 2021, 23: 33-42. |
| [35] |
Ma ML, Meng HL, Zhang W, et al. Complete chloroplast genome and phylogenetic analysis of Amomum villosum[J]. Acta Bot Bor-Occid Sin (西北植物学报), 2020, 40: 978-986. |
| [36] |
Powell W, Morgante M, Mcdevitt R, et al. Polymorphic simple sequence repeat regions in chloroplast genomes: applications to the population genetics of pines[J]. Proc Natl Acad Sci U S A, 1995, 92: 7759-7763. DOI:10.1073/pnas.92.17.7759 |
| [37] |
Pugh T, Fouet O, Risterucci AM, et al. A new cacao linkage map based on codominant markers: development and integration of 201 new microsatellite markers[J]. Theor Appl Genet, 2004, 108: 1151-1161. DOI:10.1007/s00122-003-1533-4 |
| [38] |
Wang HK, Lou XM, Zhang Z. Application in germplasm resource research using chloroplast simple sequence repeat[J]. Mol Plant Breed (分子植物育种), 2006, 4: 92-98. |
| [39] |
Kim GB, Lim CE, Kim JS, et al. Comparative chloroplast genome analysis of Artemisia (Asteraceae) in East Asia: insights into evolutionary divergence and phylogenomic implications[J]. BMC Genomics, 2020, 21: 415. DOI:10.1186/s12864-020-06812-7 |
| [40] |
Iram S, Abdullah, Furrukh M, et al. Chloroplast genome sequences of Artemisia maritima and Artemisia absinthium: comparative analyses, mutational hotspots in genus Artemisia and phylogeny in family Asteraceae[J]. Genomics, 2020, 112: 1454-1463. DOI:10.1016/j.ygeno.2019.08.016 |
| [41] |
Tyagi S, Jung JA, Kim JS, et al. Comparative analysis of the complete chloroplast genome of mainland Aster spathulifolius and other Aster species[J]. Plants, 2020, 9: 568. DOI:10.3390/plants9050568 |
| [42] |
Loeuille B, Thode V, Siniscalchi C, et al. Extremely low nucleotide diversity among thirty-six new chloroplast genome sequences from Aldama (Heliantheae, Asteraceae) and comparative chloroplast genomics analyses with closely related genera[J]. PeerJ, 2021, 9: e10886. DOI:10.7717/peerj.10886 |
| [43] |
Campos JL, Zeng K, Parker DJ, et al. Codon usage bias and effective population sizes on the X chromosome versus the autosomes in Drosophila melanogaster[J]. Mol Biol Evol, 2013, 30: 811-823. DOI:10.1093/molbev/mss222 |
| [44] |
Wang J, Wang TY, Wang LY, et al. Assembling and analysis of whole chloroplast genome sequence of Elaeagenus angustifolia and its codon usage bias[J]. Acta Bot Bor-Occid Sin (西北植物学报), 2019, 39: 1559-1572. |
| [45] |
Gichira AW, Avoga S, Li ZZ, et al. Comparative genomics of 11 complete chloroplast genomes of Senecioneae (Asteraceae) species: DNA barcodes and phylogenetics[J]. Bot Stud, 2020, 60: 17. |
| [46] |
Zhang MW, Zhang TK, Liu CY, et al. Biosynthesis pathway evolution and codon usage bias analysis of lignin in Punica granatum[J]. Mol Plant Breed (分子植物育种), 2018, 16: 2131-2138. |
| [47] |
Yuan XL, Li YQ, Zhang JF, et al. Analysis of codon usage bias in the chloroplast genome of Dalbergia odorifera[J]. Guihaia (广西植物), 2021, 41: 622-630. |
| [48] |
Zhu SX. Systematics of Chaetoseris and Stenoseris (Compositae-Lactueeae) (毛鳞菊和细莴苣属的系统学研究) [D]. Beijing: Graduate School, the Chinese Academy of Science (Institute of Botany, the Chinese Academy of Sciences), 2004.
|
| [49] |
Gu JY. A Phylogenetic Study of Anaphalis Based on Chloroplast Genomes (基于叶绿体基因组的香青属分子系统学研究) [D]. Zhengzhou: Zhengzhou University, 2019.
|
| [50] |
Wu X, Jiang M, Chen HM, et al. Comparative analysis of three complete chloroplast genomes of Inula genus with phylogenetic analysis of 49 plants from Carduoideae[J]. Acta Pharm Sin (药学学报), 2020, 55: 1042-1049. |
| [51] |
Zhu B, Hu LJ, Tian ZJ, et al. Phylogenetic relationship analysis of 50 species in Asteraceae family based on the chloroplast genome sequence[J]. J Guizhou Norm Univ (贵州师范大学学报), 2021, 39: 15-25. |
 2022, Vol. 57
2022, Vol. 57


