2. 中国医学科学院、北京协和医学院药用植物研究所生物信息中心, 北京 100193;
3. 清新天正 (北京) 国际科技有限责任公司, 北京 100097;
4. 菏泽学院药学院, 山东 菏泽 274015;
5. 湘南学院药学院, 湖南 郴州 423099;
6. 齐鲁工业大学药学院, 山东 济南 250200
2. Institute of Medicinal Plant Development, Chinese Academy of Medical Sciences and Peking Union Medical College, Beijing 100193, China;
3. Fresh Sky-Right (Beijing) International Science and Technology Co. Ltd., Beijing 100097, China;
4. College of Pharmacy, Heze University, Heze 274015, China;
5. College of Pharmacy, Xiangnan University, Chenzhou 423099, China;
6. School of Pharmaceutical Sciences, Qilu University of Technology (Shandong Academy of Sciences), Jinan 250200, China
唇形科在全世界有3 500余种, 我国有99属800余种[1]。唇形科植物多数富含具有药用的芳香油, 香气浓郁, 且大多数颜色鲜丽, 是有名的油料作物和药用植物, 比如黄芩、丹参、薰衣草、薄荷、益母草等[2]。
地笋属约14种, 分布于东半球温带及北美, 我国有4种4变种, 4种分别是地笋L. lucidus、小花地笋L. parviflorus、小叶地笋L. coreanus、欧地笋L. europaeus和4变种分别是硬毛地笋L. lucidus Turcz. var. hirtus Regel、异叶地笋L. Turcz. var. maackianus Maxim.、西南小叶地笋L. coreanus Levl. var. Cavaleriei、深裂欧地笋L. europaeus Linn. var.exaltatus Hook[3]。
国内外研究表明, 地笋属的植物主要含有挥发油、萜类与甾体、酚酸类、黄酮类和微量元素等化学成分[3]。其中硬毛地笋是地笋的变种, 分布较广, 含有黄酮、挥发油等成分[4, 5]。全草入药, 乃本草经著录的泽兰正品, 为妇科要药, 能通经利尿, 对产前产后诸病有效, 根通称地笋, 可食, 又为金疮肿毒良剂, 并治风湿关节痛[1]。欧地笋L. europaeus是唇形科地笋属多年生草本植物, 小坚果四边形, 棕褐色。欧地笋分布于河北北部、陕西及新疆等地; 生于田边、沟边、潮湿草地, 海拔700~1 000 m。欧洲至亚洲中部也有, 北美洲也有引进[1]。研究表明, 欧地笋含有黄酮、有机酸、甾类和脂溶性挥发油等化学成分[6], 全草入药, 具有活血化瘀, 清热解毒, 通经, 利尿的功效; 对产前产后诸病有效, 根可治金疮肿毒, 并治风湿关节痛[7]。
叶绿体(chloroplast) 是绿色植物细胞中重要的细胞器, 是植物进行光合作用的场所, 也即“光动力工厂”。研究中发现, 植物细胞内的叶绿体基因组分子长度约为120~160 kb, 多数DNA分子是闭合共价双链形状, 通常叶绿体基因组可编码120个以上的基因, 主要分为三类基因, 分别是遗传系统基因, 即与转录和翻译相关的基因, 与光合作用相关的光合系统基因和与氨基酸、脂肪酸等物质生物合成相关的基因。通过对叶绿体基因组的研究, 发现叶绿体基因组特有的基因序列可用于物种的鉴别[8]、遗传多样性分析[9]、物种DNA分子鉴定[10]、同科属物种分子系统发育、药用植物遗传改良转化、分子育种基因工程[11]、生物制剂的生产、植物叶绿体基因的适应性进化等方面[12]。相对于独立的核系统转化, 叶绿体转化系统为植物导入外源基因提供了新的途径, 能够高效地在对应的重复区域内自动调整拷贝数并表达[13]。
到目前为止, 唇形科地笋属硬毛地笋的叶绿体基因组序列已有研究报道[14], 而同科属的植物欧地笋的叶绿体基因组还未见报道, 为深入探讨唇形科各个属植物之间的系统发育进化关系, 且为了比较与欧地笋具有相近功效物种间叶绿体全基因组序列的特征, 本研究对欧地笋叶绿体基因组进行了测序并分析了其结构特征, 通过DNA和蛋白质序列构建不同的进化树揭示欧地笋与各个植物的进化关系及其在唇形科系统发育中的地位。
材料与方法材料 欧地笋新鲜幼嫩的叶片采自广西植物园(22°54′30.34″N, 103°13′16.39″E), 经鉴定为欧地笋, 标本存放于中国医学科学院药用植物研究所(标本号implad201910060)。
基因组DNA提取和检测 取经硅胶干燥的欧地笋叶片100 mg, 用植物基因组DNA提取试剂盒(TIANGEN生物, 北京) 提取欧地笋总基因组DNA。通过1.0%琼脂糖凝胶电泳, NanoDrop 2000微量分光光度计(Thermo scientific, 美国) 和Qubit3.0检测总DNA的纯度和浓度。
叶绿体基因组的测序、组装、注释与特征分析 将符合测序要求的叶绿体基因组总DNA提取物500 ng DNA构建文库, 在IlluminaSolexa测序平台(Hiseq 4000, 圣地亚哥, CA, 美国) 上测序, 获取2×150 bp的reads[15]。利用NGS QC ToolKit过滤去除接头及低质量reads, 得到高质量待分析reads (即clean reads)[16]。以硬毛地笋(NC_036935.1) 叶绿体基因组序列作为参考序列, 采用NOVOplasty (3.7.2) 从原始测序数据中组装欧地笋的叶绿体基因组[17]。使用Bowtie2 (2.4.4) (http://bowtie-bio.sourceforge.net/bowtie2/manual.shtml)[18]将原始reads映射到组装好的欧地笋叶绿体基因组上, 检测组装序列的正确性。应用CPGAVAS2在线工具(http://www.herbalgenomics.org/cpgavas2/)[19]对欧地笋的叶绿体基因组序列进行注释和特征分析, 使用默认参数预测蛋白质编码基因、转移RNA (tRNA) 基因和核糖体RNA (rRNA) 基因, 对注释信息采用Apollo软件进行手工编辑校正[20]。将注释好的欧地笋叶绿体基因组序列用BankIt向NCBI在线提交, 获得序列登录号为OM617843。
欧地笋叶绿体基因组的特征分析 采用CPGview-RSG在线软件(http://www.herbalgenomics.org/cpgview/) 绘制欧地笋叶绿体基因组图谱, 对顺式剪接基因和反式剪接基因的情况进行了可视化展示[21]。应用CPGAVAS2在线分析工具(http://www.herbalgenomics.org/cpgavas2/) 获得欧地笋叶绿体基因组的总长度, 大单拷贝区LSC、小单拷贝区SSC和一对反向重复区IR的长度及各个区的GC含量; 并注释出各种编码基因的情况和长度、内含子和外显子的特征及非编码区的长度。然后与同属物种硬毛地笋的叶绿体基因组特征对比分析研究。
重复序列分析 CPGAVAS2在线工具可注释出各种的重复序列, 利用MISA软件(http://pgrc.ipk-gatersleben.de/misa/) 鉴定欧地笋和硬毛地笋叶绿体基因组中的简单重复序列(simple sequence repeats, SSR), 也叫微卫星序列[22, 23], 搜索参数设置为: 重复单元碱基数为1~6, 最小重复单元个数分别为10、5、4、3、3和3, 设置2个SSR之间的最小距离为100 bp, 如果距离小于100 bp, 则2个SSR被当做一个复合微卫星; 使用TRF软件(4.09, Gary Benson, 波士顿大学, https://tandem.bu.edu/trf/trf.html) 预测欧地笋叶绿体基因串联重复序列[24], 设定重复单元大小为≥ 7; 采用VMATCH软件预测欧地笋叶绿体基因散在重复序列。
LSC、SSC、IR边界与特征基因的比较研究 使用IRscope (https://irscope.shinyapps.io/irapp/) 比较唇形科中与地笋属近缘的共计10种植物的叶绿体基因组LSC、SSC和IR区域边界和大小[25]。同时对存在于不同区域和连接位点的相同或不同的基因进行比较分析, 探讨与近似药理作用的相关性。
系统发育分析 组装后的欧地笋叶绿体基因组和唇形科同科不同属且具有相近药理作用的物种共计28种(表 1), 以瑞香科的狼毒(S. chamaejasme, NC_042714.1) 和蔷薇科的委陵菜(P. chinensis, MN871983.1) 作为外类群, 从GenBank数据库下载27个物种的叶绿体基因组序列(表 1), 利用系统发育软件Phylosuite (v1.2.2) 软件[26]提取共有蛋白质和DNA序列, 不同物种按相同排列顺序首尾连接, 采用MAFFT (v7.313)[27]软件比对。系统发育分析基于TVM+F+I+G4模型下在IQ-TREE (v1.6.8)[28]中构建蛋白质和DNA序列比对的最大似然法(maximum likelihood, ML)[29]进化树。此外, 还应用软件Mega[30]结合邻接NJ法(neighbor joining method)[31]基于蛋白质和DNA序列构建系统进化树。通过1 000次bootstrap重复分析来评估系统发育树的显著性水平, 使用MEGA-X可视化系统发育树。
| Table 1 Information of chloroplast genomes used in this study. LSC: Large single copy region; SSC: Small single copy region; IR: Inverted repeat region |
欧地笋叶绿体基因组图谱绘制如图 1, 顺式剪切基因和反式剪切基因及其内含子、外显子的情况如图 2和3, 在预测出的基因中, 12个顺式剪接基因的rps16、atpF、rpoC1、petB、petD、rpl2 (×2)、ndhB (×2)、ndhA包含一个内含子, 而ycf3、clpP各包含两个内含子(图 2)。rps12是一种反式剪接基因, 它包含3个外显子。白色区域为IRa的第2外显子, 黑色区域为IRb的第2外显子, 灰色区域为第1外显子。其5′端位于LSC区, 而3′端位于IR区(图 3)。
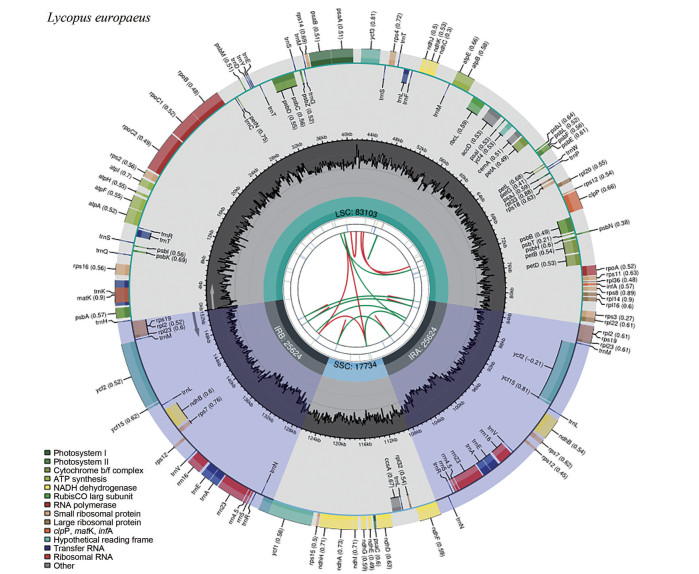
|
Figure 1 Chloroplast genome map of L. europaeus. Graphic representation of features identified in C. spicatus chloroplast genome by using CPGview-RSG (http://www.herbalgenomics.org/cpgview). The map contains seven circles. From the center going outward, the first circle shows the distributed repeats connected with red (the forward direction) and green (the reverse direction) arcs. The next circle shows the tandem repeats marked with short bars. The third circle shows the microsatellite sequences as short bars. The fourth circle shows the size of the LSC and SSC. The fifth circle shows the IRa and IRb. The sixth circle shows the GC contents along the plastome. The seventh circle shows the genes having different colors based on their functional group |
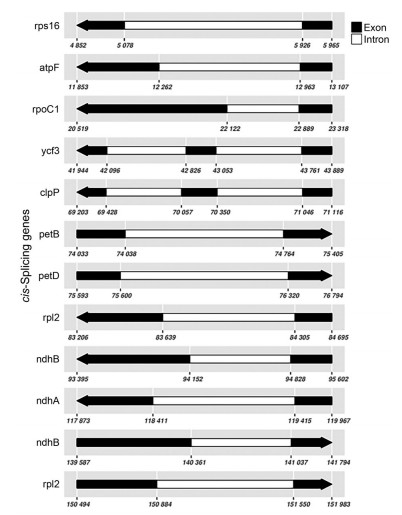
|
Figure 2 Schematic representation of the cis-splicing genome structure of the L. europaeus chloroplast genome. White and black regions are introns, exons, respectively. Arrows indicate the direction of the gene |
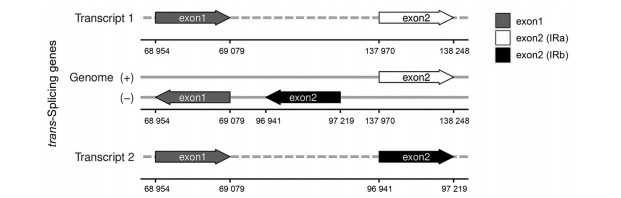
|
Figure 3 Schematic representation of the trans-splicing genome structure of the L. europaeus chloroplast genome. White, grey and black regions are exons. Arrows indicate the direction of the gene |
欧地笋总DNA测序共获得数据6.0 GB, 对应18 663 661条reads, 其原始数据(raw reads) 已经提交到GenBank, 相应的Bioproject ID为PRJNA726215。测序数据的覆盖度为100%, 其中337 628对reads (1.1%) 被精确地映射到基因组上一次, 全基因组的平均测序深度为337 628 × 151 / 152 085 = 335.2。与绝大多数被子植物叶绿体基因组一样, 欧地笋叶绿体基因组为双链环状分子, 全长152 085 bp, 包括1个大的单拷贝(LSC) 区(83 103 bp), 1个小的单拷贝(SSC) 区(17 734 bp) 和1对反向重复(IR) 区(25 624 bp)。全基因组的GC含量为38.04%, 其中IR区GC含量最高(43.16%), LSC区(36.16%) 和SSC区(32.05%) 均较低, 低于全基因组的GC含量。在欧地笋叶绿体基因组中共注释到132个基因, 包括88个蛋白质编码基因(独特基因80个), 8个rRNA基因[rrn16S (×2)、rrn23S (×2)、rrn4.5S (×2) 和rrn5S (×2)] (独特基因4个) 和36个tRNA基因(独特基因29个) (表 2)。其中8个蛋白质编码基因(ndhB、rps7、rps12、rps19、rpl2、rpl23、ycf2、ycf15)、7种tRNA编码基因(trnM-CAU、trnL-CAA、trnN-GUU、trnE-UUC、trnV-GAC、trnA-UGC、trnR-ACG)、4个rRNA编码基因(rrn16S、rrn23S、rrn4.5S、rrn5S) 位于IR区。欧地笋叶绿体基因组中有23个含内含子的基因, 包括13个蛋白质编码基因[rps16、rps19 (×2)、atpF、rpoC1、rpl2 (×2)、petB、petD、rpl16、ndhB (×2)、ndhA] 各含有1个内含子(intron), 2个蛋白质编码基因(ycf3、clpP) 各含有2个内含子, 8个tRNA编码基因各含有1个内含子。欧地笋叶绿体基因组中蛋白质编码区(coding sequence, CDS) 的长度为80 775 bp, 占整个基因组长度的53.11%。rRNA基因的长度为9 406 bp, 占整个基因组长度的6.18%。而tRNA基因的长度为2 724 bp, 占整个基因组长度的1.79%。叶绿体基因组非编码区主要包括内含子和基因间隔区, 其中基因间隔区的长度占整个基因组长度的38.92%。
| Table 2 Encoded genes in the chloroplast genome of L. europaeus. *(×2) The marker gene has two copies of the genes |
对唇形科10种植物的叶绿体基因组进行比较, 包括地笋属的欧地笋和硬毛地笋(表 1)。发现除了活血丹、硬毛地笋植物外的叶绿体基因组LSC/IRa和LSC/IRb边界的侧翼基因相同, LSC/IRb边界均在基因rpl22、rps19和rpl2基因之间, 而LSC/IRb边界位于rps19和psbA基因之间。SSC/IRa边界IRa一侧都存在一个ycf1基因且均跨越SSC/IRa区, 而活血丹叶绿体基因组比较特殊, rpl22、rps19、rpl2和psbA均未在四区出现, 多种tRNA出现, 有trnV、trnR、trnN, 还有rrn5、rrn23等, 硬毛地笋叶绿体基因组未见rpl2, SSC/IRa均位于ycf1基因中。地笋属两个物种间基因组长度有11个碱基的差距, 其中LSC区碱基长度差距为8 bp, SSC区碱基长度差距为3 bp, IR区长度相等。两个物种的叶绿体基因组均包含132个基因, 88个蛋白编码基因, 36个tRNA基因和8个rRNA基因, 且基因组的GC含量均为38.04%, 说明欧地笋与其变种硬毛地笋非常近缘(图 4)。通过比较10个物种的药理作用, 发现它们都主要用于感冒发热、清热解毒、治疗风湿性关节炎和尿路感染等方面, 可能与基因生物合成的化学成分之间的亲缘关系有一定的关联。
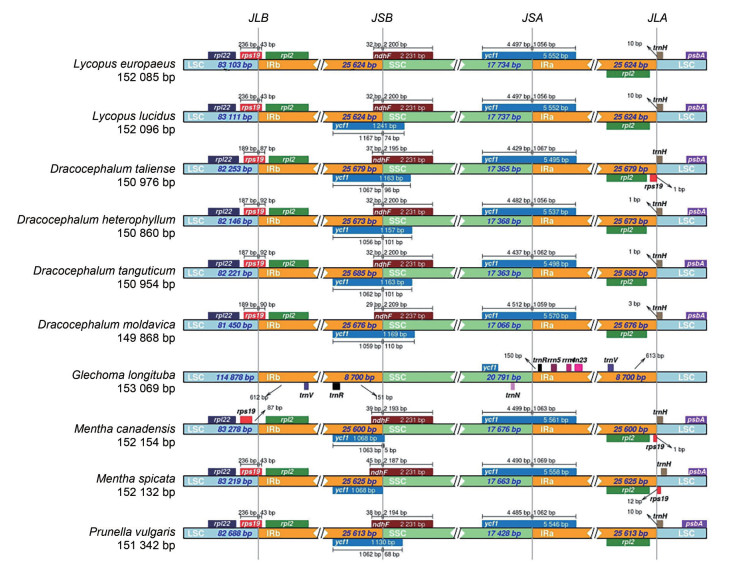
|
Figure 4 Schematic diagram showing the gene contents at the IR boundries in the chloroplast genomes from L. europaeus and its related species |
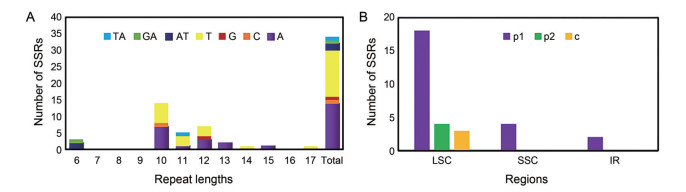
重复序列的插入、重排或缺失都会影响叶绿体基因组的长度和顺序, 且对叶绿体基因组进化方面也会有重要的作用。叶绿体基因组中重复序列一般可分为简单重复序列(SSR)、串联重复序列(tandem repeat sequence) 和分散重复序列(dispersed repeat sequence)。欧地笋叶绿体基因组中共检测到34个SSR, 其中单核苷酸重复SSR最多, 共计30个, A、C、G和T分别为14、1、1和14个; 其次是二核苷酸SSR, 共计4个, AT、GA和TA分别为2、1和1个; 三核苷酸和四核苷酸SSR未检测到。在所检测的SSR中以A/T、AT/AT为重复单元的占91.18%, 表明欧地笋叶绿体SSR偏好使用A和T碱基(表 3, 图 5), 其类型分别为P1 (24个)、P2 (4个) 和c (3个), 大部分都位于LSC区(80.65%), 位于SSC和IR区分别为12.9%和6.45%, SSRs的大小在10~22 bp之间(图 5)。以总长度超过20 bp且重复单元之间的相似性≥ 72%筛选后, 发现31个串联重复序列。以e值(e-value) 小于1E-05为阈值, 欧地笋叶绿体基因组散在重复序列包括回文重复序列17条、正向重复序列18条。
| Table 3 Microsatellite repeat sequences of L. europaeus chloroplast genome |
|
Figure 5 The number of identified SSR and their distribution. A: The number of SSR having a particular type of motif; B: Distribution of various SSR in LSC, SSC and IR regions from the L. europaeus chloroplast genome. p1: Monocleotide repeat; p2: Dinucleotide repeat; c: Composite SSR. SSR: Simple sequence repeats |
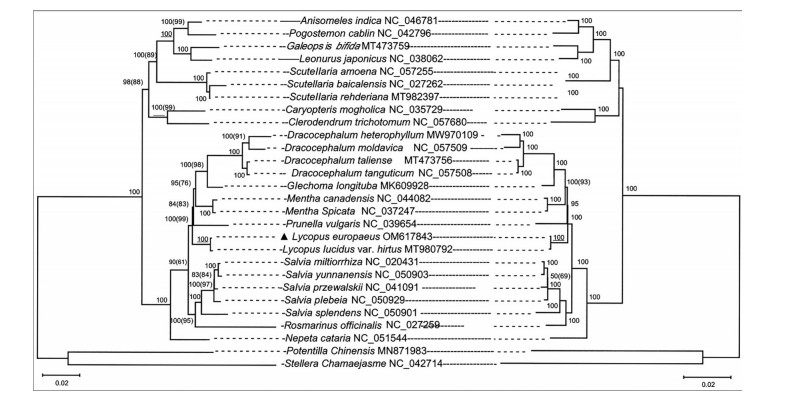
叶绿体基因组结构简单, 序列保守且基因大部分同源。研究异同科属植物叶绿体基因组之间的进化关系可提供系统发育和功效同源的有力依据。本研究选择了唇形科中常见具有类似的药理作用, 用于活血化瘀、清热解毒、抗菌消炎、抗风湿关节炎症的物种构建系统发育树。利用系统发育软件Phylosuite (v1.2.2) 软件提取81个共有蛋白质和DNA序列, 其中有26个唇形科的物种, 瑞香科的狼毒(S. chamaejasme, NC_042714.1) 和蔷薇科的委陵菜(P. chinensis, MN871983.1) 作为外类群构建最大似然法系统发育树(表 1)。从DNA和蛋白质序列构建的系统发育树中显示, 唇形科物种聚为一支, 分为4个小分支, 地笋属的两个物种与青兰属的4个物种、活血丹、薄荷属的两个物种、夏枯草聚为一支; 鼠尾草属5个物种和迷迭香聚为一支; 荆芥单独聚为一支; 蒙古莸、海州常山、黄芩属3个物种、广防风、广藿香、鼬瓣花和益母草聚为一支, 两个外类群狼毒和委陵菜单独聚为一支, 24个节点中, 应用CDS蛋白质和DNA构建的进化树bootstrap值[32]分别在84%~100%、50%~100%之间, 说明经过1 000次运算之后, 拓扑结构[33]置信度分别在84%和50%以上(图 6, 左右两侧分别为蛋白质和DNA进化树比对图)。应用软件Mega结合邻接NJ法(neighbor joining method) 基于DNA和CDS蛋白质序列构建的系统进化树, 各个物种的聚类结果与Phylosuite (v1.2.2) 结合最大似然法结果一致, 只是进化树的bootstrap值分别在69%~100%、61%~100%之间, 说明经过1 000次运算之后, 拓扑结构置信度分别在69%和61%以上(图 6), 结果表明应用Phylosuite结合最大似然法提取各个物种中的共有CDS蛋白质构建进化树的各个分支的拓扑结构可信度最高, 也说明两种方法构建的进化树都是可用的。
|
Figure 6 Phylogenetic tree based on CDS-protein (left) and DNA (right) sequences using the method of ML and NJ in the 28 species chloroplast genomes. The bootstrap values of ML and NJ methods are shown outside and inside of brackets on the nodes |
应用IlluminaSolexa测序平台首次完成了欧地笋的叶绿体基因组的测序, 使用软件NOVOplasty3.7.2组装原始数据。欧地笋叶绿体基因组中, 具有自我复制且在IR区的蛋白质编码基因有NADH脱氢酶亚单(ndhB)、核糖体小亚单位(rps7、rps12、rps19)、核糖体大亚单位(rpl2、rpl23), 还有两个未知功能具有ORF的基因(ycf2、ycf15)。在研究中比较了地笋属两个物种欧地笋和硬毛地笋的叶绿体基因组的基本特征, 虽然两者全长仅有11个碱基的差别, 经过比对发现两个物种的叶绿体基因组序列有多处的基因缺失和差异, 通过进化树构建中基因和蛋白等信息的提取后, 发现两者的共有基因110个, 其中硬毛地笋特有的tRNA基因有3种, 分别是trnI-CAU、trnI-GAU和trnV-UAC; 蛋白质序列均为80个, 其中有差异的3个, 分别是rps12、rps19和ycf1; 两个物种有68种基因间区, 其中欧地笋特定的基因间区有6个, 分别是ndhC_trnM-CAU、rpl22_ rpl2、rps15_ycf1、trnE-UUC_rrn16S、rrn16S_trnE-UUC和trnN-GUU_ndhF, 硬毛地笋特定的基因间区有5个, 分别是ndhC_trnV-UAC、rrn16S_trnI-GAU、start_rps12、trnI-GAU_rrn16S和trnN-GUU_ycf1。两个物种从进化树分支上看是近缘物种, 仅从原植物的外观性状鉴定, 硬毛地笋不同于欧地笋的地方就在于茎稜上被向上小硬毛, 节上密集硬毛; 叶披针形, 暗绿色, 上面密被细刚毛状硬毛, 叶缘具缘毛, 下面主要在肋及脉上被刚毛状硬毛, 两端渐狭, 边缘具锐齿。故今后需要结合共同基因和特定基因所表达的蛋白差异功能, 不仅从外观性状鉴定和区别, 而且结合异同基因生物化学过程合成功效成分的动态化学成分的差异, 也可从化学亲缘关系方面比较和区分两个物种。
在植物群体遗传学、种群分析、多态性研究和进化系统的研究中, 叶绿体基因组中的SSR基因起着很好的应用价值[34]。目前应用叶绿体基因组中的SSR研究的药用植物有甘草[35]、灵芝[36]、丹参[37]、明党参和川明参[38]、文冠果[39]、藠头[40]、甘西鼠尾草[41]、藠头[42]和旋覆花属等[43]。本研究结果表明欧地笋叶绿体基因组中的SSR包含高频率的A或T重复, 与很多植物叶绿体基因组SSR序列的组成相似, 比如草果[44]和丹参[45]。本研究中26个唇形科叶绿体基因组序列, 以瑞香科狼毒和蔷薇科委陵菜为外类群, 用两种方法构建的系统发育树中看到, 地笋属的两个物种与青兰属的4个物种在唇形科中的亲缘关系较近; 而且地笋属的两个物种在进化树中的节点的最大相似度为100%。从各个药用植物的药理作用分析发现, 与地笋属聚为一支的物种主要有活血化瘀、清热解毒、消炎平喘为主的功效, 当然主要是基于化学物质构效关系的不同引起的; 鼠尾草属5个物种和迷迭香主要以活血化瘀的“心脏动力药”为主聚为一类; 荆芥主要用于解暑、发汗发热、防治感冒, 故单独为一类; 蒙古莸、海州常山、黄芩属3个物种、广防风、广藿香、鼬瓣花和益母草主要以活血补血、解热解毒、止痛驱寒为主要功效, 故聚为一支。同时从分子角度分析物种的叶绿体基因组的聚类分析证实了物种之间的亲缘关系, 从而也可以一定程度说明该物种的药用成分可能具有高度的相似性, 因为植物体内的化学成分都是通过相关基因家族通过特定的生物合成途径生成的, 故相近化学成分和药理作用的物种间可以根据需要互相增补和调换使用。通过本次对欧地笋植物叶绿体基因的研究, 可为唇形科及地笋属的物种鉴定、分子进化和遗传系统发育研究提供重要的参考。
作者贡献: 第一作者杜清、王立强负责论文设计、实验、数据分析及论文撰写; 通讯作者刘昶、王彬负责指导论文的实验和数据分析; 姜梅、陈海梅参与样品采集、保存、数据分析; 陈卓尔、曾晶、刘鑫参与数据分析和文章修改。
利益冲突: 所有作者均声明不存在利益冲突。
| [1] |
Editorial Committee of Chinese Botany, Chinese Academy of Sciences. Flora of China: Vol. 66 (中国植物志: 第66卷)[M]. Beijing: Science Press, 1977: 1-18.
|
| [2] |
Editorial Department of Chinese Materia Medica. Chinese Materia Medica: Vol 19 (中华本草: 第19卷)[M]. Shanghai: Shanghai Science and Technology Press, 1999: 3-237.
|
| [3] |
Yang BC, Peng T, Kang WY. Advance on chemical constituents of Lycopus[J]. Chin J Exp Tradit Med Form (中国实验方剂学杂志), 2013, 19: 346-350. |
| [4] |
Peng T, Wang W, Zhang QJ, et al. Chemical constituents of Lycopus lucidus Turcz. var. hirtus Regel[J]. Nat Prod Res Dev (天然产物研究与开发), 2013, 25: 782-784, 806. |
| [5] |
Peng T, Wang JM, Zhang QJ, et al. Volatile oils constituents of Lycopus lucidus var. hirtus[J]. Nat Prod Res Dev (天然产物研究与开发), 2012, 24: 342-344. |
| [6] |
He J, He Y, Zhang JQ, et al. Studies on chemical constituents of Lycopus europaeus Linn.[J]. Pharm J Chin PLA (解放军药学学报), 2007, 23: 432-433. |
| [7] |
López V, Akerreta S, Casanova E, et al. In vitro antioxidant and anti-rhizopus activities of Lamiaceae herbal extracts[J]. Plant Foods Hum Nutr, 2007, 62: 151-155. DOI:10.1007/s11130-007-0056-6 |
| [8] |
Dong BR, Zhao ZL, Ni LH, et al. Comparative analysis of complete chloroplast genome sequences within Gentianaceae and significance of identifying species[J]. Chin Tradit Herb Drugs (中草药), 2020, 51: 1641-1649. |
| [9] |
Clegg MT, Gaut BS, Learn GH Jr, et al. Rates and patterns of chloroplast DNA evolution[J]. Proc Natl Acad Sci U S A, 1994, 91: 6795-6801. DOI:10.1073/pnas.91.15.6795 |
| [10] |
Zhang YJ, Li DZ. Advances in phylogenomics based on complete chloroplast genomes[J]. Plant Diver Resour (植物分类与资源学报), 2011, 33: 365-375. |
| [11] |
Ni LH, Zhao ZL, Mi M. Progress in the chloroplast genome of medicinal plants[J]. J Chin Med Mater (中药材), 2015, 38: 1990-1994. |
| [12] |
Wang B, Gao L, Su YJ, et al. Adaptive evoIutionary analysis of chloroplast genes in euphyllophytes based on complete chloroplast genome sequences[J]. J Sun Yat-sen Univ (Nat Sci Ed) (中山大学学报(自然科学版)), 2012, 51: 108-113, 146. |
| [13] |
Zhang JY, Zhang ZL, Su N, et al. Chloroplast transformation: a new way to introduce foreign genes into plant[J]. Chin Bull Bot (植物学通报), 2001, 18: 288-294. |
| [14] |
Wang Y, Wang HP, Zhou BZ, et al. The complete chloroplast genomes of Lycopus lucidus and Agastache rugosa, two herbal species in tribe Mentheae of Lamiaceae family[J]. Mitochondrial DNA Part B, 2021, 6: 89-90. DOI:10.1080/23802359.2020.1847617 |
| [15] |
Whiteford N, Skelly T, Curtis C, et al. Swift: primary data analysis for the Illumina Solexa sequencing platform[J]. Bioinformatics, 2009, 25: 2194-2199. DOI:10.1093/bioinformatics/btp383 |
| [16] |
Patel RK, Jain M. NGS QC Toolkit: a toolkit for quality control of next generation sequencing data[J]. PLoS One, 2012, 7: e30619. DOI:10.1371/journal.pone.0030619 |
| [17] |
Dierckxsens N, Mardulyn P, Smits G. NOVOPlasty: de novo assembly of organelle genomes from whole genome data[J]. Nucleic Acids Res, 2017, 45: e18. DOI:10.1093/nar/gkw1060 |
| [18] |
Langmead B, Salzberg SL. Fast gapped-read alignment with Bowtie 2[J]. Nat Methods, 2012, 9: 357-359. DOI:10.1038/nmeth.1923 |
| [19] |
Liu C, Shi L, Zhu Y, et al. CpGAVAS, an integrated web server for the annotation, visualization, analysis, and GenBank submission of completely sequenced chloroplast genome sequences[J]. BMC Genomics, 2012, 13: 715. DOI:10.1186/1471-2164-13-715 |
| [20] |
Firtina C, Kim JS, Alser M, et al. Apollo: a sequencing-technology-independent, scalable and accurate assembly polishing algorithm[J]. Bioinformatics, 2020, 36: 3669-3679. DOI:10.1093/bioinformatics/btaa179 |
| [21] |
Stothard P, Grant JR, Van Domselaar G. Visualizing and comparing circular genomes using the CGView family of tools[J]. Brief Bioinform, 2019, 20: 1576-1582. DOI:10.1093/bib/bbx081 |
| [22] |
Beier S, Thiel T, Münch T, et al. MISA-web: a web server for microsatellite prediction[J]. Bioinformatics, 2017, 33: 2583-2585. DOI:10.1093/bioinformatics/btx198 |
| [23] |
Provan J, Powell W, Hollingsworth PM. Chloroplast microsatellites: new tools for studies in plant ecology and evolution[J]. Trends Ecol Evol, 2001, 16: 142-147. DOI:10.1016/S0169-5347(00)02097-8 |
| [24] |
Benson G. Tandem repeats finder: a program to analyze DNA sequences[J]. Nucleic Acids Res, 1999, 27: 573-580. DOI:10.1093/nar/27.2.573 |
| [25] |
Amiryousefi A, Hyvönen J, Poczai P. IRscope: an online program to visualize the junction sites of chloroplast genomes[J]. Bioinformatics, 2018, 34: 3030-3031. DOI:10.1093/bioinformatics/bty220 |
| [26] |
Zhang D, Gao F, Jakovlić I, et al. PhyloSuite: an integrated and scalable desktop platform for streamlined molecular sequence data management and evolutionary phylogenetics studies[J]. Mol Ecol Resour, 2020, 20: 348-355. DOI:10.1111/1755-0998.13096 |
| [27] |
Katoh K, Standley DM. MAFFT multiple sequence alignment software version 7: improvements in performance and usability[J]. Mol Biol Evol, 2013, 30: 772-780. DOI:10.1093/molbev/mst010 |
| [28] |
Nguyen LT, Schmidt HA, von Haeseler A, et al. IQ-TREE: a fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies[J]. Mol Biol Evol, 2015, 32: 268-274. DOI:10.1093/molbev/msu300 |
| [29] |
Zhou JM. Parallelization Research on Maximum Likelihood Method in Molecular Phylogenetic Inference (分子系统发育推断中最大似然法的并行化研究) [D]. Fuzhou: Fujian Agriculture and Forestry University, 2016.
|
| [30] |
Kumar S, Stecher G, Li M, et al. MEGA X: molecular evolutionary genetics analysis across computing platforms[J]. Mol Biol Evol, 2018, 35: 1547-1549. DOI:10.1093/molbev/msy096 |
| [31] |
Jaffe A, Amsel N, Aizenbud Y, et al. Spectral neighbor joining for reconstruction of latent tree models[J]. SIAM J Math Data Sci, 2021, 3: 113-141. DOI:10.1137/20M1365715 |
| [32] |
Huang J, Liu Y, Zhu T, et al. The asymptotic behavior of bootstrap support values in molecular phylogenetics[J]. Syst Biol, 2021, 70: 774-785. DOI:10.1093/sysbio/syaa100 |
| [33] |
Pan WZ. Topology Optimization Method of Space Structures Based on Plant Growth Simulation Algorithm (基于模拟植物生长算法的空间结构拓扑优化方法研究) [D]. Guangzhou: South China University of Technolgy, 2019.
|
| [34] |
Ellegren H. Microsatellites: simple sequences with complex evolution[J]. Nat Rev Genet, 2004, 5: 435-445. |
| [35] |
Liu YL, Song ML, Hou JL, et al. Optimization and primer selections of SSR-PCR reaction system from medicinal Glycyrrhiza uralensis[J]. Lishizhen Med Mater Med Res (时珍国医国药), 2017, 28: 740-744. |
| [36] |
Qian J, Xu HB, Song JY, et al. Genome-wide analysis of simple sequence repeats in the model medicinal mushroom Ganoderma lucidum[J]. Gene, 2013, 512: 331-336. DOI:10.1016/j.gene.2012.09.127 |
| [37] |
Tian HY, Fei JQ, Zou Z, et al. Assessment of genetic diversity and genetic relationship of Salvia yunnanensis C. H. W. Wright germplasm resources based on SSR marker[J]. Mol Plant Breeding (分子植物育种), 2021, 9: 1-15. |
| [38] |
Qiu YX, Fu CX, Wu FJ. Analysis of population genetic structure and molecular identification of Changium smyrnioides and Chuanminshen violaceum with ISSR marker[J]. China J Chin Mater Med (中国中药杂志), 2003, 7: 598-603. |
| [39] |
Le LL, Yang XM, Yu WW, et al. Development and preliminary verification of SSR markers based on the genome of Xanthoceras sorbifolium Bunge[J]. Mol Plant Breeding (分子植物育种), 2021, 5: 1-11. |
| [40] |
Sathishkumar R, Lakshmi PT, Annamalai A, et al. Mining of simple sequence repeats in the genome of Gentianaceae[J]. Pharmacogn Res, 2011, 3: 19-29. DOI:10.4103/0974-8490.79111 |
| [41] |
Li WY, Wang JH, Tong L, et al. Establishment and optimization of Salvia przewalskii Maxim. ISSR-PCR reaction system[J]. J Anhui Agric Sci (安徽农业科学), 2015, 43: 37-38. |
| [42] |
Yang QQ, Jiang M, Wang LQ, et al. Complete chloroplast genome of Allium chinense: comparative genomic and phylogenetic analysis[J]. Acta Pharm Sin (药学学报), 2019, 54: 173-181. |
| [43] |
Wu X, Jiang M, Chen HM, et al. Comparative analysis of three complete chloroplast genomes of Inula genus with phylogenetic analysis of 49 plants from Carduoideae[J]. Acta Pharm Sin (药学学报), 2020, 55: 1042-1049. |
| [44] |
Ma ML, Zhang W, Meng HL, et al. Characterization and phylogenetic analysis of the complete chloroplast genome of Amomum tsao-ko[J]. Chin Tradit Herb Drugs (中草药), 2021, 52: 6023-6031. |
| [45] |
Qian J. Study on Chloroplast and Mitochondrial Genomes of Salvia miltiorrhiza (丹参的叶绿体和线粒体基因组研究) [D]. Beijing: Chinese Academy of Medical Sciences and Peking Union Medical College, 2014.
|
 2022, Vol. 57
2022, Vol. 57


