从给美洲古代三大文明之一——阿兹特克文明造成灾难性影响的天花到迄今为止严重威胁人类健康仍然缺乏根治手段的艾滋病和乙型肝炎等疾病, 再到近年来埃博拉病毒(Ebola virus, EBOV)、严重急性呼吸综合征冠状病毒(severe acute respiratory syndrome coronavirus, SARS-CoV)、中东呼吸综合征冠状病毒(Middle east respiratory syndrome coronavirus, MERS-CoV) 及新型冠状肺炎病毒(severe acute respiratory syndrome-coronavirus 2, SARS-CoV-2) 等引发的大规模疫情, 历史上病毒感染性疾病对人生命与健康的威胁从未停止[1]。虽然随着人类对传染性疾病认识的深入、良好卫生习惯的建立、疫苗和各种抗病毒药物的发展, 使得人类对病毒感染性疾病的控制力大大增强, 但是由于新病毒不断出现, 已知病毒容易发生突变, 疫苗接种覆盖率不足及抗病毒药物药效性不能满足临床需要等问题的存在, 研发新型、高效抗病毒药物一直是学术界和工业界持续关注的热点和重大课题。
抗病毒药物包括无机化合物、有机小分子、多肽和抗体类药物等, 相较于无机化合物和有机小分子, 多肽和抗体类药物因为具有更高的特异性而备受关注, 正逐渐在药物市场占据主导地位, 尤其是2020年11月, 美国、俄罗斯和印度分别批准4种抗体类药物用于治疗新冠肺炎[2]。噬菌体展示技术是发现多肽和抗体类药物的有力工具, 一些噬菌体展示文库来源的分子已经进入药物市场或正在开展临床研究, 而且随着噬菌体展示技术知识产权到期, 利用该技术研发抗病毒药物会更加便捷, 可以预见在不久的将来会有越来越多噬菌体展示技术来源的药物造福于病毒感染性患者。本文对噬菌体展示技术的原理和筛选过程进行了介绍, 对抗病毒药物研发领域常用的噬菌体展示文库及其在抗病毒药物发现中的作用进行了综述, 并对增强该技术来源的抗病毒药物活性的策略进行了总结, 在此基础上, 探讨了基于噬菌体展示技术的抗病毒药物研发领域面临的挑战和未来发展的方向。
1 噬菌体展示技术的基本原理 1.1 噬菌体展示文库的构建噬菌体展示技术起源于George P. Smith 1985年将外源多肽展示于单链噬菌体表面的研究[3], 其原理是将编码外源多肽或蛋白的DNA序列插入编码噬菌体衣壳蛋白的基因中, 从而将外源多肽或蛋白展示于噬菌体表面[4]。随着科技的发展, 包括多肽、抗体、抗体片段和cDNA在内的多种分子均可展示于噬菌体表面, 由于该技术通过在DNA水平上方便快捷的构建和测序, 实现了在蛋白水平上多样化的展示和高通量筛选, 因而成为基础研究领域和药物研发领域中的有力工具。2018年George P. Smith和Gregory P. Winter教授因在“多肽及抗体的噬菌体展示”技术方面的开创性工作获得诺贝尔化学奖。
可用于噬菌体表面展示的噬菌体载体很多, 如丝状噬菌体、T4噬菌体和λ噬菌体等。与丝状噬菌体相比, T4噬菌体和λ噬菌体作为展示载体有一些优点: ①两者均为烈性噬菌体, 通过裂解宿主菌得以释放, 这样一些难以穿过细菌细胞膜的蛋白就可以展示于噬菌体表面, 而丝状噬菌体展示的蛋白必须是一些能够穿越细菌细胞膜的蛋白, 因为对于丝状噬菌体而言, 其衣壳蛋白的组装是在细菌的周质腔中完成的, 被展示在衣壳蛋白上的外源蛋白必须穿越细菌的细胞膜到达周质腔; ② T4噬菌体和λ噬菌体可以用于展示高拷贝的外源蛋白, 而且对外源蛋白大小的限制较小; ③ T4噬菌体和λ噬菌体可以比较方便地将外源蛋白展示于C端。虽然如此, 丝状噬菌体展示系统依然是构建噬菌体展示文库的首选载体。这是因为: 首先, 由于T4噬菌体和λ噬菌体展示的蛋白是在细菌的细胞内完成的, 细胞内还原性的环境导致被展示的外源蛋白无法形成二硫键, 这样就提高了蛋白错误折叠的可能。而丝状噬菌体展示外源蛋白的组装是在周质腔中完成的, 周质腔中氧化性的环境为二硫键的形成提供了条件, 从而保证了外源蛋白折叠的准确性; 其次, T4噬菌体和λ噬菌体的基因组大而复杂, 操作麻烦, 而丝状噬菌体的基因组较小, 操作方便; 此外, T4噬菌体和λ噬菌体展示系统必须进行DNA亚克隆才能在蛋白水平上进行分析。正是由于这些原因, 丝状噬菌体展示系统仍然是目前最常用、发展最快的展示系统。如已广泛应用于基础研究和临床研究领域、由NEB和MoBitec GmbH公司开发的商品化的7肽库、12肽库、环7肽库等均属于丝状噬菌体展示系统[5]。
1.2 噬菌体展示文库的筛选从噬菌体展示文库中筛选获得阳性噬菌体的过程被称为生物淘洗(biopanning), 生物淘洗可以分为体外生物淘洗和体内生物淘洗。当靶蛋白或靶细胞易于获得时, 人们常采用体外生物淘洗法。该方法的基本流程为将靶蛋白或靶细胞固定在固相载体上, 然后加入噬菌体展示文库。与靶蛋白或靶细胞特异性结合的阳性噬菌体被捕获于固相载体上, 而不能结合的噬菌体则被洗去。用洗脱液将阳性噬菌体洗脱并感染大肠杆菌进行扩增, 扩增纯化后的噬菌体可投入下一轮循环(图 1)。通常3~4轮生物淘洗之后, 即可获得特异性识别靶标蛋白或靶标细胞的阳性噬菌体。通过对阳性噬菌体DNA的测序, 即可分析出噬菌体展示的多肽或者抗体的种类。当靶蛋白难于获得或者不明确时, 通常采用体内淘洗, 该方法是直接将噬菌体展示文库以尾静脉注射入模型动物体内, 通过心脏灌注洗去未结合的噬菌体, 然后分离目标器官或组织, 匀浆后感染大肠杆菌进行扩增并进入下一轮生物淘洗(图 1)。相较于体外淘洗, 体内淘洗最大的优点是靶蛋白可保持其天然构象, 这样获得的能够特异性识别靶器官或组织的分子具有更高的准确性[5]。
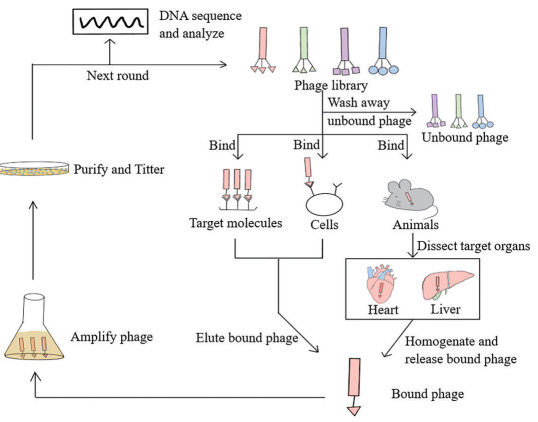
|
Figure 1 The biopanning process in vivo and in vitro. Phages specifically bound to targeted molecules, cells or organs can be obtained by in vitro and in vivo biopanning. The unbound phages are washed away and the bound phages are eluted and amplified in bacterial. The purified bound phages are put into the next round of biopanning. After 3-4 rounds of biopanning, the polypeptides or antibodies presented by the bound phages can be deduced from DNA sequencing and analysis |
根据展示对象的不同, 噬菌体展示文库可以分为噬菌体展示随机多肽文库、噬菌体展示抗体文库和噬菌体展示支架蛋白文库等, 这些文库在抗病毒药物研发领域均显示出良好的应用前景。
2.1 噬菌体展示随机多肽文库作为有机化合物, 多肽和许多有机小分子一样具有组织和器官渗透性高的优点, 同时其又比一般有机小分子的特异性高。相较于抗体类的大分子, 多肽还具有成本低、免疫原性低、生物活性高、稳定性高等优点, 因此噬菌体展示随机多肽库是抗病毒药物研究领域中最常用的文库之一。多个研究团队已分别用噬菌体展示7肽库和12肽库对多种病毒进行了生物淘洗, 包括禽传染性支气管炎病毒(IBV)、登革热病毒血清型2 (DENV-2)、罗氏沼虾结节病毒(MrNv)、日本脑炎病毒(JEV)、猪繁殖与呼吸系统疾病综合征病毒(PRRSV)、猪流行性腹泻病毒(PEDV)、传染性胃肠炎病毒(TGE)、传染性鲑鱼贫血病毒(ISAV)、牛短命热病毒(BEFV)、B型流感病毒和水貂肠炎病毒(MEV)[6-17]。Cao等[18]以抗PEDV刺突蛋白S的抗体MAb-5E12为靶蛋白, 从噬菌体展示12肽库中获得3个特异性与MAb-5E12结合的肽(LMQINPTYYQLM、WSFNPSTYTIAG和HDFVADMYQLAQ)。3个肽与天然S1蛋白的比对显示, 其与S1蛋白中一个假定的线性抗原表位(201MQYVYTPTYYML212) 具有高度的相似性。其中两个肽序列(LMQINPTYYQLM和WSFNPSTYTIAG) 具有共同的基序“PxxY”, 这两个多肽可以和细胞表面的受体结合, 阻断病毒与细胞的吸附。虽然MAb-5E12对PEDV没有中和活性, 但是两个MAb-5E12识别的肽(LMQINPTYYQLM和WSFNPSTYTIAG) 在体外可通过抑制PEDV对细胞表面受体的吸附显著降低病毒的产生。肽比抗体小, 具有与细胞表面受体更好的结合能力从而具有更好的病毒抑制活性。Lü等[19]则以基因型1a的HCV衣壳蛋白E2为靶分子对7肽库进行了淘洗。阻断实验表明, 淘洗获得的7肽(WPWHNHR) 可以阻断基因型1b和2a对Huh7.5细胞的入侵, 提示该肽在抗HCV方面具有潜在的应用前景, 而且其不受HCV基因型的限制。大量研究表明, 来自噬菌体展示多肽文库的多肽在与病毒高亲和力结合和阻断病毒活性方面表现优异, 有可能发展成为潜在的抗病毒药物(表 1)[6, 20-25]。
| Table 1 Application of phage display peptide libraries in antiviral drug research. JEV: Japanese encephalitis virus; HCV: Hepatitis C virus; H9N2 and H1N1: Avian influenza virus subtypes; WNV: West Nile virus; HIV-1: Human immunodeficiency virus 1; HBV: Hepatitis B virus; NA: Not available |
目前, 常用的噬菌体展示抗体文库主要为噬菌体展示抗体片段文库, 包括噬菌体展示Fab抗体文库、噬菌体展示ScFv文库和噬菌体展示nanobodies文库等。根据获得抗体或者抗体片段的基因来源, 又可将其分为天然文库、免疫文库、半合成和全合成文库等。天然文库构建自健康供体B细胞的IgM mRNA, 由于该文库中的抗体不偏向于任何特定的抗原, 可以针对多种疾病, 常常是进行药物筛选的首选文库; 免疫文库构建自免疫供体B细胞的IgG mRNA, 其克服了天然文库中抗体亲和力不够成熟的缺点, 主要用于筛选获得针对特定免疫原的抗体; 半合成抗体库构建自天然抗体序列和合成抗体序列的组合, 其多样性更高, 常用于筛选针对自身抗原的抗体[26, 27]。利用噬菌体展示抗体文库已经发现了多种病毒的抑制性候选药物(表 2)[28-37]。
| Table 2 Application of phage display antibody libraries in antiviral drug research. HEV: Hepatitis E virus; SARS-CoV-2: Severe acute respiratory syndrome coronavirus-2; EBOV: Ebola virus; HCMV: Human cytomegalovirus; RBD: Receptor-binding domain; Fab: Antibody-binding fragment; VH: Variable domain of heavy chain; VHH: Variable heavy chain of HCAb; HeV: Hendra virus |
抗原结合片段(antigen-binding fragment, Fab) 是抗体结构中可以与抗原结合的区域, 其含有1个可变区(V区) 和1个恒定区(C1), V区含有3个互补决定结构域(CDR) (图 2)。Schofield等[28]从1只感染了5种肝炎病毒(A到E) 的黑猩猩的骨髓构建了噬菌体展示Fab文库。通过对巴基斯坦菌株SAR-55 (SAR-55 ORF2) 重组戊型肝炎病毒(HEV) ORF2蛋白的筛选, 得到了与SAR-55 ORF2特异性结合的HEV#4和HEV#31单链抗体, Kd值分别为1.7和4.5 nmol·L-1。分别将HEV#4和HEV#31单链抗体及无关对照单链抗体和HEV SAR-55株孵育后感染恒河猴(Macaca mulatta) 以测定HEV#4和HEV#31的中和活性。研究表明, HEV预先和HEV#4或HEV#31孵育后不能使恒河猴产生感染, 而对照组中则有4只恒河猴发生了HEV感染。
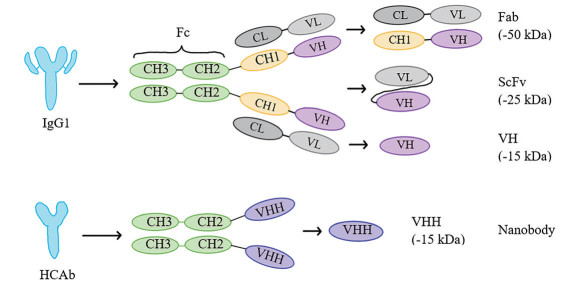
|
Figure 2 Schematic representation of antibodies and antibody fragments. IgG1: Immunoglobulin G1; HCAb: Heavy chain-only antibody; Fc: Crystallizable fragment; ScFv: Single-chain variable fragment; CH1-CH2-CH3: Constant domains of heavy chain; CL: Constant domain of light chain; VL: Variable domain of light chain. Adapted from Ref. 2 with permission. Copyright © MDPI open access journals |
单链抗体可变区(single-chain variable fragment, ScFv, 以下都称为单链抗体), 是由抗体重链可变区(heavy chain variable domain, VH) 和轻链可变区(light chain variable domain, VL) 通过一个10~25个氨基酸组成的柔性短肽(linker) 连接而成, 是最小的重组抗体形式(约27 kDa) (图 2)[38]。各种合成和天然的噬菌体展示ScFv文库已被构建并用于抗病毒药物的开发[39-42]。Van Audenhove等[43]以EBOV的VP35蛋白为靶分子, 对噬菌体展示ScFv文库进行了生物淘洗, VP35是一个双链RNA结合蛋白, 也是病毒RNA聚合酶复合体的共因子, 其通过阻断干扰素(interferon, IFN) 介导的抗病毒活性抑制宿主的免疫反应。将表达抗VP35 ScFv的质粒转染入表达IFN-β和EBOV VP35蛋白的A549细胞以评价生物淘洗获得的ScFv活性。被检测的5种ScFv中的两种F9和E1能够通过与VP35蛋白结合, 解除VP35对IFN抗病毒活性的抑制。Zhao等[44]利用狂犬病毒疫苗免疫供者外周血淋巴细胞构建了噬菌体展示ScFv文库。以狂犬病毒Gp蛋白线性中和表位为靶分子对文库进行筛选, 获得了具有中和活性的两个ScFv: T166和F21。斑块还原中和实验表明, T166和F21能够抑制3种不同狂犬病毒株(CVS-II、CQ92和SBD) 侵染Vero细胞。
2.2.3 噬菌体展示nanobodies文库纳米抗体(nanobodies) 是指骆驼科动物(如羊驼、骆驼等) 体内一种天然缺失轻链的抗体——重链抗体(heavy chain antibody, HCAb) 的Fab段。由于缺少轻链, HCAb的Fab段其实为重链上唯一的可变结构域, 即所谓的VHH (图 2)[45], 其分子质量和体积均远远小于普通抗体的Fab段, 因此被称为纳米抗体。和传统抗体相比, 纳米抗体具有分子质量小和高稳定的特点; 同时和同样具有小尺寸优势的Fab和ScFvs相比, 纳米抗体具有结构更稳定、溶解性更高和在微生物表达系统中产率更高的优点, 因此广泛应用于抗体药研究领域中[46]。
Tarr等[47]以HCV E2糖蛋白免疫的羊驼血细胞为基础, 构建了噬菌体展示VHH文库。以E2蛋白为靶分子, 对该文库进行淘洗获得了纳米体DO3。DO3特异性识别E2蛋白上与细胞受体(CD81) 结合的区域, 并与多种中和HCV E2单克隆抗体的表位重叠。DO3可以中和大部分原代HCV分离株, 其IC50值为1~10 μg·mL-1。此外, 纳米体DO3能够抑制HCV的细胞间传播, 这一点是一些中和抗体所不及的。DO3的分子质量较小(15 kDa) 以及其与E2蛋白独特的结合模式可能是其表现优异的原因。
2.3 噬菌体展示支架蛋白文库和缺少构象的线性多肽以及只能模拟单一β转角的环肽相比, 支架蛋白质因为具有确定的构象而具有更高的亲和力和特异性。因此, 近年来各种噬菌体展示支架蛋白文库引起了人们的关注。
锚蛋白重复序列是一种含有33个氨基酸残基的肽段, 在自然界400种以上的蛋白质中都发现了这段重复序列[48]。锚蛋白重复序列蛋白(designed ankyrin repeat proteins, DARPins) 是由紧密排列的重复结构域组成的, 通常由4个重复结构域组成。DARPins因为具有稳定性高、溶解性好及易于在细菌中表达等优点而被用于构建噬菌体展示支架蛋白文库[49]。Nangola等[50]以HIV-1 Gag前体的基质域(MA) 和衣壳域(CA) 组成的融合蛋白为靶分子对噬菌体展示DARPins文库进行筛选, 获得特异性针对HIV-1的锚蛋白AnkGAG1D4 (Kd: ~l µmol·L-1), 该蛋白包含3个锚蛋白重复序列, 能够特异性与Gag结合。在SupTI细胞表达外源性AnkGAG1D4能够降低HIV-1对该细胞的侵染。进一步的研究表明, AnkGAG1D4的抗病毒作用发生在HIV-1生命周期的整合后阶段, 包括干扰Gag蛋白的组装和出芽。
3 提高噬菌体展示文库来源抗病毒药物活性的策略 3.1 多价表达病毒与宿主细胞的结合是通过多个病毒表面外壳蛋白与宿主细胞表面多个受体同时结合而实现的, 噬菌体展示文库获得的常常是针对某一个分子的多肽(单价多肽), 其可能无法有效阻断病毒与宿主间的相互作用。为了克服这一问题, 在噬菌体展示文库进行筛选的基础上, 人们提出了构建多价多肽以增强多肽抗病毒活性的策略。构建多价多肽可以通过化学偶联的方法也可以通过基因工程的方法。
Hall等[51]将噬菌体展示库来源的2个靶向引起汉坦病毒肺综合征的无名病毒(Sin Nombre virus, SNV) 的环状肽CLVRNLAWC和CQATTARNC以化学偶联的方式共同连接在二氧化硅纳米颗粒上。研究结果显示, 相对于单独的CLVRNLAWC和CQATTARNC, CLVRNLAWC和CQATTARNC的偶联有效提高了对SNV病毒的抑制活性。
Zhou等[52]从噬菌体呈现12肽库中获得了靶向橙斑石斑鱼神经坏死病毒(OGNNV) 的12肽12C (LHWDFQSWVPLL), 并将12C和麦芽糖结合蛋白(MBP) 进行融合表达以增强12C的溶解性。为了提高12C与OGNNV的结合能力, 其将12C进行串联表达, 即将3份12C与MBP进行融合表达生成MBP-T12C。研究表明MBP-T12C与OGNNV的亲和性是单个12C的9倍。而且体外实验表明400 μg·mL-1 MBP-T12C蛋白可有效抑制OGNNV感染SB成纤维细胞。
3.2 骨架修饰多肽被内源性蛋白酶的快速降解导致了其生物利用度低, 对多肽骨架进行修饰可以克服这一问题。
细胞中的氨基酸除了甘氨酸外主要为L型氨基酸, 细胞中蛋白酶的主要成分也是L型氨基酸, 如果把L型氨基酸换为D型氨基酸, 则就可以免遭蛋白酶的降解。Liu等[53]以PRRSV的RNA依赖RNA聚合酶(NSP9) 蛋白为靶点对噬菌体展示12肽库进行了生物淘洗, 获得了阳性多肽P9。其分别合成了L型P9和D型P9, 并研究二者在抑制PRRSV感染MARC-145细胞中的作用, 实验结果显示D型P9具有比L型P9更高的抗病毒活性, 其IC50值分别为16.12和56.47 μmol·L-1。
3.3 完全抗体的转换噬菌体展示文库来源的抗体片段因为存在稳定性和溶解性低、半衰期短等缺点从而可能降低其体内外的中和活性。为了克服这一问题, 人们提出了在从噬菌体展示文库获得目的抗体片段的基础上, 将其转换为完全抗体的策略, 这样一方面利用了噬菌体展示文库方便、快捷、高通量的优点, 另一方面又可以发挥全长抗体稳定性好、半衰期长和中和活性强的优点。
Ohta等[32]以纯化的人巨细胞病毒(human cytomegalovirus, HCMV) 粒子和重组包膜糖蛋白B为靶分子对噬菌体展示Fab文库进行了生物淘洗, 获得了3个阳性Fab (H05、H08和H14), 体外实验表明, 这3个阳性Fab均没有中和活性, 但是当把3个阳性Fab转换为全长的IgG1时, 在添加5%豚鼠补体的情况下, 3个完整人IgG抗体均表现出了中和活性。
将噬菌体展示文库来源的ScFvs和Fab转化为完整的IgG抗体已被用于开发针对多种病毒的抗病毒药物, 包括流感病毒、呼吸道合胞病毒(respiratory syncytial virus, RSV)、HIV-1、冠状病毒和委内瑞拉马脑炎病毒(Venezuelan equine encephalitis virus, VEEV)。如MERS-CoV中和抗体是通过对由40名健康志愿者外周血单个核细胞构建的噬菌体展示Fab库筛选获得的。将MERS-CoV刺突蛋白和结合亲和力最高的3个Fab (m336、m337和m338) 转化为IgG1s后大大提高了其亲和力, 并且3种抗体均对MERS-CoV具有中和活性, 其中m336在Vero细胞中表现出最强的抑制活性, IC50为0.07 μg·mL-1。
3.4 跨膜能力的改进从噬菌体展示文库获得的多肽和蛋白通常是亲水性的, 如何顺利地通过细胞膜进入细胞以发挥抗病毒活性是噬菌体展示文库来源的药物面临的一大挑战。在这方面, 将噬菌体展示文库来源的药物与细胞穿透肽(cell-penetrating peptides, CPP) 等连接是常用的策略。CPP又称蛋白转导域(protein transduction domains, PTDs), 来源于果蝇触角同源结构域的第三螺旋(RQIKIWFQNRRMKWKK) 是一种介导抗病毒药物穿过细胞膜的多肽[54]。Yang等[55]以HIV-1复制必需的病毒粒子感染因子(Vif蛋白) 为靶蛋白对噬菌体展示肽库进行筛选, 在获得2种阳性多肽的基础上, 将多肽与CPP融合生成了2个融合多肽(Ant-VMI7和Ant-VMI9)。体外实验结果显示, 50 μmol·L-1 Ant-VMI7和Ant-VMI9融合肽能够抑制H9细胞中HIV-1的复制。
除了RQIKIWFQNRRMKWKK之外, 来源于HIV-1的Tat肽和多聚精氨酸都是广泛介导多肽或蛋白进入细胞或细胞核的CPP。如Zhuang等[56]通过将Tat与噬菌体展示Fab库来源的针对HIV-1 Rev蛋白的融合构建了FabRev1-Tat, 研究显示FabRev1-Tat可以有效进入PBMC, 并对3种不同的CCR5分离株均有抑制作用, IC50为0.09~0.44 μg·mL-1。
3.5 抗体亲和力成熟如何提高抗体类抗病毒药物, 特别是小分子抗体的亲和力一直是抗病毒药物研究领域需要解决的关键问题, 这一问题对于噬菌体展示文库来源的抗体或抗体片段而言同样备受关注。在噬菌体抗体展示技术中, 重链可变区和轻链可变区基因的随机组合在一定程度上模拟了抗体体内亲和力成熟的过程, 随着科学技术的不断进步, 借助于各种突变技术和计算机模拟等手段则可以进一步促进抗体亲和力的成熟。噬菌体展示技术中常见的突变技术包括错配PCR技术、定向CDR重组技术、热点突变技术、链替换技术和利用大肠杆菌高变株等。Huo等[57]通过PCR诱导抗体CDR3区域突变以促进抗体亲和力成熟, 获得了两个与SARS-CoV-2亲和力更高的纳米体H11-H4和H11-D4, 这两种纳米体的Fc融合物均显示出对SARS-CoV-2的中和活性(H11-H4为4~6 nmol·L-1, H11-D4为18 nmol·L-1)。
4 展望噬菌体展示技术是药物发现领域中非常重要的研究手段, 目前FDA已经批准了多个来源于此技术的多肽类和抗体类药物用于治疗不同的疾病, 如遗传性血管水肿、免疫性血小板减少性紫癜、类风湿性关节炎和葡萄膜炎等[58-60]。这些药物在临床上的优异表现和市场需求使噬菌体展示技术成为抗病毒药物发现领域备受关注的技术之一。特别是在新冠肺炎暴发以来, 全球多个研究小组已经分别以SARS-CoV-2的刺突糖蛋白S蛋白(spike glycoprotein, S potein)、S蛋白S1亚基的受体结合域(receptor binding domain, RBD) 及S蛋白的受体——血管紧张素转换酶2 (angiotensin converting enzyme 2, ACE2) 为靶点对噬菌体展示文库进行了筛选, 并且已经获得了多个靶向SARS-CoV-2的重组中和抗体[37, 61-67]。噬菌体展示文库最大的特点之一在于其库容的多样性、筛选的高通量和成本的低价性, 其库容通常大于1.0×109, 远远高于目前常用于筛选SARS-CoV-2抑制剂的化合物库如MDL公司的化合物库(MDL-CMC化合物库, 多样性约为8.0×103)、NIH分子库(多样性约2.9×105) 和ChemBridge Corporation化合库(多样性约为5.2×104), 可以预见随着人们对SARS-CoV-2结构和生命周期解析的进一步深入, 噬菌体展示技术在新冠病毒药物筛选的各个方面, 如基于病毒蛋白/酶的高通量筛选、基于细胞水平的表型筛选、基于靶标相似性的精准药物设计、基于靶标的全新药物设计等方面都将发挥巨大作用[68]。
除了筛选获得特异性抗病毒药物之外, 噬菌体展示技术在筛选广谱抗病毒药物方面同样具有广阔的应用前景。广谱抗病毒药物可作用于多种病毒或同一病毒的多种基因型, 具有一箭多雕的作用, 尤其对于应对忽然大规模暴发的疫情具有重要的“备选药物库”作用, 因而越来越受到抗病毒研究者的关注。以病毒复制过程中通用的一些病毒蛋白(如病毒聚合酶、病毒包膜等) 和宿主蛋白(如溶酶体、人腺苷三磷酸酶、宿主激酶和Toll样受体等) 为靶点对噬菌体展示文库进行筛选, 利用噬菌体展示文库组合多样性和筛选高通量性的优势, 必将有助于加快广谱抗病毒药物的开发进程[69]。
利用噬菌体展示技术获得的抗病毒药物主要以多肽和抗体片段为主, 但是目前多肽和抗体片段在进入临床应用中仍然面临很多挑战。如相对于完整的抗体分子, 多肽和抗体片段具有对酶的抵抗力较差、口服生物利用度低、血液半衰期短等缺点[70, 71]。但是正如前文所述, 研究者们正采用多种方法对噬菌体展示技术来源的多肽和抗体片段进行优化。对多肽进行化学修饰提高其对酶的抵抗性, 将筛选获得的抗体片段还原为全长的抗体分子来提高其血清半衰期等。多肽类药物被误认为具有成本高、药代动力学差和缺少口服生物利用度的劣势, 这可能是影响多肽类药物市场前景的重要原因。虽然多肽的大规模生产的确成本较高, 但是Otvos和Wade[72]强调研发具有活性药物成分的成本只占据药物开发总成本很小一部分比例(< 3%), 后期高的临床成功率完全可以对这部分成本给予补偿。此外, 多肽在进行了化学或者物理的修饰之后完全可以用于不同的给药方式, 包括鼻饲或口服, 药物研发者对这一点应该提高认识。另一方面, 虽然相对于抗体的全长分子, 抗体片段具有很多优点, 如可以适用于新的给药途径、可以靶向传统抗体无法靶向的表位、生产成本低等, 但是目前药物市场上的抗体片段类药物很少[73]。2019年, FDA批准caplacizumab用于治疗获得性血栓性血小板减少性紫癜, 这是FDA批准的第一个单域抗体, 其在临床上优于传统抗体的表现不仅增加了研发人员对这类药物的兴趣, 而且可以预见在不久的将来, 这类药物将在药物市场上快速推广[70]。
除了前文所述的挑战之外, 多肽和抗体类药物的广泛应用还面临一些其他难题, 如病毒突变引起的免疫逃逸, 研发可以形成终身免疫力的疫苗等[74, 75]。病毒感染性疾病的一过性也是导致抗病毒药物在药物市场吸引力低的原因。但是接种疫苗的覆盖率低以及目前一些病毒尚缺乏有效疫苗的事实, 使得研发有效的抗病毒药物仍然是十分必要的; 此外, 引发许多新发流行性传染疾病的病毒都是在曾经流行过的病毒的基础上变异而来, 与原来病毒具有很高的相似度, 持续做好现有病毒的药物储备, 是预防新发流行性传染疾病的有效措施之一。研究者期待在不久的将来, 噬菌体展示技术来源的多肽和抗体类药物能够加速抗病毒药物的研发从而造福人类。
作者贡献: 许世琦、贺子涵、陶炳灼完成本综述的文献检索、论文撰写、图表绘制、文章修改; 秦鑫负责了文章选题、写作指导、修改及校对。
利益冲突: 所有作者均声明不存在利益冲突。
| [1] |
Xu SJ, Zhang XJ, Ding D, 等. Bioinorganic chemistry strategies in antiviral drug discovery[J]. Acta Pharm Sin (药学学报), 2022, 57: 576-592. |
| [2] |
Sokullu E, Gauthier MS, Coulombe B. Discovery of antivirals using phage display[J]. Viruses, 2021, 13: 1120. DOI:10.3390/v13061120 |
| [3] |
Delhalle S, Schmit JC, Chevigné A. Phages and HIV-1: from display to interplay[J]. Int J Mol Sci, 2012, 13: 4727-4794. DOI:10.3390/ijms13044727 |
| [4] |
Reperant LA, Osterhaus AD. AIDS, avian flu, SARS, MERS, Ebola, Zika… what next?[J]. Vaccine, 2017, 35: 4470-4474. DOI:10.1016/j.vaccine.2017.04.082 |
| [5] |
Qin X. Screening and Identification of A Novel Peptide Ligand of Human Vascular Endothelia Growth Factor Receptor 3 (人血管内皮生长因子受体3结合肽的筛选和鉴定)[D]. Xi'an: Fourth Military Medical University, 2007.
|
| [6] |
Zu X, Liu Y, Wang S, et al. Peptide inhibitor of Japanese encephalitis virus infection targeting envelope protein domain Ⅲ[J]. Antivir Res, 2014, 104: 7-14. DOI:10.1016/j.antiviral.2014.01.011 |
| [7] |
Peng B, Chen H, Tan Y, et al. Identification of one peptide which inhibited infectivity of avian infectious bronchitis virus in vitro[J]. Sci China C Life Sci, 2006, 49: 158-163. |
| [8] |
Thong QX, Wong CL, Ooi MK, et al. Peptide inhibitors of Macrobrachium rosenbergii nodavirus[J]. J Gen Virol, 2018, 99: 1227-1238. DOI:10.1099/jgv.0.001116 |
| [9] |
Wei J, Hameed M, Wang X, et al. Antiviral activity of phage display-selected peptides against Japanese encephalitis virus infection in vitro and in vivo[J]. Antivir Res, 2020, 174: 104673. DOI:10.1016/j.antiviral.2019.104673 |
| [10] |
Wang H, Liu R, Cui J, et al. Characterization and utility of phages bearing peptides with affinity to porcine reproductive and respiratory syndrome virus NSP7 protein[J]. J Virol Methods, 2015, 222: 231-241. DOI:10.1016/j.jviromet.2015.04.026 |
| [11] |
Zou H, Zarlenga DS, Sestak K, et al. Transmissible gastroenteritis virus: identification of M protein-binding peptide ligands with antiviral and diagnostic potential[J]. Antivir Res, 2013, 99: 383-390. DOI:10.1016/j.antiviral.2013.06.015 |
| [12] |
Zhang Q, Wang Y, Ji Q, et al. Selection of antiviral peptides against mink enteritis virus using a phage display peptide library[J]. Curr Microbiol, 2013, 66: 379-384. DOI:10.1007/s00284-012-0284-3 |
| [13] |
Chew MF, Tham HW, Rajik M, et al. Anti-dengue virus serotype 2 activity and mode of action of a novel peptide[J]. J Appl Microbiol, 2015, 119: 1170-1180. DOI:10.1111/jam.12921 |
| [14] |
de la Guardia C, Quijada M, Lleonart R. Phage-displayed peptides selected to bind envelope glycoprotein show antiviral activity against Dengue virus serotype 2[J]. Adv Virol, 2017, 2017: 1827341. |
| [15] |
Ojeda N, Cárdenas C, Guzmán F, et al. Chemical synthesis and in vitro evaluation of a phage display-derived peptide active against infectious salmon anemia virus[J]. Appl Environ Microbiol, 2016, 82: 2563-2571. DOI:10.1128/AEM.00184-16 |
| [16] |
Hou P, Zhao G, He C, et al. Biopanning of polypeptides binding to bovine ephemeral fever virus G1 protein from phage display peptide library[J]. BMC Vet Res, 2018, 14: 3. DOI:10.1186/s12917-017-1315-x |
| [17] |
Wang Y, Xu H, Wu N, et al. Monoclonal antibody, but not synthetic peptide, targeting the ectodomain of influenza B virus M2 proton channel has antiviral activity[J]. New Microbiol, 2010, 33: 311-317. |
| [18] |
Cao L, Ge X, Gao Y, et al. Putative phage-display epitopes of the porcine epidemic diarrhea virus S1 protein and their anti-viral activity[J]. Virus Genes, 2015, 51: 217-224. DOI:10.1007/s11262-015-1234-5 |
| [19] |
Lü X, Yao M, Zhang JM, et al. Identification of peptides that bind hepatitis C virus envelope protein E2 and inhibit viral cellular entry from a phage-display peptide library[J]. Int J Mol Med, 2014, 33: 1312-1318. DOI:10.3892/ijmm.2014.1670 |
| [20] |
Amin A, Zaccardi J, Mullen S, et al. Identification of constrained peptides that bind to and preferentially inhibit the activity of the hepatitis C viral RNA-dependent RNA polymerase[J]. Virology, 2003, 313: 158-169. DOI:10.1016/S0042-6822(03)00313-1 |
| [21] |
Rajik M, Jahanshiri F, Omar AR, et al. Identification and characterisation of a novel anti-viral peptide against avian influenza virus H9N2[J]. Virol J, 2009, 6: 74. DOI:10.1186/1743-422X-6-74 |
| [22] |
Mertinková P, Mochnáčová E, Bhide K, et al. Development of peptides targeting receptor binding site of the envelope glycoprotein to contain the West Nile virus infection[J]. Sci Rep, 2021, 11: 20131. DOI:10.1038/s41598-021-99696-w |
| [23] |
Thenin-Houssier S, Valente ST. HIV-1 capsid inhibitors as antiretroviral agents[J]. Curr HIV Res, 2016, 14: 270-282. DOI:10.2174/1570162X14999160224103555 |
| [24] |
Wang W, Liu Y, Zu X, et al. Blocking peptides against HBV: preS1 protein selected from a phage display library[J]. Biochem Biophys Res Commun, 2011, 412: 633-637. DOI:10.1016/j.bbrc.2011.08.014 |
| [25] |
Matsubara T, Sumi M, Kubota H, et al. Inhibition of influenza virus infections by sialylgalactose-binding peptides selected from a phage library[J]. J Med Chem, 2009, 52: 4247-4256. DOI:10.1021/jm801570y |
| [26] |
Wang JL, He SQ, Kang ZX, 等. Antibody phage display technology and its application in the discovery of anti-SARS-CoV-2 antibodies[J]. Biotechnol Bull (生物技术通讯), 2021. DOI:10.13560/j.cnki.biotech.bull.1985.2021-0862 |
| [27] |
Flego M, Frau A, Accardi L, et al. Intracellular human antibody fragments recognizing the VP35 protein of Zaire Ebola filovirus inhibit the protein activity[J]. BMC Biotechnol, 2019, 19: 64. DOI:10.1186/s12896-019-0554-2 |
| [28] |
Schofield DJ, Purcell RH, Nguyen HT, et al. Monoclonal antibodies that neutralize HEV recognize an antigenic site at the carboxyterminus of an ORF2 protein vaccine[J]. Vaccine, 2003, 22: 257-267. DOI:10.1016/j.vaccine.2003.07.008 |
| [29] |
Pan Y, Du J, Liu J, et al. Screening of potent neutralizing antibodies against SARS-CoV-2 using convalescent patients-derived phage-display libraries[J]. Cell Discov, 2021, 7: 57. |
| [30] |
Wang R, Zhang H, Peng C, et al. Identification and characterization of a novel single domain antibody against Ebola virus[J]. Virol Sin, 2021, 36: 1600-1610. DOI:10.1007/s12250-021-00454-z |
| [31] |
Desimmie BA, Humbert M, Lescrinier E, et al. Phage display-directed discovery of LEDGF/p75 binding cyclic peptide inhibitors of HIV replication[J]. Mol Ther, 2021, 29: 887. DOI:10.1016/j.ymthe.2020.12.021 |
| [32] |
Ohta A, Fujita A, Murayama T, et al. Recombinant human monoclonal antibodies to human cytomegalovirus glycoprotein B neutralize virus in a complement-dependent manner[J]. Microbes Infect, 2009, 11: 1029-1036. DOI:10.1016/j.micinf.2009.07.010 |
| [33] |
Amatya P, Wagner N, Chen G, et al. Inhibition of Marburg virus RNA synthesis by a synthetic anti-VP35 antibody[J]. ACS Infect Dis, 2019, 5: 1385-1396. DOI:10.1021/acsinfecdis.9b00091 |
| [34] |
Anand T, Virmani N, Bera BC, et al. Phage display technique as a tool for diagnosis and antibody selection for coronaviruses[J]. Curr Microbiol, 2021, 78: 1124-1134. DOI:10.1007/s00284-021-02398-9 |
| [35] |
Chen C, Sun Z, Liu X, et al. Protocol for constructing large size human antibody heavy chain variable domain (VH) library and selection of SARS-CoV-2 neutralizing antibody domains[J]. STAR Protoc, 2021, 2: 100617. DOI:10.1016/j.xpro.2021.100617 |
| [36] |
Frenzel A, Schirrmann T, Hust M. Phage display-derived human antibodies in clinical development and therapy[J]. MAbs, 2016, 8: 1177-1194. DOI:10.1080/19420862.2016.1212149 |
| [37] |
Hong J, Kwon HJ, Cachau R, et al. Camel nanobodies broadly neutralize SARS-CoV-2 variants[J]. bioRxiv, 2021. DOI:10.1101/2021.10.27.465996 |
| [38] |
Maneewatch S, Thanongsaksrikul J, Songserm T, et al. Human single-chain antibodies that neutralize homologous and hetero-logous strains and clades of influenza A virus subtype H5N1[J]. Antivir Ther, 2009, 14: 221-230. DOI:10.1177/135965350901400204 |
| [39] |
Bannas P, Koch-Nolte F. Perspectives for the development of CD38-specific heavy chain antibodies as therapeutics for multiple myeloma[J]. Front Immunol, 2018, 9: 2559. DOI:10.3389/fimmu.2018.02559 |
| [40] |
Yang EY, Shah K. Nanobodies: next generation of cancer diagnostics and therapeutics[J]. Front Oncol, 2020, 10: 1182. DOI:10.3389/fonc.2020.01182 |
| [41] |
Bannas P, Hambach J, Koch-Nolte F. Nanobodies and nanobody-based human heavy chain antibodies as antitumor therapeutics[J]. Front Immunol, 2017, 8: 1603. DOI:10.3389/fimmu.2017.01603 |
| [42] |
Hassanzadeh-Ghassabeh G, Devoogdt N, De Pauw P, et al. Nanobodies and their potential applications[J]. Nanomedicine, 2013, 8: 1013-1026. DOI:10.2217/nnm.13.86 |
| [43] |
Van Audenhove I, Gettemans J. Nanobodies as versatile tools to understand, diagnose, visualize and treat cancer[J]. EBioMedicine, 2016, 8: 40-48. DOI:10.1016/j.ebiom.2016.04.028 |
| [44] |
Zhao XL, Yin J, Chen WQ, et al. Generation and characterization of human monoclonal antibodies to G5, a linear neutralization epitope on glycoprotein of rabies virus, by phage display technology[J]. Microbiol Immunol, 2008, 52: 89-93. DOI:10.1111/j.1348-0421.2008.00016.x |
| [45] |
Muyldermans S. Applications of nanobodies[J]. Annu Rev Anim Biosci, 2021, 9: 401-421. DOI:10.1146/annurev-animal-021419-083831 |
| [46] |
Liu H, Liang C, Duan H, et al. Intracellularly expressed nanobodies against non-structural protein 4 of porcine reproductive and respiratory syndrome virus inhibit virus replication[J]. Biotechnol Lett, 2016, 38: 1081-1088. DOI:10.1007/s10529-016-2086-3 |
| [47] |
Tarr AW, Lafaye P, Meredith L, et al. An alpaca nanobody inhibits hepatitis C virus entry and cell-to-cell transmission[J]. Hepatology, 2013, 58: 932-939. DOI:10.1002/hep.26430 |
| [48] |
Caputi AP, Navarra P. Beyond antibodies: ankyrins and DARPins. From basic research to drug approval[J]. Curr Opin Pharmacol, 2020, 51: 93-101. DOI:10.1016/j.coph.2020.05.004 |
| [49] |
Evans BJ, King AT, Katsifis A, et al. Methods to enhance the metabolic stability of peptide-based PET[J]. Molecules, 2020, 25: 2314. DOI:10.3390/molecules25102314 |
| [50] |
Nangola S, Urvoas A, Valerio-Lepiniec M, et al. Antiviral activity of recombinant ankyrin targeted to the capsid domain of HIV-1 Gag polyprotein[J]. Retrovirology, 2012, 9: 17. DOI:10.1186/1742-4690-9-17 |
| [51] |
Hall PR, Hjelle B, Brown DC, et al. Multivalent presentation of antihantavirus peptides on nanoparticles enhances infection blockade[J]. Antimicrob Agents Chemother, 2008, 52: 2079-2088. DOI:10.1128/AAC.01415-07 |
| [52] |
Zhou Q, Zhang J, Huang R, et al. An affinity peptide exerts antiviral activity by strongly binding nervous necrosis virus to block viral entry[J]. Fish Shellfish Immunol, 2019, 86: 465-473. DOI:10.1016/j.fsi.2018.12.003 |
| [53] |
Liu K, Feng X, Ma Z, et al. Antiviral activity of phage display selected peptides against porcine reproductive and respiratory syndrome virus in vitro[J]. Virology, 2012, 432: 73-80. DOI:10.1016/j.virol.2012.05.010 |
| [54] |
Schneider AFL, Kithil M, Cardoso MC, et al. Cellular uptake of large biomolecules enabled by cell-surface-reactive cell-penetrating peptide additives[J]. Nat Chem, 2021, 13: 530-539. DOI:10.1038/s41557-021-00661-x |
| [55] |
Yang B, Gao L, Li L, et al. Potent suppression of viral infectivity by the peptides that inhibit multimerization of human immunodeficiency virus type 1 (HIV-1) Vif proteins[J]. J Biol Chem, 2003, 278: 6596-6602. DOI:10.1074/jbc.M210164200 |
| [56] |
Zhuang X, Stahl SJ, Watts NR, et al. A cell-penetrating antibody fragment against HIV-1 Rev has high antiviral activity: characterization of the paratope[J]. J Biol Chem, 2014, 289: 20222-20233. DOI:10.1074/jbc.M114.581090 |
| [57] |
Huo J, Le Bas A, Ruza RR, et al. Neutralizing nanobodies bind SARS-CoV-2 spike RBD and block interaction with ACE2[J]. Nat Struct Mol Biol, 2020, 27: 846-854. DOI:10.1038/s41594-020-0469-6 |
| [58] |
Mimmi S, Maisano D, Quinto I, et al. Phage display: an overview in context to drug discovery[J]. Trends Pharmacol Sci, 2019, 40: 87-91. DOI:10.1016/j.tips.2018.12.005 |
| [59] |
Nixon AE, Sexton DJ, Ladner RC. Drugs derived from phage display: from candidate identification to clinical practice[J]. MAbs, 2014, 6: 73-85. DOI:10.4161/mabs.27240 |
| [60] |
Parray HA, Chiranjivi AK, Asthana S, et al. Identification of an anti-SARS-CoV-2 receptor-binding domain-directed human monoclonal antibody from a naïve semisynthetic library[J]. J Biol Chem, 2020, 295: 12814-12821. DOI:10.1074/jbc.AC120.014918 |
| [61] |
Temsamani J, Vidal P. The use of cell-penetrating peptides for drug delivery[J]. Drug Discov Today, 2004, 9: 1012-1019. DOI:10.1016/S1359-6446(04)03279-9 |
| [62] |
Ravichandran S, Coyle EM, Klenow L, et al. Antibody signature induced by SARS-CoV-2 spike protein immunogens in rabbits[J]. Sci Transl Med, 2020, 12: eabc3539. DOI:10.1126/scitranslmed.abc3539 |
| [63] |
Zeng X, Li L, Lin J, et al. Isolation of a human monoclonal antibody specific for the receptor binding domain of SARS-CoV-2 using a competitive phage biopanning strategy[J]. Antib Ther, 2020, 3: 95-100. |
| [64] |
Bertoglio F, Meier D, Langreder N, et al. SARS-CoV-2 neutralizing human recombinant antibodies selected from pre-pandemic healthy donors binding at RBD-ACE2 interface[J]. Nat Commun, 2021, 12: 1577. DOI:10.1038/s41467-021-21609-2 |
| [65] |
Gai J, Ma L, Li G, 等. A potent neutralizing nanobody against SARS-CoV-2 with inhaled delivery potential[J]. MedComm(2020), 2021, 2: 101-113. |
| [66] |
Kim YJ, Lee MH, Lee SR, et al. Neutralizing human antibodies against severe acute respiratory syndrome coronavirus 2 isolated from a human synthetic Fab phage display library[J]. Int J Mol Sci, 2021, 22: 1913. DOI:10.3390/ijms22041913 |
| [67] |
Lim SA, Gramespacher JA, Pance K, et al. Bispecific VH/Fab antibodies targeting neutralizing and non-neutralizing spike epitopes demonstrate enhanced potency against SARS-CoV-2[J]. MAbs, 2021, 13: 1893426. DOI:10.1080/19420862.2021.1893426 |
| [68] |
Li J, Jiang XY, Xu SJ, 等. Medicinal chemistry strategies in seeking coronavirus inhibitors[J]. Acta Pharm Sin (药学学报), 2020, 55: 537-553. |
| [69] |
Huang TG, Sun L, Zhan P, 等. Recent advances in the research of broad-spectrum antiviral agents[J]. Acta Pharm Sin (药学学报), 2020, 55: 679-693. |
| [70] |
Duan H, Ma Z, Xu L, et al. A novel intracellularly expressed NS5B-specific nanobody suppresses bovine viral diarrhea virus replication[J]. Vet Microbiol, 2020, 240: 108449. DOI:10.1016/j.vetmic.2019.108449 |
| [71] |
Gupta V, Hwang BH, Lee J, et al. Mucoadhesive intestinal devices for oral delivery of salmon calcitonin[J]. J Control Release, 2013, 172: 753-762. DOI:10.1016/j.jconrel.2013.09.004 |
| [72] |
Otvos L Jr, Wade JD. Current challenges in peptide-based drug discovery[J]. Front Chem, 2014, 2: 62. |
| [73] |
Lu RM, Hwang YC, Liu IJ, et al. Development of therapeutic antibodies for the treatment of diseases[J]. J Biomed Sci, 2020, 27: 1. DOI:10.1186/s12929-019-0592-z |
| [74] |
Marasco WA, Sui J. The growth and potential of human antiviral monoclonal antibody therapeutics[J]. Nat Biotechnol, 2007, 25: 1421-1434. DOI:10.1038/nbt1363 |
| [75] |
Salazar G, Zhang N, Fu TM, et al. Antibody therapies for the prevention and treatment of viral infections[J]. NPJ Vaccines, 2017, 2: 19. DOI:10.1038/s41541-017-0019-3 |
 2022, Vol. 57
2022, Vol. 57


