2. 石河子大学甘草研究所, 新疆 石河子 832003
2. Licorice Research Institute of Shihezi University, Shihezi 832003, China
甘草在多种中药复方中常作为佐使药, 具有多种功效和应用[1], 在《新型冠状病毒感染的肺炎诊疗方案(试行第1至7版)》中, 甘草的使用频次位居榜首[2]。甘草(Glycyrrhiza uralensis Fisch.)、胀果甘草(Glycyrrhiza inflata Bat.)、光果甘草(Glycyrrhiza glabra L.) 作为药用甘草资源植物被收录于《中国药典2020版》[3]。由于3种药用甘草存在种间自然杂交现象[4-8], 形成多种变异和种间杂交类型[9, 10], 给野生药用甘草的种质资源分类鉴定带来困难[11], 造成人工栽培种源混乱、药材质量参差不齐, 严重影响药用甘草资源利用和甘草产业发展。
黄甘草(Glycyrrhiza eurycarpa P.C.Li) 为甘草属药用植物资源, 其形态性状介于胀果甘草、甘草或光果甘草之间, 不同的形态变异类群主要分布于新疆和甘肃[7, 12-14]; 相关研究表明黄甘草是同域分布种的种间杂交、基因渐渗的产物[15], 基于形态性状和核DNA遗传结构的分析均表明与3种药用甘草密切关联, 常混于道地甘草药材中, 是目前中药材甘草资源分类鉴定的疑难种, 也是药材种源和种苗混杂的类群之一。
叶绿体基因组具有单系或兼系遗传、小分子量、多拷贝、分子进化速率慢等特点[16-18]。近年来, 通过叶绿体全基因组解析和特征比较研究, 解析种及种下单元“困难”植物类群系统发育关系, 在中药材分类鉴定研究中得到广泛应用, 如秦艽[19]、柴胡[20]等。目前, 有关甘草属叶绿体基因组的研究主要集中于胀果甘草、甘草、光果甘草这3种药用甘草的初步解析和对比[21-26], 但基于药材资源中疑难种群的分类、DNA指纹鉴定和甘草属种间叶绿体比较基因组学和系统发育的研究还未见报道, 尤其是有关黄甘草叶绿体基因组的特征研究。
本研究测序获得新疆产黄甘草及其同域分布的甘草、胀果甘草3个物种的叶绿体全基因组序列; 并与已发表的3种药用甘草的叶绿体基因组序列进行比较基因组学分析, 旨在全面解析黄甘草以及甘草属种间叶绿体全基因组序列与结构特征差异, 筛选种间高变异序列区段和物种候选特异DNA指纹; 构建系统发育树, 阐明甘草属种间亲缘关系。为药用甘草资源物种鉴定、药材特异DNA指纹开发、分子植物育种等研究奠定基础。
材料与方法实验材料 黄甘草、甘草采自石河子大学引种种植30余年甘草资源圃, 原产地为新疆巴楚, 胀果甘草采自新疆巴楚。标本采集压制后, 以《甘草属(Glycyrrhiza L.) 分类系统与实验生物学研究》[27]、《中国药典》[3]、《中国植物志》[28]为基础, 从中筛选出具有典型物种形态特征, 经石河子大学生命科学学院陆嘉惠教授鉴定确认后的3份样本(对应3种甘草), 3份标本必需满足的条件有: 具有相应物种的典型特征; 植株的叶片无破损、虫蛀、霉斑; 叶片完全舒展压制并脱水及时, 叶色新绿, 保证叶绿体DNA提取质量; 标本的采集年份、采集地区等信息完整。将标本完整叶片置于自封袋干燥低温保存, 送往上海天昊生物公司进行叶绿体全基因组测序, 凭证标本(采集号: 201309013、201309306、201809036) 保存于石河子大学植物标本馆甘草标本室。从NCBI数据库下载光果甘草序列, 序列号为: KU891817。
基因组DNA的提取和测序 利用TGuide植物基因组DNA提取试剂盒(天根, OSR-M301) 提取总DNA。胀果甘草叶绿体全基因组通过第三代高通量测序(PacBio RSⅡ) 技术获得: 利用Qubit3.0检测基因组DNA浓度, G-tubes方法将基因组DNA处理成8~10 K的片段。两端分别连接环装单链, 将文库单链环退火, 结合到固定的零模板波导孔(zero-modewaveguides, ZMW) 后用PacBio测序平台进行测序。黄甘草、甘草的叶绿体全基因组利用第二代高通量测序(Illumina Hiseq) 技术获得: 完成基因组DNA抽提后, 利用1%琼脂糖凝胶电泳检测收集基因组DNA。使用Covaris ME220预设程序超声打断cpDNA的同时在片段两端加上接头, 快速高效构建文库, PCR扩增等获得测序文库。应用Agilent 2100 Bioanalyzer确定文库片段大小分布, 质检合格后用IlluminaHiSeq平台, 以2×150 bp双端测序模式进行高通量测序。
叶绿体基因组的组装与注释 先将IlluminaHiSeq平台测序得到的原始序列(raw reads或raw data) 进行数据过滤。利用SOAPdenovo v2.04 (http://soap.genomics.org.cn/) 拼接软件将二代和三代测序的高质量数据进行拼接。运用GapCloser v1.12软件对组装结果进行局部内洞填充和碱基校正。使用metaSPAdes软件将序列组装成Contigs。根据参照基因组(MT120789) 对组装结果进一步分析, 判断是否成环、校正Contig方向和判断起始碱基位置等。用CPGAVAS2软件注释叶绿体基因组序列, ogdraw软件将Genebank文件绘制成基因圈图。注释并组装完成后的黄甘草、甘草、胀果甘草的叶绿体全基因组序列提交至GenBank数据库, 检索号分OK545539、OK509081、OK509082。
重复序列分析和密码子使用分析 简单重复序列(simple sequence repeats, SSRs) 检测通过MISA软件完成, 参数设置为单核苷酸重复单元不少于10个, 二核苷酸重复单元不少于5个, 三核苷酸和四核苷酸重复单元不少于4个, 五核苷酸和六核苷酸重复单元不少于3个, 且两个SSRs之间的最小距离为100 bp, 若距离小于100 bp, 则两个SSR被当作一个复合微卫星[29]。
甘草、黄甘草、胀果甘草、光果甘草的叶绿体基因组的密码子运用软件CodonW1.4.2分析获得相关参数: 有效密码子数(effective number of codon, Nc)、GC含量和第三位上的GC碱基的含量(GC3s)。RSCU值采用python CAI包中的RSCU模块分别计算。
叶绿体全基因组比较分析 利用在线基因组比对工具mVISTA, 以黄甘草(OK545539) 为参照, 将本研究所测甘草(OK509081) 和胀果甘草(OK509082) 与已公开报道的甘草(MN199032、KU862308)、胀果甘草(MH321931、MN064852)、光果甘草(NC024038、KU891817、KF201590) 的叶绿体全基因组序列进行全基因组对比。
序列变异分析 为进一步分析黄甘草叶绿体全基因组序列间的变异情况, 将本研究所测黄甘草(OK545539)、甘草(OK509081) 和胀果甘草(OK509082) 与已公开报道的甘草(MN199032、KU862308)、胀果甘草(MH321931、MN064852)、光果甘草(NC024038、KU891817、KF201590) 的叶绿体全基因组序列比对和检查调整之后, 利用DnaSP软件进行滑动窗口分析(sliding window analysis), 计算核苷酸多样性指数Pi, 并根据分析结果筛选种间高变异序列。滑动窗口(windows length) 大小设置为200 bp, 步长(step size) 为100 bp。
系统发育关系 将本研究测序的黄甘草、甘草、胀果甘草和从NCBI下载的16条甘草属植物序列(甘草: MN199032、KU862308; 胀果甘草: MH321931、MN064852; 光果甘草: KU891817、NC024038、KF201590; 刺毛甘草G. echinata: MT120761; 云南甘草G. yunnansis: MT120794; 臭甘草G. foetida: MT120765; 粗毛甘草G. aspera: MT120751、欧洲甘草G. lepidota: MT120781、直甘草G. astragalina: MT120756、三叶甘草G. triphylla: MT120805、圆果甘草G. squamulosa: MT120788、刺果甘草G. pallidiflora: MT120785) 以及1个外类群(紫藤W. Sinensis: KT200359) 的完整叶绿体基因组序列利用Mafft在线进行比对, 将比对结果采用IQtree软件ML法构建系统进化树。
结果 1 叶绿体基因组基本特征本研究首次测序获得黄甘草的叶绿体基因组, 并新测序甘草、胀果甘草的叶绿体基因组, 光果甘草为NCBI下载序列。黄甘草叶绿体基因组为典型环状结构(图 1), 因缺失1个反向重复IR区, 形成IRLC群体。总长127 864 bp, 和甘草、胀果甘草、光果甘草长度相似, 仅相差24~31 bp (表 1)。黄甘草GC含量为34.25%, 与3种药用甘草相近。甘草、黄甘草、胀果甘草共注释得到110个编码基因, 包括蛋白编码基因76个、tRNA基因30个和rRNA基因4个(表 1), 基因组成可分为4类: 与光合作用有关的基因、与自我复制有关的基因、成熟酶基因(matK)、囊膜蛋白基因(cemA)、蛋白酶(clpP1)、乙酰辅酶A羧化酶(accD)、c型细胞色素合成基因(ccsA) 和未知功能的蛋白基因(ycf1、ycf2、ycf4) (表 2)。
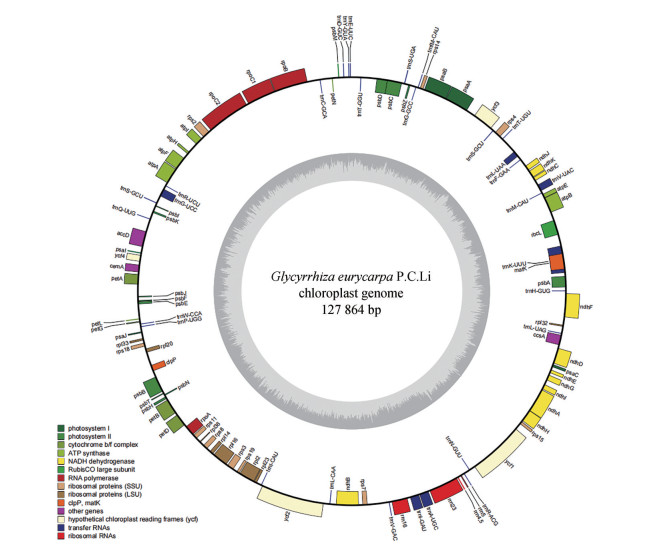
|
Figure 1 Chloroplast genome map of Glycyrrhiza eurycarpa P.C.Li. Genes inside and outside the circle are transcribed in a clockwise and counter clockwise direction, respectively. Genes are color-coded based on their functions. The grey area in the inner circle indicates the GC content of the chloroplast genome |
| Table 1 Comparison of chloroplast genome characteristics of four Glycyrrhiza species |
| Table 2 Gene composition in the chloroplast genome of Glycyrrhiza species |
甘草、胀果甘草、黄甘草和光果甘草的叶绿体基因组分别检测出79、75、74、75个SSR。SSR位点包括单核苷酸重复、二核苷酸重复、三核苷酸重复、四核苷酸重复以及复合型SSR。其中单核苷酸重复最多, 占比达到40%以上。其次是二核苷酸、复合型SSR、四核苷酸, 三核苷酸重复最少。4种甘草的1~4核苷酸重复基序的组成存在一定的碱基偏好性, 以富含A-T的重复基序为主(表 3)。
| Table 3 SSR motifs and numbers in chloroplast genomes of four Glycyrrhiza species |
利用CodonW1.4.2对黄甘草、甘草、胀果甘草、光果甘草叶绿体基因组的密码子组成进行分析。由表 4可知, 4种甘草叶绿体基因组的Nc值范围在53.72~53.99, 说明叶绿体基因组的密码子偏好性较弱。叶绿体基因组的GC3s含量范围为32.6%~33.0%, GC含量也在50%以下, 说明4种甘草叶绿体基因的密码子偏向使用A和T这两种碱基。由于遗传密码子的简并性, 除Trp和Met只有1个密码子外, 其他18种氨基酸均有2个及2个以上的密码子, 如Phe、Tyr、His、Gln、Glu、Asn、Asp、Lys、Cys、IleVal、Pro、Thr、Ala、Gly、Leu、Arg、Ser; 除甲硫氨酸(Met, M) 和色氨酸(Trp, W) 外, 大多数氨基酸密码子具有偏好性(图 2)。4种叶绿体基因组中蛋白编码基因的密码子数量分别是21 909、22 036、21 909、22 025个; 其中编码率最高的氨基酸是亮氨酸(Leu, L) (2 310~2 318个, 10.5%), 编码率最低的氨基酸是半胱氨酸(Cys, C) (256~259个, 1.2%) (图 3)。
| Table 4 Overall characteristics of codon usage of four Glycyrrhiza species chloroplast genome |

|
Figure 2 Codon relative usage of 20 amino acids in chloroplast genome protein encoding genes of four Glycyrrhiza species |
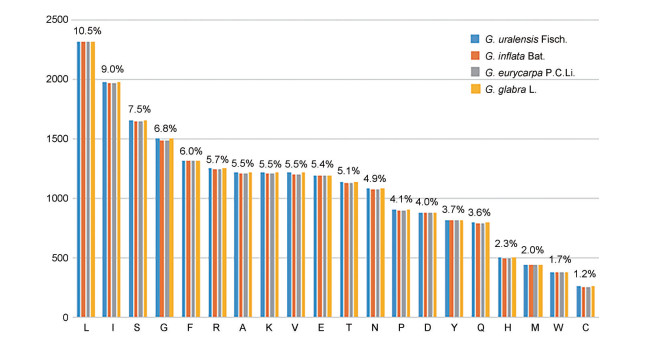
|
Figure 3 The number and coding rate of 20 amino acids in chloroplast genome protein coding genes of four Glycyrrhiza species |
本研究以黄甘草(OK545539) 的叶绿体基因组序列作为参考, 比对结果显示甘草属种间叶绿体基因组的相似性和差异性(图 4)。经比对发现本研究测序黄甘草和胀果甘草的序列一致性、同源性较甘草高。黄甘草在3个基因区域(trnL-UAA~trnT-UGU、trnG-GCC~psbZ、ycf2) 与3种药用甘草的差异较大, 具有特异性, 其中2个非编码区, 1个编码区。

|
Figure 4 Collinearity analysis of chloroplast genome sequences of Glycyrrhiza species. The vertical scale, ranging from 50% to 100%, indicates the percentage of identity calculated in sliding windows. The horizontal axis indicates the coordinates within the chloroplast genome. Different colors correspond to the types of the genome regions. Blue: Regions coding for proteins; Pink: Regions that are non-coding; Light blue: Regions coding for tRNAs and rRNAs |
使用软件DnaSP分析检测叶绿体基因组中的高突变区, 计算核苷酸变异值(Pi) 以显示在序列水平上的变异程度。结果如图 5所示, Pi值检测范围在1~128 411 bp之间, 甘草属叶绿体全基因组序列的核苷酸多样性指数Pi平均值为0.000 921 144。通过Pi值可以筛选出6个核苷酸高突变(psbZ~psbC、trnC-GCA~rpoB、trnR-UCU~trnG-UCC、ycf2、trnN-GUU~ycf1、ndhA), 其中ycf2的核苷酸多样性最高(0.02)。
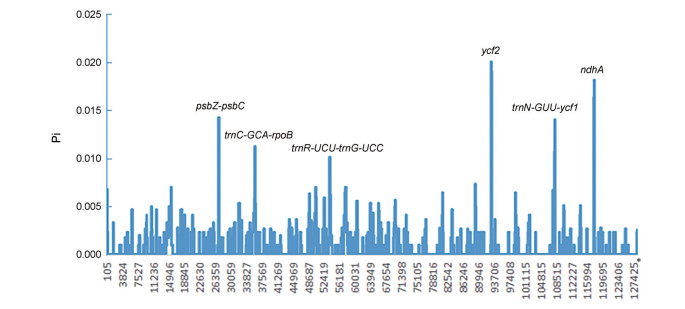
|
Figure 5 High nucleotide mutation sites of Glycyrrhiza species. Window length: 200 bp; Step size: 100 bp. *bp |
为确定黄甘草的系统发育地位, 基于20条叶绿体全基因组序列构建ML系统进化树(图 6)。结果显示: 第Ⅲ组外类群紫藤单独成为一支; 甘草属在99%以上支持率的进化支上可以分为Ⅰ、Ⅱ组: 第Ⅰ组在100%的支持率上, 3种主要的药用甘草(甘草、胀果甘草、光果甘草) 和黄甘草、粗毛甘草、三叶甘草、臭甘草聚为一支。第Ⅱ组的刺果甘草、圆果甘草、刺毛甘草、云南甘草、欧洲甘草、直甘草聚为一大支。
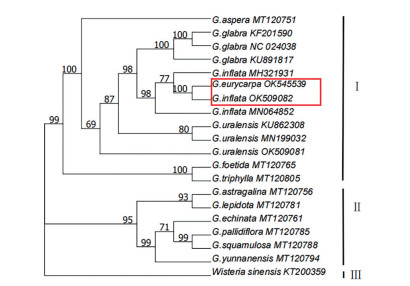
|
Figure 6 ML phylogenetic tree was constructed based on the whole chloroplast genome sequence |
由以上系统进化树的聚类结果可知: 黄甘草与胀果甘草、光果甘草、甘草亲缘关系较近, 其中黄甘草和本研究同域分布的胀果甘草的遗传关系最近。
讨论本研究基于二代高通量测序技术和生物信息学分析方法首次对黄甘草叶绿体基因组进行测序研究。结果表明黄甘草叶绿体基因组长为127 864 bp, 与甘草、胀果甘草、光果甘草相比长度相似。同时, 黄甘草与甘草、胀果甘草、光果甘草皆缺失一个方向重复的IR区。IR缺失现象不仅出现在甘草属植物的研究中[21-26], 蝶形花亚科其他植物, 如黄芪[30]、苦参[31]等植物中也有被报道。有研究指出IR丢失事件致使蝶形花科植物在进化上形成IRLC (缺失IR序列的蝶形花科植物) 分支[24, 32], 黄甘草是IRLC群体的新成员。
本研究新测序的甘草、胀果甘草、黄甘草共得到注释基因110个, 与已公开报道的甘草属叶绿体基因组的注释基因结果相同[21-26]。本研究ycf3基因属于光系统Ⅰ亚基, 这与Kang等[25]的注释结果一致。雷万钧[33]在统计蝶形花亚科植物的叶绿体基因组基因丢失情况时发现包括光果甘草在内的36种植物皆丢失rps16基因, 本研究4种甘草也存在相同的情况, rps16基因丢失现象在甘草属以及蝶形花亚科是否具有规律性还需更多的物种进行分析说明。
叶绿体基因组中的SSRs因含量丰富、多态性高, 且兼具单亲遗传模式等优点, 被作为分子标记广泛用于群体遗传学、谱系地理学等研究[34, 35]。从4种甘草的叶绿体全基因组中共检测出301个SSRs, 其中单核苷酸重复最多, 占比达到40%以上。其次是二核苷酸、复合型SSR、四核苷酸, 三核苷酸重复最少。1~4核苷酸重复富含A-T碱基, 这与其他被子植物叶绿体基因组中的SSRs序列构成一致[36-38], 也进一步支持了叶绿体SSR主要是由短的polyA和polyT重复所构成, 较少含有C或G串联重复的观点[39]。另外, 辛倩基于转录组数据的SSR研究发现, 胀果甘草的SSR重复以三核苷酸(5 111, 47.51%) 最多, 重复类型以CAA/GAA为主; 其次是二核苷酸(5 102, 47.51%) 重复, 重复类型以CT/TC为主[10], 这与本研究中4种甘草叶绿体基因组SSR特征不同。
对4种甘草叶绿体基因组的密码子进行分析, 获得相关参数Nc、RSCU、GC和GC3s。Nc值的范围在20~61之间, 越靠近数值20表明密码子的偏好性越强, 反之, 越偏向数值61密码子的偏好性越弱[40]。经分析发现4种甘草叶绿体基因组的Nc值大于40, 说明叶绿体基因组的密码子使用偏好性较弱。经测定4种甘草叶绿体基因组的GC含量和GC3s含量均小于50%, 说明叶绿体基因组的密码子偏好使用A和T碱基, 与蒺藜苜蓿[41]和苦参[31]的研究结果一致。4种甘草叶绿体基因组的密码子相关参数比较发现, 不同种的甘草植物叶绿体基因组的蛋白编码基因具有很大的相似性, 尤其是黄甘草和胀果甘草的同源性更高。
在甘草属植物研究中, Kondo等[42]利用ITS和叶绿体基因组的3个高突变区(rbcL, matK, trnH-psbA) 进行了药用甘草的鉴定, 成功将3种甘草鉴定出来, 但是由于位点数目有限, 对存在杂交性状的疑似杂交类群并没有得到很好的结论。Chen等[9] 2016年通过3种药用甘草的人工杂交组合, 获得亲本和F1代的ITS和cpDNA的rbcL、matk、trnH-psbA序列, 发现甘草属的质体遗传方式主要为父系遗传, 为甘草属疑似杂交类群的亲本鉴定和杂交起源研究提供了科学依据。本文基于mVISTA的叶绿体全基因组序列比较获得3个黄甘草特异性指纹片段(trnL-UAA~trnT-UGU、trnG-GCC~psbZ、ycf2), 可用于DNA条形码的开发, 为甘草属药材种质资源和正伪品鉴别、种苗筛选, 解决药材种源混乱问题提供分子鉴定依据。
此外, 核苷酸多样性分析获得甘草属的6个高突变区(psbZ~psbC、trnC-GCA~rpoB、trnR-UCU~trnG-UCC、ycf2、trnN-GUU~ycf1、ndhA)。叶绿体基因组高突变区的发现, 在属下种间、种内不同居群的系统发育中具有重要价值[43]。如猕猴桃研究中, Tang等[44]对9个猕猴桃种的叶绿体基因组进行全基因组范围内的差异位点的挖掘, 鉴定得到24个高变异的基因间区域; 基于其中4个区域[ndhF-rps32、ndhC-trnV (UAC)、rps16-trnQ (UUG) 和trnE (UUC)-trnT (GGU)] 的扩增结果, 对46个不同猕猴桃种进行了亲缘关系的系统进化分析, 物种的聚类结果和基于核基因组SNP的聚类结果一致, 支持了猕猴桃属的系统发育关系[45]。本研究获得的6个高突变区可为今后甘草属亲缘关系和系统发育分析增加新的候选分子标记。
本研究所获得的系统发育树中各类群间的拓扑结构与经典分类学观点基本一致: 首先外类群紫藤单独被分出, 甘草属分为2支。黄甘草与3种药用甘草聚为一大支, 遗传关系近, 这与陆嘉惠等[14]、陈超南等[9]、宋凤[15]的研究结果一致。在有关黄甘草分类地位研究中, 相关研究通过形态特征、幼苗发育、SCoT分子标记、SSR分子标记探索了黄甘草的分类地位和系统发育关系[6, 8, 13, 14], 认为黄甘草的出现是建立在甘草属种间不断分化, 并伴有一定的基因交流和渐渗的过程中逐渐产生的, 是物种形成的邻域、同域模式下的杂交产物。在甘草属物种同域分布区, 由于相遇的亲本种存在花期重叠、杂交亲和现象[5, 6], 种间杂交、回交渐渗等事件频繁发生, 产生形态和基因型不同的杂交类群。黄甘草是甘草、胀果甘草、光果甘草的种间杂交类群的统称, 在自然分布区, 目前研究发现存在多种杂交类型[9]。本研究中, 新疆产黄甘草(OK545539) 和同域分布的巴楚产胀果甘草(OK509082) 在100%的支持率下聚为一支, 根据甘草属的叶绿体DNA遗传模式主要表现为父系遗传的结论[9], 推测新疆巴楚产胀果甘草为同域分布黄甘草的父本。本研究通过甘草属关键物种的叶绿体全基因组测序和分析, 进一步验证新疆产黄甘草的杂交身份和父系来源。但在新疆和甘肃的其他地区还存在不同亲本同域分布区, 所出现的杂交类群-黄甘草在形态和遗传性状上与新疆巴楚产黄甘草不同, 需要进一步进行相关野生资源的调查和收集、测序工作, 为甘草药材资源鉴定和甘草产业化发展提供理论依据。
作者贡献: 第一作者张洁负责论文设计、实验、数据分析及论文撰写; 通讯作者陆嘉惠负责论文设计、实验材料鉴定、数据分析和论文指导; 王倩倩参与实验及数据分析; 刘梦楠、徐可参与样品采集。所有作者参与论文修改。
利益冲突: 本文所有的作者之间不存在利益冲突。
| [1] |
Zhao YK, Li L, Liu X, et al. Explore pharmacological mechanism of glycyrrhizin based on systems pharmacology[J]. China J Chin Mater Med (中国中药杂志), 2016, 41: 1916-1920. |
| [2] |
Chen R, Wang TT, Li KL, et al. Characteristics and application of immune-regulating and antiviral Chinese materia medica[J]. Chin Tradit Herb Drugs (中草药), 2020, 51: 1412-1426. |
| [3] |
Chinese Pharmacopoeia Commission. Pharmacopoeia of the People's Republic of China (中华人民共和国药典)[J]. Beijing: China Medical Science Press, 2020, 1088. |
| [4] |
Zhang XL, Li XY, Wei LJ, et al. Interspecific hybridization of Glycyrrhiza in Xinjiang[J]. Acta Bot Bor-Occid Sin (西北植物学报), 1998, 18: 132-136. |
| [5] |
Liao YH. Study on Breeding System of Two Medicinal Glycyrrhiza (两种药用甘草繁育系统的研究)[D]. Shihezi: Shihezi University, 2011.
|
| [6] |
Tian RW. Effects of Stamens Differentiations on Pollination Mechanism and Breeding System of two Glycyrrhiza Species (两种药用甘草花内雄蕊分化对传粉机制和繁育系统影响的研究)[D]. Shihezi: Shihezi University, 2013.
|
| [7] |
Xie LB, Lu JH, Li XL, et al. The cross compatibility and hybrid seed vigor among three Glycyrrhiza species[J]. Plant Diver Resour (植物分类与资源学报), 2014, 36: 342-348. |
| [8] |
Li XL, Lu JH, Xie LB, et al. Development of EST-SSR primers and genetic relationship analysis in four Glycyrrhiza L. Species[J]. Acta Bot Bor-Occid Sin (西北植物学报), 2015, 35: 480-485. |
| [9] |
Chen CN, Lu JH, Li XY, et al. Inheritance analysis and discovery of chloroplast paternal inheritance in interspecific crossing of Glycyrrhiza[J]. Guihaia (广西植物), 2017, 37: 162-168, 138. |
| [10] |
Xin Q. Hybridization Area Composition and Interspecies Introgression Glycyrrhiza L. Revealed by Transcriptome Microsatellite Marker (基于转录组SSR分子标记的甘草属植物杂交区组成及种间基因渐渗研究)[D]. Shihezi: Shihezi University, 2019.
|
| [11] |
Chen WQ, Lu JH, Wang QQ, et al. Numerica ltaxonomic study of morphological characteristics of species in Glycyrrhiza hybridzones[J]. Acta Pratac Sin (草业学报), 2020, 29: 14-26. |
| [12] |
Zhang PY, Peng ZX. Glycyrrhiza of northwest China - one of the resource plant information of northwest China[J]. J Lanzhou Univ (兰州大学学报), 1960, 6: 7-8. |
| [13] |
Ma M, Li XY. Allozyme Analysis of Glycyrrhiza inflata Complex. Chinese Society of Botany. Abstracts of the second China Licorice Symposium and the second Xinjiang Plant Resources Development, Utilization and Protection Symposium (全国第二届中国甘草学术研讨会暨第二届植物资源开发、利用与保护学术研讨会会议论文摘要集)[C]. Beijing: Chinese Society of Botany, 2004: 7.
|
| [14] |
Lu JH, Li XY, Ma M, et al. Analysis and classification of Glycyrrhiza L. plants in China by RAPD[J]. Acta Bot Bor-Occid Sin (西北植物学报), 2006, 26: 527-531. |
| [15] |
Song F. The Genetic Diversity and Introgression among Natural Populations of Glycyrrhiza L. Revealed by Start Codon Targeted (SCoT) (基于SCoT分子标记的甘草属植物自然居群的遗传多样性与基因渐渗研究)[D]. Shihezi: Shihezi University, 2017.
|
| [16] |
Palmer J, Stein D. Conservation of chloroplast genome structure among vascular plants[J]. Curr Genet, 1986, 10: 823-833. DOI:10.1007/BF00418529 |
| [17] |
Wu LW, Cui YX, Nie LP, et al. The characteristics of complete chloroplast genome sequence and phylogenetic analysis of Dendrobium moniliforme[J]. Acta Pharm Sin (药学学报), 2020, 55: 1056-1066. |
| [18] |
Wu Q, Jiang M, Chen HM, et al. Comparative analysis of three complete chloroplast genomes of Inula genus with phylogenetic analysis of 49 plants from Carduoideae[J]. Acta Pharm Sin (药学学报), 2020, 55: 1042-1049. |
| [19] |
Dong BR, Zhao ZL, Ni LH, et al. Molecular markers based upon whole chloroplast genomes and identifying alpine Gentiana waltonii and G. lhassica (Gentianaceae)[J]. Acta Pharm Sin (药学学报), 2021, 56: 2584-259. |
| [20] |
Zhang MY, Zhang YQ, Li YM, et al. Complete plastid genomes of Bupleurum chinense DC. and B. boissieuanum H. Wolff, with comparative and phylogenetic analyses of medicinal Bupleurum species[J]. Acta Pharm Sin (药学学报), 2021, 56: 618-629. |
| [21] |
Sabir J, Schwarz E, Ellison N, et al. Evolutionary and biotechnology implications of plastid genome variation in the inverted-repeat-lacking clade of legumes[J]. Plant Biotechnol J, 2014, 12: 743-754. DOI:10.1111/pbi.12179 |
| [22] |
Duan L, Zhang ZR, Deng SW, et al. The complete chloroplast genomes of rare medical herb Glycyrrhiza inflata and its relative G. aspera (Fabaceae)[J]. Mitochondrial DNA B, 2019, 4: 4083-4084. DOI:10.1080/23802359.2019.1691067 |
| [23] |
Jiang WL, Tan W, Gao H, et al. Transcriptome and complete chloroplast genome of Glycyrrhiza inflata and comparative analyses with the other two licorice species[J]. Genomics, 2020, 112: 4179-4188. DOI:10.1016/j.ygeno.2020.07.007 |
| [24] |
Lee C, Choi IS, Cardoso D, et al. The chicken or the egg? Plastome evolution and an independent loss of the inverted repeat in papilionoid legumes[J]. Plant J, 2021, 107: 861-875. DOI:10.1111/tpj.15351 |
| [25] |
Kang SH, Lee JH, Lee HO, et al. Complete chloroplast genome and 45S nrDNA sequences of the medicinal plant species Glycyrrhiza glabra and Glycyrrhiza uralensis[J]. Genes Genet Syst, 2018, 93: 83-89. DOI:10.1266/ggs.17-00002 |
| [26] |
Jia GL, Li P, Zhu Q, et al. Characterization of the complete chloroplast genome of Glycyrrhiza uralensis (Leguminosae), a traditional Chinese medicine[J]. Mitochondrial DNA B, 2019, 4: 3040-3041. DOI:10.1080/23802359.2019.1666050 |
| [27] |
Li XY, Lu JH. Taxonomy and Experimental Biology of the Genus Glycyrrhiza L (甘草属分类系统与实验生物学研究)[M]. Shanghai: Fudan University Press, 2015: 3-50.
|
| [28] |
Cui HB, Li PQ. Flora of China: Vol 42 (中国植物志第42卷)[M]. Beijing: Science Press, 1998: 168-174.
|
| [29] |
Weber JL. Informativeness of human (dC-dA)n·(dG-dT)n polymorphisms[J]. Genomics, 1990, 7: 524-530. DOI:10.1016/0888-7543(90)90195-Z |
| [30] |
Wang B, Chen HM, Ma HQ, et al. Complete plastid genome of Astragalus membranaceus (Fisch.) Bunge var. Membranaceus[J]. Mitochondrial DNA B, 2016, 1: 517-519. DOI:10.1080/23802359.2016.1197057 |
| [31] |
Qiao YG, He JX, Wang JX, et al. Analysis of chloroplast genome and its characteristics of medicinal plant Sophora flavescen[J]. Acta Pharm Sin (药学学报), 2019, 54: 2106-2112. |
| [32] |
Palmer JD, Nugent JM, Herbon LA. Unusual structure of geranium chloroplast DNA: a triple-sizedinverted repeat, extensive gene duplications, multiple inversions, and two repeat families[J]. Proc Natl Acad Sci U S A, 1987, 84: 769-773. DOI:10.1073/pnas.84.3.769 |
| [33] |
Lei WJ. Study on Chloroplast Genome of A. membranaceus (Fisch.) Bunge var. mongholicus (Bunge) P.K. (Hsiao) (蒙古黄芪叶绿体基因组研究)[D]. Taiyuan: Shanxi Agricultural University, 2016.
|
| [34] |
Huo YX, Zhao YK, Xu LW, et al. An integrated strategy for target SSR genotyping with toleration of nucleotide variations in the SSRs and flanking regions[J]. BMC Bioinform, 2021, 22: 1-14. DOI:10.1186/s12859-020-03881-z |
| [35] |
Zhang MY, Wang XF, Gao J, et al. Complete chloroplast genome of Paeonia mairei H. Lév: characterization and phylogeny[J]. Acta Pharm Sin (药学学报), 2020, 55: 168-176. |
| [36] |
Guo HJ, Liu JS, Luo L, et al. Complete chloroplast genome sequences of Schisandra chinensis: genome structure, comparative analysis, and phylogenetic relationship of basal angiosperms[J]. Sci China C (中国科学: 生命科学), 2017, 60: 1286-1290. DOI:10.1007/s11427-017-9098-5 |
| [37] |
Yang QQ, Jiang M, Wang LQ, et al. Complete chloroplast genome of Allium chinense: comparative genomic and phylogenetic analysis[J]. Acta Pharm Sin (药学学报), 2019, 54: 173-181. |
| [38] |
Tian N, Han LM, Chen C, et al. The complete chloroplast genome sequence of Epipremnum aureum and its comparative analysis among eight Araceae species[J]. PLoS One, 2018, 13: e0192956. DOI:10.1371/journal.pone.0192956 |
| [39] |
Kuang DY, Wu H, Wang TL, et al. Complete chloroplast genome sequence of Magnolia kwangsiensis (Magnoliaceae): implication for DNA barcoding and population genetics[J]. Genome, 2011, 54: 663-673. DOI:10.1139/g11-026 |
| [40] |
Ikemura T. Correlation between the abundance of Escherichia coli transfer RNAs and the occurrence of the respective codons in its protein genes: a proposal for a synonymous codon choice that is optimal for the E. coli translational system[J]. J Mol Biol, 1981, 151: 389-409. DOI:10.1016/0022-2836(81)90003-6 |
| [41] |
Yang GF, Su KL, Zhao YR, et al. Analysis of codon usage in the chloroplast genome of Medicago truncatula[J]. Acta Pratac Sin (草业学报), 2015, 24: 171-179. |
| [42] |
Kondo K, Shiba M, Yamaji HY, et al. Species identifi-cation of licorice using nr DNA and cp DNA genetic marker[J]. Biol Pharm Bull, 2007, 30: 1497-1502. DOI:10.1248/bpb.30.1497 |
| [43] |
Yang JL, Zhu ZL, Fan YJ, et al. Comparative plastomic analysis of three Bulbophyllum medicinal plants and its significance in species identification[J]. Acta Pharm Sin (药学学报), 2020, 55: 2736-2745. |
| [44] |
Tang P, Xu Q, Shen RN, et al. Phylogenetic relationship in Actinidia (Actinidiaceae) based on four noncoding chloroplast DNA sequences[J]. Plant Syst Evol, 2019, 305: 787-796. DOI:10.1007/s00606-019-01607-0 |
| [45] |
Liu YF, Li DW, Zhang Q, et al. Rapid radiations of both kiwifruit hybrid lineages and their parents shed light on a two-layer mode of species diversification[J]. New Phytol, 2017, 215: 877-890. DOI:10.1111/nph.14607 |
 2022, Vol. 57
2022, Vol. 57


