2. 中国医学科学院、北京协和医学院药用植物研究所, 国家中医药管理局中药资源保护重点研究室, 北京 100193;
3. 中药资源教育部工程研究中心, 北京 100193
2. Key Laboratory of Chinese Medicine Resources Conservation, State Administration of Traditional Chinese Medicine of the People's Republic of China, Institute of Medicinal Plant Development, Chinese Academy of Medical Sciences & Peking Union Medical College, Beijing 100193, China;
3. Engineering Research Center of Chinese Medicine Resource of Ministry of Education, Beijing 100193, China
《中国药典》收载药材大黄Rhei Radix et Rhizoma为蓼科植物掌叶大黄Rheum palmatum L.、唐古特大黄Rh. tanguticum Maxim. ex Balf.和药用大黄Rh. officinale Baill.的干燥根及根茎[1]。大黄作为药材的记载始见于东汉的《神农本草经》, 药用历史悠久[2]。现代药理研究表明大黄有抗病毒、抗肿瘤、抗炎、抗氧化应激、抗细胞凋亡和调节机体免疫等功能[3]。国家食品药品监督管理总局公开中成药配方有900余种中成药含有大黄[4]。大黄具有广泛药效作用主要与其含有大量活性成分有关, 大黄主要化学成分为蒽醌类、蒽酮类、二苯乙烯类、鞣质类等化合物[5]。不同基原的大黄药材在化学物质上存在差别[6], 在不同功效上表现出专长, 研究表明掌叶大黄专长于“清热泻火、利湿退黄”, 而唐古特大黄专长于“泻下攻积、逐瘀通经”[4]。因此大黄基原物种鉴别对于大黄临床精准用药具有重要意义。
大黄药材基原物种鉴定方法主要有性状鉴定、显微鉴定、理化鉴定及分子鉴定。大黄药材3个基原物种区别主要在于叶片分裂程度[7], 药用大黄掌状浅裂, 呈大齿形或宽三角形; 掌叶大黄叶片浅裂至半裂, 呈较窄三角形; 唐古特大黄叶深裂, 裂片狭长呈三角状披针形或狭长形[8]。性状和显微鉴定对大黄药材基原物种或药材特征的完整性要求较高, 鉴别过程需依靠长期经验积累, 且经过深加工的大黄饮片及中成药, 形态特征破坏会增加基原物种鉴定难度。理化鉴定(HPLC、UPLC等)[9, 10]依据大黄药材主要成分进行鉴定, 但操作较为繁琐。与传统鉴定方法相比, DNA分子能为物种鉴定提供丰富信息, 通过ITS2、psbA-trnH、matK等DNA条形码[11-13]可有效鉴别正品大黄与华北大黄Rh. franzenbachii Munt.、河套大黄Rh. hotaoense C. Y. Cheng et Kao等混伪品, 但难以精确到基原物种[14]。
叶绿体基因组可作为“超级条形码”进行物种鉴定[15]。绝大多数被子植物叶绿体基因组表现为典型双链环状四分体结构[16], 基因组长度通常介于110~170 kb之间[17], 其编码基因数一般为110~150个, 且在基因组成、基因含量及排列顺序上高度保守[18]。与传统相对较短并易扩增的DNA条形码相比, 叶绿体基因组具有更丰富变异位点信息和更强物种分辨率, 可更准确反应各物种遗传特性。因此本研究拟基于叶绿体基因组开发有效鉴别唐古特大黄、掌叶大黄和药用大黄的特异DNA条形码, 以期用于大黄基原植物、药材、饮片及相关中成药的基原鉴定, 为保证大黄药材临床用药安全、规范大黄药材市场及保护大黄种质资源提供依据。
材料与方法植物材料 用于叶绿体基因组测序的样品, 唐古特大黄新鲜叶片采自四川省阿坝州红原县邛溪镇, 掌叶大黄新鲜叶片采自甘肃省陇南市宕昌县理川镇, 药用大黄新鲜叶片采自四川省凉山州冕宁县冶勒乡。用于验证特异DNA条形码的样品除大黄药材3个基原物种外, 还包括食用大黄Rheum rhaponticum、波叶大黄Rh. rhabarbarum Linnaeus、苞叶大黄Rh. alexandrae Batal.、塔黄Rh. nobile Hook. f. et Thoms.及皱叶酸模Rumex crispus L.共42份, 详见表 1。样品由中国医学科学院药用植物研究所林余霖研究员鉴定, 凭证标本保存于中国医学科学院药用植物研究所。
| Table 1 Samples used to verify the specific DNA barcodes |
基因组DNA提取与测序 利用E.Z.N.A® Plant DNA kit (D3485, OMEGA, USA) 提取总DNA, 1.0%的琼脂糖凝胶电泳检测DNA质量, Qubit 3.0 (Thermo Fisher Scientific, USA) 检测DNA浓度。构建插入片段为350 bp的文库, 利用Illumina NovaSeq测序平台进行双末端测序, 双端各测150 bp。
叶绿体基因组组装、拼接与注释 利用FastQC对测序所得原始序列进行质量检测, 采用Trimmomatic软件[19]过滤去除接头序列和低质量区。使用NOVOPlasty[20]将原始读长组装成完整的叶绿体基因组。采用BWA将高质量读长比对回叶绿体基因组序列, 并在IGV中进行人工检查以确保组装无误。利用CPGAVAS软件[21]和GeSeq软件[22]对完整叶绿体基因组序列进行注释, tRNA基因用tRNAscan-SE软件[23]进行鉴定及人工校正。利用Organellar Genome DRAW (http://ogdraw.mpimp-golm.mpg.de/cgi-bin/ogdraw.pl) 在线绘制叶绿体基因组图谱。
密码子偏好性分析 采用软件CodonW (http://codonw.sourceforge.net) 分析密码子使用情况。通过计算Relative synonymous codon usage (RSCU) 获得密码子的偏好性值。密码子RSCU值等于1.00, 表示该密码子无偏好性; RSCU值大于1.00则表示该密码子使用频率较高, 反之亦然。
长重复序列 (long repeat) 和简单序列重复 (SSR) 分析 使用软件REPuter (http://bibiserv.techfak.uni-bielefeld.de/reputer/) 进行长重复序列分析, 查找正向重复F (forward)、反向重复R (reverse)、互补重复C (complement)、回文重复P (palindromic) 四种重复类型。所有重复序列识别长度不小于30 bp, 且序列相似度不小于90%。利用MISA软件(http://pgrc.ipk-gatersleben.de/misa/) 测定SSR位点类型与数目, 对于单、二、三、四、五和六核苷酸重复序列, 重复次数的最小值分别设置为10、5、4、3、3和3, 两个SSR之间最小距离设置为0 bp。
叶绿体基因组比较分析 应用在线比对工具mVISTA对测序获得的唐古特大黄、药用大黄、掌叶大黄叶绿体基因组进行全局比对分析, 利用DnaSP软件检测3条叶绿体基因组的核苷酸变异程度(Pi)。
特异DNA条形码鉴定分析 为评估叶绿体基因组所筛选高变区的实际物种鉴定效率, 以筛选的高变区序列为模板设计引物(表 2), 并利用收集的42份样本分别进行PCR扩增验证。PCR扩增采用25 μL反应混合体系, 包括2×Taq MasterMix (北京艾德莱生物科技) 12.5 μL, 上下游引物(2.5 μmol·L-1) 各1 μL, DNA模板约50 ng, 加灭菌双蒸水补足体积至25 μL。PCR程序为94 ℃, 5 min; (94 ℃, 30 s; 55 ℃, 30 s; 72 ℃, 45 s), 35个循环; 72 ℃, 10 min。扩增产物在ABI3730XL测序仪上进行Sanger测序。利用CodonCode Aligner软件对测序所得峰图文件进行序列拼接。为评估这些序列的鉴定能力, 利用MEGA 7.0软件进行序列比对、计算K2P遗传距离和构建邻接法(neighbor-joining, NJ) 系统进化树, 设置bootstrap重复值为1 000。
| Table 2 Primer pairs used to distinguish original species of Rhei Radix et Rhizoma |
唐古特大黄、药用大黄、掌叶大黄分别得到18 266 357 400、11 124 793 200、5 717 253 900 bp的原始数据, 组装拼接后的叶绿体基因组大小为161 039~161 136 bp (GenBank登录号见表 3), 为常见四分体结构, 包含一对反向重复区域IRa和IRb (30 929~30 963 bp), 一个大单拷贝区域LSC (86 426~86 523 bp) 和一个小单拷贝区域SSC (12 696~12 747 bp) (图 1)。叶绿体基因组总GC含量为37.3%~37.4%, 且GC含量在不同区域分布不均衡, IR区GC含量最高(41.1%), LSC区GC含量次之(35.3%~35.4%), SSC区GC含量最低(32.6%)。3个物种完整叶绿体基因组均编码131个基因, 包括86个蛋白质编码基因、37个tRNA基因和8个rRNA基因。3个物种叶绿体基因组的基因分布完全相同, 均发现18个具有内含子的基因, 其中15个基因包含一个内含子, 3个基因(rps12、ycf3、clpP) 包含两个内含子。在这18个含有内含子的基因中, 12个基因分布在LSC区, 4个基因(rpl2、ndhB、trnI-GAU、trnA-UGC) 分布在IR区, 1个基因(ndhA) 分布在SSC区, 还有rps12基因是一个特殊的反式剪切基因, 5′端位于LSC区, 3′端位于IR区(表 4)。
| Table 3 The basic information of chloroplast genomes of the three Rheum species |

|
Figure 1 Chloroplast genome map of the three Rheum species. The length of the chloroplast genome was shown in the circle. The different colors on the circle represented different types of genes. Represented with arrows, the transcription directions for the inner and outer genes were listed clockwise and anticlockwise, respectively |
| Table 4 Gene composition in chloroplast genomes of the three Rheum species. *Contains one intron; **Contains two introns |
根据RSCU值计算唐古特大黄、掌叶大黄和药用大黄叶绿体基因组蛋白编码基因的密码子使用模式。结果表明3个物种叶绿体基因组包含64种密码子, 其中61种密码子编码20种氨基酸, 其余3个为终止密码子。在所有氨基酸密码子中, 编码亮氨酸(Leu, L) 的密码子数最多, 半胱氨酸(Cys, C) 的密码子数最少。RSCU为 > 1的密码子共30个, 其中以A/U结尾的密码子29个。RSCU ≤ 1的密码子共34个密码子, 其中31个为G/C结尾的密码子。这些结果表明3个物种叶绿体基因组对A/U结尾密码子的偏好性高于G/C结尾密码子。编码蛋氨酸(Met, M) 和色氨酸(Trp, W) 的密码子均只有一种, RSCU值为1, 无使用偏好性。编码亮氨酸的密码子UUA和编码精氨酸(Arg, R) 的密码子CGC的RSCU值分别为最高和最低。此外, 药用大黄和掌叶大黄的叶绿体基因组密码子使用比较接近(图 2)。

|
Figure 2 Heat map of the RSCU values among the three Rheum species |
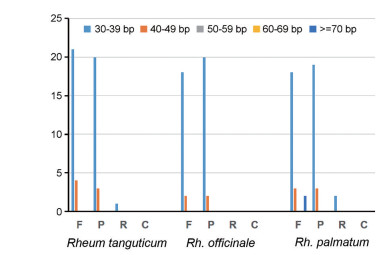
一些长度大于等于30 bp的重复序列称为长重复序列, 这些重复结构有利于叶绿体基因组重排, 增加种群遗传多样性。长重复序列数量分布结果显示, 唐古特大黄叶绿体基因组共鉴定到49个长重复序列, 包括25个正向重复F (21个30~39 bp, 4个40~49 bp)、23个回文重复P (20个30~39 bp, 3个40~49 bp)、1个反向重复R (30~39 bp); 药用大黄叶绿体基因组共鉴定到42个长重复序列, 包括20个正向重复F (18个30~39 bp, 2个40~49 bp)、22个回文重复P (20个30~39 bp, 2个40~49 bp); 掌叶大黄叶绿体基因组共鉴定47个长重复序列, 包括23个正向重复F (18个30~39 bp, 3个40~49 bp, 2个大于70 bp)、22个回文重复P (19个30~39 bp, 3个40~49 bp)、2个反向重复R (30~39 bp)。在这3个物种中, 大多数重复序列长度都在30~39 bp之间, 只有掌叶大黄叶绿体基因组存在2个长度超过70 bp的长重复序列, 均未鉴定到互补重复序列(图 3)。
|
Figure 3 Repeat sequences in the chloroplast genomes of the three Rheum species. F: Forward repeats; P: Palindrome repeats; R: Reverse repeats; C: Complement repeats |
简单重复序列(SSR), 是一段短而串联的DNA序列, 大小约为1~6 bp。SSR广泛分布于叶绿体基因组中, 通常被用作物种鉴定的重要分子标记。唐古特大黄、药用大黄及掌叶大黄分别检测到71、84、80个SSR位点, 其中单核苷酸重复序列分别为35、46、44个, 二核苷酸重复序列分别为16、18、14个, 三核苷酸重复序列分别为11、10、11个, 四核苷酸重复序列均为8个, 五核苷酸重复序列分别为1、2、3个, 未检测到六核苷酸重复序列(表 5)。重复最多的序列是单碱基重复, 且全部是A/T重复。在所有类型的SSRs中, A和T始终是最常用碱基, 叶绿体基因组SSRs通常由ployA或ployT重复序列组成, 这是叶绿体基因组A/T碱基偏好性的结果。
| Table 5 Types and amounts of SSRs in the chloroplast genomes of the three Rheum species |
为检测大黄药材3个基原物种叶绿体基因组的种间变异情况, 以注释过的掌叶大黄叶绿体基因组Rh. palmatum_MZ958818为参考, 利用mVISTA在线软件将获得的叶绿体基因组Rh. tanguticum_MZ958816、Rh. officinale_MZ958817、Rh. palmatum_MZ958818进行全局比对(图 4)。结果显示, 3条叶绿体基因组序列中非编码区变异高于保守蛋白编码区域, LSC区、SSC区变异明显大于IR区, rRNA基因(rrn4.5、rrn5、rrn16、rrn23) 高度保守几乎没有变异。基因的保守程度非常高, 绝大多数的基因相似度都在90%以上。基因间区变异大于基因区, 如rps16-trnQ、trnT-psbD、psaA-ycf3、trnT-trnL、psbE-petL、ndhF-rpl32、trnN-ycf1、ccsA-ndhD等。这些高变区可能是唐古特大黄、药用大黄和掌叶大黄物种鉴定的潜在分子标记, 具有较高研究价值。

|
Figure 4 Global alignment in the chloroplast genomes of the three Rheum species |
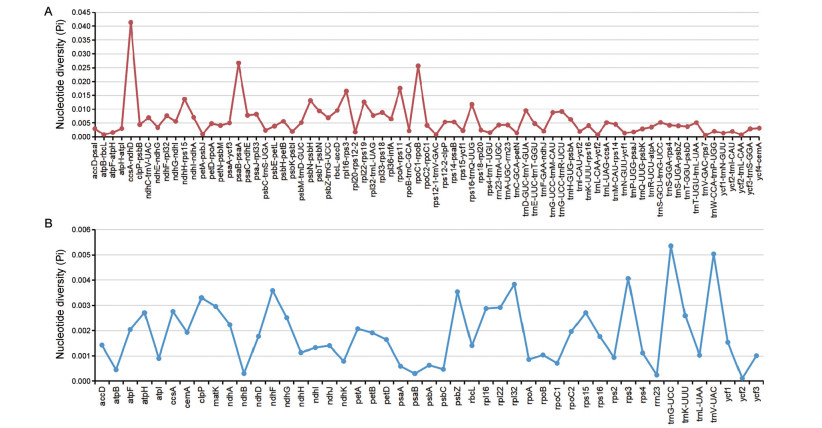
使用软件DnaSP计算3个物种叶绿体基因组所有基因和基因间区的核苷酸变异值(Pi), 进行高变区检测和序列差异水平分析。结果显示3个物种叶绿体基因组的基因间区Pi平均值(0.004) 变异大于基因Pi平均值(0.0008), 突变热点通常位于LSC区和SSC区(图 5), 与mVISTA分析结果一致。psaB-psaA (25 bp)、rpoC1-rpoB (26 bp) 等基因间区Pi值较高与自身序列长度较短有关, 因此在筛选可作为潜在分子标记的高变热点区域时, 应保证序列长度大于100 bp, 结合Pi值与mVISTA结果分析, 本研究筛选rps16-trnQ、psaA-ycf3、psbE-petL、ndhF-rpl32、trnT-trnL为高变区。
|
Figure 5 Nucleotide diversity of shared various regions with Pi > 0 in the chloroplast genomes of the three Rheum species. A: Pi values in the intergenic spacer regions; B: Pi values in the gene regions |
以样品总DNA为模板对筛选的叶绿体基因组高变区进行PCR扩增, 并对测序结果进行分析。实验结果表明基于高变区序列所设计的5对引物均可对42份样品进行有效扩增, 条带单一明亮(图 6), 1F/1R扩增区域为rps16-trnQ, 扩增长度为1 176 bp; 2F/2R扩增区域为psaA-ycf3, 扩增长度为751 bp; 3F/3R扩增区域为psbE-petL, 扩增长度为706 bp; 4F/4R扩增区域为ndhF-rpl32, 扩增长度为600 bp; 5F/5R扩增区域为trnT-trnL, 扩增长度为630 bp。根据MEGA 7.0计算测序所获高质量序列的种内及种间K2P距离, 结果显示种间最小遗传距离大于种内最大遗传距离, 不存在重叠, 表明所筛选片段符合DNA条形码的要求(表 6)。NJ系统进化树分析证实唐古特大黄单独聚为一支, 药用大黄单独聚为一支, 掌叶大黄单独聚为一支(图 7)。因此rps16-trnQ、psaA-ycf3、psbE-petL、ndhF-rpl32、trnT-trnL可以作为特异DNA条形码用于大黄药材3个基原物种的鉴定。psaA-ycf3、ndhF-rpl32、trnT-trnL的NJ系统发育树显示唐古特大黄PS2904MT01~PS2904MT06单独聚为一支, 采集地点为四川红原; PS2904MT07~PS2904MT12单独聚为一支, 采集地点为青海林川, 表明psaA-ycf3、ndhF-rpl32与trnT-trnL可作为潜在特异DNA条形码用于同一物种不同产区的鉴定。

|
Figure 6 Agarose gel electrophoresis of five specific DNA barcodes of the original species of Rhei Radix et Rhizoma. a: rps16-trnQ; b: psaA-ycf3; c: psbE-petL; d: ndhF-rpl32; e: trnT-trnL. M: DL2000 DNA marker; 0: Negative control; 1: Rheum officinale_PS2902MT01; 2: Rh. palmatum_PS2903MT01; 3: Rh. tanguticum_PS2904MT01; 4: Rh. tanguticum_PS2904MT07; 5: Rh. rhaponticum_PS2905MT01; 6: Rh. rhabarbarum_PS2906MT01; 7: Rh. alexandrae_PS2907MT01; 8: Rh. nobile_PS2908MT01; 9: Rumex crispus_PS2909MT01 |
| Table 6 Intra/Inter-specific K2P distance of five specific DNA barcodes of the original species of Rhei Radix et Rhizoma |

|
Figure 7 NJ phylogenetic tree based on five specific DNA barcodes of the original species of Rhei Radix et Rhizoma. The bootstrap scores (1 000 replicates) are shown (≥50%) for each branch |
长期以来大黄药材基原物种分类与鉴定主要依据外观性状, 但地理和生态因素可能引起大黄药材基原植物不同物种间形态特征的持续变化[13], 因而开发大黄药材3个物种的特异DNA条形码具有巨大价值。采用Illumina高通量测序和比较基因组学对大黄药材3个基原物种进行叶绿体基因组分析, 结果显示叶绿体基因组可作为超级条形码准确鉴定唐古特大黄、药用大黄和掌叶大黄。叶绿体基因组的一些高变异区序列可作为物种鉴定及系统发育关系分析等相关研究的特异DNA条形码, 本研究在唐古特大黄、药用大黄和掌叶大黄叶绿体高变基因间区筛选到5条序列, 对42份样品进行PCR实验验证, 扩增效率高, 扩增片段长度为600~1 200 bp, Sanger测序后所得高质量序列利用MEGA软件计算K2P距离和构建NJ系统进化树, 结果证明所筛选的片段rps16-trnQ、psaA-ycf3、psbE-petL、ndhF-rpl32与trnT-trnL能作为特异DNA条形码鉴定大黄药材三个基原物种。
尽管已有近百篇研究基于叶绿体基因组筛选高变区尝试解决一些近缘物种的鉴定问题, 但这些研究都均未进行实验验证。如刘昶等[24]对川贝母、丹参等53种重要药用植物叶绿体基因组图谱及应用进行详细阐述, 并利用ecoPrimers软件基于叶绿体基因组设计能够区分贝母属、葱属、芍药属及百合属等物种的DNA条形码序列, 可作为潜在分子标记开发区域。武立伟等[25]分析检测出细茎石斛复合种叶绿体基因组中的trnF-trnV、ndhF-rpl32等高变异区。杨嘉鹏等[26]推荐石豆兰属植物叶绿体基因组高变区psbI-trnS、psbC-trnS、clpP-ex1-psbB、psaJ-rpl33、rpl33-rps18组合用于设计鉴定石豆兰属药用植物的专属DNA条形码。另外, 在中国知网仅检索到10余篇文献对于叶绿体基因组所筛选高变区进行实验验证, 如董博然等[27]基于叶绿体基因组筛选trnT-trnL和ycf1两个高变区, 91份样品实验验证后构建长梗秦艽、全萼秦艽的特异性引物PCR两步鉴定法, 方法易于操作且准确性高。崔英贤[28]对筛选的高变区atpH-atpI设计两对特异性引物, 但扩增仍不成功, 表明该区段扩增效率低, 并不适合于豆蔻属分子标记开发; accD-psaI和ycf4-cemA作为阳春砂、绿壳砂和海南砂的潜在分子标记, 但其实验所获序列构建的NJ树各分支支持率普遍较低, 鉴定效率不理想。由此可见, 对所筛选高变区进行实验验证的研究非常少, 结果不尽理想。综合文献研究, 建议在筛选出高变区域后, 应对实际扩增效率进行实验验证, 对所筛选高变区的物种鉴定能力进行确定。本研究对大黄药材3个基原物种叶绿体基因组进行测序分析, 开发特异DNA条形码并进行PCR验证, 从K2P遗传距离和NJ进化树两个方面评价所筛选高变区实际鉴定效率, 表明所筛选5个特异DNA条形码可以有效实施大黄基原物种鉴定, 为大黄基原物种分类鉴定奠定基础。
研究表明唐古特大黄、药用大黄及掌叶大黄在地理分布上出现重叠与替换[29], ISSR分析发现3个物种展示出丰富的遗传多样性[30, 31]。采用核基因组序列ITS的研究结果表明唐古特大黄存在种内遗传多样性[32], 同一物种在不同地区间遗传物质有所差异。针对高变区设计的5对引物扩增片段长度为600~1 200 bp, 旨在筛选包含丰富变异位点的特异DNA条形码, NJ进化树结果证明四川与青海两处的唐古特大黄各自聚为一支, 表明所筛选特异DNA条形码可以用于大黄基原植物同一物种不同产区的分类鉴定。本研究实验采集对象为大黄药材基原植物, 基因组DNA较为完整, 对所筛选片段可进行有效扩增。DNA条形码在中药全产业链各关键环节发挥重要作用[33], 因此可采集不同产区的大黄药材基原植物样本, 基于本文筛选的5个特异DNA条形码, 进一步丰富大黄药材三个基原物种的遗传变异信息, 用于市售饮片与相关中成药的分子鉴定, 为保证大黄药材临床用药安全、规范大黄药材市场提供新依据。
致谢: 感谢中国中医科学院中药研究所孙伟研究员提供苞叶大黄和塔黄样品。
作者贡献: 李冉郡负责实验工作、数据分析、文章撰写与修改; 武立伟负责数据分析; 辛天怡负责样本收集及稿件修改; 廖海参与样本收集及稿件修改; 林余霖负责基原植物性状鉴定; 姚辉参与数据分析; 周嘉裕和宋经元负责实验设计、总体把关和稿件修改等工作。
利益冲突: 本文的研究内容无任何利益冲突。
| [1] |
State Pharmacopoeia Committee. Pharmacopoeia of the People's Republic of China (中华人民共和国药典)[S]. 2020 Ed. Part I. Beijing: China Medical Science Press, 2020: 24.
|
| [2] |
Chen JQ, Chen YY, Tang YP, et al. Multifunctional regularity of Rhei Radix et Rhizoma in ancient and modern medicine[J]. Chin Tradit Herb Drugs (中草药), 2019, 50: 1485-1492. |
| [3] |
Xu LH, Huang XJ, Li YR, et al. Effect and potential mechanism of rhubarb on COVID-19 based on the "wenbingzaixiaqiyure" theory[J]. Pharmacol Clin Chin Mater Med (中药药理与临床), 2020, 36: 85-90. |
| [4] |
Chen YY, Tang YP, Chen JQ, et al. Research progress and utilization strategy on resource chemistry of Rhei Radix et Rhizoma[J]. Chin Tradit Herb Drugs (中草药), 2018, 49: 5170-5178. |
| [5] |
Wang Y, Yang X, Xia P, et al. Research progress on chemical composition and pharmacological effects of Rhei Radix et Rhizoma and predictive analysis on quality markers[J]. Chin Tradit Herb Drugs (中草药), 2019, 50: 4821-4837. |
| [6] |
Komatsu K, Nagayama Y, Tanaka K, et al. Comparative study of chemical constituents of rhubarb from different origins[J]. Chem Pharm Bull (Tokyo), 2006, 54: 1491-1499. DOI:10.1248/cpb.54.1491 |
| [7] |
Wang XM, Hou XQ, Zhang YQ, et al. Morphological variation in leaf dissection of Rheum palmatum complex (Polygonaceae)[J]. PLoS One, 2014, 9: e110760. DOI:10.1371/journal.pone.0110760 |
| [8] |
Lin YL, Chen SL. Illustrated Handbook of Chinese Medicinal Plants (中国药用植物原色图鉴)[M]. Fuzhou: Fujian Science and Technology Press, 2016.
|
| [9] |
Ge JH, Liu XH, Xu H, et al. Identification of different varieties of Rhei Radix et Rhizoma based on chemical analysis[J]. China J Chin Mater Med (中国中药杂志), 2015, 40: 2309-2313. |
| [10] |
Du QT, Wen JL, Yan YS, et al. UPLC fingerprint of Rhei Radix et Rhizoma from different habitats[J]. J Chin Med Mater (中药材), 2013, 36: 725-731. |
| [11] |
Li MN, Han RL, Han JP, et al. Identification of original plants of Radix et Rhizoma Rhei from its adulterants Rhizoma et Radix Polygoni Cuspidati and Radix Rumicis Obtusifolii by ITS2 sequences[J]. Global Tradit Chin Med (环球中医药), 2012, 5: 185-189. |
| [12] |
Song JY, Yao H, Li Y, et al. Authentication of the family Polygonaceae in Chinese pharmacopoeia by DNA barcoding technique[J]. J Ethnopharmacol, 2009, 124: 434-439. DOI:10.1016/j.jep.2009.05.042 |
| [13] |
Zhang XQ, Liu CS, Yan XL, et al. Sequence analysis and identification of a chloroplast matK gene in Rhei Rhizoma from different botanical origins[J]. Acta Pharm Sin (药学学报), 2013, 48: 1722-1728. |
| [14] |
Li RJ, Xin TY, Song LK, et al. Research progress in original species identification in industry chain of Rhei Radix et Rhizoma[J]. China J Chin Mater Med (中国中药杂志), 2021, 46: 1060-1066. |
| [15] |
Kane NC, Cronk Q. Botany without borders: barcoding in focus[J]. Mol Ecol, 2008, 17: 5175-5176. DOI:10.1111/j.1365-294X.2008.03972.x |
| [16] |
Jansen RK, Raubeson LA, Boore JL, et al. Methods for obtaining and analyzing whole chloroplast genome sequences[J]. Methods Enzymol, 2005, 395: 348-384. |
| [17] |
Zhu AD, Guo WH, Gupta S, et al. Evolutionary dynamics of the plastid inverted repeat: the effects of expansion, contraction, and loss on substitution rates[J]. New Phytol, 2016, 209: 1747-1756. DOI:10.1111/nph.13743 |
| [18] |
Green BR. Chloroplast genomes of photosynthetic eukaryotes[J]. Plant J, 2011, 66: 34-44. DOI:10.1111/j.1365-313X.2011.04541.x |
| [19] |
Bolger AM, Lohse M, Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data[J]. Bioinformatics, 2014, 30: 2114-2120. DOI:10.1093/bioinformatics/btu170 |
| [20] |
Dierckxsens N, Mardulyn P, Smits G. Unraveling heteroplasmy patterns with NOVOPlasty[J]. NAR Genom Bioinform, 2019, 2: lqz011. |
| [21] |
Liu C, Shi LC, Zhu YJ, et al. CpGAVAS, an integrated web server for the annotation, visualization, analysis, and GenBank submission of completely sequenced chloroplast genome sequences[J]. BMC Genomics, 2012, 13: 715. DOI:10.1186/1471-2164-13-715 |
| [22] |
Tillich M, Lehwark P, Pellizzer T, et al. GeSeq-versatile and accurate annotation of organelle genomes[J]. Nucleic Acids Res, 2017, 45: W6-11. DOI:10.1093/nar/gkx391 |
| [23] |
Schattner P, Brooks AN, Lowe TM. The tRNAscan-SE, snoscan and snoGPS web servers for the detection of tRNAs and snoRNAs[J]. Nucleic Acids Res, 2005, 33: W686-689. DOI:10.1093/nar/gki366 |
| [24] |
Liu C, Huang LF. Chloroplast Genome Illustrated Handbook of Chinese Medicinal Plants (中国药用植物叶绿体基因组图谱第一册)[M]. Beijing: Science Press, 2020.
|
| [25] |
Wu LW, Cui YX, Nie LP, et al. The characteristics of complete chloroplast genome sequence and phylogenetic analysis of Dendrobium moniliforme[J]. Acta Pharm Sin (药学学报), 2020, 55: 1056-1066. |
| [26] |
Yang JP, Zhu ZL, Fan YJ, et al. Comparative plastomic analysis of three Bulbophyllum medicinal plants and its significance in species identification[J]. Acta Pharm Sin (药学学报), 2020, 55: 2736-2745. |
| [27] |
Dong BR, Zhao ZL, Ni LH, et al. Molecular markers based upon whole chloroplast genomes and identifying alpine Gentiana waltonii and G. lhassica (Gentianaceae)[J]. Acta Pharm Sin (药学学报), 2021, 56: 2584-2591. |
| [28] |
Cui YX. Structural Analysis of Complete Chloroplast Genome of Medicinal and Edible Original Plants of Amomum, Chinese Wolfberry, Hawthorn and Ginger (药食两用药材砂仁、枸杞、山楂和姜基原植物叶绿体基因组结构解析)[D]. Beijing: Peking Union Medical College, 2020.
|
| [29] |
Wang XM, Hou XQ, Zhang YQ, et al. Distribution pattern of genuine species of rhubarb as traditional Chinese medicine[J]. J Med Plants Res, 2010, 4: 1865-1876. |
| [30] |
Wang XM, Yang R, Feng SF, et al. Genetic variation in Rheum palmatum and Rheum tanguticum (Polygonaceae), two medicinally and endemic species in China using ISSR markers[J]. PLoS One, 2012, 7: e51667. DOI:10.1371/journal.pone.0051667 |
| [31] |
Wang XM, Hou XQ, Zhang YQ, et al. Genetic diversity of the endemic and medicinally important plant Rheum officinale as revealed by Inter-Simpe Sequence Repeat (ISSR) markers[J]. Int J Mol Sci, 2012, 13: 3900-3915. DOI:10.3390/ijms13033900 |
| [32] |
Ma XC, Xie CX, Guan M, et al. High levels of genetic diversity within one population of Rheum tanguticum on the Qinghai-Tibet Plateau have implications for germplasm conservation[J]. Pharm Crops, 2014, 5: 1-8. DOI:10.2174/2210290601405010001 |
| [33] |
Lou Q, Xin TY, Song JY. Application of DNA barcoding technology in the whole industrial chain of traditional Chinese medicine[J]. Acta Pharm Sin (药学学报), 2020, 55: 1784-1791. |
 2022, Vol. 57
2022, Vol. 57


