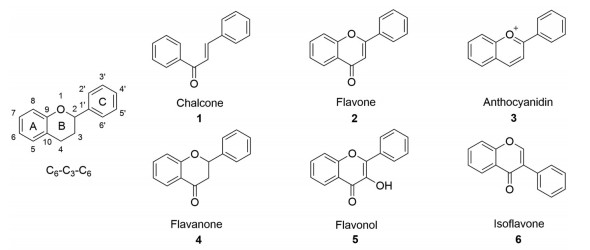
黄酮类化合物(flavonoids) 是广泛分布于植物中的一大类次级代谢产物。目前已表征结构的黄酮类化合物超过10 000种[1], 其基本母体是由两个具有酚羟基的苯环(A环和B环) 通过3个碳原子相互连接而成的C6-C3-C6单元。其中, C3可为脂链, 也可与C6部分形成五元或六元氧杂环。根据C3部分是否成环及其氧化程度、取代方式等差异, 黄酮类化合物又可分为查尔酮类(chalcones, 1)、黄酮类(flavones, 2)、花青素类(anthocyanidins, 3)、二氢黄酮类(flavanones, 4)、黄酮醇类(flavonols, 5)、异黄酮类(isoflavones, 6) 等多个亚类[2] (图 1)。
|
Figure 1 The chemical structures of main sub-classes of flavonoids[2] |
黄酮类化合物具有多种生物学活性。作为植物次级代谢产物之一, 其在植物生长发育过程中具有重要作用, 研究发现: 豆科植物产生的异黄酮类化合物可诱导结瘤基因表达从而参与调节植物-根瘤菌共生过程[3]; 花青素作为天然色素, 可使花瓣及种皮呈现不同颜色, 从而吸引授粉昆虫以帮助植物授粉及传播种子等[4]。同时, 黄酮类化合物还具有显著的药理活性, 如二氢黄酮类化合物柚皮素(naringenin) 具有抗炎、抗癌活性[5]; 黄酮醇类化合物山柰酚(kaempferol) 具有抗焦虑、抗过敏[6]活性; 异黄酮类化合物染料木素(genistein) 对前列腺癌、乳腺癌等癌症的发生发展具有抑制作用[7]等。
近年来, 黄酮类化合物由于在医药、农业等领域的应用不断拓展, 市场需求不断扩大, 其可持续性生产制造问题引起了广泛关注。传统上, 黄酮类化合物主要通过植物提取法获得; 然而在植物中, 黄酮类化合物含量较低、分离困难、成本高, 且过度采伐对物种和生态环境可能造成不可逆的破坏[8]。另一方面, 黄酮类化合物也可通过化学全合成或半合成的方法进行生产制备, 但存在毒性试剂如硝酸铊的使用、反应条件难以控制等不足[9, 10], 成本高且环境不友好。因此, 尚需要寻找其他更优的生产途径以大量、稳定、绿色可持续地供应活性黄酮类化合物。
随着各种组学、生物信息学、分子生物学等相关领域基础理论及技术的发展, 药用天然产物生物合成和合成生物学领域的研究不断取得新突破, 为其获取提供了新模式[11]。通过在大肠杆菌(Escherichia coli) 或酿酒酵母(Saccharomyces cerevisiae) 等微生物细胞中异源重构生物合成途径来生产具有高附加值的目标化合物, 并通过优化基因元器件及其适配性、调节相关代谢通路等方法来提高其产量已成为可能。与传统的植物提取法和化学合成法相比, 这种经人工设计构建获得的可按照预设线路生成目标化合物的“细胞工厂(cell factory)”拥有成本低、耗时短、环境友好等优势, 已成为该领域的研究热点[12]; 同时, “造物致知, 造物致用”, 具有理论和应用价值。目前, 药用天然产物细胞工厂相关研究有诸多报道, 如: 经设计优化的产白藜芦醇(resveratrol) 大肠杆菌细胞工厂[13], 在外源添加对香豆酸时, 产量可达到2.3 g·L-1; 抗疟药物青蒿素(artemisinin) 的前体青蒿酸(artemisinic acid) 的酿酒酵母细胞工厂产量已达到25 g·L-1 [14]; 在人参皂苷类(ginsenosides) 化合物合成生物学研究方面, 以高产前体原人参二醇(protopanaxadiol, PPD) 的酿酒酵母作为底盘细胞, 经改造优化可得到产人参皂苷Rh2细胞工厂, 产量达到2.2 g·L-1 [15]。相比之下, 黄酮类化合物细胞工厂的最高产量仅达1.2 g·L-1 [16]。
由于黄酮类化合物种类繁多, 构建细胞工厂的方式方法也各有不同, 为了全面地了解黄酮类化合物细胞工厂的研究进展, 本文首先概述了黄酮类化合物的生物合成过程, 随之介绍了二氢黄酮、黄酮、黄酮醇、异黄酮等代表性黄酮类化合物的合成生物学研究进展, 以期为该领域的研究提供参考。
1 黄酮类化合物的生物合成黄酮类化合物虽然结构多样, 但其基本骨架的早期形成过程是相同的, 即经莽草酸-聚酮途径杂合衍生而来, 随后经多种修饰反应生成不同的衍生物。以下将分别从黄酮类化合物前体、骨架的生物合成以及后修饰三个阶段对黄酮类化合物的生物合成过程进行简单介绍。
1.1 黄酮类化合物前体的生物合成黄酮类化合物有两种生物合成起始前体, 分别是经聚酮途径生成的丙二酸单酰辅酶A (malonyl-CoA) 以及经莽草酸-苯丙素途径生成的4-香豆酰辅酶A (4-coumaryl coenzyme A)。
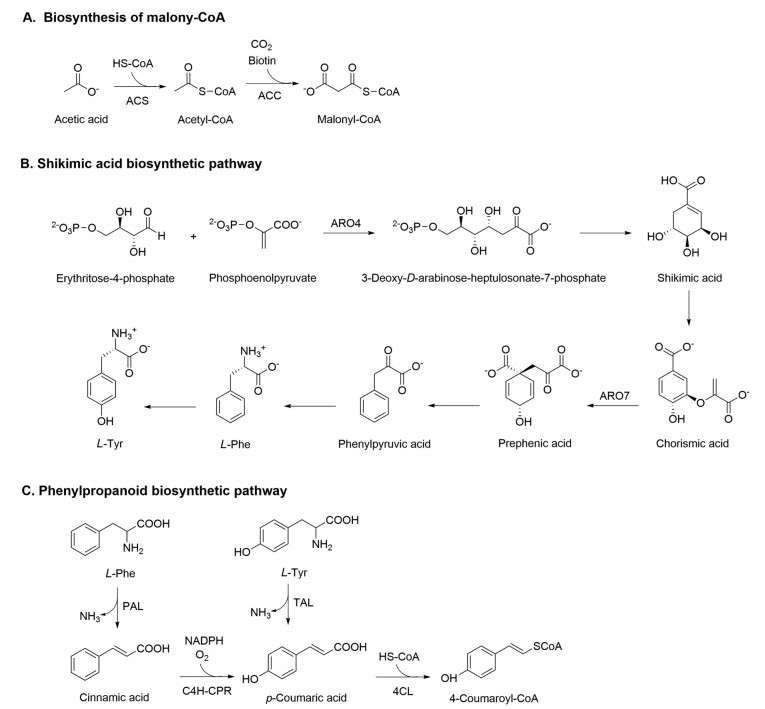
在生物体内, 经糖酵解途径生成的或由乙酸在乙酰辅酶A合酶(acetyl-CoA synthase, ACS) 催化下生成的乙酰辅酶A (acetyl-CoA) 在辅酶生物素(biotin) 的参与下, 由乙酰辅酶A羧化酶(acetyl-CoA carboxylase, ACC) 催化生成丙二酸单酰辅酶A[2] (图 2A)。该化合物可经不同的酶促反应参与多种细胞生命过程, 尤其是在脂肪酸类化合物的生物合成过程中可作为重要的碳链延伸单元参与代谢。由于细胞内大部分丙二酸单酰辅酶A用于脂肪酸合成, 因此, 在对以其为前体的目标化合物进行异源途径重构时, 可通过下调脂肪酸合成代谢通量来重新分配丙二酸单酰辅酶A的代谢流, 从而提高目标化合物的产量。
|
Figure 2 The upstream biosynthetic pathways of flavonoids [2]. A: Biosynthesis of malony-CoA. ACS: Acetyl-CoA synthase; ACC: Acetyl-CoA carboxylase; B: Shikimic acid biosynthetic pathway. ARO4: 3-Deoxy-D-arabinose-heptulosonate-7-phosphate (DAHP) synthase; ARO7: Chorismate mutase; C: Phenylpropanoid biosynthetic pathway. PAL: Phenylalanine ammonia-lyase; TAL: Tyrosine ammonia-lyase; C4H: Cinnamate-4-hydroxylase; CPR: Cytochrome P450 reductase; 4CL: 4-Coumarate: coenzyme A ligase |
4-香豆酰辅酶A的生物合成按先后顺序可分为莽草酸途径(shikimic acid pathway) 和苯丙素途径(phenylpropanoid pathway) 两个阶段。植物体内的葡萄糖(glucose) 先在莽草酸途径相关酶的催化下, 依次经3-脱氧-D-阿拉伯糖-庚酮酸-7-磷酸(3-deoxy-D-arabinose-heptulosonate-7-phosphate, DAHP)、预苯酸(prephenic acid) 等重要中间体, 转化生成芳香族氨基酸(aromatic amino acid), 如L-苯丙氨酸(L-Phe)、L-酪氨酸(L-Tyr) 等[17] (图 2B)。其中, 负责催化生成DAHP的DAHP合酶以及负责催化生成预苯酸的分支酸变位酶(chorismate mutase) 可受到莽草酸途径终产物L-Tyr的负反馈调节, 是莽草酸途径的重要限速步骤, 也是常用的代谢改造靶点。
随后, 生成的芳香族氨基酸进入苯丙素代谢途径(图 2C): L-Phe可在苯丙氨酸解氨酶(phenylalanine ammonia-lyase, PAL) 的催化下生成反式肉桂酸(trans-cinnamic acid)[18], 随即经CYP450酶系的肉桂酸-4-羟化酶(cinnamate-4-hydroxylase, C4H) 催化, 消耗NADPH, 生成对香豆酸(p-coumaric acid)[19]。上述两步反应也可由酪氨酸解氨酶(tyrosine ammonia-lyase, TAL) 以L-Tyr为底物, 经一步酶促反应完成[20]。继而, 对香豆酸可在4-香豆酸: 辅酶A连接酶(4-coumarate: coenzyme A ligase, 4CL) 作用下生成活化形式4-香豆酰辅酶A [21], 参与下游代谢。研究表明, L-Phe代谢过程在植物中更为普遍, 但其生成的中间体肉桂酸可诱导PAL失活, 且抑制PAL编码基因的转录; 同时, 该途径还涉及常被认为是黄酮类化合物生物合成途径限速酶之一的C4H[22]。因此, 在异源重构黄酮类化合物生物合成途径时, 常采用TAL直接催化底物L-Tyr反应生成对香豆酸的策略避免生成具有负反馈调节作用的中间体肉桂酸、规避C4H限速反应, 同时通过减少通路涉及的酶数量来降低工程菌的代谢负担[23]。
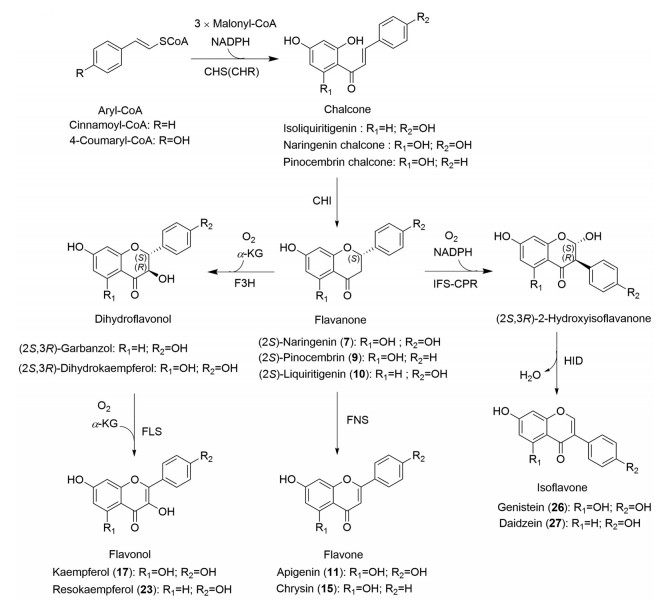
1.2 黄酮类化合物骨架的生物合成查尔酮合酶(chalcone synthase, CHS) 是Ⅲ型聚酮合酶家族(polyketone synthase Ⅲ, PKS Ⅲ) 成员之一, 也是黄酮类化合物生物合成途径中的特征性酶。该酶可催化1分子4-香豆酰辅酶A与3分子丙二酸单酰辅酶A生成查尔酮类化合物柚皮素查尔酮(naringenin chalcone) [24]; 相似地, 也可催化反式肉桂酰辅酶A与丙二酸单酰辅酶A生成松属素查尔酮(pinocembrin chalcone)[25]。此外, 在异甘草素(isoliquiritigenin, 与柚皮素查尔酮的结构相比, C-6′无羟基) 的生物合成过程中, 该反应除了CHS外, 还需要查尔酮还原酶(chalcone reductase, CHR) 协同催化完成[26, 27] (图 3)。随即, 查尔酮类化合物可由具有立体选择性的查尔酮异构酶(chalcone isomerase, CHI) 催化生成具有光学活性的(2S)-二氢黄酮。其中, CHI根据作用底物不同, 可分成Ⅰ型和Ⅱ型。这两类CHI均能催化6′-羟基查尔酮生成(2S)-5-羟基二氢黄酮, 如催化柚皮素查尔酮生成(2S)-柚皮素(naringenin), 但只有Ⅱ型CHI才可催化6′-脱氧查尔酮生成(2S)-5-脱氧二氢黄酮, 如催化异甘草素生成(2S)-甘草素(liquiritigenin)[28] (图 3)。
|
Figure 3 Biosynthetic pathways of representative flavonoids. CHS: Chalcone synthase; CHR: Chalcone reductase; CHI: Chalcone isomerase; IFS: Isoflavone synthase; CPR: Cytochrome P450 reductase; HID: 2-Hydroxyisoflavanone dehydratase; FNS: Flavone synthase; F3H: Flavanone-3-hydroxylase; FLS: Flavonol synthase; α-KG: α-Ketoglutaric acid |
二氢黄酮作为黄酮类化合物生物合成过程中的重要中间体, 可经多种反应生成其他黄酮亚类(图 3), 主要包括: ①黄酮类化合物: 如柚皮素、松属素(pinocembrin) 在黄酮合酶(flavone synthase, FNS) 催化下, C-2/C-3单键去饱和, 分别生成芹菜素(apigenin)、白杨素(chrysin)[29]; ②黄酮醇类化合物: 如柚皮素在二氢黄酮-3-羟化酶(flavanone-3β-hydroxylase, F3H) 和黄酮醇合酶(flavonol synthase, FLS) 的相继作用下, 经二氢山柰酚(dihydrokaempferol) 生成黄酮醇类化合物山柰酚(kaempferol)[30, 31]; ③异黄酮类化合物: 如甘草素、柚皮素在CYP450酶系的异黄酮合酶(isoflavone synthase, IFS) 和细胞色素P450还原酶(cytochrome P450 reductase, CPR) 的协同作用下, 发生芳基迁移反应, 生成不稳定的2-羟基二氢异黄酮类化合物(2-hydroxyisoflavanones), 随即又在2-羟基二氢异黄酮脱水酶(2-hydroxyisoflavanone dehydratase, HID) 催化下脱水, 生成相应的异黄酮类化合物大豆苷元(daidzein)、染料木素[32, 33]。
1.3 黄酮类化合物的后修饰经上述过程形成各类黄酮类化合物基本骨架后, 可经甲基化、羟基化、糖基化、异戊烯基化等后修饰反应, 生成结构多样的衍生物。例如, 芹菜素经7-O-甲基化修饰生成芫花素(genkwanin)[34]; 芹菜素、松属素在CYP450家族的黄酮-6-羟化酶(flavone 6-hydroxylase, F6H) 的催化下, 在C-6位引入羟基, 分别生成野黄芩素(scutellarein) 和黄芩素(baicalein)[35]; 柚皮素在CYP450家族的二氢黄酮-3′-羟化酶(flavonoid-3′-hydroxylase, F3′H) 的催化下, C-3′发生羟基化形成圣草酚(eriodictyol)[36]; 同属于P450酶家族的黄酮3′-单加氧酶(flavonoid 3′-monooxygenase, FMO) 协同CPR可催化山奈酚、5-脱氧山柰酚(resokaempferol) 发生羟基化反应, 分别生成槲皮素(quercetin)、非瑟酮(fisetin)[37]; 大豆苷元可在具有区域选择性的8-C-糖基转移酶(8-C-glycosyltransferase, 8-CGT) 的催化下生成葛根素(puerarin)[38], 也可在7-O-糖基转移酶(7-O-glycosyl-transferase, 7-OGT) 的催化下生成大豆苷(daidzin)[39]; SfFPT可区域选择性地催化多种黄酮类化合物在C-8位发生异戊烯基化反应[40]; GuILDT可催化查尔酮类化合物进行C-3′异戊烯基化反应[41]; GuA6DT则可特异性地催化黄酮类化合物的C-6异戊烯基化反应[42]; 来自桑科植物桑(Morus alba) 和柘(Cudrania tricuspidata) 的异戊烯基转移酶MaIDT、CtIDT具有区域选择性, 可催化查尔酮类化合物在C-3′、异黄酮和黄酮类化合物在C-6发生异戊烯基化反应[43]等。此外, 部分衍生物需经过多步后修饰反应生成, 如: 山柰酚先在8-异戊烯基转移酶(8-prenyltransferase) 催化下生成8-异戊烯基山柰酚(8-prenylkaempferol, 8P-KAE), 进一步经3-O-鼠李糖基转移酶催化生成宝藿苷Ⅱ (baohuoside Ⅱ); 或经4′-O-甲基转移酶(4′-O-methyltransferase) 催化生成淫羊藿素(icaritin), 淫羊藿素经3-O-鼠李糖基转移酶、7-O-葡萄糖基转移酶相继催化生成淫羊藿苷(icariin)[44-46]等。
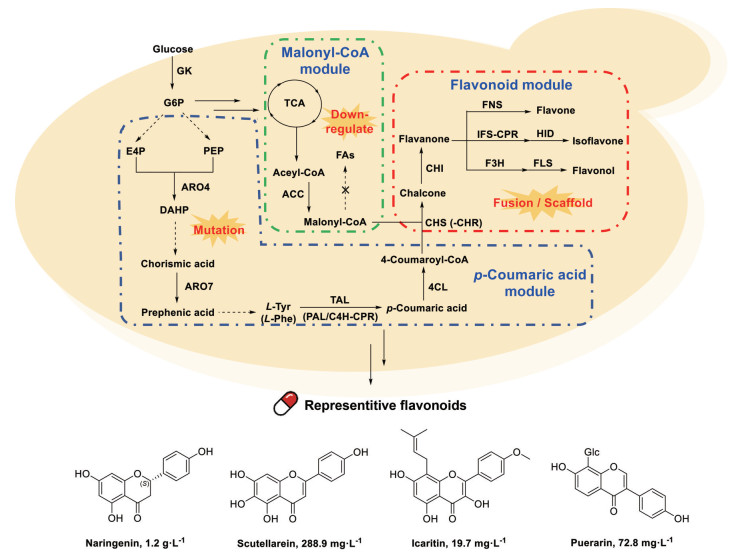
2 黄酮类化合物的合成生物学研究进展过去几十年间, 黄酮类化合物生物合成相关基因的鉴定和功能表征有诸多报道, 生物合成途径已基本阐明, 为开展合成生物学研究奠定了基础(图 4)。目前药用天然产物合成生物学研究的主要思路为: 在微生物细胞中异源表达生物合成基因, 构建得到目标化合物的生物全合成或半合成工程菌, 并在此基础上, 通过基因工程、代谢工程相关方法进行优化改造以提高产量, 如: ①通过增加关键基因拷贝数、进行密码子优化等手段来提高酶的异源表达水平; ②采用定点突变、利用linker或蛋白质-多肽相互作用构建酶复合体、尝试不同基因元器件组合等方法来提高酶的催化活性、催化效率及适配性等。除此之外, 产黄酮类化合物工程菌代谢瓶颈还可通过调节代谢流, 即增强底物供应、削弱竞争途径等方式进行优化。其中, 增强底物供应主要针对莽草酸-苯丙素途径的L-Tyr和聚酮途径的丙二酸单酰辅酶A, 前者常采用过表达DAHP合酶、分支酸变位酶的突变体来解除L-Tyr对其上游通路产生的负反馈抑制作用, 从而提高L-Tyr的合成代谢通量; 后者可通过过表达ACC和ACS、醛脱氢酶(aldehyde dehydrogenase, ALD)、醇脱氢酶(alcohol dehydrogenase, ADH) 等丙二酸单酰辅酶A、乙酰辅酶A的生物合成基因来增加其供应; 而削弱竞争途径主要是指通过敲除脂肪酸合成途径中某些关键酶的编码基因或下调其表达水平, 来降低脂肪酸合成的代谢通量, 使乙酰辅酶A、丙二酸单酰辅酶A等重要底物更多地流向目标途径, 进而使下游目标化合物的产量增加。另外, 优化发酵条件如温度、pH值、培养基的碳源种类、底物投料比等, 也有助于提高目标化合物的产量。
|
Figure 4 Biosynthesis of flavonoids in microbial cell factory[16-37]. GK: Glucokinase; ARO4: 3-Deoxy-D-arabinose-heptulosonate-7-phosphate (DAHP) synthase; ARO7: Chorismate mutase; TAL: Tyrosine ammonia-lyase; PAL: Phenylalanine ammonia-lyase; C4H: Cinnamate-4-hydroxylase; CPR: Cytochrome P450 reductase; 4CL: 4-Coumarate: coenzyme A ligase; CHS: Chalcone synthase; CHR: Chalcone reductase; CHI: Chalcone isomerase; FNS: Flavone synthase; IFS: Isoflavone synthase; HID: 2-Hydroxyisoflavanone dehydratase; F3H: Flavanone-3-hydroxylase; FLS: Flavonol synthase; TCA: Tricarboxylic acid cycle; FAs: Fatty acid derivatives |
近来, 随着各种组学、生物信息学、分子生物学等相关领域的基础理论及技术的不断更新, 黄酮类化合物合成生物学研究亦得以创新和推进。在底盘细胞选择方面, 除了大肠杆菌和酿酒酵母等传统微生物外, 不断挖掘新型微生物作为基因改造平台, 其中以产油酵母中的解脂耶氏酵母(Yarrowia lipolytica) 研究较多。这种酵母细胞具有丰富的膜结构, 可产生大量脂质; 其乙酰辅酶A、丙二酸单酰辅酶A、ATP等多种重要前体的代谢通量高于常用的酿酒酵母[47], 有利于黄酮类化合物生物合成。同时, 由于在单一宿主细胞中重构较长的异源生物合成途径可能存在多个基因之间适配性差、工程菌代谢负担过大等问题, 导致细胞生长受到抑制、目标化合物产量降低。对此, 有人提出“微生物共培养(microbial co-culture)”的概念, 即将目标化合物生物合成途径人为分成两个或多个生物合成模块, 分别构建到不同的宿主细胞中, 得到相应工程菌后进行共培养发酵, 由此高效获得目标化合物[48]。这种共培养策略也常用于构建产黄酮类化合物工程菌。
在发酵起始底物方面, 除了外源饲喂L-Tyr或对香豆酸等苯丙酸类底物进行生物半合成外, 近年多项研究对莽草酸途径进行了优化改造, 使黄酮类化合物以葡萄糖为底物进行从头合成成为可能。这些工作主要采用过表达DAHP合酶和分支酸变位酶突变体等方式降低两者对可产生负反馈抑制作用的下游产物L-Tyr的敏感性, 从而增加L-Tyr、L-Phe的供应, 以此构建得到高产对香豆酸工程菌, 产量达到每升克级水平[49, 50], 为下游生物合成提供较充足的底物, 从而提高目标黄酮类化合物的产量。
基于此, 下文将分述二氢黄酮、黄酮、黄酮醇、异黄酮4种类型中代表性化合物的合成生物学研究进展。
2.1 二氢黄酮二氢黄酮如柚皮素、圣草酚、松属素和甘草素等(图 5) 是黄酮类化合物重要亚类, 具有多种药理活性, 同时也是许多其他类型的黄酮类化合物生物合成中间体。因此, 二氢黄酮类化合物的合成生物学研究具有关键意义, 引起了广泛关注。
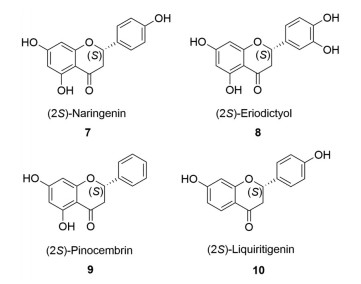
|
Figure 5 The chemical structures of representative flavanones |
柚皮素(naringenin, 7) 作为多种黄酮类化合物生物合成的共同中间体, 其合成生物学研究较早且报道较多, 以下主要从起始底物、底盘细胞两方面对其进行介绍。
在发酵起始底物方面, 通过外源饲喂L-Tyr或对香豆酸等苯丙酸类底物进行生物半合成可避开葡萄糖到L-Tyr的限速转化过程, 构建获得的产柚皮素工程菌产量相对更高, 如: Gao等[51]通过在酿酒酵母中异源表达柚皮素生物合成基因而构建获得的工程菌, 在外源添加2.5 g·L-1对香豆酸时, 柚皮素产量达648.6 mg·L-1; 随后, Gao等[16]在产柚皮素工程酿酒酵母基础上, 采用优化启动子、生物反应器发酵条件等方法, 在外源饲喂对香豆酸时, 检测到柚皮素产量达1.2 g·L-1, 这是目前报道的柚皮素细胞工厂的最高产量。相比之下, 以葡萄糖为底物从头合成柚皮素的工程菌由于涉及的酶促反应更多, 工程菌的代谢负担更大、转化过程中能量和中间体损耗更多, 其柚皮素产量通常低于半合成法。Zhou等[52]通过过表达DAHP合酶、分支酸变位酶的突变体ARO4K229L和ARO7G141, 同时敲除一个抑制子编码基因tyrR等方法对可从头合成柚皮素的工程大肠杆菌进行改造, 后又经发酵条件优化, 检测到柚皮素产量最高达588 mg·L-1。Koopman等[53]以酿酒酵母为底盘细胞, 通过过表达ARO3、ARO4的突变体以及提高chs、tal的拷贝数来增加底物代谢通量, 敲除苯丙酮酸脱羧酶(phenylpyruvate decarboxylase, ARO10) 和丙酮酸脱羧酶(pyruvate decarboxylase, PDC) 5、PDC6的编码基因来抑制竞争通路活性, 最终检测到优化后的工程酿酒酵母的柚皮素从头合成产量达400 μmol·L-1 (108 mg·L-1)。Lyu等[54]在上述过表达柚皮素生物合成基因、敲除竞争性苯乙醇通路相关基因的基础上, 主要采用优化培养基碳源、添加表面活性剂Tween 80等方法提高产量, 在最优条件下, 构建得到可从头合成柚皮素的工程酿酒酵母的产量达220 mg·L-1。
在底盘细胞的选择方面, 一些研究成功构建了产柚皮素的细胞共培养体系, 如: Ganesan等[55]构建的产柚皮素大肠杆菌共培养体系经调节接种比例等优化改造后, 柚皮素产量达41.5 mg·L-1; Zhang等[56]构建了一个可催化D-木糖(D-xylose) 生成柚皮素的大肠杆菌-酿酒酵母共培养体系, 主要通过基因组整合兼用高拷贝质粒的方法提高限速酶Pc4CL的表达量, 同时调节接种量和接种比例对该体系进行优化, 检测到柚皮素终产量为21.2 mg·L-1。这种共培养策略虽然在理论上可减轻细胞代谢负担, 增加目标化合物产量, 而且可以更加灵活地通过改变不同工程菌的接种比例等方式来协调上下游的代谢不平衡问题, 但在具体实例中发现了一些新的难点: 共培养体系中不同菌株的最适生物合成条件可能不同, 在单一培养条件下难以达到最佳产量; 中间体在菌株之间的转运难以控制等, 尚需要更多研究与探索。此外, 另一些研究尝试以解脂耶氏酵母为底盘细胞进行柚皮素生物合成途径的异源重构及优化, 如Lv等[57]构建了可从头合成柚皮素的工程解脂耶氏酵母, 同时又过表达了acc、acs等可进一步增加丙二酸单酰辅酶A供应的编码基因, 调整了chs、cpr的基因拷贝数, 检测到柚皮素产量达252.4 mg·L-1; Palmer等[58]通过联合β-氧化相关优化策略提高了乙二胺四乙酸的代谢通量, 由此获得的工程解脂耶氏酵母在生物反应器条件下的柚皮素从头合成产量达898 mg·L-1。
2.1.2 圣草酚和松属素圣草酚(eriodictyol, 8) 细胞工厂相关研究表明: 经基因改造得到的可从头合成圣草酚的工程解脂耶氏酵母[57]和未经优化的工程酿酒酵母[59]的圣草酚产量可分别达到134.2、152 mg·L-1。Leonard等[60]构建了一个可催化生成多种二氢黄酮类化合物的工程大肠杆菌, 经丙二酸单酰辅酶A代谢通量优化后, 在外源添加乙酸、肉桂酸时, 其松属素(pinocembrin, 9) 产量可达到429 mg·L-1; 随后, 通过进一步抑制脂肪酸合成通路[61], 松属素产量可提高到710 mg·L-1; Wu等[62]采用CRISPER/Cas9技术增加了产松属素工程大肠杆菌中丙二酸单酰辅酶A的代谢通量, 并采用了pH阶段性调控策略, 检测到松属素从头合成产量达到525.8 mg·L-1。
2.1.3 甘草素如前所述, 甘草素(liquiritigenin, 10) 前体异甘草素的生物合成需要CHS和CHR协同催化完成。相较于上述二氢黄酮类化合物, 目前报道的甘草素细胞工厂产量较低, 且通常伴有副产物柚皮素的生成, 这可能是由CHR的底物特异性及其需要与CHS协同发挥催化作用导致。因此, 甘草素细胞工厂的代谢改造难点除提高产量外, 还需控制副产物的产生。Yan等[63]在2007年采用来自苜蓿(Medicago sativa) 的mschr以及其他相关生物合成基因(pc4cl2、phchs、mschi), 成功构建了产甘草素工程酿酒酵母, 该菌在添加1.5 mmol·L-1对香豆酸时可生成13.5 mg·L-1甘草素, 但副产物柚皮素的产量达118.9 mg·L-1。Rodriguez等[64]在高产对香豆酸工程酿酒酵母基础上选用来自蒙古黄芪(Astragalus mongholicus) 的amchr以及与上述报道[63]相同的其他相关生物合成基因, 首次获得了可从头合成甘草素的工程菌, 产量为5.3 mg·L-1, 而副产物柚皮素产量约为1 mg·L-1。Stahlhut等[65]为了提高CHS反应中间体到CHR催化活性位点的转运效率, 将删除了终止密码子的phchs的C端通过柔性linker与amchr的N端相连, 获得可表达CHS-CHR融合蛋白(fusion protein) 的产甘草素工程大肠杆菌, 在外源添加L-Tyr的条件下, 该菌的甘草素产量比独立表达CHS、CHR时约提高4倍, 达到1.8 mg·L-1, 而副产物柚皮素产量为0.1 mg·L-1。2019年, Akram等[66]采用丙二酸单酰辅酶A代谢通量更高的解脂耶氏酵母作为底盘细胞, 选用zmpal、pc4cl2、phchs、mschr、mschi等基因, 综合运用调节基因元件组合搭配等策略, 构建了可从头合成甘草素的工程菌, 并在生物反应器发酵条件下检测到产量达62.4 mg·L-1, 这是目前报道的甘草素细胞工厂的最高产量。因此, 在甘草素生物合成途径的异源重构中, 构建CHS-CHR融合蛋白、选择适宜的底盘细胞可能是提高产量、减少副产物的有效策略。
2.2 黄酮目前, 黄酮类化合物的合成生物学研究报道主要集中于芹菜素(apigenin, 11) 衍生物(图 6), 如芫花素(genkwanin, 12)、黄芩素(baicalein, 13)、野黄芩素(scutellarein, 14) 等。
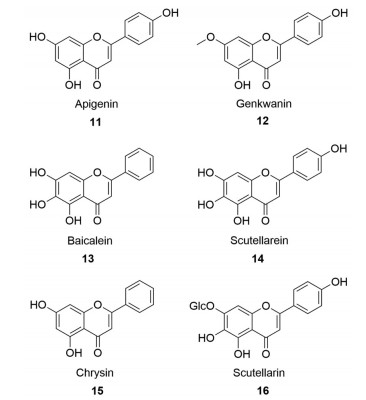
|
Figure 6 The chemical structures of representative flavones |
芫花素是中药芫花(Daphne genkwa) 中主要的非糖基化黄酮类化合物, 由芹菜素经7-O-甲基化衍生而来。研究表明, 芫花素具有抗炎、抗肿瘤活性, 可作为直肠癌的潜在治疗药物[67]。Lee等[68]构建的产芹菜素工程大肠杆菌, 在添加对香豆酸时可合成30 mg·L-1芹菜素; 在此基础上, 通过增加L-Tyr的生物合成通量并引入具有区域选择性的7-O-甲基转移酶(7-O-methyltransferases), 构建获得可从头合成芫花素的工程菌, 产量达41 mg·L-1, 同时芹菜素产量达55 mg·L-1。
2.2.2 野黄芩素和黄芩素野黄芩素和黄芩素(图 6) 是中药黄芩(Scutellaria baicalensis) 中的主要活性化合物, 具有抗癌、抗氧化、促进细胞凋亡等药理活性, 近年相关的合成生物学研究报道较多。Li等[69]选用rtpal、pc4cl、phchs、mschi、pcfnsⅠ、sbf6h等基因, 构建了一个可产黄芩素和野黄芩素的工程大肠杆菌, 经丙二酸单酰辅酶A代谢通量优化, 该工程菌能以L-Phe、L-Tyr、丙二酸为底物合成芹菜素、白杨素(chrysin, 15)、野黄芩素和黄芩素, 产量分别达到109.7、29.2、106.5和23.6 mg·L-1。在此基础上, 他们利用一对蛋白质-多肽结构域, PSD95/Dlg1/zo-1(PDZ) 及其配体, 分别采用刚性linker (ER/K)、柔性linker (GGGGS)2与选定的rtpal的C端、pc4cl的N端相连, 使这两个酶在表达后可通过蛋白质-多肽相互作用进行自组装形成双酶复合物, 以提高目标化合物的转化效率。经进一步优化, 在分批发酵条件下检测到黄芩素及野黄芩素的产量分别达271.6、288.9 mg·L-1 [70, 71]。
灯盏花素(breviscapine) 是中药灯盏花(Erigeron breviscapus) 的总黄酮提取物, 已在临床作为处方药物用于心脑血管疾病治疗, 其主要成分是野黄芩苷(scutellarin, 16, 也称灯盏花乙素) 和少量的芹菜素-7-O-葡糖醛酸苷(apigenin-7-O-glucuronide, 也称灯盏花甲素)。2018年, Liu等[72]从灯盏花中鉴定得到两个参与灯盏花素生物合成的关键酶——黄酮-7-O-葡糖醛酸基转移酶(flavonoid-7-O-glucuronosyltransferase, F7GAT) 和黄酮-6-羟化酶(flavone-6-hydroxylase, F6H), 阐明了灯盏花素的生物合成途径, 并在酿酒酵母细胞中完成了途径重构, 实现了野黄芩苷及芹菜素-7-O-葡糖醛酸苷的从头合成。随后, 通过增加上述工程菌的乙酰辅酶A代谢通量, 野黄芩苷及芹菜素-7-O-葡糖醛酸苷的产量分别提高到108、185 mg·L-1。
2.3 黄酮醇黄酮醇类化合物的合成生物学研究报道以山柰酚(kaempferol, 17) 及其衍生物为主(图 7)。尤其近年对传统中草药淫羊藿(Epimedium) 中的活性黄酮醇类化合物的生物合成和合成生物学研究取得了较大进展, 简述如下。
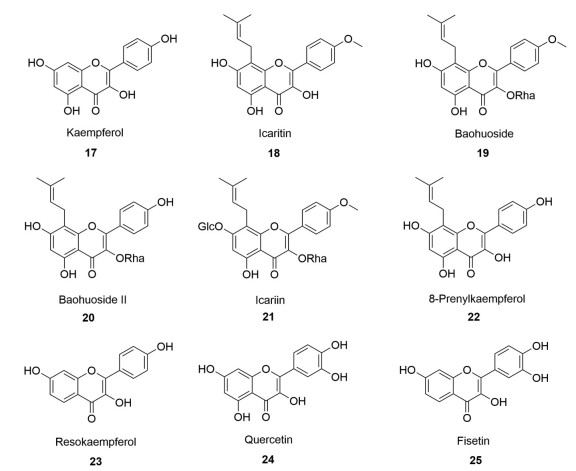
|
Figure 7 The chemical structures of representative flavonols |
山柰酚具有镇痛、抗焦虑、抗过敏、抗肿瘤等多种药理活性。Rodriguez等[64]通过在高产对香豆酸的工程酿酒酵母中重构对香豆酸到山柰酚的生物合成途径, 实现了山柰酚的从头合成, 产量达26.6 mg·L-1。2017年, Duan等[73]在酿酒酵母中重构了山柰酚的从头生物合成途径并提高了丙二酸单酰辅酶A的代谢通量, 检测到该工程菌的山柰酚产量达8.6 mg·L-1; 而后在分批饲喂对香豆酸的条件下, 山柰酚产量提高到66.3 mg·L-1。Lyu等[54]通过调节培养基的碳源组成、增加底物磷酸烯醇式丙酮酸(phosphoenolpyruvic acid, PEP) 供应等策略优化产山柰酚工程菌的发酵条件, 山柰酚产量达到86 mg·L-1。
淫羊藿是应用历史悠久的传统中草药, 具有抗炎、抗氧化、治疗骨质疏松等多种药理作用, 其主要活性成分是以山柰酚为母核的黄酮醇类衍生物, 如淫羊藿素(icaritin, 18)、宝藿苷(baohuoside, 19)、宝藿苷Ⅱ (baohuoside Ⅱ, 20)、淫羊藿苷(icariin, 21) 等(图 7)。2018年, Feng等[44]从拟巫山淫羊藿(E. pseudowushanense) 中鉴定得到一个具有底物特异性的3-O-鼠李糖基转移酶(3-O-rhamnosyltransferase) EpPF3RT和一个UDP-鼠李糖合酶EpRhS, 并以此构建了一个可催化8-异戊烯基山柰酚(8-prenylkaempferol, 8P-KAE, 22) 转化生成宝藿苷Ⅱ的工程大肠杆菌, 产量为96.7 μg·mL-1, 转化率达100%。次年, Feng等[45]又鉴定了一个具有糖基供体杂泛性的7-O-葡萄糖基转移酶Es7GT, 该酶可催化宝藿苷的7-OH发生葡萄糖基化反应, 生成淫羊藿苷, 以此构建得到的工程大肠杆菌的淫羊藿苷产量达到10.1 μg·mL-1, 转化率为11.7%。另外, Wang等[46]从箭叶淫羊藿(E. sagittatum) 中鉴定了一个可催化山柰酚生成8P-KAE的异戊烯基转移酶EsPT2, 结合源自大豆(Glycine max) 的一个可催化8P-KAE进行4′-O-甲基化反应的O-甲基转移酶GmOMT2, 以酿酒酵母为底盘细胞, 通过同时引入酪氨酸和苯丙氨酸途径、提高关键基因拷贝数、下调竞争性通路的代谢通量等方法, 构建并优化得到产山柰酚工程酿酒酵母, 产量达到151.5 mg·L-1; 继而在此工程菌中引入N端截除了30个氨基酸的EsPT2, 同时过表达7个甲羟戊酸途径(mevalonate pathway) 相关酶以提高异戊烯基供体DMAPP的供应, 在酿酒酵母中实现了8P-KAE的从头合成, 产量达到25.9 mg·L-1。在此基础上, 鉴于GmOMT2的最适pH高于胞质pH, 作者采用两种策略来解决GmOMT2的pH适配性问题: ①在GmOMT2的N端添加信号肽, 使其定位至pH高于胞质的线粒体中, 有效发挥其催化功能; ②将淫羊藿素的生物合成途径分成两个模块, 即在酿酒酵母中完成葡萄糖到8P-KAE的生物合成, 在大肠杆菌中表达GmOMT2以完成8P-KAE到淫羊藿素的转化, 最后通过共培养的方式实现淫羊藿素的从头合成, 产量分别达到7.2、19.7 mg·L-1。
2.3.2 槲皮素和非瑟酮山柰酚和5-脱氧山柰酚(resokaempferol, 23) 的C-3′经羟基化可分别衍生成槲皮素(quercetin, 24) 和非瑟酮(fisetin, 25), 这两种化合物在自然界中分布广泛, 具有抗炎、抗氧化、神经保护等多种药理活性。Rodriguez等[64]在酿酒酵母中重构了槲皮素、非瑟酮的从头生物合成途径, 并将FMO-CPR以融合蛋白的方式进行表达, 检测到工程菌的槲皮素、非瑟酮产量分别达到20.4、1.6 mg·L-1; 随后又通过增加CHS、CHR的基因拷贝数等方法优化非瑟酮途径, 其产量提高到2.3 mg·L-1。此外, Stahlhut等[65]以大肠杆菌为底盘细胞, 通过构建融合蛋白表达框、截去不溶性蛋白的跨膜区等优化策略得到可表达CHS-CHR融合蛋白以及可溶性增强的缩短FMO的产非瑟酮工程菌, 在添加L-Tyr时, 产量达0.3 mg·L-1。Du等[74]构建了一个由柚皮素合成模块和柚皮素消耗模块组成的工程酿酒酵母共培养体系, 经72 h分批饲喂发酵, 该体系的山柰酚、槲皮素产量分别达到168.1、154.2 mg·L-1。
2.4 异黄酮异黄酮类化合物主要分布在豆科植物中, 由于结构与人类雌激素相似, 有植物雌激素(phytoestrogen) 之称, 在临床上可用于缓解围绝经期症状, 如防治骨质疏松、潮热等[75]。常见的异黄酮类化合物主要有染料木素(genistein, 26)、大豆苷元(daidzein, 27) 及其衍生物(图 8) 等。
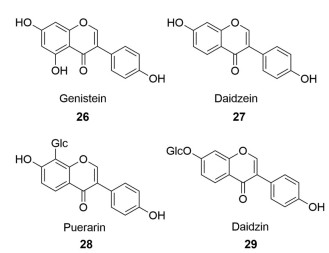
|
Figure 8 The chemical structures of representative isoflavones |
2009年, Kim等[76]将tpifs和oscpr的跨膜区截除, 并采用GST (Gly-Ser-Thr) 作为linker将两者构建成融合蛋白表达框, 由此得到相应的工程大肠杆菌可催化柚皮素生成染料木素, 转化率达75%。随后, 他们在此基础上构建了一个工程大肠杆菌共培养体系[77], 经细胞密度、温度、pH值等发酵条件优化, 该体系以柚皮素、对香豆酸为底物时可分别合成染料木素35、18.6 mg·L-1。此外, Horinouchi等[78]采用可将L-Tyr转化为柚皮素的工程大肠杆菌与可在半乳糖诱导条件下表达GeIFS的酿酒酵母构建成大肠杆菌-酿酒酵母共培养体系, 在外源添加L-Tyr时, 染料木素产量达6 mg·L-1。
2.4.2 大豆苷元Wang等[38]在野葛(Pueraria lobata) 转录组中鉴定得到对异黄酮类化合物具有底物特异性的8-C-糖基转移酶PlUGT43, 该酶可催化大豆苷元生成葛根素(puerarin, 28), 以此构建得到的PlUGT43-转基因大豆发根的葛根素产量达到0.6 mg·L-1。Leonard等[79]通过在大肠杆菌中表达截去跨膜区并经信号肽修饰的IFS-CPR融合蛋白而得到的工程菌可成功催化底物甘草素生成大豆苷元, 产量达到18 mg·g-1细胞干重(dry cell weight, DCW)。2021年, Liu等[80]综合应用生物合成途径重构、底物代谢通量增强、竞争性通路下调等多种合成生物学技术和优化策略, 构建得到首个可从头合成异黄酮类衍生物的酿酒酵母细胞工厂; 在摇瓶条件下, 该工程菌的大豆苷元、葛根素、大豆苷(daidzin, 29) 产量分别达到85.4、72.8、73.2 mg·L-1。这项工作不仅大大推进了大豆苷元相关的合成生物学研究, 而且对异黄酮类化合物的细胞工厂构建和优化具有重要参考价值。
3 总结与展望近年来, 黄酮类化合物的合成生物学研究取得较大进展, 但相比于青蒿酸、人参皂苷等化合物, 其细胞工厂产量仍较低。除了柚皮素、圣草酚等二氢黄酮类化合物的产量可超过500 mg·L-1外, 其他类型黄酮类化合物细胞工厂最高产量仅为100 mg·L-1左右, 且随着催化反应步骤增多, 产量相应降低, 尤其是需要CYP450酶系参与合成的黄酮醇类、异黄酮类化合物等, 产量则更低(表 1)。这可能是由黄酮类化合物的生物合成途径与其他多种代谢通路共享生物合成前体、某些生物合成关键酶在异源微生物中表达不佳、活性不强以及各元器件之间适配性较差等因素所致, 尚需要进一步探究。
| Table 1 Metabolic engineering of flavonoids in microorganisms. a: The scale of fermentation; BR: Bioreactor; FLK: Shake flask; PLT: 96/24-well plates; b: 18 mg·g-1 dry cell weight (DCW) |
高通量检测、计算机辅助设计等方法技术的开发应用推动合成生物学进入快速、高效、智能化的发展轨道。采用合成生物学策略构建细胞工厂是高效生产药用化学成分的新模式, 黄酮类化合物的生物合成途径解析和重构相对较易, 而如何提高产量是实现其高效可持续工业化生产的主要瓶颈问题, 通过代谢工程、酶工程、发酵工程、人工智能等多学科交叉合作有可能解决这一难题。
作者贡献: 林春草负责本综述的文献检索、稿件撰写与修改; 陈大伟负责指导本综述的稿件修改; 戴均贵负责指导本综述的稿件撰写与修改。
利益冲突: 本文无利益冲突。
| [1] |
Dixon RA, Pasinetti GM. Flavonoids and isoflavonoids: from plant biology to agriculture and neuroscience[J]. Plant Physiol, 2010, 154: 453-457. DOI:10.1104/pp.110.161430 |
| [2] |
Walsh CT, Tang Y. Natural Product Biosynthesis: Chemical Logic and Enzymatic Machinery[M]. London: The Royal Society of Chemistry, 2020.
|
| [3] |
Novák K, Lisá L, Skrdleta V. Rhizobial nod gene-inducing activity in pea nodulation mutants: dissociation of nodulation and flavonoid response[J]. Physiol Plant, 2004, 120: 546-555. DOI:10.1111/j.0031-9317.2004.0278.x |
| [4] |
Harborne JB, Williams CA. Advances in flavonoid research since 1992[J]. Phytochemistry, 2000, 55: 481-504. DOI:10.1016/S0031-9422(00)00235-1 |
| [5] |
Salehi B, Fokou PVT, Sharifi-Rad M, et al. The therapeutic potential of naringenin: a review of clinical trials[J]. Pharmaceuticals, 2019, 12: 11. |
| [6] |
Chen AY, Chen YC. A review of the dietary flavonoid, kaempferol on human health and cancer chemoprevention[J]. Food Chem, 2013, 138: 2099-2107. DOI:10.1016/j.foodchem.2012.11.139 |
| [7] |
Ko KP. Isoflavones: chemistry, analysis, functions and effects on health and cancer[J]. Asian Pac J Cancer Prev, 2014, 15: 7001-7010. DOI:10.7314/APJCP.2014.15.17.7001 |
| [8] |
Lee MH, Lin CC. Comparison of techniques for extraction of isoflavones from the root of Radix Puerariae: ultrasonic and pressurized solvent extractions[J]. Food Chem, 2007, 105: 223-228. DOI:10.1016/j.foodchem.2006.11.009 |
| [9] |
Lee DYW, Zhang WY, Karnati VVR. Total synthesis of puerarin, an isoflavone C-glycoside[J]. Tetrahedron Lett, 2003, 44: 6857-6859. DOI:10.1016/S0040-4039(03)01715-5 |
| [10] |
Wang YC, Chen S, Yu O. Metabolic engineering of flavonoids in plants and microorganisms[J]. Appl Microbiol Biotechnol, 2011, 91: 949-956. DOI:10.1007/s00253-011-3449-2 |
| [11] |
Liu XY, Luo LL, Ma Y, et al. Biopathway construction of plant natural products[J]. Acta Pharm Sin (药学学报), 2021, 56: 3285-3299. |
| [12] |
Ko YS, Kim JW, Lee JA, et al. Tools and strategies of systems metabolic engineering for the development of microbial cell factories for chemical production[J]. Chem Soc Rev, 2020, 49: 4615-4636. DOI:10.1039/D0CS00155D |
| [13] |
Lim CG, Fowler ZL, Hueller T, et al. High-yield resveratrol production in engineered Escherichia coli[J]. Appl Environ Microbiol, 2011, 77: 3451-3460. DOI:10.1128/AEM.02186-10 |
| [14] |
Paddon CJ, Westfall PJ, Pitera DJ, et al. High-level semi-synthetic production of the potent antimalarial artemisinin[J]. Nature, 2013, 496: 528-532. DOI:10.1038/nature12051 |
| [15] |
Wang PP, Wei W, Ye W, et al. Synthesizing ginsenoside Rh2 in Saccharomyces cerevisiae cell factory at high-efficiency[J]. Cell Discov, 2019, 5: 5. DOI:10.1038/s41421-018-0075-5 |
| [16] |
Gao S, Zhou HR, Zhou JW, et al. Promoter-library-based pathway optimization for efficient (2S)-naringenin production from p-coumaric acid in Saccharomyces cerevisiae[J]. J Agric Food Chem, 2020, 68: 6884-6891. DOI:10.1021/acs.jafc.0c01130 |
| [17] |
Wu JJ, Du GC, Zhou JW, et al. Metabolic engineering of Escherichia coli for (2S)-pinocembrin production from glucose by a modular metabolic strategy[J]. Metab Eng, 2013, 16: 48-55. DOI:10.1016/j.ymben.2012.11.009 |
| [18] |
Zhu QL, Xie XR, Lin HX, et al. Isolation and functional characterization of a phenylalanine ammonia-lyase gene (SsPAL1) from Coleus (Solenostemon scutellarioides (L.) Codd)[J]. Molecules, 2015, 20: 16833-16851. DOI:10.3390/molecules200916833 |
| [19] |
Wang ZW, Jian XY, Zhao YC, et al. Functional characterization of cinnamate 4-hydroxylase from Helianthus annuus Linn using a fusion protein method[J]. Gene, 2020, 758: 144950. DOI:10.1016/j.gene.2020.144950 |
| [20] |
Jendresen CB, Stahlhut SG, Li MJ, et al. Highly active and specific tyrosine ammonia-lyases from diverse origins enable enhanced production of aromatic compounds in bacteria and Saccharomyces cerevisiae[J]. Appl Environ Microbiol, 2015, 81: 4458-4476. DOI:10.1128/AEM.00405-15 |
| [21] |
Li ZB, Li CF, Li J, et al. Molecular cloning and functional characterization of two divergent 4-coumarate: coenzyme A ligases from kudzu (Pueraria lobata)[J]. Biol Pharm Bull, 2014, 37: 113-122. DOI:10.1248/bpb.b13-00633 |
| [22] |
Trantas E, Panopoulos N, Ververidis F. Metabolic engineering of the complete pathway leading to heterologous biosynthesis of various flavonoids and stilbenoids in Saccharomyces cerevisiae[J]. Metab Eng, 2009, 11: 355-366. DOI:10.1016/j.ymben.2009.07.004 |
| [23] |
Li MJ, Kildegaard KR, Chen Y, et al. De novo production of resveratrol from glucose or ethanol by engineered Saccharomyces cerevisiae[J]. Metab Eng, 2015, 32: 1-11. DOI:10.1016/j.ymben.2015.08.007 |
| [24] |
Nakajima O, Akiyama T, Hakamatsuka T, et al. Isolation, sequence and bacterial expression of a cDNA for chalcone synthase from the cultured cells of Pueraria lobata[J]. Chem Pharm Bull, 1991, 39: 1911-1913. DOI:10.1248/cpb.39.1911 |
| [25] |
Leonard E, Lim KH, Saw PN, et al. Engineering central metabolic pathways for high-level flavonoid production in Escherichia coli[J]. Appl Environ Microbiol, 2007, 73: 3877-3886. DOI:10.1128/AEM.00200-07 |
| [26] |
Mameda R, Waki T, Kawai Y, et al. Involvement of chalcone reductase in the soybean isoflavone metabolon: identification of GmCHR5, which interacts with 2-hydroxyisoflavanone synthase[J]. Plant J, 2018, 96: 56-74. DOI:10.1111/tpj.14014 |
| [27] |
Ayabe S, Udagawa A, Furuya T. NAD(P)H-dependent 6'-deoxychalcone synthase activity in Glycyrrhiza echinata cells induced by yeast extract[J]. Arch Biochem Biophys, 1988, 261: 458-462. DOI:10.1016/0003-9861(88)90362-1 |
| [28] |
Ralston L, Subramanian S, Matsuno M, et al. Partial reconstruction of flavonoid and isoflavonoid biosynthesis in yeast using soybean type I and type II chalcone isomerases[J]. Plant Physiol, 2005, 137: 1375-1388. DOI:10.1104/pp.104.054502 |
| [29] |
Zhao Q, Zhang Y, Wang G, et al. A specialized flavone biosynthetic pathway has evolved in the medicinal plant, Scutellaria baicalensis[J]. Sci Adv, 2016, 2: e1501780. DOI:10.1126/sciadv.1501780 |
| [30] |
Prescott AG, Stamford NPJ, Wheeler G, et al. In vitro properties of a recombinant flavonol synthase from Arabidopsis thaliana[J]. Phytochemistry, 2002, 60: 589-593. DOI:10.1016/S0031-9422(02)00155-3 |
| [31] |
Salanoubat M, Lemcke K, Rieger M, et al. Sequence and analysis of chromosome 3 of the plant Arabidopsis thaliana[J]. Nature, 2000, 408: 820-822. DOI:10.1038/35048706 |
| [32] |
Jung W, Yu O, Lau SM, et al. Identification and expression of isoflavone synthase, the key enzyme for biosynthesis of isoflavones in legumes[J]. Nat Biotechnol, 2000, 18: 208-212. DOI:10.1038/72671 |
| [33] |
Akashi T, Aoki T, Ayabe S. Molecular and biochemical characterization of 2-hydroxyisoflavanone dehydratase. Involvement of carboxylesterase-like proteins in leguminous isoflavone biosynthesis[J]. Plant Physiol, 2005, 137: 882-891. DOI:10.1104/pp.104.056747 |
| [34] |
Kim BG, Kim H, Hur HG, et al. Regioselectivity of 7-O-methyltransferase of poplar to flavones[J]. J Biotechnol, 2006, 126: 241-247. DOI:10.1016/j.jbiotec.2006.04.019 |
| [35] |
Zhao Q, Cui MY, Levsh O, et al. Two CYP82D enzymes function as flavone hydroxylases in the biosynthesis of root-specific 4'-deoxyflavones in Scutellaria baicalensis[J]. Mol Plant, 2018, 11: 135-148. DOI:10.1016/j.molp.2017.08.009 |
| [36] |
Amor ILB, Hehn A, Guedone E, et al. Biotransformation of naringenin to eriodictyol by Saccharomyces cerevisiea functionally expressing flavonoid 3'-hydroxylase[J]. Nat Prod Commun, 2010, 5: 1893-1898. |
| [37] |
Thill J, Miosic S, Gotame TP, et al. Differential expression of flavonoid 3'-hydroxylase during fruit development establishes the different B-ring hydroxylation patterns of flavonoids in Fragaria × ananassa and Fragaria vesca[J]. Plant Physiol Biochem, 2013, 72: 72-78. DOI:10.1016/j.plaphy.2013.03.019 |
| [38] |
Wang X, Li CF, Zhou C, et al. Molecular characterization of the C-glucosylation for puerarin biosynthesis in Pueraria lobata[J]. Plant J, 2017, 90: 535-546. DOI:10.1111/tpj.13510 |
| [39] |
Noguchi A, Saito A, Homma Y, et al. A UDP-glucose: isoflavone 7-O-glucosyltransferase from the roots of soybean (Glycine max) seedlings. Purification, gene cloning, phylogenetics, and an implication for an alternative strategy of enzyme catalysis[J]. J Biol Chem, 2007, 282: 23581-23590. DOI:10.1074/jbc.M702651200 |
| [40] |
Chen RD, Liu X, Zou JH, et al. Regio- and stereospecific prenylation of flavonoids by Sophora flavescens prenyltransferase[J]. Adv Synth Catal, 2013, 355: 1817-1828. DOI:10.1002/adsc.201300196 |
| [41] |
Li JH, Chen RD, Wang RS, et al. Biocatalytic access to diverse prenylflavonoids by combining a regiospecific C-prenyltransferase and a stereospecific chalcone isomerase[J]. Acta Pharm Sin B, 2018, 8: 678-686. DOI:10.1016/j.apsb.2018.01.009 |
| [42] |
Li JH, Chen RD, Wang RS, et al. GuA6DT, a regiospecific prenyltransferase from Glycyrrhiza uralensis, catalyzes the 6-prenylation of flavones[J]. Chembiochem, 2014, 15: 1673-1681. DOI:10.1002/cbic.201402160 |
| [43] |
Wang RS, Chen RD, Li JH, et al. Molecular characterization and phylogenetic analysis of two novel regio-specific flavonoid prenyltransferases from Morus alba and Cudrania tricuspidate[J]. J Biol Chem, 2014, 289: 35815-35825. DOI:10.1074/jbc.M114.608265 |
| [44] |
Feng KP, Chen RD, Xie KB, et al. A regiospecific rhamnosyltransferase from Epimedium pseudowushanense catalyzes the 3-O-rhamnosylation of prenylflavonols[J]. Org Biomol Chem, 2018, 16: 452-458. DOI:10.1039/C7OB02763J |
| [45] |
Feng KP, Chen RD, Xie KB, et al. Ep7GT, a glycosyltransferase with sugar donor flexibility from Epimedium pseudowushanense, catalyzes the 7-O-glycosylation of baohuoside[J]. Org Biomol Chem, 2019, 17: 8106-8114. DOI:10.1039/C9OB01352K |
| [46] |
Wang PP, Li CJ, Li XD, et al. Complete biosynthesis of the potential medicine icaritin by engineered Saccharomyces cerevisiae and Escherichia coli[J]. Sci Bull, 2021, 66: 1906-1916. DOI:10.1016/j.scib.2021.03.002 |
| [47] |
Muhammad A, Feng XD, Rasool A, et al. Production of plant natural products through engineered Yarrowia lipolytica[J]. Biotechnol Adv, 2020, 43: 107555. DOI:10.1016/j.biotechadv.2020.107555 |
| [48] |
Chen TT, Zhou YY, Lu YH, et al. Advances in heterologous biosynthesis of plant and fungal natural products by modular co-culture engineering[J]. Biotechnol Lett, 2019, 41: 27-34. DOI:10.1007/s10529-018-2619-z |
| [49] |
Rodriguez A, Kildegaard KR, Li MJ, et al. Establishment of a yeast platform strain for production of p-coumaric acid through metabolic engineering of aromatic amino acid biosynthesis[J]. Metab Eng, 2015, 31: 181-188. DOI:10.1016/j.ymben.2015.08.003 |
| [50] |
Liu QL, Yu T, Li XW, et al. Rewiring carbon metabolism in yeast for high level production of aromatic chemicals[J]. Nat Commun, 2019, 10: 4976. DOI:10.1038/s41467-019-12961-5 |
| [51] |
Gao S, Lyu YB, Zeng WZ, et al. Efficient biosynthesis of (2S)-naringenin from p-coumaric acid in Saccharomyces cerevisiae[J]. J Agric Food Chem, 2020, 68: 1015-1021. DOI:10.1021/acs.jafc.9b05218 |
| [52] |
Zhou SH, Hao TT, Zhou JW. Fermentation and metabolic pathway optimization to de novo synthesize (2S)-naringenin in Escherichia coli[J]. J Microbiol Biotechnol, 2020, 30: 1574-1582. DOI:10.4014/jmb.2008.08005 |
| [53] |
Koopman F, Beekwilder J, Crimi B, et al. De novo production of the flavonoid naringenin in engineered Saccharomyces cerevisiae[J]. Microb Cell Fact, 2012, 11: 155. DOI:10.1186/1475-2859-11-155 |
| [54] |
Lyu XM, Zhao GL, Ng KR, et al. Metabolic engineering of Saccharomyces cerevisiae for de novo production of kaempferol[J]. J Agric Food Chem, 2019, 67: 5596-5606. DOI:10.1021/acs.jafc.9b01329 |
| [55] |
Ganesan V, Li ZH, Wang XN, et al. Heterologous biosynthesis of natural product naringenin by co-culture engineering[J]. Synth Syst Biotechnol, 2017, 2: 236-242. DOI:10.1016/j.synbio.2017.08.003 |
| [56] |
Zhang W, Liu H, Li X, et al. Production of naringenin from D-xylose with co-culture of E. coli and S. cerevisiae[J]. Eng Life Sci, 2017, 17: 1021-1029. DOI:10.1002/elsc.201700039 |
| [57] |
Lv YK, Marsafari M, Koffas M, et al. Optimizing oleaginous yeast cell factories for flavonoids and hydroxylated flavonoids biosynthesis[J]. ACS Synth Biol, 2019, 8: 2514-2523. DOI:10.1021/acssynbio.9b00193 |
| [58] |
Palmer CM, Miller KK, Nguyen A, et al. Engineering 4-coumaroyl-CoA derived polyketide production in Yarrowia lipolytica through a β-oxidation mediated strategy[J]. Metab Eng, 2020, 57: 174-181. DOI:10.1016/j.ymben.2019.11.006 |
| [59] |
Eichenberger M, Hansson A, Fischer D, et al. De novo biosynthesis of anthocyanins in Saccharomyces cerevisiae[J]. FEMS Yeast Res, 2018. DOI:10.1093/femsyr/foy046 |
| [60] |
Leonard E, Lim KH, Saw PN, et al. Engineering central metabolic pathways for high-level flavonoid production in Escherichia coli[J]. Appl Environ Microbiol, 2007, 73: 3877-3886. DOI:10.1128/AEM.00200-07 |
| [61] |
Leonard E, Yan YJ, Fowler ZL, et al. Strain improvement of recombinant Escherichia coli for efficient production of plant flavonoids[J]. Mol Pharm, 2008, 5: 257-265. DOI:10.1021/mp7001472 |
| [62] |
Wu JJ, Zhang X, Zhou JW, et al. Efficient biosynthesis of (2S)-pinocembrin from D-glucose by integrating engineering central metabolic pathways with a pH-shift control strategy[J]. Bioresour Technol, 2016, 218: 999-1007. DOI:10.1016/j.biortech.2016.07.066 |
| [63] |
Yan YJ, Huang LX, Koffas MAG. Biosynthesis of 5-deoxyflavanones in microorganisms[J]. Biotechnol J, 2007, 2: 1250-1262. DOI:10.1002/biot.200700119 |
| [64] |
Rodriguez A, Strucko T, Stahlhut SG, et al. Metabolic engineering of yeast for fermentative production of flavonoids[J]. Bioresour Technol, 2017, 245: 1645-1654. DOI:10.1016/j.biortech.2017.06.043 |
| [65] |
Stahlhut SG, Siedler S, Malla S, et al. Assembly of a novel biosynthetic pathway for production of the plant flavonoid fisetin in Escherichia coli[J]. Metab Eng, 2015, 31: 84-93. DOI:10.1016/j.ymben.2015.07.002 |
| [66] |
Akram M, Rasool A, An T, et al. Metabolic engineering of Yarrowia lipolytica for liquiritigenin production[J]. Chem Eng Sci, 2021, 230: 116117. |
| [67] |
Wang X, Song ZJ, He X, et al. Antitumor and immunomodulatory activity of genkwanin on colorectal cancer in the APC(Min/+) mice[J]. Int Immunopharmacol, 2015, 29: 701-707. DOI:10.1016/j.intimp.2015.09.006 |
| [68] |
Lee H, Kim BG, Kim M, et al. Biosynthesis of two flavones, apigenin and genkwanin, in Escherichia coli[J]. J Microbiol Biotechnol, 2015, 25: 1442-1448. DOI:10.4014/jmb.1503.03011 |
| [69] |
Li JH, Tian CF, Xia YH, et al. Production of plant-specific flavones baicalein and scutellarein in an engineered E. coli from available phenylalanine and tyrosine[J]. Metab Eng, 2019, 52: 124-133. DOI:10.1016/j.ymben.2018.11.008 |
| [70] |
Ji DN, Li JH, Xu FL, et al. Improve the biosynthesis of baicalein and scutellarein via manufacturing self-assembly enzyme reactor in vivo[J]. ACS Synth Biol, 2021, 10: 1087-1094. DOI:10.1021/acssynbio.0c00606 |
| [71] |
Dueber JE, Wu GC, Malmirchegini GR, et al. Synthetic protein scaffolds provide modular control over metabolic flux[J]. Nat Biotechnol, 2009, 27: 753-759. DOI:10.1038/nbt.1557 |
| [72] |
Liu XN, Cheng J, Zhang GH, et al. Engineering yeast for the production of breviscapine by genomic analysis and synthetic biology approaches[J]. Nat Commun, 2018, 9: 448. DOI:10.1038/s41467-018-02883-z |
| [73] |
Duan LJ, Ding WT, Liu XN, et al. Biosynthesis and engineering of kaempferol in Saccharomyces cerevisiae[J]. Microb Cell Fact, 2017, 16: 165. DOI:10.1186/s12934-017-0774-x |
| [74] |
Du Y, Yang BR, Yi ZQ, et al. Engineering Saccharomyces cerevisiae coculture platform for the production of flavonoids[J]. J Agric Food Chem, 2020, 68: 2146-2154. DOI:10.1021/acs.jafc.9b07916 |
| [75] |
Dixon RA, Ferreira D. Genistein[J]. Phytochemistry, 2002, 60: 205-211. DOI:10.1016/S0031-9422(02)00116-4 |
| [76] |
Kim DH, Kim BG, Jung NR, et al. Production of genistein from naringenin using Escherichia coli containing isoflavone synthase-cytochrome P450 reductase fusion protein[J]. J Microbiol Biotechnol, 2009, 19: 1612-1616. DOI:10.4014/jmb.0905.05043 |
| [77] |
Kim BG. Biological synthesis of genistein in Escherichia coli[J]. J Microbiol Biotechnol, 2019, 30: 770-776. |
| [78] |
Horinouchi S. Combinatorial biosynthesis of plant medicinal polyketides by microorganisms[J]. Curr Opin Chem Biol, 2009, 13: 197-204. DOI:10.1016/j.cbpa.2009.02.004 |
| [79] |
Leonard E, Koffas MAG. Engineering of artificial plant cytochrome P450 enzymes for synthesis of isoflavones by Escherichia coli[J]. Appl Environ Microbiol, 2007, 73: 7246-7251. DOI:10.1128/AEM.01411-07 |
| [80] |
Liu QL, Liu Y, Li G, et al. De novo biosynthesis of bioactive isoflavonoids by engineered yeast cell factories[J]. Nat Commun, 2021, 12: 6085. DOI:10.1038/s41467-021-26361-1 |
 2022, Vol. 57
2022, Vol. 57


