艾滋病、乙肝等病毒感染导致的慢性传染性疾病严重危害人类的健康与生命[1-3]。新发病毒在人类历史上不断出现, 已累计造成数千万人死亡。近年来气候变化和全球化都为病毒传播创造了更有利的条件。与其他微生物相比, 病毒具有极高突变率, 使其迅速适应新宿主并对疫苗和抗病毒药物产生耐药性[4-6]。
与DNA病毒相比, RNA病毒的突变率更高, 可以跨种传播感染人类, 这导致RNA病毒占人畜共患病病毒的80%以上, 是过去20年中重大流行病的罪魁祸首。除了高遗传变异性, RNA病毒可以通过气溶胶传播, 人传人的传播性很高。自2000年以来, 所有主要的流行病和大流行性暴发都是由RNA病毒引起的。例如, 甲型H1N1流感病毒(2009/2010) 疫情、埃博拉病毒(Ebola virus, EBOV) 疫情(2014~2016)、寨卡病毒(Zika virus, ZIKV) 疫情(2015~)、基孔肯雅热病毒(chikungunya fever, CHIKF) (2015/2016) 及3次高致病性冠状病毒(coronavirus, CoV) 感染疫情, 包括2002/2003年SARS-CoV (severe acute respiratory syndrome-CoV) 感染导致的非典肺炎疫情、2012年MERS-CoV (middle east respiratory syndrome-CoV) 感染引起的中东呼吸综合征疫情和2019/2020年SARS-CoV-2 (severe acute respiratory syndrome-CoV 2) 感染所造成的新冠肺炎(corona virus disease 2019, COVID-19) 疫情。特别是, CoV引发的瘟疫呈现出越来越频繁的趋势, 由于缺乏疗效确切的特效药物, 给人类社会造成了极大的危害[4]。
不容忽视的是, 未来随时可能暴发的新型病毒是人类一直面临的巨大威胁, 也让全球公共卫生体系面临严峻的挑战。近期, 世界卫生组织(WHO) 提出要防御“Disease X”, 即由目前未知的病原体(包括SARS-CoV-2) 引起的严重国际大流行的人类疾病(http://www.who.int/blueprint/priority-diseases/en/)。因此, 研发广谱、高效的抗病毒备选药物对于应对当前疫情以及将来可能发生的新型病毒感染来说都是十分重要的[7-9]。
本文从药物化学的角度, 精选近年经典案例, 从抗病毒药物研究的共同靶标、共性环节、通用策略以及广谱抗病毒分子等方面总结了广谱抗病毒药物研发的普适性策略, 期望对当下及未来的抗病毒药物研发提供参考。
1 抗病毒药物研究的共同靶标 1.1 合成糖受体包膜病毒通常在进入细胞之前, 通过聚糖介导的相互作用与宿主细胞膜上的蛋白质对接。研究者提出了一种通过使用合成糖受体(synthetic carbohydrate receptors, SCRs) 来破坏这些相互作用, 进而抑制病毒生命周期来降低病毒传染性的方法。近期, SCRs作为高糖基化包膜病毒的有效抑制剂, 受到普遍关注。该类分子不但可以抑制病毒侵入, 而且可以通过干扰包膜的糖分子, 使病毒暴露于宿主免疫系统中得以清除, 具有双重作用模式[10-13]。SCRs已报道具有抗ZIKV、流感病毒和艾滋病毒(human immunodeficiency virus, HIV) 活性, 有望成为抗登革病毒(dengue virus, DENV) 及SARS-CoV-2等其他包膜病毒的广谱抑制剂[14-16]。苯基硼酸作为顺式邻二醇的可逆结合基团, 是糖分子的有效配体, 可作为SCRs的关键药效团元素, 用于设计广谱抗病毒分子[17]。
1.2 靶向病毒膜的广谱抗病毒策略由于脂质成分对于细胞膜的膜曲率和流动性至关重要, 因此通过改变(降低或增加) 脂质成分有望成为广谱抗病毒策略[18-20]。例如, 一些阳离子的抗病毒肽(antiviral peptides, AVPs) 在高浓度下具有类似洗涤剂的性质, 可以导致病毒膜孔的形成及胶束化。多不饱和内质网-靶向脂质体(polyunsaturated endoplasmic reticulum-targeting liposomes, PERL) 通过耗竭细胞和病毒膜的胆固醇发挥广谱抗病毒作用; 胆固醇耗尽会降低膜的流动性, 影响病毒和细胞膜融合所必需的负曲率(图 1)。
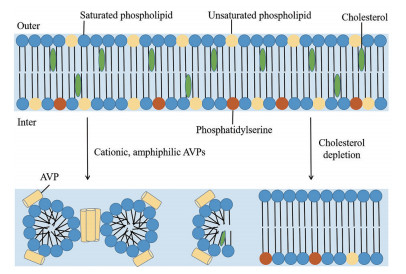
|
Figure 1 Broad-spectrum antivirals targeting viral membranes |
楔子状或倒锥状分子和一些嗜碱性抗病毒肽可以增加病毒膜脂双链的自发正曲率, 提高病毒融合蛋白介导的膜融合所需的能垒。同样, 膜靶向Ⅱ型光敏剂在病毒膜平面上产生的单层氧能够氧化不饱和磷脂并诱导病毒膜纳米结构的变化。氧化磷脂的簇合物导致脂质包装的差异化, 降低流动性, 增加正曲率, 增加分子脂质面积, 减小膜的厚度。
磷脂特异性抗体可以靶向病毒膜中丰富的特定磷脂家族, 例如磷脂酰丝氨酸, 进而阻止病毒的吸附和侵入。
大多数致病性病毒病原体都是包膜病毒。“分子钳”是靶向病毒膜的特殊化合物, 该类化合物的发现是受天然的“锁钥模型”的启发[21, 22]。“分子钳”选择性地与病毒包膜的脂筏区域作用, 代表性的“分子钳”为CLR01和CLR05 (图 2)。CLR01是赖氨酸和精氨酸特异性的配体, 可以破坏HIV、EBOV和ZIKV等包膜病毒。CLR01和CLR05对单纯疱疹病毒(herpes simplex virus, HSV)、梅斯勒病毒、流感病毒和SARS-CoV-2等具有广谱抑制活性, 但对非包膜病毒无效[23-25]。

|
Figure 2 Chemical structures of CLR01 and CLR05 |
近日, 清华大学研究人员从埃及伊蚊肠道内分离出的具有抗蚊媒病毒活性的色素杆菌新菌株Chromobacterium sp. Beijing入手, 筛选并鉴定了两个对多种包膜病毒(DENV、ZIKV、SARS-CoV-2、HIV和HSV) 均有较强抑制作用的抗病毒效应因子CbAE-1和CbAE-2。机制研究表明, CbAE-1和CbAE-2通过其脂酶活性, 直接破坏病毒包膜结构导致其失活。同时, CbAE-2在人类细胞和小鼠上均表现出了较强的安全性, 具有作为广谱抗病毒药物的潜力[26]。
此外, 病毒聚合酶镁离子螯合区域[27]、铁硫簇(iron-sulfur cluster)[28]、锌指结构[29]等也可作为广谱抗病毒药物发现的共性靶标。
2 抗病毒药物研究的共性环节 2.1 靶向病毒膜融合过程在包膜病毒的复制周期中, 需要病毒和细胞膜融合才能进入细胞。病毒通过受体识别以及膜融合或内吞等步骤进入靶细胞是首要环节。在该过程中, 介导病毒与细胞受体识别的病毒表面蛋白(surface protein, SP) 的受体结合亚基、介导膜融合的病毒SP跨膜亚基、细胞上的受体、切割SP所需的宿主细胞蛋白酶等均是常见的抗病毒靶点[30]。
CoV是Ⅰ型包膜病毒, 位于包膜表面的S蛋白介导病毒入侵宿主细胞过程, 包括受体结合及膜融合等步骤。在膜融合的过程中, 形成六螺旋束(six-helix bundle, 6-HB) 是一个非常保守且关键的机制。目前发现感染人的冠状病毒(HCoV) 中, 其HR1 (heptad repeat-1) 三聚体与HR2 (heptad repeat-2) 作用的表面氨基酸大都为保守的疏水性氨基酸, 因此HR1是CoV S蛋白上非常保守的药物靶点[30]。
2018年, 姜世勃与刘克良团队发现靶向病毒融合蛋白的α-螺旋脂肽具有广谱抗MERS-CoV (EC50 = 0.11 μmol·L-1, CC50>100 μmol·L-1) 及甲型流感病毒(influenza A virus, IAV) 活性(H1N1 EC50 = 1.73 μmol·L-1, CC50>100 μmol·L-1)[31]。近日, 复旦大学姜世勃/陆路团队与上海科技大学杨贝/Wilson团队合作, 通过系统地筛选与结构修饰, 发现了能够广谱抑制多种HCoV感染的多肽类融合抑制剂EK1及脂肽EK1C4, 并揭示了其作用靶点与分子机制[32, 33]。该研究同时证明了CoV刺突蛋白的HR1区域是一个重要且保守的药物靶点, 为后续广谱抗HCoVs药物研发提供了思路。
2.2 核酸复制病毒进入靶细胞后, 病毒基因组DNA/RNA被释放到细胞中, 作为模板指导病毒蛋白的合成。RNA病毒的基因组复制需要RNA依赖的RNA或DNA聚合酶(RNA-dependent RNA polymerase, RdRp; RNA-dependent DNA polymerase, RdDp), 这类酶在人体中不存在且相对保守, 成为抗病毒药物研发的重要靶点。不同病毒聚合酶的结构和功能有许多相似之处, 因此针对某一种病毒聚合酶设计的抑制剂往往对其他病毒也有较好的抑制作用[34, 35]。
自从1962年世界第一个抗病毒药物碘苷被批准上市以来, 全球已有众多抗病毒核苷类似物药物获批上市。在病毒疫情暴发时, 核苷类药物往往成为人们的首选。早在2014年西非暴发的大规模EBOV疫情中, 部分核苷类似物药物在临床阶段均表现出一定的抗病毒活性——例如日本富山化学的新型抗流感药物法匹拉韦(favipiravir) 以及瑞德西韦(remdesivir, 图 3), 特别是瑞德西韦目前已经完成EBOV的试验药物Ⅲ期临床试验。随着研究的深入, 瑞德西韦被发现具有广谱抗病毒活性, 涵盖丝状病毒科病毒(EBOV和马尔堡病毒等)、沙粒病毒科病毒(拉沙病毒和胡宁病毒等)、CoV科病毒(SARS、MERS和猫科冠状病毒等) 和黄病毒科病毒(ZIKV等) 等, 因此也成为了治疗SARS-CoV-2的首个小分子药物[36]。
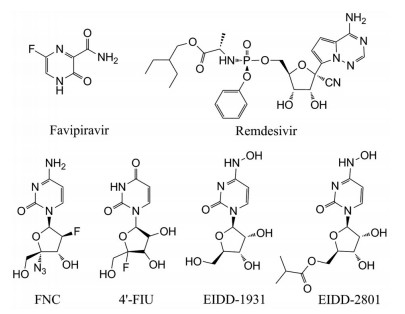
|
Figure 3 Chemical structures of some representative broad-spectrum antiviral nucleosides |
阿兹夫定(azvudine, FNC, 图 3) 具有抑制HIV、丙型肝炎病毒(hepatitis C virus, HCV)、肠道病毒71型等RNA病毒复制的功能, 2021年7月, 已在中国上市用于治疗高病毒载量的成年HIV-1感染者。此外, 阿兹夫定在新冠肺炎临床研究中也取得显著效果[37]。
瑞德西韦进入临床研究后, 其抗病毒效果与预期有一定差距, 原因可能是: 疾病的病程及动物模型与人体药动学差异、药物之间的相互作用和个体差异。此外, CoV特有的“复制矫正” (proofreading) 机制, 即将掺入RNA产物链的核苷药物“剔除”, 进而逃逸核苷类抗病毒药物的抑制, 可能是此类抗病毒药物效果不佳的一个重要原因[38]。
近日, 美国乔治亚州立大学的研究人员报道了一种抑制呼吸道合胞病毒(respiratory syncytial virus, RSV)、相关RNA病毒和SARS-CoV-2的广谱抗病毒核苷分子——4'-氟尿啶(4'-FlU, EIDD-2749, 图 3), 它在细胞和分化良好的人气道上皮中具有高选择性指数。RSV和SARS-CoV-2体外RdRp聚合酶抑制显示掺入后i或i+3/4位出现转录暂停。每日一次的口服治疗对RSV感染的小鼠或SARS-CoV-2感染的雪貂非常有效[39]。
EIDD-1931 (即NHC, 图 3), 是一种核苷酸类似物。NHC上的肟形式模仿尿苷, 与腺苷匹配, 而另一个互变异构体模仿胞苷, 与鸟苷匹配。它的原理是通过给病毒RNA引入大量的突变, “瘫痪”病毒的基因组, 进而导致遗传信息大量错误使病毒无法存活[40-45]。目前仅有NHC及其衍生物能够躲避病毒复制矫正机制的干扰。在体外模型中, NHC对RSV、流感病毒、CHIKF、EBOV、委内瑞拉马脑炎病毒、东部马脑炎病毒、MERS-CoV、SARS-CoV以及SARS-CoV-2 (多数变异毒株) 等具有广谱抗病毒活性, 无明显细胞毒性[46-48]; 但在食蟹猕猴中口服生物利用度较差。EIDD-2801 (molnupiravir, 图 3) 是NHC的异丙酯前体药物, 旨在改善NHC体内药代动力学以及在人类和非人类灵长类动物的口服生物利用度。Molnupiravir在雪貂和非人类灵长类动物中具有较好的口服生物利用度。对感染流感病毒的雪貂进行molnupiravir口服治疗, 可将大流行流感和季节性甲型流感的病毒载量降低数个数量级, 并可减轻发热、呼吸道上皮组织病变和炎症[39, 49]。Molnupiravir使轻中度新冠肺炎患者的住院率或死亡风险降低了约50%。2021年11月4日, 英国药品和保健产品监管局(MHRA) 已在英国批准molnupiravir上市, 用于治疗重症和住院风险较高的轻至中度新冠肺炎成人患者(http://www.21jingji.com/article/20211104/herald/f0b15254b2fcc17b70b26b839e32b1c6.html)。
除了molnupiravir之外, 法匹拉韦也可以掺入到病毒RNA链, 诱发病毒的基因组突变, 并通过累积这种突变, 导致病毒失活或失去感染能力[50]。
总之, 靶向病毒最为保守的RdRp是一种开发广谱抗病毒药物非常有前景的策略。目前处于临床研究阶段的多个新冠病毒RdRp抑制剂类药物结构差异较大, 靶向RdRp影响病毒复制的机制也不尽相同, 特别需要从结构生物学角度解析抑制剂与RdRp复合物结构, 明确作用机制, 为精准开发高效特异的、以RdRp为靶标的广谱抗病毒药物提供理论基础。
2.3 核糖体移码(ribosomal frameshifting)在正常细胞内, 核糖体(ribosome) 以3个碱基为单位(即密码子codon) 由5到3端单向、连续地读取mRNA中的遗传信息, 合成蛋白质[51]。由于体积的限制, 病毒的基因组通常较小, 所携带的遗传信息较少。包括SARS-CoV-2在内的各种RNA病毒在复制过程中会利用一些特殊的机制调控病毒基因表达, 扩展其所携带遗传信息的利用率, 其中一种常用的机制是称为程序性“移码”的蛋白质合成重编码机制(programmed ribosomal frameshifting, PRF)[52-54]。即核糖体不遵循常规读取3个字母的步骤, 而是会漏掉一两个RNA字母。核糖体发生的这种错位被称为“移码”, 会导致核糖体错误读取遗传密码。例如, SARS-CoV-2严重依赖其RNA折叠引起的“移码”来合成蛋白[52-54]。
理论上, 任何通过靶向RNA折叠来抑制“移码”的化合物都可能作为一种治疗感染的药物。“移码”现象在人类自身基因的表达中极为罕见, 因此靶向读码框“移码”是一个可行的抗病毒策略。
研究者通过运用荧光蛋白报告基因系统联合高通量筛选技术, 鉴定出了一个可以高效抑制读码框“移码”的小分子化合物美拉沙星(merafloxacin, 图 4), 它能在细胞水平(Vero E6细胞) 显著抑制SARS-CoV-2复制[55]。美拉沙星抑制读码框“移码”的机制尚不清楚, 可能直接作用于核糖体与病毒RNA的结合, 或者抑制内源性调控蛋白。
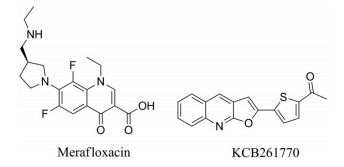
|
Figure 4 Chemical structures of merafloxacin and KCB261770 |
近期, Ahn等[56]从9 689个小分子中发现了一种新型的呋喃[2, 3-b]喹啉类化合物KCB261770 (图 4), 它能够抑制MERS-CoV的“移码”和细胞水平MERS-CoV的复制。此外, 该化合物还能抑制SARS-CoV和SARS-CoV-2的“移码”, 具有广谱抗病毒活性。
3 抗病毒药物研究的通用策略 3.1 细胞纳米“海绵”SARS-CoV-2的细胞结合和进入是由其刺突糖蛋白(S蛋白) 介导的, S蛋白不仅与人类血管紧张素转换酶2 (angiotensin converting enzyme Ⅱ, ACE2) 受体结合, 还与肝素等糖胺聚糖结合。近期研究发现细胞膜包被的纳米颗粒(细胞纳米“海绵”) 模拟宿主细胞, 通过自然的细胞受体吸引和中和SARS-CoV-2, 可作为一种广谱抗病毒策略, 还发现增加细胞纳米海绵表面肝素密度可以提高抗SARS-CoV-2作用[57]。
3.2 抗体募集/杀死细胞2009年, 研究者设计了一种新的小分子ARM-H, 它有可能通过两种机制抑制HIV: ①通过招募抗体到gp120表达病毒颗粒和受感染的人类细胞, 从而增强其吸收和人类免疫系统的破坏; ②通过结合病毒糖蛋白gp120, 抑制其与人CD4结合和防止病毒进入。研究人员通过实验证明了ARM-H能够同时结合gp120和抗2, 4-二硝基苯抗体(DNP, 存在于人血液中)[58]。
抗体、ARM-H和gp120之间形成的三元复合物具有免疫活性, 导致补体介导的表达env细胞的破坏。此外, ARM-H可以阻止病毒进入人类T细胞, 因此应该能够通过两种相互强化的机制(抑制病毒进入和抗体介导的杀伤) 来抑制病毒复制。这些研究表明, 通过抗体招募的小分子具有可行的抗艾滋病毒活性, 并有可能启动HIV治疗的新范式。
2020年, Low团队通过将神经氨酸酶抑制剂扎那米韦与高免疫原性半抗原2, 4-二硝基苯(DNP) 结合, 设计并合成了一种双功能小分子, DNP专门针对游离病毒和病毒感染细胞的表面。该类分子抑制病毒释放的同时, 通过免疫介导清除游离病毒和病毒感染的细胞, 对感染100倍MLD50病毒的小鼠进行鼻内或腹腔注射单剂量药物, 可以根除A型和B型流感毒株的晚期感染[59]。
近期研究发现, 抗生素分子concanamycin A可让免疫系统杀死被HIV感染的人体细胞[60]。DDX3抑制剂可以让HIV-1感染的细胞选择性死亡, 进而耗竭病毒潜伏库[61], 为根治艾滋病提供了新思路。
3.3 多价结合——靶向病毒表面的非特异作用细胞表面的糖链是细菌、病毒、免疫细胞的接触点。病毒进入宿主细胞的过程涉及与不同细胞表面受体稳定但短暂的多价相互作用。几种病毒的最初接触始于在细胞表面附着硫酸肝素蛋白聚糖, 最终导致病毒进入。已经开发出的广谱抗病毒药物如肝素或类肝素材料模拟细胞表面糖负责最初的病毒附着, 如硫酸乙酰肝素(heparan sulfate)。高磺化金纳米粒子具有广谱杀病毒性能。然而, 由于未知的清除机制和潜在的长期毒性是金纳米颗粒成药性的不利因素。环糊精(cyclodextrins, CDs) 是天然的葡萄糖衍生物, 具有一种刚性的环状结构, 由α (1-4) 连接的吡喃葡萄糖组成。磺化环糊精对HIV具有可逆及特异的抑制活性。
最近, 英国曼彻斯特大学研究小组对天然葡萄糖衍生物环糊精进行磺化修饰开发出了一种能够破坏病毒的外壳且对耐药性病毒也有效的新的广谱抗病毒分子, 其有望治疗HSV、RSV、HCV、HIV和ZIKV等多种病毒感染[62]。
基于多价相互作用的抗病毒药物, 如柔性纳米凝胶, 通过干扰病毒颗粒和阻断与细胞受体的初始相互作用已经成为广谱抗病毒药物研究的有效策略。
负电荷多硫酸盐可以结合SARS-CoV-2受体结合区域(receptor binding domain, RBD) 上的正电荷斑块(patches), 阻止病毒与宿主细胞相互作用进而抑制感染。与肝素相比, 合成的线型聚甘油硫酸酯(linear polyglycerol sulfate, 图 5) 的抗病毒活性更高, 且抗凝血活性较低[63]。
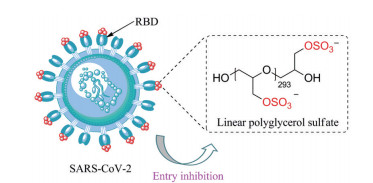
|
Figure 5 Polysulfates block SARS-CoV-2 uptake through electrostatic interactions |
巨大球状多价糖富勒烯、糖基化碳纳米管能抑制EBOV、ZIKV和DENV的感染, 活性可达皮摩尔水平[64-66]。多价唾液化(sialylated) 聚甘油对甲型流感毒株(含耐药株) 具有广谱抑制活性[67]。
3.4 基于拓扑匹配的药物设计IAV颗粒表面均匀分布血凝素和神经氨酸酶。近期, Nie等[68]运用拓扑匹配(topology-matching design) 的药物设计理念, 设计了一种纳米颗粒抑制剂(纳米抑制剂, 图 6A), 它与IAV病毒粒子的纳米拓扑结构匹配, 对血凝素和神经氨酸酶具有多价抑制作用, 可以在细胞外中和病毒颗粒, 阻断其附着和进入宿主细胞。病毒复制显著减少了6个数量级, 即使在感染24 h后使用, 仍能达到99.999%以上的抑制作用。2020年, 该团队用类似的思路, 发现了与IAV表面空间匹配的尖峰纳米抑制剂(spiky nanoinhibitor, 图 6B), 峰值在5~10 nm之间的纳米结构与病毒粒子的结合明显优于平滑的纳米粒子, 获得的红细胞膜(erythrocyte membrane, EM) 包覆的纳米结构可以有效地阻止IAV病毒粒子与细胞的结合, 并抑制随后的感染。EM包覆的纳米结构在细胞无毒剂量下降低了>99.9%的病毒复制[69]。
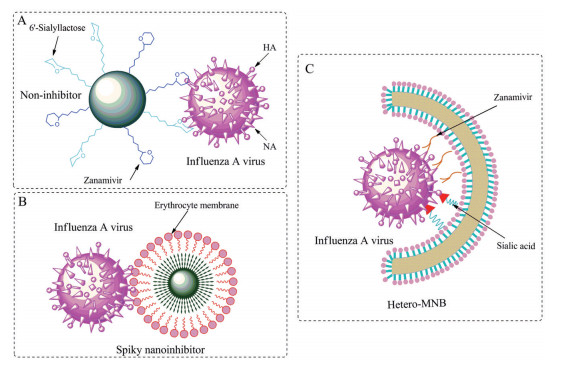
|
Figure 6 (A) Proposed binding patterns between nano-inhibitor and influenza A virus (IAV) particles; (B) Proposed binding patterns between spiky nanoparticle-based inhibitor and IAV particles; (C) Proposed binding patterns between IAV and the heteromultivalent nanobowl (Hetero-MNB), where sialic acid and Zanamivir bind to hemagglutinin (HA) and neuraminidase (NA), respectively, and the bowl shape with facilitating the capping to the surface of the virus particle |
2021年, 该课题组运用拓扑匹配设计理念, 基于宿主红细胞膜设计了与病毒状球面相匹配的碗状纳米结构(“纳米碗”, heteromultivalent nanobowl, Hetero-MNB, 图 6C), 可作为广谱病毒进入抑制剂。与传统的同多价抑制剂不同, 该类异多价抑制剂由于协同多价效应和拓扑匹配的形状, 其半最大抑制浓度为32.4 ± 13.7 μg·mL-1。在不引起细胞毒性的剂量下, 可减少>99.99%的病毒传播。由于在SARS-CoV-2的S蛋白上也发现了多个结合位点, 因此, 异多价纳米结构有望为开发一种有效的预防CoV感染提供新思路[70]。
3.5 靶向病毒核酸病毒RNA会折叠成复杂的RNA结构, 在病毒的生命过程调控中起重要作用, 为开发抗病毒疗法的靶标提供了新的机会。很多研究已经发现多种病毒的非编码区RNA结构可以调控病毒的翻译、复制以及稳定性, 它们通常在相关病毒中高度保守[71-73]。例如, 黄病毒中5'UTR和3'UTR之间的分子内RNA-RNA相互作用促进基因组环化并帮助协调复制; HCV 5'UTR内部核糖体进入位点的结构对于翻译至关重要; 并且ZIKV和其他黄病毒的3'UTR中的多假性结构已显示出使RNA外切核酸酶Xrn1失速, 从而产生了亚基因组黄病毒RNA, 有助于病毒逃避细胞抗病毒过程[74, 75]。
需要指出的是, 与蛋白质类药物靶标相比, RNA结构的动态性与复杂性为药物筛选增加了困难, 往往需要借助于高通量筛选。例如, SARS-CoV-2的RNA基因组含有15个独立的RNA调节元件。研究者通过基于NMR的片段筛选, 从含有768个小分子的片段库中发现了SARS-CoV-2的RNA配体[76]。
近日, 新加坡科学家使用多种RNA分子结构探测方法以及RNA-RNA相互作用分析技术, 解析了SARS-CoV-2基因组RNA的二级结构信息和病毒-宿主之间的RNA相互作用; 同时发现在SARS-CoV-2基因组RNA上广泛存在2'-O-甲基化修饰, 推测可能有助于新冠病毒逃避宿主免疫攻击, 揭示病毒逃避宿主免疫的潜在机制[77]。
G-四链体是由G-quartet层叠而形成的DNA或RNA四链构象, 是最重要的非典型核酸二级结构之一, 因其独特的构象、重要的基因功能和生物学意义而备受关注, 是很有前途的药物靶点[78]。中国科学院长春应用化学研究所曲晓刚团队使用多种生物物理技术和分子生物学技术, 发现SARS-CoV-2基因组中存在G-四链体结构RNA, 证实SARS-CoV-2中的富G序列(位于SARS-CoV-2核衣壳磷酸化蛋白N编码序列区域) 可以在活细胞中折叠成稳定的单分子RNA G-四链体结构。该G-四链体RNA可以被G-四链体特异结合配体吡啶他汀吡咯烷(pyridostatin pyrrolidine, PDP) (图 7) 等识别并稳定, 进而影响G-四链体RNA的生物功能。因此, 该G-四链体可能是抗SARS-CoV-2药物新靶点[79]。
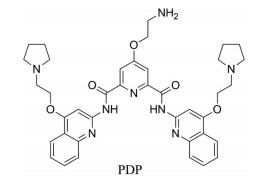
|
Figure 7 Chemical structure of PDP (pyridostatin pyrrolidine) |
病毒基因组的未翻译区域(the untranslated regions, UTR) 包含多种保守和动态结构, 这些功能性的RNA结构对病毒复制至关重要, 为广谱抗病毒研发提供了药物靶点。然而, 计算机对接筛选对于具有内在柔性特征的RNA结构仍存在较大挑战。研究者将体外RNA分析与分子动力学模拟相结合, 构建SARS-CoV-2基因组5'UTR关键区域结构和动力学的3D模型, 进而确定了圆柱形金属超分子螺旋([Ni2L3]4+、[Fe2L3]4+) 对这种RNA结构的约束。这些纳米尺寸的金属超分子螺旋分子可以与核酸结合, 并且在细胞水平具有抗SARS-CoV-2等病毒复制作用[80, 81]。
3.7 核糖核酸酶靶向嵌合体核糖核酸酶靶向嵌合体(ribonuclease targeting chimeras, RIBOTACs) 是降解RNA的新策略, RIBOTACs基于小分子选择性结合RNA (特别是形成复杂的二级和三级结构的RNA), 进而激活核糖核酸酶L (ribonuclease L, RNase L)。RNase L是一种在脊椎动物细胞中广泛表达、具有单链RNA内切活性的蛋白质。该技术已被用于靶向SARS-CoV-2的RNA基因组, 抑制RNA的移码, 并且募集细胞核糖核酸酶彻底杀死SARS-CoV-2。该策略有望用于抗其他病毒药物研发[82]。
3.8 反义核酸技术反义核酸(antisense oligonucleotides) 可以序列特异性地与靶标RNA结合, 实现高效的寻靶和抑制活性。近期, 北京大学的研究人员构建了一类靶向SARS-CoV-2包膜蛋白RNA (E-RNA) 和刺突蛋白RNA (S-RNA) 的单链嵌合反义寡聚核苷酸, 通过在2'甲氧基修饰的反义核酸序列5'端缀合RNase L招募基团2-5A, 可实现有效的病毒RNA降解并抑制病毒增殖[83]。
3.9 核酸适配体技术核酸适配体(nucleic acid aptamers) 是一小段经体外筛选得到的寡核苷酸序列(单链DNA或RNA分子), 能与相应的配体进行高亲和力和强特异性的结合[84]。适配体已经在抗病毒药物开发方面(含SARS-CoV-2) 展现出巨大的潜力[85-87]。
3.10 基于蛋白自组装的配体发现动态组合化学(dynamic combinatorial chemistry, DCC) 融合了组合化学和分子自组装过程两个领域的特点, 开辟了使用相对较小的库组装很多的物质的途径, 而不必单独合成每一个物质。早在2003年, 研究者通过基于点击化学的蛋白模板诱导片段组装, 发现了高活性的HIV蛋白酶抑制剂[88]。2008年, 研究者通过动态连接筛选(dynamic link screening, DLS) 开发了一种潜在的抗SARS药物, 其亲核片段通过与醛抑制剂的可逆反应将亲核片段指向蛋白质的活性位点。它们的抑制作用可以通过与荧光酶底物的竞争检测到。有了这一概念, 与活性位点特异性结合的低亲和力片段在功能酶分析中迅速被识别出来[89]。2021年, 基于Knoevenagel反应的蛋白模板诱导片段组装策略用于Enterovirus D68蛋白酶抑制剂的发现[90]。总之, 动态组合化学在抗病毒药物发现领域仍具有广阔的前景。
4 “万金油”式广谱抗病毒分子在抗新冠药物研发中, 一些“万金油”式的分子引起了足够的重视, 例如具有抗HIV及SARS-CoV-2活性的依布硒(ebselen)[91-95]、具有抗HIV、CHIKF、EBOV及SARS-CoV-2活性的苏拉明(suramin)[96-100]。这类分子往往具有多重抗病毒机制, 应该给予广泛挖掘, 并做好深入的成药性评价。此外, 金属配合物药物[101-105]、肽与聚合物[106]等也应引起足够的重视。
宿主是重要的广谱抗病毒药物靶标[107, 108], 近期中国科学家发现利用小分子N- (对氨基肉桂酰) 邻氨基苯甲酸(ACA) 可抑制包括SARS-CoV-2、HIV和IAV在内的一系列病毒, 作用靶标是TGF-β信号通路[109]。笔者已在近期综述对广谱抗病毒活性分子进行了总结[4, 5], 本文不再赘述。
5 总结与展望近年来, 无论是暴发流行的SARS、EBOV、新冠肺炎, 还是持续蔓延的DENV、HIV, 病毒导致的感染病例数以亿计, 其中数百万患者患有严重的临床症状甚至死亡, 给全球公共卫生和经济带来了沉重负担。
特别是这次新冠疫情为人类敲响了警钟, 迫切需要广谱抗病毒药物来有效控制突发公共卫生危机, 加强抗击新兴病毒性疾病的技术储备。在此背景下, 本文从抗病毒药物研究的共同靶标、共性环节、通用策略、广谱分子等层次总结了发现广谱抗病毒药物的一些策略与方法(图 8)。各类策略的应用势必会加快抗病毒药物研发的进程, 但是一些策略同时也存在着局限性, 例如有些病毒通过内吞作用进入细胞, 不涉及膜融合过程; 多价结合分子由于普遍较大的相对分子质量面临着成药性问题; 核糖体移码、靶向病毒核酸等技术的普遍应用还局限于对病毒结构的进一步认识; 基于宿主红细胞膜设计与病毒状球面相匹配的碗状纳米结构存在一定的技术挑战。尽管如此, 在抗SARS-CoV-2药物研发中, 各行各业也将自己的专长发挥到极大, 各种技术路线齐上阵, 进行了饱和式研发, 体现了人类应对巨大危机时的智慧和力量。本文篇幅所限, 难免挂一漏万, 例如未展开论述的虚拟筛选新技术[110]、自由能扰动计算[111]、高效合成技术[112]、DNA编码库技术[113]、变构调节技术[114]等。随着分子生物学与结构生物学的发展, 不同病毒中的共性机制逐渐被揭示, 如病毒的RNA盖帽机制(RNA capping machinery)[115-119]、病毒中普遍存在的疏水性蛋白结构viroporins (如IAV M2、HIV-1 Vpu、HCV p7)[120]、核转运[121]等, 将为广谱抗病毒药物的研发提供新的重要靶标。特别是, 中国科学家抗SARS-CoV-2药物研发的典型案例不胜枚举, 例如主蛋白酶抑制剂[122, 123]、肽类侵入抑制剂[7, 33, 124, 125]及铋类抑制剂[126-128]等, 从非典到新冠, 每一个团队无一不是跨越十多年之久。可见抗病毒药物研发需要久久为功、厚积薄发。唯有未雨绸缪, 才能在疫情来临时做好准备从容应对。
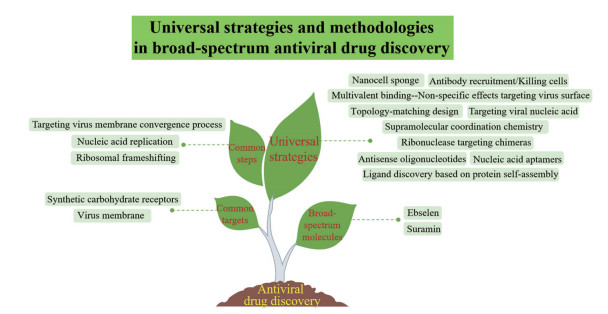
|
Figure 8 Universal strategies and methodologies in broad-spectrum antiviral drug discovery |
病毒的高度变异性和致病机制的复杂性一直是生命医学领域的研究重点和难点。针对病毒的高变异性, 笔者近期总结了发现抗耐药性抗病毒药物的药物化学策略[1], 其中, 蛋白降解、靶向宿主等策略有望对广谱抗病毒药物研发提供参考。针对病毒致病机制的复杂性, 特别是世界范围内多次出现的病毒性呼吸道传染病, 中医药在病毒性肺炎防治中显示出一定的特色和优势。总之, 本文中的一些总结与思索期待能为更好地抗击仍处于流行的SARS-CoV-2及其突变株和未来可能出现的新发病毒提供指引和启发。
作者贡献: 徐淑静和丁当负责全文的撰写; 刘新泳和展鹏对论文进行整体的指导和修改。
利益冲突: 所有作者均声明不存在利益冲突。
| [1] |
Ma Y, Frutos-Beltrán E, Kang D, et al. Medicinal chemistry strategies for discovering antivirals effective against drug-resistant viruses[J]. Chem Soc Rev, 2021, 50: 4514-4540. DOI:10.1039/D0CS01084G |
| [2] |
Fu ZP, Kang DW, Liu XY, et al. Advances of research on target-based anti-AIDS drugs[J]. Prog Pharm Sci (药学进展), 2020, 44: 681-697. |
| [3] |
Huo ZP, Zuo XF, Kang DW, et al. Research progress on new targets of anti-AIDS drugs and their small molecule inhibitors[J]. Acta Pharm Sin (药学学报), 2018, 53: 356-374. |
| [4] |
Song LT, Cheng YS, Gao SH, et al. Research progress of human coronavirus broad-spectrum inhibitors[J]. Chin J Med Chem (中国药物化学杂志), 2021, 31: 721-738. |
| [5] |
Huang TG, Sun L, Zhan P, et al. Recent advances in the research of broad-spectrum antiviral agents[J]. Acta Pharm Sin (药学学报), 2020, 55: 679-693. |
| [6] |
Li J, Jiang XY, Xu SJ, et al. Medicinal chemistry strategies in seeking coronavirus inhibitors[J]. Acta Pharm Sin (药学学报), 2020, 55: 537-553. |
| [7] |
Jiang S, He Y, Liu S. SARS vaccine development[J]. Emerg Infect Dis, 2005, 11: 1016-1020. DOI:10.3201/eid1107.050219 |
| [8] |
Xu SJ, Zhang XJ, Ding D, et al. Bioinorganic chemistry strategies in antiviral drug discovery[J]. Acta Pharm Sin (药学学报), 2022, 57: 576-592. |
| [9] |
Xu SJ, Ding D, Zhang XJ, et al. Novel targets and strategies in antiviral drug discovery[J]. Acta Pharm Sin (药学学报), 2022, 57: 903-916. |
| [10] |
Bravo MF, Lema MA, Marianski M, et al. Flexible synthetic carbohydrate receptors as inhibitors of viral attachment[J]. Biochemistry, 2021, 60: 999-1018. DOI:10.1021/acs.biochem.0c00732 |
| [11] |
Balzarini J. Carbohydrate-binding agents: a potential future cornerstone for the chemotherapy of enveloped viruses?[J]. Antivir Chem Chemother, 2007, 18: 1-11. DOI:10.1177/095632020701800101 |
| [12] |
Gupta RK, Apte GR, Lokhande KB, et al. Carbohydrate-binding agents: potential of repurposing for COVID-19 therapy[J]. Curr Protein Pept Sci, 2020, 21: 1085-1096. DOI:10.2174/1389203721666200918153717 |
| [13] |
François KO, Balzarini J. Potential of carbohydrate-binding agents as therapeutics against enveloped viruses[J]. Med Res Rev, 2012, 32: 349-387. DOI:10.1002/med.20216 |
| [14] |
Palanichamy K, Joshi A, Mehmetoglu-Gurbuz T, et al. Anti-zika activity of a library of synthetic carbohydrate receptors[J]. J Med Chem, 2019, 62: 4110-4119. DOI:10.1021/acs.jmedchem.9b00142 |
| [15] |
Vanderlinden E, Van Winkel N, Naesens L, et al. In vitro characterization of the carbohydrate-binding agents HHA, GNA, and UDA as inhibitors of influenza A and B virus replication[J]. Antimicrob Agents Chemother, 2021, 65: e01732-20. |
| [16] |
Francesconi O, Nativi C, Gabrielli G, et al. Antiviral activity of synthetic aminopyrrolic carbohydrate binding agents: targeting the glycans of viral gp120 to inhibit HIV entry[J]. Chemistry, 2015, 21: 10089-10093. DOI:10.1002/chem.201501030 |
| [17] |
Trippier PC, McGuigan C, Balzarini J. Phenylboronic-acid-based carbohydrate binders as antiviral therapeutics: monophenylboronic acids[J]. Antivir Chem Chemother, 2010, 20: 249-257. DOI:10.3851/IMP1632 |
| [18] |
Vigant F, Santos NC, Lee B. Broad-spectrum antivirals against viral fusion[J]. Nat Rev Microbiol, 2015, 13: 426-437. DOI:10.1038/nrmicro3475 |
| [19] |
Jackman JA. Antiviral peptide engineering for targeting membrane-enveloped viruses: recent progress and future directions[J]. Biochim Biophys Acta Biomembr, 2021, 1864: 183821. |
| [20] |
Yoon BK, Jeon WY, Sut TN, et al. Stopping membrane-enveloped viruses with nanotechnology strategies: toward antiviral drug development and pandemic preparedness[J]. ACS Nano, 2021, 15: 125-148. DOI:10.1021/acsnano.0c07489 |
| [21] |
Regen SL. Membrane-disrupting molecules as therapeutic agents: a cautionary note[J]. JACS Au, 2020, 1: 3-7. |
| [22] |
Mbarek A, Moussa G, Chain JL. Pharmaceutical applications of molecular tweezers, clefts and clips[J]. Molecules, 2019, 24: 1803. DOI:10.3390/molecules24091803 |
| [23] |
Weil T, Groß R, Röcker A, et al. Supramolecular mechanism of viral envelope disruption by molecular tweezers[J]. J Am Chem Soc, 2020, 142: 17024-17038. DOI:10.1021/jacs.0c06400 |
| [24] |
Lump E, Castellano LM, Meier C, et al. A molecular tweezer antagonizes seminal amyloids and HIV infection[J]. Elife, 2015, 18: e05397. |
| [25] |
Röcker AE, Müller JA, Dietzel E, et al. The molecular tweezer CLR01 inhibits Ebola and Zika virus infection[J]. Antiviral Res, 2018, 152: 26-35. DOI:10.1016/j.antiviral.2018.02.003 |
| [26] |
Yu X, Zhang L, Tong L, et al. Broad-spectrum virucidal activity of bacterial secreted lipases against flaviviruses, SARS-CoV-2 and other enveloped viruses [J]. bioRxiv, 2020.05.22.109900. DOI: https://doi.org/10.1101/2020.05.22.109900.
|
| [27] |
Xu HT, Colby-Germinario SP, Hassounah S, et al. Identification of a pyridoxine-derived small-molecule inhibitor targeting dengue virus RNA-dependent RNA polymerase[J]. Antimicrob Agents Chemother, 2015, 60: 600-608. |
| [28] |
Maio N, Lafont BAP, Sil D, et al. Fe-S cofactors in the SARS-CoV-2 RNA-dependent RNA polymerase are potential antiviral targets[J]. Science, 2021, 373: 236-241. DOI:10.1126/science.abi5224 |
| [29] |
García CC, Damonte EB. Zn finger containing proteins as targets for the control of viral infections[J]. Infect Disord Drug Targets, 2007, 7: 204-212. DOI:10.2174/187152607782110004 |
| [30] |
Wang X, Xia S, Zhu Y, et al. Pan-coronavirus fusion inhibitors as the hope for today and tomorrow[J]. Protein Cell, 2021, 12: 84-88. DOI:10.1007/s13238-020-00806-7 |
| [31] |
Wang C, Zhao L, Xia S, et al. Design of α-helical lipopeptides targeting viral fusion proteins: a promising strategy for relatively broad-spectrum antiviral drug discovery[J]. J Med Chem, 2018, 61: 8734-8745. DOI:10.1021/acs.jmedchem.8b00890 |
| [32] |
Xia S, Yan L, Xu W, et al. A pan-coronavirus fusion inhibitor targeting the HR1 domain of human coronavirus spike[J]. Sci Adv, 2019, 5: eaav4580. DOI:10.1126/sciadv.aav4580 |
| [33] |
Xia S, Liu M, Wang C, et al. Inhibition of SARS-CoV-2 (previously 2019-nCoV) infection by a highly potent pan-coronavirus fusion inhibitor targeting its spike protein that harbors a high capacity to mediate membrane fusion[J]. Cell Res, 2020, 30: 343-355. DOI:10.1038/s41422-020-0305-x |
| [34] |
Zhang Y, Hou LX, Ju H, et al. Research progress of antiviral drugs targeting polymerase[J]. Chin J Med Chem (中国药物化学杂志), 2021, 31: 657-679. |
| [35] |
Sun YY, Kang DW, Gao SH, et al. Recent advances in the research of nucleoside antiviral agents[J]. Chin J Med Chem (中国药物化学杂志), 2021, 31: 55-75. |
| [36] |
Seley-Radtke KL, Thames JE, Waters CD. Broad spectrum antiviral nucleosides-our best hope for the future[J]. Annu Rep Med Chem, 2021, 57: 109-132. |
| [37] |
Sun L, Peng Y, Yu W, et al. Mechanistic insight into antiretroviral potency of 2'-deoxy-2'-β-fluoro-4'-azidocytidine (FNC) with a long-lasting effect on HIV-1 prevention[J]. J Med Chem, 2020, 63: 8554-8566. DOI:10.1021/acs.jmedchem.0c00940 |
| [38] |
Yan L, Yang Y, Li M, et al. Coupling of N7-methyltransferase and 3'-5' exoribonuclease with SARS-CoV-2 polymerase reveals mechanisms for capping and proofreading[J]. Cell, 2021, 184: 3474-3485. DOI:10.1016/j.cell.2021.05.033 |
| [39] |
Sourimant J, Lieber CM, Aggarwal M, et al. 4'-Fluorouridine is an oral antiviral that blocks respiratory syncytial virus and SARS-CoV-2 replication[J]. Science, 2021, 2: eabj5508. |
| [40] |
Meng H, Yu F, He YP, et al. A novel orally bioavailable broad-spectrum antiviral[J]. J Clin Med (临床药物治疗杂志), 2020, 18: 12-15. |
| [41] |
Shilatifard A. COVID-19: rescue by transcriptional inhibition[J]. Sci Adv, 2020, 6: eabc6891. DOI:10.1126/sciadv.abc6891 |
| [42] |
Robson F, Khan KS, Le TK, et al. Coronavirus RNA proofreading: molecular basis and therapeutic targeting[J]. Mol Cell, 2020, 79: 710-727. DOI:10.1016/j.molcel.2020.07.027 |
| [43] |
Kabinger F, Stiller C, Schmitzová J, et al. Mechanism of molnupiravir-induced SARS-CoV-2 mutagenesis[J]. Nat Struct Mol Biol, 2021, 28: 740-746. DOI:10.1038/s41594-021-00651-0 |
| [44] |
Gordon CJ, Tchesnokov EP, Schinazi RF, et al. Molnupiravir promotes SARS-CoV-2 mutagenesis via the RNA template[J]. J Biol Chem, 2021, 297: 100770. DOI:10.1016/j.jbc.2021.100770 |
| [45] |
Malone B, Campbell EA. Molnupiravir: coding for catastrophe[J]. Nat Struct Mol Biol, 2021, 28: 706-708. DOI:10.1038/s41594-021-00657-8 |
| [46] |
Hampton T. New flu antiviral candidate may thwart drug resistance[J]. JAMA, 2020, 323: 17. DOI:10.1001/jama.2019.20225 |
| [47] |
Toots M, Yoon JJ, Cox RM, et al. Characterization of orally efficacious influenza drug with high resistance barrier in ferrets and human airway epithelia[J]. Sci Transl Med, 2019, 11: eaax5866. DOI:10.1126/scitranslmed.aax5866 |
| [48] |
Sheahan TP, Sims AC, Zhou S, et al. An orally bioavailable broad-spectrum antiviral inhibits SARS-CoV-2 in human airway epithelial cell cultures and multiple coronaviruses in mice[J]. Sci Transl Med, 2020, 12: eabb5883. DOI:10.1126/scitranslmed.abb5883 |
| [49] |
Cox RM, Wolf JD, Plemper RK. Therapeutically administered ribonucleoside analogue MK-4482/EIDD-2801 blocks SARS-CoV-2 transmission in ferrets[J]. Nat Microbiol, 2021, 6: 11-18. DOI:10.1038/s41564-020-00835-2 |
| [50] |
Zhao L, Zhong W. Mechanism of action of favipiravir against SARS-CoV-2: mutagenesis or chain termination?[J]. Innovation, 2021, 2: 100165. |
| [51] |
Anokhina VS, Miller BL. Targeting ribosomal frameshifting as an antiviral strategy: from HIV-1 to SARS-CoV-2[J]. Acc Chem Res, 2021, 54: 3349-3361. DOI:10.1021/acs.accounts.1c00316 |
| [52] |
Bhatt PR, Scaiola A, Loughran G, et al. Structural basis of ribosomal frameshifting during translation of the SARS-CoV-2 RNA genome[J]. Science, 2021, 372: 1306-1313. DOI:10.1126/science.abf3546 |
| [53] |
Schlick T, Zhu Q, Dey A, et al. To knot or not to knot: multiple conformations of the SARS-CoV-2 frameshifting RNA element[J]. J Am Chem Soc, 2021, 143: 11404-11422. DOI:10.1021/jacs.1c03003 |
| [54] |
Neupane K, Zhao M, Lyons A, et al. Structural dynamics of single SARS-CoV-2 pseudoknot molecules reveal topologically distinct conformers[J]. Nat Commun, 2021, 12: 4749. DOI:10.1038/s41467-021-25085-6 |
| [55] |
Sun Y, Abriola L, Niederer RO, et al. Restriction of SARS-CoV-2 replication by targeting programmed-1 ribosomal frameshifting[J]. Proc Natl Acad Sci U S A, 2021, 118: e2023051118. DOI:10.1073/pnas.2023051118 |
| [56] |
Ahn DG, Yoon GY, Lee S, et al. A novel frameshifting inhibitor having antiviral activity against zoonotic coronaviruses[J]. Viruses, 2021, 13: 1639. DOI:10.3390/v13081639 |
| [57] |
Ai X, Wang D, Honko A, et al. Surface glycan modification of cellular nanosponges to promote SARS-CoV-2 inhibition[J]. J Am Chem Soc, 2021, 143: 17615-17621. DOI:10.1021/jacs.1c07798 |
| [58] |
Parker CG, Domaoal RA, Anderson KS, et al. An antibody-recruiting small molecule that targets HIV gp120[J]. J Am Chem Soc, 2009, 131: 16392-16394. DOI:10.1021/ja9057647 |
| [59] |
Liu X, Zhang B, Wang Y, et al. A universal dual mechanism immunotherapy for the treatment of influenza virus infections[J]. Nat Commun, 2020, 11: 5597. DOI:10.1038/s41467-020-19386-5 |
| [60] |
Painter MM, Zimmerman GE, Merlino MS, et al. Concanamycin A counteracts HIV-1 Nef to enhance immune clearance of infected primary cells by cytotoxic T lymphocytes[J]. Proc Natl Acad Sci U S A, 2020, 117: 23835-23846. DOI:10.1073/pnas.2008615117 |
| [61] |
Rao S, Lungu C, Crespo R, et al. Selective cell death in HIV-1-infected cells by DDX3 inhibitors leads to depletion of the inducible reservoir[J]. Nat Commun, 2021, 12: 2475. DOI:10.1038/s41467-021-22608-z |
| [62] |
Jones ST, Cagno V, Janeček M, et al. Multivalent flexible nanogels exhibit broad-spectrum antiviral activity by blocking virus entry[J]. ACS Nano, 2018, 12: 6429-6442. DOI:10.1021/acsnano.8b01616 |
| [63] |
Nie C, Pouyan P, Lauster D, et al. Polysulfates block SARS-CoV-2 uptake through electrostatic interactions[J]. Angew Chem Int Ed Engl, 2021, 60: 15870-15878. DOI:10.1002/anie.202102717 |
| [64] |
Muñoz A, Sigwalt D, Illescas BM, et al. Synthesis of giant globular multivalent glycofullerenes as potent inhibitors in a model of Ebola virus infection[J]. Nat Chem, 2016, 8: 50-57. DOI:10.1038/nchem.2387 |
| [65] |
Ramos-Soriano J, Reina JJ, Illescas BM, et al. Synthesis of highly efficient multivalent disaccharide/[60]fullerene nanoballs for emergent viruses[J]. J Am Chem Soc, 2019, 141: 15403-15412. DOI:10.1021/jacs.9b08003 |
| [66] |
Rodríguez-Pérez L, Ramos-Soriano J, Pérez-Sánchez A, et al. Nanocarbon-based glycoconjugates as multivalent inhibitors of ebola virus infection[J]. J Am Chem Soc, 2018, 140: 9891-9898. DOI:10.1021/jacs.8b03847 |
| [67] |
Stadtmueller MN, Bhatia S, Kiran P, et al. Evaluation of multivalent sialylated polyglycerols for resistance induction in and broad antiviral activity against influenza A viruses[J]. J Med Chem, 2021, 64: 12774-12789. DOI:10.1021/acs.jmedchem.1c00794 |
| [68] |
Nie C, Parshad B, Bhatia S, et al. Topology-matching design of an influenza-neutralizing spiky nanoparticle-based inhibitor with a dual mode of action[J]. Angew Chem Int Ed Engl, 2020, 59: 15532-15536. DOI:10.1002/anie.202004832 |
| [69] |
Nie C, Stadtmüller M, Yang H, et al. Spiky nanostructures with geometry-matching topography for virus inhibition[J]. Nano Lett, 2020, 20: 5367-5375. DOI:10.1021/acs.nanolett.0c01723 |
| [70] |
Nie C, Stadtmüller M, Parshad B, et al. Heteromultivalent topology-matched nanostructures as potent and broad-spectrum influenza A virus inhibitors[J]. Sci Adv, 2021, 7: eabd3803. DOI:10.1126/sciadv.abd3803 |
| [71] |
Falese JP, Donlic A, Hargrove AE. Targeting RNA with small molecules: from fundamental principles towards the clinic[J]. Chem Soc Rev, 2021, 50: 2224-2243. DOI:10.1039/D0CS01261K |
| [72] |
Meyer SM, Williams CC, Akahori Y, et al. Small molecule recognition of disease-relevant RNA structures[J]. Chem Soc Rev, 2020, 49: 7167-7199. DOI:10.1039/D0CS00560F |
| [73] |
Costales MG, Childs-Disney JL, Haniff HS, et al. How we think about targeting RNA with small molecules[J]. J Med Chem, 2020, 63: 8880-8900. DOI:10.1021/acs.jmedchem.9b01927 |
| [74] |
Li P, Wei Y, Mei M, et al. Integrative analysis of zika virus genome RNA structure reveals critical determinants of viral infectivity[J]. Cell Host Microbe, 2018, 24: 875-886. DOI:10.1016/j.chom.2018.10.011 |
| [75] |
Sun L, Xu K, Huang W, et al. Predicting dynamic cellular protein-RNA interactions by deep learning using in vivo RNA structures[J]. Cell Res, 2021, 31: 495-516. DOI:10.1038/s41422-021-00476-y |
| [76] |
Sreeramulu S, Richter C, Berg H, et al. Exploring the druggability of conserved RNA regulatory elements in the SARS-CoV-2 genome[J]. Angew Chem Int Ed Engl, 2021, 60: 19191-19200. DOI:10.1002/anie.202103693 |
| [77] |
Yang SL, DeFalco L, Anderson DE, et al. Comprehensive mapping of SARS-CoV-2 interactions in vivo reveals functional virus-host interactions[J]. Nat Commun, 2021, 12: 5113. DOI:10.1038/s41467-021-25357-1 |
| [78] |
Ruggiero E, Richter SN. Viral G-quadruplexes: new frontiers in virus pathogenesis and antiviral therapy[J]. Annu Rep Med Chem, 2020, 54: 101-131. |
| [79] |
Zhao C, Qin G, Niu J, et al. Targeting RNA G-quadruplex in SARS-CoV-2: a promising therapeutic target for COVID-19?[J]. Angew Chem Int Ed Engl, 2021, 60: 432-438. DOI:10.1002/anie.202011419 |
| [80] |
Melidis L, Hill HJ, Coltman NJ, et al. Supramolecular cylinders target bulge structures in the 5' UTR of the RNA genome of SARS-CoV-2 and inhibit viral replication[J]. Angew Chem Int Ed Engl, 2021, 60: 18144-18151. DOI:10.1002/anie.202104179 |
| [81] |
Melidis L, Styles IB, Hannon MJ. Targeting structural features of viral genomes with a nano-sized supramolecular drug[J]. Chem Sci, 2021, 12: 7174-7184. DOI:10.1039/D1SC00933H |
| [82] |
Haniff HS, Tong Y, Liu X, et al. Targeting the SARS-CoV-2 RNA genome with small molecule binders and ribonuclease targeting chimera (RIBOTAC) degraders[J]. ACS Cent Sci, 2020, 6: 1713-1721. DOI:10.1021/acscentsci.0c00984 |
| [83] |
Su X, Ma W, Feng D, et al. Efficient inhibition of SARS-CoV-2 using chimeric antisense oligonucleotides through RNase L activation[J]. Angew Chem Int Ed Engl, 2021, 60: 21662-21667. DOI:10.1002/anie.202105942 |
| [84] |
Chen XF, Zhao X, Yang Z. Aptamer-based antibacterial and antiviral therapy against infectious diseases[J]. J Med Chem, 2021, 64: 17601-17626. DOI:10.1021/acs.jmedchem.1c01567 |
| [85] |
Sun M, Liu S, Wei X, et al. Aptamer blocking strategy inhibits SARS-CoV-2 virus infection[J]. Angew Chem Int Ed Engl, 2021, 60: 10266-10272. DOI:10.1002/anie.202100225 |
| [86] |
Schmitz A, Weber A, Bayin M, et al. A SARS-CoV-2 spike binding DNA aptamer that inhibits pseudovirus infection by an RBD-independent mechanism[J]. Angew Chem Int Ed Engl, 2021, 60: 10279-10285. DOI:10.1002/anie.202100316 |
| [87] |
Sun M, Liu S, Song T, et al. Spherical neutralizing aptamer inhibits SARS-CoV-2 infection and suppresses mutational escape[J]. J Am Chem Soc, 2021, 143: 21541-21548. DOI:10.1021/jacs.1c08226 |
| [88] |
Whiting M, Muldoon J, Lin YC, et al. Inhibitors of HIV-1 protease by using in situ click chemistry[J]. Angew Chem Int Ed Engl, 2006, 45: 1435-1439. DOI:10.1002/anie.200502161 |
| [89] |
Schmidt MF, Isidro-Llobet A, Lisurek M, et al. Sensitized detection of inhibitory fragments and iterative development of non-peptidic protease inhibitors by dynamic ligation screening[J]. Angew Chem Int Ed Engl, 2008, 47: 3275-3278. DOI:10.1002/anie.200704594 |
| [90] |
Tauber C, Wamser R, Arkona C, et al. Chemical evolution of antivirals against enterovirus D68 through protein-templated knoevenagel reactions[J]. Angew Chem Int Ed Engl, 2021, 60: 13294-13301. DOI:10.1002/anie.202102074 |
| [91] |
Thun-Hohenstein STD, Suits TF, Malla TR, et al. Structure-activity studies reveal scope for optimisation of ebselen-type inhibition of SARS-COV-2 main protease[J]. ChemMedChem, 2022, 17: e202100582. |
| [92] |
Sies H, Parnham MJ. Potential therapeutic use of ebselen for COVID-19 and other respiratory viral infections[J]. Free Radic Biol Med, 2020, 156: 107-112. DOI:10.1016/j.freeradbiomed.2020.06.032 |
| [93] |
Menéndez CA, Byléhn F, Perez-Lemus GR, et al. Molecular characterization of ebselen binding activity to SARS-CoV-2 main protease[J]. Sci Adv, 2020, 6: eabd0345. DOI:10.1126/sciadv.abd0345 |
| [94] |
Zhang DW, Yan HL, Xu XS, et al. The selenium-containing drug ebselen potently disrupts LEDGF/p75-HIV-1 integrase interaction by targeting LEDGF/p75[J]. J Enzyme Inhib Med Chem, 2020, 35: 906-912. DOI:10.1080/14756366.2020.1743282 |
| [95] |
Thenin-Houssier S, de Vera IM, Pedro-Rosa L, et al. A small-molecule capsid inhibitor of HIV-1 replication[J]. Antimicrob Agents Chemother, 2016, 60: 2195-2208. DOI:10.1128/AAC.02574-15 |
| [96] |
De Clercq E. 1984-Discovery of the first anti-HIV drug, suramin[J]. Viruses, 2021, 13: 1646. DOI:10.3390/v13081646 |
| [97] |
Henß L, Beck S, Weidner T, et al. Suramin is a potent inhibitor of Chikungunya and Ebola virus cell entry[J]. Virol J, 2016, 13: 149. DOI:10.1186/s12985-016-0607-2 |
| [98] |
Yin W, Luan X, Li Z, et al. Structural basis for inhibition of the SARS-CoV-2 RNA polymerase by suramin[J]. Nat Struct Mol Biol, 2021, 28: 319-325. DOI:10.1038/s41594-021-00570-0 |
| [99] |
Bertolin AP, Weissmann F, Zeng J, et al. Identifying SARS-CoV-2 antiviral compounds by screening for small molecule inhibitors of nsp12/7/8 RNA-dependent RNA polymerase[J]. Biochem J, 2021, 478: 2425-2443. DOI:10.1042/BCJ20210200 |
| [100] |
Zeng J, Weissmann F, Bertolin AP, et al. Identifying SARS-CoV-2 antiviral compounds by screening for small molecule inhibitors of nsp13 helicase[J]. Biochem J, 2021, 478: 2405-2423. DOI:10.1042/BCJ20210201 |
| [101] |
De Paiva REF, Marçal Neto A, Santos IA, et al. What is holding back the development of antiviral metallodrugs? A literature overview and implications for SARS-CoV-2 therapeutics and future viral outbreaks[J]. Dalton Trans, 2020, 4: 16004-16033. |
| [102] |
Rothan HA, Stone S, Natekar J, et al. The FDA-approved gold drug auranofin inhibits novel coronavirus (SARS-COV-2) replication and attenuates inflammation in human cells[J]. Virology, 2020, 547: 7-11. DOI:10.1016/j.virol.2020.05.002 |
| [103] |
Gil-Moles M, Basu U, Büssing R, et al. Gold metallodrugs to target coronavirus proteins: inhibitory effects on the spike-ACE2 interaction and on PLpro protease activity by auranofin and gold organometallics[J]. Chemistry, 2020, 26: 15140-15144. DOI:10.1002/chem.202004112 |
| [104] |
Chuong C, DuChane CM, Webb EM, et al. Noble metal organometallic complexes display antiviral activity against SARS-CoV-2[J]. Viruses, 2021, 13: 980. DOI:10.3390/v13060980 |
| [105] |
Karges J, Kalaj M, Gembicky M, et al. ReI tricarbonyl complexes as coordinate covalent inhibitors for the SARS-CoV-2 main cysteine protease[J]. Angew Chem Int Ed Engl, 2021, 60: 10716-10723. DOI:10.1002/anie.202016768 |
| [106] |
Kuroki A, Tay J, Lee GH, et al. Broad-spectrum antiviral peptides and polymers[J]. Adv Healthc Mater, 2021, 10: e2101113. DOI:10.1002/adhm.202101113 |
| [107] |
Galindo I, Garaigorta U, Lasala F, et al. Antiviral drugs targeting endosomal membrane proteins inhibit distant animal and human pathogenic viruses[J]. Antiviral Res, 2021, 186: 104990. DOI:10.1016/j.antiviral.2020.104990 |
| [108] |
Weston S, Baracco L, Keller C, et al. The SKI complex is a broad-spectrum, host-directed antiviral drug target for coronaviruses, influenza, and filoviruses[J]. Proc Natl Acad Sci U S A, 2020, 117: 30687-30698. DOI:10.1073/pnas.2012939117 |
| [109] |
Yuan S, Chu H, Huang J, et al. Viruses harness YxxØ motif to interact with host AP2M1 for replication: a vulnerable broad-spectrum antiviral target[J]. Sci Adv, 2020, 6: eaba7910. DOI:10.1126/sciadv.aba7910 |
| [110] |
Huang R, Xu M, Zhu H, et al. Biological activity-based modeling identifies antiviral leads against SARS-CoV-2[J]. Nat Biotechnol, 2021, 39: 747-753. DOI:10.1038/s41587-021-00839-1 |
| [111] |
Zhang CH, Stone EA, Deshmukh M, et al. Potent noncovalent inhibitors of the main protease of SARS-CoV-2 from molecular sculpting of the drug perampanel guided by free energy perturbation calculations[J]. ACS Cent Sci, 2021, 7: 467-475. DOI:10.1021/acscentsci.1c00039 |
| [112] |
Sutanto F, Shaabani S, Oerlemans R, et al. Combining high-throughput synthesis and high-throughput protein crystallography for accelerated hit identification[J]. Angew Chem Int Ed Engl, 2021, 60: 18231-18239. DOI:10.1002/anie.202105584 |
| [113] |
Chamakuri S, Lu S, Ucisik MN, et al. DNA-encoded chemistry technology yields expedient access to SARS-CoV-2 Mpro inhibitors[J]. Proc Natl Acad Sci U S A, 2021, 118: e2111172118. DOI:10.1073/pnas.2111172118 |
| [114] |
El-Baba TJ, Lutomski CA, Kantsadi AL, et al. Allosteric inhibition of the SARS-CoV-2 main protease: insights from mass spectrometry based assays[J]. Angew Chem Int Ed Engl, 2020, 59: 23544-23548. DOI:10.1002/anie.202010316 |
| [115] |
Ferron F, Decroly E, Selisko B, et al. The viral RNA capping machinery as a target for antiviral drugs[J]. Antiviral Res, 2012, 96: 21-31. DOI:10.1016/j.antiviral.2012.07.007 |
| [116] |
Kasprzyk R, Spiewla TJ, Smietanski M, et al. Identification and evaluation of potential SARS-CoV-2 antiviral agents targeting mRNA cap guanine N7-methyltransferase[J]. Antiviral Res, 2021, 193: 105142. DOI:10.1016/j.antiviral.2021.105142 |
| [117] |
Abdelnabi R, Kovacikova K, Moesslacher J, et al. Novel class of chikungunya virus small molecule inhibitors that targets the viral capping machinery[J]. Antimicrob Agents Chemother, 2020, 64: e00649-20. |
| [118] |
Gigante A, Gómez-SanJuan A, Delang L, et al. Antiviral activity of [1, 2, 3]triazolo[4, 5-d]pyrimidin-7(6H)-ones against chikungunya virus targeting the viral capping nsP1[J]. Antiviral Res, 2017, 144: 216-222. DOI:10.1016/j.antiviral.2017.06.003 |
| [119] |
Delang L, Li C, Tas A, et al. The viral capping enzyme nsP1: a novel target for the inhibition of chikungunya virus infection[J]. Sci Rep, 2016, 6: 31819. DOI:10.1038/srep31819 |
| [120] |
Nieva JL, Madan V, Carrasco L. Viroporins: structure and biological functions[J]. Nat Rev Microbiol, 2012, 10: 563-574. DOI:10.1038/nrmicro2820 |
| [121] |
Caly L, Wagstaff KM, Jans DA. Nuclear trafficking of proteins from RNA viruses: potential target for antivirals?[J]. Antiviral Res, 2012, 95: 202-206. DOI:10.1016/j.antiviral.2012.06.008 |
| [122] |
Jin Z, Du X, Xu Y, et al. Structure of Mpro from SARS-CoV-2 and discovery of its inhibitors[J]. Nature, 2020, 582: 289-293. DOI:10.1038/s41586-020-2223-y |
| [123] |
Xiong B, Gui CS, Xu XY, et al. A 3D model of SARS_CoV 3CL proteinase and its inhibitors design by virtual screening[J]. Acta Pharmacol Sin, 2003, 24: 497-504. |
| [124] |
Du L, He Y, Zhou Y, et al. The spike protein of SARS-CoV-a target for vaccine and therapeutic development[J]. Nat Rev Microbiol, 2009, 7: 226-236. DOI:10.1038/nrmicro2090 |
| [125] |
Su S, Du L, Jiang S. Learning from the past: development of safe and effective COVID-19 vaccines[J]. Nat Rev Microbiol, 2021, 19: 211-219. DOI:10.1038/s41579-020-00462-y |
| [126] |
Yuan S, Wang R, Chan JF, et al. Metallodrug ranitidine bismuth citrate suppresses SARS-CoV-2 replication and relieves virus-associated pneumonia in Syrian hamsters[J]. Nat Microbiol, 2020, 5: 1439-1448. DOI:10.1038/s41564-020-00802-x |
| [127] |
Yang N, Tanner JA, Wang Z, et al. Inhibition of SARS coronavirus helicase by bismuth complexes[J]. Chem Commun (Camb), 2007, 12: 4413-4415. |
| [128] |
Lin MCM, Kung HF, Sun H, et al. Bismuth complexes inhibit the SARS coronavirus[J]. Angew Chem Weinheim Bergstr Ger, 2007, 119: 6584-6588. |
 2022, Vol. 57
2022, Vol. 57


