2. 中国中医科学院中药研究所, 北京 100700;
3. 云南大麻产业投资有限公司, 云南 昆明 650217;
4. 武汉理工大学化学化工与生命科学学院, 湖北 武汉 430070
2. Institute of Chinese Materia Medica, China Academy of Chinese Medical Sciences, Beijing 100700, China;
3. Yunnan Hemp Industrial Investment Co. Ltd., Kunming 650217, China;
4. School of Chemistry, Chemical Engineering and Life Sciences, Wuhan University of Technology, Wuhan 430070, China
生物和非生物胁迫, 如病原微生物、干旱、盐、重金属和高温等胁迫, 不利于植物的生长发育[1]。为避免外界胁迫造成的不利影响, 植物进化出了一套有效的自我防御机制, 如热激蛋白(heat shock protein 20, Hsp20), 主要在植物发育过程和应对非生物胁迫中起作用[2, 3]。热激蛋白的分子质量介于10~200 kDa, 依据分子质量和作用机制可将其分成6个亚家族: Hsp20、Hsp40、Hsp60、Hsp70、Hsp90和Hsp100, 由于Hsp20蛋白的分子质量介于15~42 kDa之间, 因此, Hsp20蛋白又被称为小热激蛋白(sHsp)[4]。Hsp20是由植物在高温等相关胁迫下产生的主要热激蛋白家族[5]。Hsp20蛋白是ATP非依赖型分子伴侣, 可形成200~800 kDa的低聚蛋白复合物, 由9至50个亚单位组成[6, 7]。Hsp20可以阻止真核细胞和原核细胞中蛋白质变性, 从而维持蛋白质的稳定性和正常功能[2, 8]。α-晶体蛋白结构域(ACD) 由大约80~100个氨基酸残基构成, 该结构域主要由β-sandwich结构组成, 其N端具有相对多样化的结构, 是Hsp20蛋白的特征结构域[9, 10]。ACD在底物相互作用中发挥功能, 其N端区域参与底物结合, C端延伸区域负责同源齐聚化[11-14]。ACD包含两个保守区域: 一个在N端共识区, 另一个在C端共同区通过疏水β6环连接, 这两个保守区分别由4个反平行片和3条β链组成[8, 15]。
植物Hsp20蛋白由核内多基因家族共同编码, 在不同种类的植物中, 该蛋白的数量各不相同[16]。例如拟南芥有19个Hsp20蛋白[17], 水稻有39个Hsp20蛋白[18], 大豆有51个Hsp20蛋白[19], 辣椒有35个Hsp20蛋白[20], 番茄有42个Hsp20蛋白[21]。Hsp20基因家族可响应多种环境胁迫。如在水稻中过表达OsHSP16.9基因可提高其抗盐和抗旱能力[22]。将MsHSP17.7基因转入拟南芥中并过表达, 发现转基因拟南芥对热、盐和氧化胁迫的耐受性有所提高[23]。此外, Hsp20基因还能调控植物的生长发育过程, 如VvHsp20基因参与了葡萄果实的发育[24]; 在拟南芥中, Hsp20基因会影响拟南芥种子早期发育[25]; 在小麦中, 研究人员发现叶绿体小热激蛋白(sHSP26) 不仅参与种子的成熟和萌发, 还能提高种子对高温的耐受性[26]。因此, 植物Hsp20蛋白是一类多功能的小分子蛋白。
大麻(Cannabis sativa L.) 属于大麻科(Cannabinaceae) 大麻属(Cannabis), 一年生草本植物, 在医药、食品、纺织等工业领域应用广泛[27]。陈士林团队率先提出了药用大麻(non-psychoactive medicinal cannabis) 的定义: 即大麻植株中四氢大麻酚(THC) 含量小于0.3%, 大麻二酚(CBD) 含量高[28]。药用大麻富含酯类、萜类和黄酮类等活性物质, 其中的大麻素成分具有神经保护作用[29]。研究表明CBD不仅具有抗炎、抗焦虑和镇痛等功效, 而且对神经精神性疾病具有一定的疗效[29, 30]。大麻的种子, 又称火麻仁, 富含大量的营养成分, 如蛋白质、不饱和脂肪酸和微量元素, 是一种良好的药食同源材料, 火麻仁已被开发成火麻油、饮料、高蛋白营养品等[31]。火麻仁味甘性平, 临床应用广泛, 不仅能用于润燥、滑肠、通淋和便秘, 还有助于缓解月经紊乱、癫痫等临床症状[31]。现代研究表明, 火麻仁富含多种活性物质, 其中独特的酚类和生物活性肽, 具有抗氧化、抗炎、神经保护、降血压等药理作用[32]。因此, 通过培育高油高蛋白含量的火麻仁基原植物, 进而获得优质的火麻仁资源是目前大麻品种选育的研究重点。随着大麻全基因组测序的完成, 大麻遗传信息获得解析[33], 然而, 在大麻中, Hsp20基因家族成员尚未确定且功能缺少系统研究。所以基于基因结构及功能预测分析, 挖掘调控大麻生长发育的功能基因至关重要。
本研究采用生物信息学技术在大麻中鉴定了CsHsp20基因家族成员, 分析了其序列特征、系统发育、基因结构及保守基序(motif)、共线性关系、顺式作用元件、蛋白互作网络及表达模式, 并利用同源建模法预测了CsHsp20基因家族的蛋白3D结构。为后续CsHsp20基因家族功能研究和火麻仁基原植物的定向培育奠定了基础。
材料与方法CsHsp20基因的全基因组鉴定 从拟南芥数据库TAIR (https://www.arabidopsis.org/) 中检索拟南芥Hsp20蛋白序列并下载, 然后用作查询, 对大麻蛋白质数据库进行BLASTP搜索(value为1e-5)。从PFAM数据库(http://pfam.xfam.org/) 中下载Hsp20蛋白保守域(PF00011), 基于大麻蛋白质数据库, 通过HMMER 3.0软件搜索具有该结构域的蛋白(value为1e-5)。将上述结果进行合并去重, 剩下的序列作为大麻Hsp20候选蛋白并提交到NCBI-CDD (https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi) 和InterPro (http://www.ebi.ac.uk/interpro/) 数据库中来验证Hsp20的保守域。没有Hsp20的保守结构域或分子质量在15~42 kDa外的序列被排除。所有非冗余和高可信度的序列均被确定为大麻Hsp20 (CsHsp20) 基因家族成员。依据它们的染色体位置命名。蛋白质的理化性质如分子质量(Mw)、等电点(pI) 和亲水性指数(GRAVY) 基于ExPASy (https://web.expasy.org/protparam/) 进行研究, 利用CELLO v2.5 (http://cello.life.nctu.edu.tw/) 进行亚细胞定位预测。
CsHsp20基因的系统发育分析 利用源自拟南芥、水稻、大豆的Hsp20蛋白的全长氨基酸序列[7]与本研究鉴定的35个CsHsp20蛋白序列联合进行系统发育分析。所有Hsp20蛋白序列均采用ClustalW工具进行多序列比对, 然后利用MGEA 7.0软件构建系统发育树(采用NJ邻接法), bootstrap值设置为1 000。
CsHsp20基因结构及保守motif分析 大麻基因组注释文件来自NCBI数据库(https://www.ncbi.nlm.nih.gov/)。使用在线软件Gene Structure Display Server (GSDS2.0) (http://gsds.gao-lab.org/index.php) 来展示基因的结构。MEME在线工具(http://meme-suite.org/) 用来分析CsHsp20蛋白的保守基序。最大motif数设置为10。
CsHsp20家族成员在染色体上的分布及共线性分析 基于本课题组自测的大麻基因组数据, 使用MapChart软件来绘制CsHsp20基因的位置。利用TBtools工具中的MCScanX程序来分析基因复制事件, 使用默认参数。TBtools工具来展示大麻和其他4个物种(拟南芥、水稻、葡萄和玉米) 之间的Hsp20基因共线性关系。
CsHsp20基因启动子区调控元件分析 从大麻全基因组数据库中提取35个CsHsp20基因启动子区域的序列(上游1.5 kb), 然后提交到PlantCARE (http://bioinformatics.psb.ugent.be/webtools/plantcare/html/) 中来预测顺式作用元件。利用TBtools工具进行可视化。
CsHsp20基因互作网络分析 由于拟南芥和大麻之间的Hsp20蛋白序列具有同源性, 因此基于二者之间的同源序列, 使用STRING数据库(https://string-db.org/) 研究CsHsp20蛋白的互作网络。利用Cytoscape软件来展示预测的互作网络。
CsHsp20基因家族成员表达模式分析 利用课题组前期自测的高CBD含量品种大麻(AA126) 的根、茎、叶、雌花、雄花和种子的转录组数据及5个不同发育时期(5个时期依据种子的发育状态来定义, S1: 第一阶段, 顶端分生组织, 雌花未出现; S2: 第二阶段, 此时雌花已出现, 柱头呈现白色; S3: 第三阶段, 此时授粉完成, 柱头呈现橙色; S4: 第四阶段, 此时种子呈现绿色尚未成熟; S5: 第五阶段, 此时种子成熟且呈现出棕褐色) 的数据进行CsHsp20基因的表达模式分析, 基于FPKM值, 利用在线云平台工具(https://www.omicstudio.cn/index) 绘制热图, 进行可视化分析。
CsHsp20基因家族成员蛋白3D结构分析 三维结构决定着蛋白质所能够执行的特定功能, 对于理解蛋白质的作用至关重要。根据CsHsp20蛋白序列, 基于同源建模法, 利用Swiss-Model网站(https://swissmodel.expasy.org/) 获取了CsHsp20蛋白的三维结构模型。
结果与分析 1 CsHsp20基因的鉴定及理化性质分析通过HMM分析和BLASTP比对分别鉴定到了41个和47个大麻Hsp20基因, 将两次鉴定的结果合并、去除重复序列后提交到NCBI-CDD和InterPro数据库中验证ACD结构域。排除没有ACD结构域及分子质量不在15~42 kDa范围内的序列, 最终得到35个CsHsp20基因, 并依据它们的染色体定位进行命名。对这些CsHsp20蛋白的序列特征进行分析(表 1), 发现这些CsHsp20蛋白的氨基酸长度变化范围在133~324个氨基酸之间, 平均长度为184个氨基酸。此外, 蛋白质分子质量介于15.1~36.49 kDa, 理论等电点介于4.62~9.37, 等电点的平均值为6.45, GRAVY均为负值, 表明这些CsHsp20蛋白均为亲水性蛋白。亚细胞定位结果显示, 21个CsHsp20蛋白定位在细胞质中, 6个CsHsp20蛋白定位在叶绿体中、4个CsHsp20蛋白定位在线粒体中, 还有4个CsHsp20蛋白定位在细胞核中。
| Table 1 Basic information of CsHsp20 genes identified in Cannabis sativa |
为了研究CsHsp20基因家族的系统发育关系, 选取了19个拟南芥Hsp20蛋白序列、20个水稻Hsp20蛋白序列(其中1个序列差异太大被排除) 和46个大豆Hsp20蛋白序列, 用来构建系统发育树(图 1), 系统发育关系表明4个物种共121个Hsp20基因被分成12个亚家族, 这12个亚家族cytosol Is (CI)、(CII)、(CIII)、(CIV)、(CV)、(CVI)、(CVII)、mitochondria Is (MI)、(MII)、plastids (P)、peroxisomes (Po) 和endoplasmic reticulum (ER) 分别含有45、12、6、4、5、2、1、7、5、12、5和9个Hsp20基因。然而, 仍然有8个CsHsp20基因不能归到任何亚家族中。除了8个未分类的CsHsp20基因, 剩下的27个CsHsp20基因被分成10个亚家族。其中16个CsHsp20基因在CI-CV中, 这表明细胞质是CsHsp20家族成员的主要功能场所。
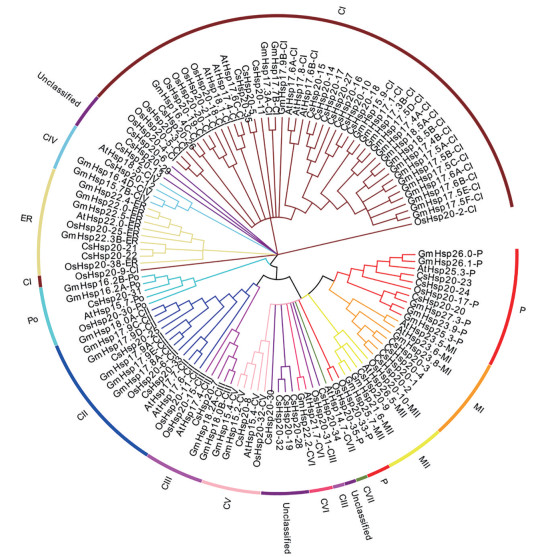
|
Figure 1 Phylogenetic tree of Hsp20 proteins from Cannabis sativa and other plants including Arabidopsis, rice and soybean was constructed using MEGA7.0 based on the NJ method; bootstrap was 1 000 replicates |
外显子-内含子结构不仅能反映基因进化的特征, 而且还为基因功能分化提供了重要的线索。CsHsp20家族成员的外显子-内含子结构分析结果显示(图 2), 19个CsHsp20基因无内含子, 15个CsHsp20基因仅含有1个内含子, 一个CsHsp20基因(CsHsp20-30) 含有6个内含子。处在同一亚家族中的CsHsp20家族成员, 它们的基因结构也相似。

|
Figure 2 Phylogenetic relationship, gene structure (A) and conserved motifs (B) of Hsp20 genes in Cannabis sativa. The yellow boxes and the black lines represent the exons and introns respectively. The conserved motifs analysis of the Hsp20 gene based on their phylogenetic relationship were identified using MEME software. Different color boxes represented the different types of motifs |
通过MEME网站预测了35个CsHsp20蛋白的保守motif, 鉴定到了10个保守的motif (图 2)。这些保守的motif长度介于11~50个氨基酸之间(图 3), 基于NCBI-CDD数据库对这10个motif进行注释发现, motif 1、motif 2和motif 3被注释为保守的ACD结构域。其中, 34个CsHsp20蛋白成员含有motif1, 含有motif 2和motif 5的CsHsp20成员各有32个, 此外, 18个CsHsp20蛋白成员含有motif 8, 16个CsHsp20蛋白成员含有motif 3。CsHsp20-5、CsHsp20-10、CsHsp20-11、CsHsp20-14、CsHsp20-15、CsHsp20-16、CsHsp20-17、CsHsp20-18、CsHsp20-27和CsHsp20-35蛋白所含的保守motif相似, 这10个成员在系统发育关系上同属CI亚家族, 这可能与其某些特定的功能相关。同一亚家族成员之间的motif组成相似, 这些结果表明CsHsp20基因家族成员在序列和功能上没有显著差异。
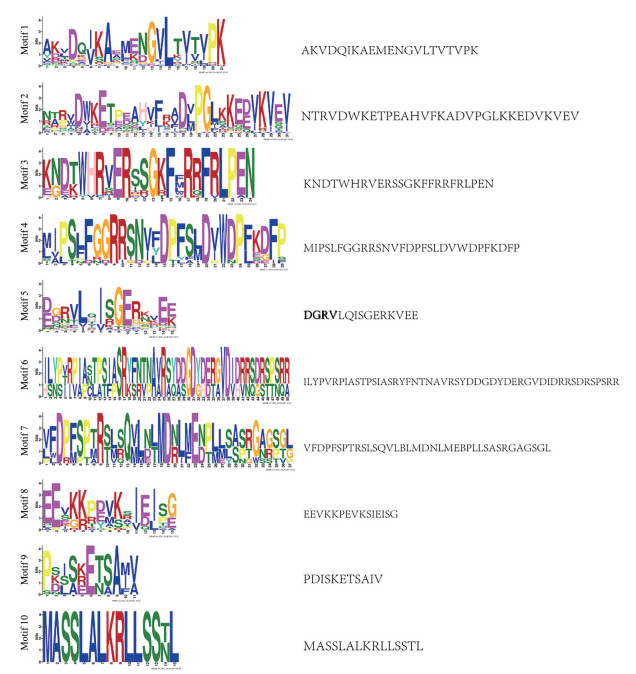
|
Figure 3 Ten conserved motifs of the CsHsp20 proteins |
由于在基因组注释文件中, CsHsp20-35基因无法定位在染色体上, 所以只有34个CsHsp20基因在9条大麻染色体上不均匀地分布(图 4)。1号和2号染色体上CsHsp20基因的数量最多, 分别有9个, 而3号染色体上只有1个CsHsp20基因, 这些结果表明, CsHsp20基因的数量与染色体长度之间无相关性。一个染色体区域内长度为200 kb的范围内包含两个或两个以上的基因被称为串联复制事件[34]。本研究发现2号染色体上CsHsp20-13和CsHsp20-14是一对串联复制基因; CsHsp20-14和CsHsp20-15是一对串联复制基因; CsHsp20-16和CsHsp20-17是一对串联复制基因; 4号染色体上CsHsp20-21和CsHsp20-22是一对串联复制基因。此外, 还鉴定到两对片段复制基因, 它们分别是CsHsp20-11和CsHsp20-26; CsHsp20-12和CsHsp20-27 (表 2)。
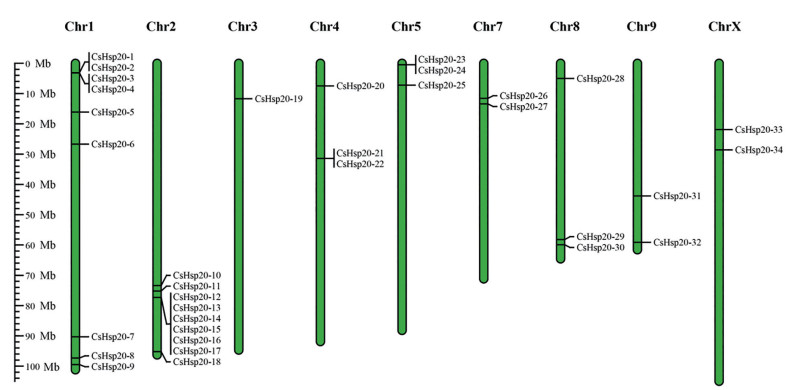
|
Figure 4 Chromosomal locations of CsHsp20 genes on Cannabis sativa chromosomes |
| Table 2 Segmentally and tandemly duplicated CsHsp20 gene pairs |
为了进一步研究CsHsp20基因家族的系统发育机制, 构建了4个大麻同源比较图谱, 其中包括两个单子叶植物(水稻和玉米) 和两个双子叶植物(拟南芥和葡萄) (图 5)。分别有6个、3个、12个和2个CsHsp20s基因与拟南芥、水稻、葡萄和玉米之间存在共线性关系。和这4个物种(拟南芥、水稻、葡萄和玉米) 之间的同源对分别有8对、3对、12对和3对。在大麻和拟南芥之间, CsHsp20-3和CsHsp20-10都有两个共线性基因对, 在大麻和玉米之间, CsHsp20-10有两个共线性基因对, 推测CsHsp20-3和CsHsp20-10可能在Hsp20基因家族进化过程中有着重要的作用。
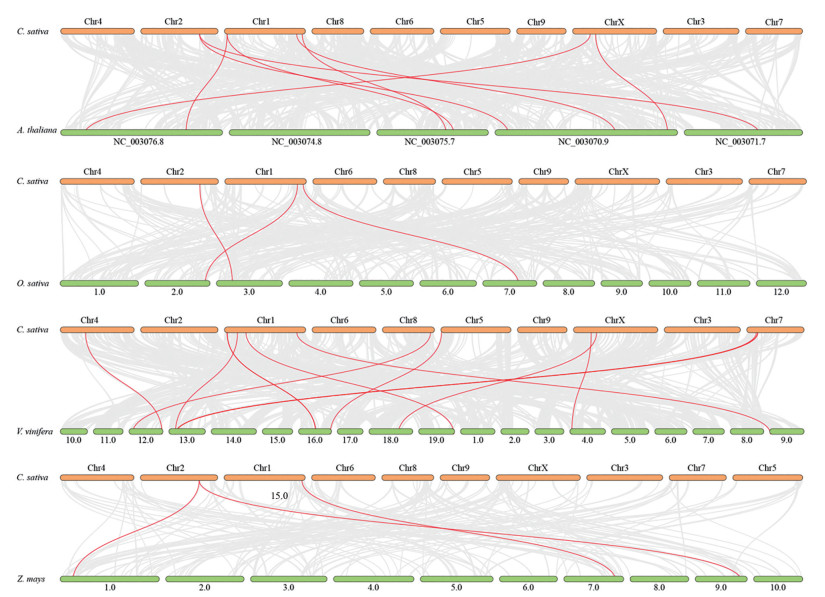
|
Figure 5 Synteny analysis of Hsp20 genes between Cannabis sativa and four representative plant species. Gray lines in the background indicate the collinear blocks within Cannabis sativa and other plant genomes, while the red lines highlight the syntenic Hsp20 gene pairs. The specie names with the prefixes 'C. sativa', 'A. thaliana', 'V. vinifera', 'O. sativa' and 'Z. mays' indicate Cannabis sativa, Arabidopsis thaliana, Vitis vinifera, Oryza sativa, and Zea mays, respectively |
基因启动子区的顺式作用元件往往能够反映出基因的功能。结果发现在这些CsHsp20基因的启动子上存在大量的与激素相关的元件, 然而, 在CsHsp20-22和CsHsp20-32两个基因上没有鉴定到筛选的这8个特定的顺式作用元件(图 6)。其中在7个CsHsp20基因中鉴定到了脱落酸响应元件(ABRE); 在5个CsHsp20基因中鉴定到了生长素响应元件(TGA); 在5个CsHsp20基因中鉴定到了茉莉酸甲酯响应元件(MeJA-responsive); 在4个CsHsp20基因中鉴定到了水杨酸响应元件(TCA); 在1个CsHsp20基因中鉴定到了赤霉素响应元件(gibberellin-responsive)。表明在大麻的生长发育过程中, Hsp20基因广泛参与激素代谢过程和信号转导过程。此外, 在这些CsHsp20基因中还鉴定到一些与胁迫相关的元件(图 6), 包括低温响应元件(LTR)、抗性与胁迫相应元件(TC-rich repeats) 和参与干旱胁迫诱导的MYB结合位点元件(MBS), 表明CsHsp20基因不仅参与大麻的生长发育, 而且还能响应生物与非生物胁迫。
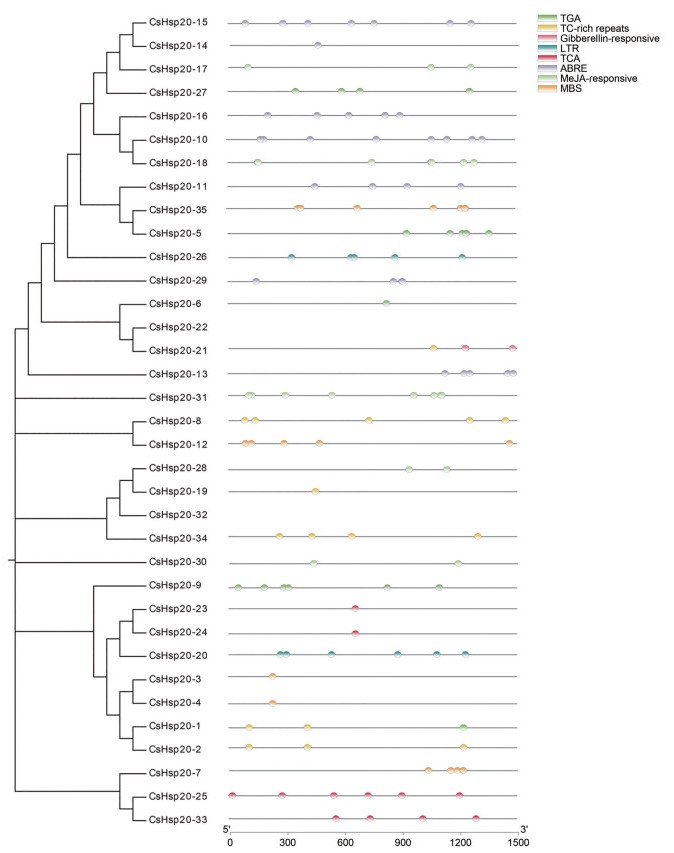
|
Figure 6 Predicted cis-elements in CsHsp20 promoters |
为了更好地了解CsHsp20基因家族参与的生物学功能和调控网络, 采用基于同源分析方法预测了它们之间的蛋白-蛋白相互作用(protein-protein interaction, PPI) (图 7)。发现了5个与拟南芥同源的CsHsp20蛋白和10个对应的互作蛋白。大部分与CsHsp20相互作用的蛋白是Hsp超家族成员。例如Hsp20、Hsp70、Hsp90和Hsp100。此外, 也发现了与CsHsp20蛋白相互作用的转录因子, 如HSFA2和Hop3, 这些互作蛋白可能在调控大麻Hsp20蛋白的功能上有着重要的作用。

|
Figure 7 Predicted protein-protein interaction networks of CsHsp20 proteins with other proteins in Cannabis sativa using STRING tool. The blue rectangle represent CsHsp20 proteins, and the circles on the inside represent proteins that interact with CsHsp20. Different colors including red, orange, green, pink, purple, and cyan represent HSFA2 transcription factor, Hsp20 family proteins, Hsp70 family proteins, Hsp90 family proteins, Hsp100 family proteins and Hop3 protein respectively |
为了研究CsHsp20基因的表达模式, 基于大麻的不同组织和不同生长时期的转录组数据绘制了热图(图 8)。结果表明在大麻的不同组织器官中, CsHsp20基因表达水平不同(图 8A)。另外发现绝大部分CsHsp20基因高表达的部位是种子, 例如, CsHsp20-10、CsHsp20-16、CsHsp20-29, 说明这些基因的表达与种子有关。此外, 还发现不同的发育时期中CsHsp20基因的表达存在差异(图 8B)。在S1~S5时期, 分别有5、7、5、13和19个CsHsp20基因高表达。从S1到S5时期, 种子逐渐发育成熟, 表达的CsHsp20基因的数量在增加。表明CsHsp20基因参与了大麻种子发育。
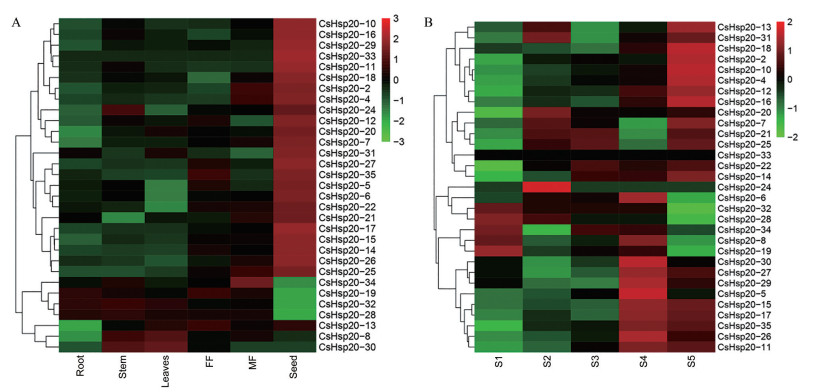
|
Figure 8 Expression profiles of CsHsp20 genes in different tissues including root, stem, leaves, female flower (FF), male flower (MF) and seed (A). Expression profiles of CsHsp20 genes in different stages, S1: Phase Ⅰ; S2: Phase Ⅱ; S3: Phase Ⅲ; S4: Phase Ⅳ; S4: Phase Ⅴ (B) |
基于同源建模法来预测CsHsp20基因家族的蛋白3D结构。28个CsHsp20蛋白序列与模板蛋白序列的一致度超过了30%, 35个CsHsp20蛋白序列与模板蛋白序列的平均一致度为51.98%, 表明预测结果是可靠的。这些CsHsp20蛋白的3D结构描述都是sHsp (Hsp20) 或ACD结构域。进一步说明之前所鉴定的结果是可信的。除了8个无法归类的CsHsp20蛋白外, 其他的CsHsp20蛋白可以依据系统发育树来分类, 同一亚家族成员的3D结构相似(图 9)。

|
Figure 9 The 3D structure modeling of CsHsp20 proteins |
本研究鉴定到了35个CsHsp20基因, 并通过生物信息学手段对其进行系统性研究, 解析了CsHsp20基因家族成员的理化性质、结构特点及潜在功能。转录组数据表明在大麻的不同组织器官及不同发育时期中, CsHsp20基因家族成员的表达水平均有差异, 它们主要在火麻仁及其成熟期高表达, 表明CsHsp20基因家族成员能调控火麻仁的生长发育。
35个CsHsp20基因被分成10个亚家族(CI、CII、CIII、CIV、CV、MI、MII、ER、P和Po)。而拟南芥的Hsp20基因家族成员可分为12个亚家族(CI-CVII、MI、MII、ER、P和Po)[17]。研究发现辣椒中Hsp20缺乏CIV、CV和CVIII亚家族[20], 水稻Hsp20家族缺乏CIV和CVII亚家族[35]。由此可见, 在植物中Hsp20亚家族存在基因缺失事件, CsHsp20亚家族的缺失可能是由于该家族成员在进化过程中被丢失。
基因结构分析表明97.14%的CsHsp20基因没有内含子或只有一个较短的内含子, 植物倾向于保留那些没有内含子或含有短内含子的基因[35]。CI、CII和ER亚家族的CsHsp20基因均无内含子, CIII、CIV、CV、MI、MII、P和Po亚家族成员只有一个内含子。这与之前的辣椒[20]和番茄[21]的报告基本一致。内含子的数量与基序排列进一步证实了CsHsp20基因分类的可靠性。研究表明含有较少或没有内含子的基因在植物中具有更高的表达水平[36, 37], 为了及时响应各种胁迫, 基因必须被快速激活, 而含有较少或没有内含子结构的基因可以被快速激活[38]。Hsp20基因能响应多种胁迫, 尤其是在热胁迫下高表达[7], 更进一步说明本研究是可靠的。
基因的复制事件影响基因家族的扩张和基因组的进化机制[39], 其中串联复制和片段复制是主要的复制模式[40]。在本次研究中, 35个CsHsp20基因不均匀地分布在9条大麻染色体上, 大麻基因组大小约是拟南芥的6.5倍, 但是CsHsp20基因的数量(35个) 是拟南芥(19个) 的1.8倍, 这种差异可能是由于两个物种的全基因组中基因复制事件导致的, 这与马铃薯中Hsp20基因的研究相似[7]。在大麻中有12个Hsp20基因存在复制事件, 包括2对片段复制和4组串联复制, 说明在CsHsp20基因家族的扩张和进化中, 串联复制和片段复制起着重要作用。物种间共线性分析结果显示, 一些同源基因对只出现在大麻和双子叶植物(拟南芥和葡萄) 之间, 而在大麻和单子叶植物(水稻和玉米) 之间不存在, 如CsHsp20-3、CsHsp20-33、CsHsp20-34与其他4个物种间的同源对只出现在拟南芥和葡萄中。这些结果说明在单子叶植物和双子叶植物开始分化形成以后才出现这些同源基因对。
Hsp20基因的表达模式已在许多物种中有过报道, 如水稻[18]、拟南芥[41]、辣椒[20]和番茄[21], 本研究首次在大麻中对CsHsp20基因的表达模式进行研究。植物的Hsp20基因没有统一的基因表达模式[7]。基于转录组数据, 发现CsHsp20基因的表达在大麻的不同组织器官及不同发育时期中存在差异, 其中绝大多数CsHsp20基因在火麻仁中有较高的表达, 少数基因在其他的组织器官中高表达。随着火麻仁的逐渐成熟, 越来越多的Hsp20基因在火麻仁中高表达, 说明CsHsp20基因参与了火麻仁的发育过程。基因启动子区的顺式作用元件在调控基因表达过程中起着重要作用。在CsHsp20基因的启动子上, 鉴定到了大量与激素响应和胁迫响应相关的作用元件, 说明CsHsp20基因家族成员参与了逆境胁迫响应。在CsHsp20基因的启动子区没有鉴定到与高温胁迫相关的顺式作用元件, 具体原因有待深入研究。激素能调节植物的生长和发育, 当植物受到环境胁迫时, 胁迫响应元件就会起作用来调节植物以应对周围的环境胁迫[24]。前人对于植物Hsp20基因的研究主要体现在其对逆境胁迫的响应上, 尤其是热胁迫响应, 如马铃薯[7]和小麦[42]等。本研究通过顺式作用元件分析预测了CsHsp20基因的潜在功能, 发现CsHsp20基因不仅能响应一些逆境胁迫, 如温度、干旱等, 而且可能会参与大麻生长发育, 转录组数据在一定程度上证明了该预测的可靠性。
Hsp超家族成员可以相互作用来调节植物的生长、发育和响应多种胁迫[9]。本研究中大部分与CsHsp20蛋白相互作用的是Hsp超家族成员, 这与茶树中的研究结果一致[9]。在CsHsp20蛋白互作网络分析中, 发现了蛋白Hop3。研究表明Hop能够连接Hsp70和Hsp90蛋白来调控拟南芥响应胁迫[42]。因此, Hop3可能作为一个分子伴侣连接CsHsp20和其他的Hsp超家族成员来调控大麻响应各种胁迫。此外, 研究已证实HSF转录因子能调控Hsp基因的表达, 参与植物的发育和响应非生物胁迫[43, 44]。本研究发现5个CsHsp20蛋白与HSFA2转录因子之间存在互作关系, 这表明CsHsp的表达可能也受转录因子HSFA2的调控。上述结果表明以Hop和HSF为主的大麻Hsp互作网络普遍存在, 且在大麻生长发育和胁迫响应中发挥重要作用。
本研究基于中药火麻仁基原植物大麻的基因组和转录组数据来鉴定CsHsp20基因家族成员, 并利用生物信息学手段对其进行系统性研究, 解析了CsHsp20基因家族成员的理化性质、结构特点、系统发育关系及潜在功能, 并通过转录组数据分析了CsHsp20基因家族成员的表达模式, 推测部分CsHsp20基因在调控火麻仁的生长发育中发挥着重要的作用。由于目前实验条件限制, 这些CsHsp20基因的生物学功能尚未得到验证。本研究为CsHsp20基因家族功能研究和优质火麻仁基原植物的定向培育奠定了基础。
作者贡献: 怀浩负责文章撰写及数据分析; 董林林、宁康负责实验设计及论文修改; 侯聪、代飞负责数据分析和实验材料的收集; 刘霞、汪鋆植指导文章撰写并提出修改意见; 陈士林负责论文设计及项目开展。
利益冲突: 所有作者均声明不存在利益冲突。
| [1] |
Fan K, Pan XF, Mao ZJ, et al. Identification and analysis of sHSP gene family in Gossypioides kirkii[J]. Acta Agron Sin (作物学报), 2021, 47: 1913-1926. |
| [2] |
Waters ER. The evolution, function, structure, and expression of the plant sHSPs[J]. J Exp Bot, 2013, 64: 391-403. DOI:10.1093/jxb/ers355 |
| [3] |
Mogk A, Bukau B. Role of sHsps in organizing cytosolic protein aggregation and disaggregation[J]. Cell Stress Chaperones, 2017, 22: 493-502. DOI:10.1007/s12192-017-0762-4 |
| [4] |
Zhao X, Zhang TT, Xing WT, et al. Genome-wide identification and expression analysis under temperature stress of HSP70 gene family in Dendrobium catenatum[J]. Acta Hort Sin (园艺学报), 2021, 48: 1743-1754. |
| [5] |
Sung DY, Kaplan F, Lee KJ, et al. Acquired tolerance to temperature extremes[J]. Trends Plant Sci, 2003, 8: 179-187. DOI:10.1016/S1360-1385(03)00047-5 |
| [6] |
Lee GJ, Vierling E. A small heat shock protein cooperates with heat shock protein 70 systems to reactivate a heat-denatured protein[J]. Plant Physiol, 2000, 122: 189-198. DOI:10.1104/pp.122.1.189 |
| [7] |
Zhao P, Wang D, Wang R, et al. Genome-wide analysis of the potato Hsp20 gene family: identification, genomic organization and expression profiles in response to heat stress[J]. BMC Genomics, 2018, 19: 61. DOI:10.1186/s12864-018-4443-1 |
| [8] |
Haslbeck M, Vierling E. A first line of stress defense: small heat shock proteins and their function in protein homeostasis[J]. J Mol Biol, 2015, 427: 1537-1548. DOI:10.1016/j.jmb.2015.02.002 |
| [9] |
Chen J, Gao T, Wan S, et al. Genome-wide identification, classification and expression analysis of the HSP gene superfamily in tea plant (Camellia sinensis)[J]. Int J Mol Sci, 2018, 19: 2633. DOI:10.3390/ijms19092633 |
| [10] |
Waters ER, Vierling E. Plant small heat shock proteins - evolutionary and functional diversity[J]. New Phytol, 2020, 227: 24-37. DOI:10.1111/nph.16536 |
| [11] |
Kirschner M, Winkelhaus S, Thierfelder JM, et al. Transient expression and heat-stress-induced co-aggregation of endogenous and heterologous small heat-stress proteins in tobacco protoplasts[J]. Plant J, 2000, 24: 397-411. DOI:10.1046/j.1365-313x.2000.00887.x |
| [12] |
Giese KC, Vierling E. Mutants in a small heat shock protein that affect the oligomeric state. Analysis and allele-specific suppression[J]. J Biol Chem, 2004, 279: 32674-32683. DOI:10.1074/jbc.M404455200 |
| [13] |
Basha E, Friedrich KL, Vierling E. The N-terminal arm of small heat shock proteins is important for both chaperone activity and substrate specificity[J]. J Biol Chem, 2006, 281: 39943-39952. DOI:10.1074/jbc.M607677200 |
| [14] |
Jaya N, Garcia V, Vierling E. Substrate binding site flexibility of the small heat shock protein molecular chaperones[J]. Proc Natl Acad Sci U S A, 2009, 106: 15604-15609. DOI:10.1073/pnas.0902177106 |
| [15] |
Bondino HG, Valle EM, Ten HA. Evolution and functional diversification of the small heat shock protein/alpha-crystallin family in higher plants[J]. Planta, 2012, 235: 1299-1313. DOI:10.1007/s00425-011-1575-9 |
| [16] |
Cui F, Taier G, Wang X, et al. Genome-wide analysis of the HSP20 gene family and expression patterns of HSP20 genes in response to abiotic stresses in Cynodon transvaalensis[J]. Front Genet, 2021, 12: 732812. DOI:10.3389/fgene.2021.732812 |
| [17] |
Scharf KD, Siddique M, Vierling E. The expanding family of Arabidopsis thaliana small heat stress proteins and a new family of proteins containing α-crystallin domains (Acd proteins)[J]. Cell Stress Chaperones, 2001, 6: 225-237. DOI:10.1379/1466-1268(2001)006<0225:TEFOAT>2.0.CO;2 |
| [18] |
Ouyang Y, Chen J, Xie W, et al. Comprehensive sequence and expression profile analysis of Hsp20 gene family in rice[J]. Plant Mol Biol, 2009, 70: 341-357. DOI:10.1007/s11103-009-9477-y |
| [19] |
Lopes-Caitar VS, de Carvalho MCCG, Darben LM, et al. Genome-wide analysis of the Hsp20 gene family in soybean: comprehensive sequence, genomic organization and expression profile analysis under abiotic and biotic stresses[J]. BMC Genomics, 2013, 14: 577. DOI:10.1186/1471-2164-14-577 |
| [20] |
Guo M, Liu JH, Lu JP, et al. Genome-wide analysis of the CaHsp20 gene family in pepper: comprehensive sequence and expression profile analysis under heat stress[J]. Front Plant Sci, 2015, 6: 806. |
| [21] |
Yu J, Cheng Y, Feng K, et al. Genome-wide identification and expression profiling of tomato Hsp20 gene family in response to biotic and abiotic stresses[J]. Front Plant Sci, 2016, 7: 1215. |
| [22] |
Jung YJ, Nou IS, Kang KK. Overexpression of Oshsp16.9 gene encoding small heat shock protein enhances tolerance to abiotic stresses in rice[J]. Plant Breed Biotech, 2014, 2: 370-379. DOI:10.9787/PBB.2014.2.4.370 |
| [23] |
Li ZY, Long RC, Zhang TJ, et al. Molecular cloning and characterization of the MsHSP17.7 gene from Medicago sativa L.[J]. Mol Biol Rep, 2016, 43: 815-826. DOI:10.1007/s11033-016-4008-9 |
| [24] |
Ji XR, Yu YH, Ni PY, et al. Genome-wide identification of small heat-shock protein (HSP20) gene family in grape and expression profile during berry development[J]. BMC Plant Biol, 2019, 19: 433. DOI:10.1186/s12870-019-2031-4 |
| [25] |
Dafny-Yelin M, Tzfira T, Vainstein A, et al. Non-redundant functions of sHSP-CIs in acquired thermotolerance and their role in early seed development in Arabidopsis[J]. Plant Mol Biol, 2008, 67: 363-373. DOI:10.1007/s11103-008-9326-4 |
| [26] |
Chauhan H, Khurana N, Nijhavan A, et al. The wheat chloroplastic small heat shock protein (sHSP26) is involved in seed maturation and germination and imparts tolerance to heat stress[J]. Plant Cell Environ, 2012, 35: 1912-1931. DOI:10.1111/j.1365-3040.2012.02525.x |
| [27] |
Bonini SA, Premoli M, Tambaro S, et al. Cannabis sativa: a comprehensive ethnopharmacological review of a medicinal plant with a long history[J]. J Ethonpharmacol, 2018, 227: 300-315. DOI:10.1016/j.jep.2018.09.004 |
| [28] |
Zhang JQ, Chen SL, Wei GF, et al. Cultivars breeding and production of non-psychoactive medicinal cannabis with high CBD content[J]. China J Chin Mater Med (中国中药杂志), 2019, 44: 4772-4780. |
| [29] |
Burstein S. Cannabidiol (CBD) and its analogs: a review of their effects on inflammation[J]. Bioorg Med Chem, 2015, 23: 1377-1385. DOI:10.1016/j.bmc.2015.01.059 |
| [30] |
Wu J, Yu HB. Recent advances in understanding the roles and molecular mechanisms of cannabidiol in neuropsychiatric disorders[J]. Acta Pharm Sin (药学学报), 2020, 55: 2800-2810. |
| [31] |
Wei F, Tu DP, Wang LP. Research progress in edible development and pharmacological action of hemp seed[J]. Chin J Gerontol (中国老年学杂志), 2015, 35: 3486-3488. |
| [32] |
Farinon B, Molinari R, Costantini L, et al. The seed of industrial hemp (Cannabis sativa L.): nutritional quality and potential functionality for human health and nutrition[J]. Nutrients, 2020, 12: 1935. DOI:10.3390/nu12071935 |
| [33] |
Hurgobin B, Tamiru-Oli M, Welling MT, et al. Recent advances in Cannabis sativa genomics research[J]. New Phytol, 2021, 230: 73-89. DOI:10.1111/nph.17140 |
| [34] |
Holub EB. The arms race is ancient history in Arabidopsis, the wildflower[J]. Nat Rev Genet, 2001, 2: 516-527. DOI:10.1038/35080508 |
| [35] |
Sarkar NK, Kim YK, Grover A. Rice sHsp genes: genomic organization and expression profiling under stress and development[J]. BMC Genomics, 2009, 10: 393. DOI:10.1186/1471-2164-10-393 |
| [36] |
Mattick JS, Gagen MJ. The evolution of controlled multitasked gene networks: the role of introns and other noncoding RNAs in the development of complex organisms[J]. Mol Biol Evol, 2001, 18: 1611-1630. DOI:10.1093/oxfordjournals.molbev.a003951 |
| [37] |
Ren XY, Vorst O, Fiers MW, et al. In plants, highly expressed genes are the least compact[J]. Trends Genet, 2006, 22: 528-532. DOI:10.1016/j.tig.2006.08.008 |
| [38] |
Chung BY, Simons C, Firth AE, et al. Effect of 5'UTR introns on gene expression in Arabidopsis thaliana[J]. BMC Genomics, 2006, 7: 120. DOI:10.1186/1471-2164-7-120 |
| [39] |
Jeffares DC, Penkett CJ, Bahler J. Rapidly regulated genes are intron poor[J]. Trends Genet, 2008, 24: 375-378. DOI:10.1016/j.tig.2008.05.006 |
| [40] |
Vision TJ, Brown DG, Tanksley SD. The origins of genomic duplications in Arabidopsis[J]. Science, 2000, 290: 2114-2117. DOI:10.1126/science.290.5499.2114 |
| [41] |
Siddique M, Gernhard S, von Koskull-Döring P, et al. The plant sHSP superfamily: five new members in Arabidopsis thaliana with unexpected properties[J]. Cell Stress Chaperones, 2008, 13: 183-197. DOI:10.1007/s12192-008-0032-6 |
| [42] |
Muthusamy SK, Dalal M, Chinnusamy V, et al. Genome-wide identification and analysis of biotic and abiotic stress regulation of small heat shock protein (HSP20) family genes in bread wheat[J]. J Plant Physiol, 2017, 211: 100-113. DOI:10.1016/j.jplph.2017.01.004 |
| [43] |
Liu H, Charng Y. Common and distinct functions of Arabidopsis class A1 and A2 heat shock factors in diverse abiotic stress responses and development[J]. Plant Physiol, 2013, 163: 276-290. DOI:10.1104/pp.113.221168 |
| [44] |
Hahn A, Bublak D, Schleiff E, et al. Crosstalk between Hsp90 and Hsp70 chaperones and heat stress transcription factors in tomato[J]. Plant Cell, 2011, 23: 741-755. DOI:10.1105/tpc.110.076018 |
 2022, Vol. 57
2022, Vol. 57


