2. 山东省中药质量控制与全产业链建设协同创新中心, 山东 济南 250355
2. Shandong Provincial Collaborative Innovation Center for Quality Control and Construction of the Whole Industrial Chain of Traditional Chinese Medicine, Jinan 250355, China
近年来, CRISPR/Cas9技术因其操作简便、实验周期短、效率高、脱靶率低等特点成为最常用的基因编辑技术, 在作物遗传育种、植物基因改造及农作物品种改良方面展现巨大的潜力[1-3]。CRISPR/Cas9基因编辑系统包括能够作为核酸酶切割双链DNA的Cas9蛋白以及一个人工融合的单向导RNA (single guide RNA, sgRNA), sgRNA决定靶序列的特异性[4, 5], 在CRISPR/Cas9基因编辑系统起着重要作用。前人研究表明, CRISPR/Cas9技术编辑效率依赖于Cas9和sgRNA的高表达[6, 7], 强组成型RNA聚合酶Ⅱ启动子如花椰菜花叶病毒35S (CaMV35S), 通常用于驱动Cas9表达, 而sgRNA通常由U6启动子驱动。U6启动子是一种RNA聚合酶Ⅲ启动子, 具有明确的转录起始位点, 能识别从G开始的高度保守的起始位点, 已被频繁用于驱动植物小RNA的高表达水平[8, 9], 并已成为驱动CRISPR/Cas9载体中sgRNA表达的首选。
研究表明, 同一物种基因组中存在多个不同表达的U6基因, 并非所有的U6启动子都能驱动基因表达且不同启动子转录效率并不相同[10, 11], U6启动子的高度保守性使其在不同的物种之间仍具有转录活性, 但在同源关系较远的不同物种之间中转录活性存在一定差异[12], 有文献报道内源性U6启动子可使sgRNA表达增加, 从而提高编辑效率[13, 14], 在大豆中, 使用大豆内源性U6启动子GmU6-10构建pCas9-GmU6-sgRNA载体突变率远高于使用拟南芥AtU6-26构建的pCas9-AtU6-sgRNA载体的突变率[15]。Long等[16]使用棉花内源性GhU6.3启动子驱动sgRNA发现其表达量是拟南芥AtU6-29启动子的6~7倍, 编辑的突变效率提高了4~6倍。目前, 已在拟南芥[17]、水稻[18]、小麦[19]、大豆[20]、玉米[21]、棉花[22]、番茄[23]等多种植物中利用植物自身的U6启动子建立CRISPR/Cas9基因组编辑系统, 实现目的基因的高效编辑。
金银花为忍冬科植物忍冬(Lonicera japonica Thunb.) 的干燥花蕾或带初开的花, 具有清热解毒、疏散风热的功效, 主要活性成分为黄酮类、有机酸、挥发油、三萜皂苷等, 开发利用前景广阔, 需求量逐年增加[24, 25]。然而, 关于忍冬U6启动子的研究尚未见报道, 严重制约了忍冬基因编辑技术的开展。本文通过PCR及农杆菌侵染烟草叶片瞬时转化方法克隆并筛选高转录活性的忍冬U6启动子, 对于构建忍冬CRISPR/Cas9基因编辑系统, 实现忍冬的分子育种具有重要意义。
材料与方法材料 忍冬、烟草植株栽培于山东中医药大学, 经山东中医药大学蒲高斌教授鉴定为忍冬Lonicera japonica Thunb.、烟草Nicotiana tabacum。pBI121双元表达载体由山东中医药大学药学院实验室保存。植物DNA提取试剂盒和5 minTM TA/Blunt-Zero Cloning Kit (南京诺唯赞生物科技股份有限公司); 通用型DNA纯化回收试剂盒、质粒小提试剂盒和DNA分子量Marker DL2000 (天根生化科技(北京) 有限公司); T4 DNA连接酶(NEB北京有限公司); Trans 5α Chemically Competent Cell (全式金生物公司); 限制性内切酶HindⅢ和BamHⅠ (赛默飞世尔科技公司); LBA4404 Chemically Competent Cell (上海唯地生物技术有限公司); 纤维素酶R-10、离析酶R-10、X-Gluc、10×PBS缓冲液(北京索莱宝科技有限公司); 引物由铂尚生物技术(上海) 有限公司负责合成。
忍冬U6启动子的克隆 取忍冬叶片, 采用植物基因组DNA提取试剂盒提取忍冬的DNA, 用保守的U6 RNA序列在忍冬基因组中搜索候选U6 RNA对应的U6启动子序列, 并设计PCR扩增引物(表 1), 进行PCR扩增。PCR反应程序为① 95 ℃ 3 min; 95 ℃ 30 s, 55 ℃ 30 s, 72 ℃ 1 min, 32个循环; 72 ℃ 5 min; 4 ℃保存。② 95 ℃ 3 min; 95 ℃ 30 s, 52 ℃ 30 s, 72 ℃ 1 min, 32个循环; 72 ℃ 5 min; 4 ℃保存。反应体系为ddH2O 11 μL、2×M5 Hiper plus Taq HiFi 10 μL、DNA模板1 μL、上下游引物各0.5 μL, 共20 μL体系; 1%琼脂糖凝胶电泳进行检测, 切胶回收获得目的DNA片段, 根据序列不同及长短差异将启动子分别命名为LjU61-F1、LjU61-F2、LjU62-F1、LjU62-F2。将启动子片段与pCE2 TA/Blunt-Zero载体连接并转化到Trans 5α感受态细胞中, 涂板并挑单克隆, 37 ℃、200 r·min-1摇菌, 将一次活化菌液送至铂尚生物技术有限公司测序, 随后用DNAMAN软件对测序结果进行序列比对分析, 正确的质粒分别命名为T-LjU61-F1、T-LjU61-F2、T-LjU62-F1、T-LjU62-F2。
| Table 1 Primer sequences used in this study |
启动子顺式作用元件分析 将测序结果提交启动子元件在线分析网站PlantCARE, 对忍冬U6启动子中的顺式作用元件进行分析。
融合表达载体的构建 分别用HindⅢ和BamHⅠ对T-LjU61-F1、T-LjU61-F2、T-LjU62-F1、T-LjU62-F2质粒及植物表达载体pBI121进行双酶切, 切胶回收目的片段, 将克隆得到的LjU6启动子取代pBI121载体中的CaMV35S启动子, 并与GUS基因连接, 从而构建pBI121融合表达载体。将融合表达载体转化Trans 5α感受态细胞, 其一次活化菌液经PCR验证正确后, 二次活化菌液并提取质粒。质粒转入LBA4404农杆菌感受态细胞中, 28 ℃倒置培养2 d, 挑取单克隆于LB液体培养基[卡那霉素(Kan) 50 μg·mL-1, 利福平(Rif) 20 μg·mL-1] 中进行一次活化, 对一次活化菌液进行PCR验证, 验证成功后, 保存一次活化菌液于-80 ℃备用。
不同启动子在烟草叶片中的瞬时表达分析 参考蒲艳等[23]的方法, 以LBA4404农杆菌感受态细胞作为阴性对照, CaMV35S-pBI121作为阳性对照。将构建的4个LjU6-pBI121融合表达载体以及阳性对照、阴性对照的一次活化农杆菌菌液划线培养, 挑取单克隆于LB液体培养基中(Kan 50 μg·mL-1, Rif 20 μg·mL-1) 活化, 28 ℃、200 r·min-1摇菌至菌液A600值在0.6~0.8左右; 分别吸取不同体积的活化菌液接种到5 mL LB液体培养基(含50 μg·mL-1 Kan和20 μg·mL-1 Rif) 中, 使每个农杆菌起始菌液A值相同, 再次28 ℃、200 r·min-1摇菌至所有菌液的A600值为1.2时, 12 000 ×g离心5 min收集菌体, 弃上清液, 重悬于缓冲液(50 mmol·L-1 MgCl2、200 mmol·L-1吗啉乙磺酸、20 mmol·L-1乙酰丁香酮) 至1 mL, 室温放置3 h后分别注射到生长2~3周的烟草叶片中, 注射完成后做好标记, 进行过夜暗培养。用剪刀分别剪下暗培养过夜后的烟草叶片, 浸泡在100 μL β-葡萄糖苷酸酶(GUS) 染液中(20 mmol·L-1 X-Gluc, 50 mmol·L-1 PBS缓冲液), 抽真空30 min。每种农杆菌转化进行技术重复3次, 生物学重复3次。
将抽完真空后的烟草叶片置于37 ℃、180 r·min-1振荡染色6~7 h, 吸尽GUS染液。最后加入300 μL 95%乙醇进行脱色处理, 脱色时, 每3~4 h更换一次脱色液, 直至叶片绿色褪去, 观察脱色后的叶片并拍照, 根据染色情况筛选出高效转录的U6启动子。
结果与分析 1 忍冬启动子片段鉴定从忍冬基因组DNA中成功克隆出LjU61-F1、LjU61-F2、LjU62-F1、LjU62-F2共4个LjU6启动子片段, 长度分别为336、708、359、602 bp, 测序结果显示4个LjU6启动子片段序列正确。
2 启动子序列分析通过序列分析(表 2、3), 可以发现4个启动子中均含有TATA框以及CAAT框等典型的启动子顺式元件, LjU61中还存在AT富集区、脱落酸反应顺式作用元件ABRE (ACGTG)、防御和应激反应顺式作用元件TC-rich repeats (ATTCTCTAAC)、生长素反应元件AuxRE (TGTCTCAATAAG) 等[26], 此外还含有多个光反应或光反应相关的元件ACE (GACACGTATG)、G-box (TACGTG)、Box Ⅱ (TGGTAATAA)、GT1-motif (GGTTAA) 等, 与LjU61相比, LjU62中含有顺式作用元件种类较少, 主要为光反应或光反应相关的元件4cl-CMA1b (ATTCCGATAAACT)、Box4 (ATTAAT) 及GA-motif (ATAGATAA) 等, 此外还含有参与逆境反应元件CCAAT-box。
| Table 2 Analysis of the cis-acting elements of LjU61 promoter |
| Table 3 Analysis of the cis-acting elements of LjU62 promoter |
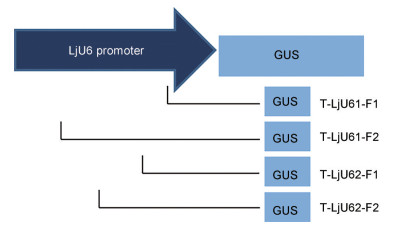
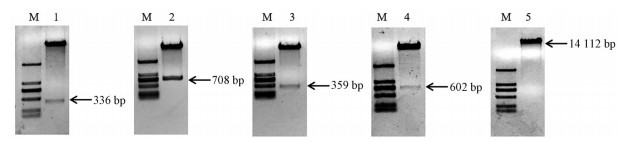
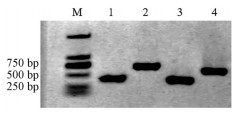
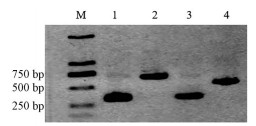
忍冬不同启动子驱动GUS基因的融合表达载体示意图如图 1。HindⅢ和BamHⅠ双酶切T-LjU61-F1、T-LjU61-F2、T-LjU62-F1、T-LjU62-F2质粒和植物表达载体pBI121, 1%琼脂糖凝胶电泳得到目的片段, 其大小与预测片段大小相符(图 2); 切胶回收不同LjU6片段及切除CaMV35S启动子后pBI121载体大片段, 将目的片段用T4连接酶连接后转化Trans 5α感受态细胞, 利用其一次活化菌液进行PCR鉴定, 1%琼脂糖凝胶电泳显示可扩增出启动子条带且大小与预期相符(图 3), 表明融合表达载体构建成功, 可进行下一步实验; 菌液二次活化后提取质粒, 将融合表达载体质粒转化LBA4404农杆菌感受态细胞, 对其一次活化菌液进行PCR鉴定, 1%琼脂糖凝胶电泳显示可扩增出目的条带且大小与预期相符(图 4), 表明转化成功。
|
Figure 1 Construction of GUS reporter gene fusion expression vector driven by LjU6 promoter |
|
Figure 2 Identification of different truncated T-LjU6 expression vectors by restriction enzyme digestion. M: 2 kb Plus DNA marker; 1: T-LjU61-F1; 2: T-LjU61-F2; 3: T-LjU62-F1; 4: T-LjU62-F2; 5: 35S-pBI121 enzyme section |
|
Figure 3 PCR detection of LjU6-pBI121 expression vector in Trans 5α competent cells. M: 2 kb Plus DNA marker; 1: LjU61-F1-pBI121 (336 bp); 2: LjU61-F2-pBI121 (708 bp); 3: LjU62-F1-pBI121 (359 bp); 4: LjU62-F2-pBI121 (602 bp) |
|
Figure 4 PCR detection of LjU6-pBI121 expression vector in LBA4404 competent cells. M: 2 kb Plus DNA marker; 1: LjU61-F1-pBI121 (336 bp); 2: LjU61-F2-pBI121 (708 bp); 3: LjU62-F1-pBI121 (359 bp); 4: LjU62-F2-pBI121 (602 bp) |
将各LjU6-pBI121融合表达载体通过农杆菌注射法瞬时转化烟草叶片, 并以CaMV35S-pBI121为阳性对照、LBA4404农杆菌为阴性对照, 通过GUS组织化学染色脱色后可发现, 阴性对照及LjU62-F1启动子处理叶片未观察到蓝色, CaMV35S、LjU61-F1、LjU61-F2、LjU62-F2启动子处理的烟草叶片可观察到蓝色, 说明克隆的4个LjU6启动子中有3个能驱动报告基因GUS的表达, 且转录活性并不相同(图 5)。

|
Figure 5 Expression of GUS driven by different LjU6 promoters in Nicotiana tabacum leaves. A: CaMV35S-pBI121 positive control; B: LjU61-F1:GUS; C: LjU61-F2:GUS; D: Agrobacterium negative control; E: LjU62-F1:GUS; F: LjU62-F2:GUS |
本研究首次克隆得到忍冬LjU6启动子, 并对其进行顺式作用元件分析, 前人研究表明启动子的结构包括顺式作用元件的排列位置及距离影响其与RNA聚合酶的识别、结合, 从而影响基因表达水平[27]; 同时, 顺式作用元件CAAT框对于基因的转录具有较强的激活作用, 在TATA框上游含有CAAT框将使基因的表达量大幅度增加[28]。在本研究中发现相较于LjU62, LjU61中含有更多的CAAT框, 这可能是其转录活性高于LjU62的一个原因。
本研究利用GUS作为报告基因, 通过瞬时转化烟草叶片后染色颜色深浅判断启动子活性, GUS组织化学染色结果显示, 克隆的4个U6启动子在烟草叶片中的转录效率并不相同。已有研究表明, 在保证U6启动子具有高转录活性的基础上, 较短的U6启动子更有利于构建CRISPR/Cas9基因编辑载体[29]。本实验结果也表明相对于序列较长的LjU61-F2, 截短的LjU61-F1的转录活性更高。Lei等[29]测定棉花6个不同长度U6启动子转录活性也发现启动子的序列越短, 其转录活性越高, 猜测其原因一方面可能是顺式作用元件的数目与位置影响其转录活性, 另一方面有可能是启动子序列过长, 在其上存在一些抑制因子, 使较长的U6启动子的转录活性下降[30], 此问题有待深入研究。LjU62-F1在GUS染色中未显蓝色, 观察序列发现LjU62-F1中除少数TATA以及光响应序列无其他结构, 其是否因为缺少某些元件而丧失了转录活性仍有待进一步研究。
作者贡献: 第一作者许小涵负责DNA提取、启动子克隆、烟草转化及论文撰写, 李小丽、唐志强和刘谦构建融合表达载体; 李佳、刘振华和张永清参与实验材料的准备及数据分析, 通讯作者蒲高斌指导选题及方案实施, 并对论文进行了修改。
利益冲突: 本文所有作者声明不存在利益冲突。
| [1] |
Cao HX, Wang W, Le HT, et al. The power of CRISPR-Cas9-induced genome editing to speed up plant breeding[J]. Int J Genomics, 2016, 2016: 5078796. |
| [2] |
Wang Y, Huang G, Yang H, et al. Advances on nonviral vectors of CRISPR/Cas9 system forgenome editing[J]. Acta Pharm Sin (药学学报), 2020, 55: 2606-2617. |
| [3] |
Nie LZ, Fang YY. Research progress of genome editing technology CRISPR/Cas9 and CRISPR/Cpf1 in crop[J]. J North Agric (北方农业学报), 2019, 47: 27-32. |
| [4] |
Shan QW, Gao CX. Research progress of genome editing and derivative technologies in plants[J]. Hereditas (遗传), 2015, 37: 953-973. |
| [5] |
Su YK, Qiu JR, Zhang H, et al. Advances in technology improvement and innovation of CRISPR/Cas9 system in plant genome editing[J]. Chin Bull Bot (植物学报), 2019, 54: 385-395. |
| [6] |
Ng H, Dean N. Dramatic improvement of CRISPR/Cas9 editing in Candida albicans by increased single guide RNA expression[J]. mSphere, 2017, 2: e00385-16. |
| [7] |
Jiang W, Zhou H, Bi H, et al. Demonstration of CRISPR/Cas9/sgRNA-mediated targeted gene modification in Arabidopsis, tobacco, sorghum and rice[J]. Nucleic Acids Res, 2013, 41: e188. DOI:10.1093/nar/gkt780 |
| [8] |
Preece R, Georgiadis C, Gkazi SA, et al. 'Mini' U6 Pol Ⅲ promoter exhibits nucleosome redundancy and supports multiplexed coupling of CRISPR/Cas9 effects[J]. Gene Ther, 2020, 27: 451-458. DOI:10.1038/s41434-020-0142-z |
| [9] |
Miyagishi M, Taira K. U6 promoter-driven siRNAs with four uridine 3' overhangs efficiently suppress targeted gene expression in mammalian cells[J]. Nat Biotechnol, 2002, 20: 497-500. DOI:10.1038/nbt0502-497 |
| [10] |
Ren C, Liu Y, Guo Y, et al. Optimizing the CRISPR/Cas9 system for genome editing in grape by using grape promoters[J]. Hort Res, 2021, 8: 52. DOI:10.1038/s41438-021-00489-z |
| [11] |
Wang MB, Helliwell CA, Wu LM, et al. Hairpin RNAs derived from RNA polymerase Ⅱ and polymerase Ⅲ promoter-directed transgenes are processed differently in plants[J]. RNA, 2008, 14: 903-913. DOI:10.1261/rna.760908 |
| [12] |
Bian SX, Han XL, Yuan GP, et al. Cloning and functional analysis of U6 promoter in apple[J]. Sci Agric Sin (中国农业科学), 2019, 52: 4364-4373. |
| [13] |
Wang C, Rollins JA. Efficient genome editing using endogenous U6 snRNA promoter-driven CRISPR/Cas9 sgRNA in Sclerotinia sclerotiorum[J]. Fungal Genet Biol, 2021, 154: 103598. DOI:10.1016/j.fgb.2021.103598 |
| [14] |
Ni XY, Lu WJ, Qiao X, et al. Genome editing efficiency of four Drosophila suzukii endogenous U6 promoters[J]. Insect Mol Biol, 2021, 30: 420-426. DOI:10.1111/imb.12707 |
| [15] |
Sun X, Hu Z, Chen R, et al. Targeted mutagenesis in soybean using the CRISPR-Cas9 system[J]. Sci Rep, 2015, 5: 10342. DOI:10.1038/srep10342 |
| [16] |
Long L, Guo DD, Gao W, et al. Optimization of CRISPR/Cas9 genome editing in cotton by improved sgRNA expression[J]. Plant Methods, 2018, 14: 85. DOI:10.1186/s13007-018-0353-0 |
| [17] |
Wu GF, Cheng HB, Wu YJ, et al. Identification of knockout of BRI1 mutant in Arabidopsis mediated by CRISPR/Cas9[J]. Bull Bot Res (植物研究), 2021, 41: 362-371. |
| [18] |
Yi Y, Zheng R, Yang B, et al. CRISPR/Cas9 mutants construction of rice zinc finger protein gene and mutation analysis[J]. J South Agric (南方农业学报), 2020, 51: 2607-2613. |
| [19] |
Sanchez-Leon S, Gil-Humanes J, Ozuna CV, et al. Low-gluten, nontransgenic wheat engineered with CRISPR/Cas9[J]. Plant Biotechnol J, 2018, 16: 902-910. DOI:10.1111/pbi.12837 |
| [20] |
Di YH, Sun XJ, Hu Z, et al. Enhancing the CRISPR/Cas9 system based on multiple GmU6 promoters in soybean[J]. Biochem Biophys Res Commun, 2019, 519: 819-823. DOI:10.1016/j.bbrc.2019.09.074 |
| [21] |
Zhang X, Shi YX, Lu BS, et al. Creation of new maize variety with fragrant rice like flavor by editing BADH2-1 and BADH2-2 using CRISPR/Cas9[J]. Sci Agric Sin (中国农业科学), 2021, 54: 2064-2075. |
| [22] |
Zang XY, Dai PH, Li JY, et al. Functional identification of cotton U6 promoters in the CRISPR/Cas9 genome editing system[J]. J Xinjiang Agric Univ (新疆农业大学学报), 2018, 41: 31-35. |
| [23] |
Pu Y, Liu C, Li JY, et al. Different SIU6 promoters cloning and establishment of CRISPR/Cas9 mediated gene editing system in tomato[J]. Sci Agric Sin (中国农业科学), 2018, 51: 315-326. |
| [24] |
Chinese Pharmacopoeia Commission. Pharmacopeia of People's Republic of China (中华人民共和国药典) [S]. Beijing: China Medical Science Press, 2020: 52, 230.
|
| [25] |
Qi LJ, Yuan Y, Wu C, et al. Bioinformatics analysis of DNA demethylase genes in Lonicera japonica Thunb[J]. Acta Pharm Sin (药学学报), 2015, 50: 367-371. |
| [26] |
Xie DJ, Ye YJ, Yang DM, et al. Cloning and analysis of the DXR gene and its promoter in Morinda officinalis[J]. Acta Pharm Sin (药学学报), 2020, 55: 335-344. |
| [27] |
Tang F, Tu HZ. Research progress of eukaryotic promoter[J]. China Forest Sci Technol (林业科技开发), 2015, 29: 7-12. |
| [28] |
Li T, Sun JK, Liu JT. Research advances on plant promote[J]. Biotechnol Bull (生物技术通报), 2015, 31: 18-25. |
| [29] |
Lei JF, Li Y, Xu XX, et al. Cloning and functional analysis of different truncated GbU6 promoters in cotton[J]. Acta Agron Sin (作物学报), 2016, 42: 675-683. |
| [30] |
Domitrovich AM, Kunkel GR. Multiple, dispersed human U6 small nuclear RNA genes with varied transcriptional efficiencies[J]. Nucleic Acids Res, 2003, 31: 2344-2352. DOI:10.1093/nar/gkg331 |
 2022, Vol. 57
2022, Vol. 57


