G蛋白偶联受体(G protein-coupled receptors, GPCRs) 是人体最大的膜蛋白受体家族, 人类基因组编码约800个GPCRs。GPCRs在各种生理和病理过程中起关键作用, 也是多种疾病的治疗靶点, 如精神疾病、免疫性疾病、心血管疾病和肿瘤等[1]。重链抗体(heavy chain antibody, hcAb) 是一种天然存在于驼科动物、仅由两条重链组成的抗体。重链抗体的重链可变区VHH (variable domain of heavy chain of heavy chain antibody) 分子质量较小, 约15 kDa, 又称为纳米抗体或单域抗体。纳米抗体具有良好的热稳定性及化学稳定性[2], 与常规抗体相比, 纳米抗体有更长的互补决定区3 (complementarity determining region 3, CDR3) 环, 因而具有更强结合“裂缝或空腔”的能力。2019年, 美国FDA批准了第1个纳米抗体类药物caplacizumab, 是纳米抗体类药物发展中一个重要的里程碑[3, 4]。本文就纳米抗体独特的生物物理特性及其在GPCRs研究中的应用进行详细阐述。
1 纳米抗体的结构特征及其生化特性 1.1 纳米抗体的结构特征1993年, Hamers-Casterman等[5]首次报道, 在骆驼的外周血中发现了一种天然没有轻链(light chain) 的抗体, 并将其重链可变区(variable region, VH) 称为纳米抗体。近些年来, 纳米抗体在基础医学研究、生物技术开发及分子成像等多个领域中扮演着越来越重要的角色[3]。
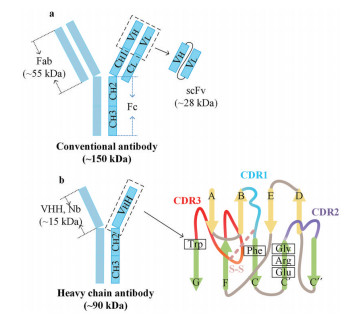
常规抗体是由两条轻链和两条重链(heavy chain) 组成的异源四聚体。轻链由可变区和恒定区(constant region, CL) 组成。重链由可变区和3个恒定区(constant region, CH1, CH2, CH3) 组成。轻链(VL和CL) 和重链中的VH和CH1构成了常规抗体的抗原结合片段(fragment antigen-binding, Fab), 重链中的CH2和CH3构成了常规抗体的可结晶片段(fragment crystallizable, Fc)。在一些抗体类药物中, 通过寡肽将VL和VH连接起来, 称为单链抗体(single-chain antibody fragment, scFv) (图 1a)[6, 7]。在羊驼、骆马和骆驼等驼科动物中, 发现了一种仅由两条重链组成的抗体, 由重链可变区VHH和恒定区CH2、CH3组成, 称为重链抗体。其中, VHH分子质量约15 kDa, 其半径仅2.5 nm, 长4 nm, 故又称为纳米抗体或单域抗体, 是自然界最小的完整抗原结合单元(图 1b)[8, 9]。与人类的VH区相似, VHH由3个互补决定区CDR和4个恒定框架区(framework region, FR) 交替排列组成, 且CDR3通常与CDR1或FR2之间形成二硫键[10]。与VH相比, VHH的CDR3环更长且FR2中几个高度保守的疏水氨基酸被替换为亲水氨基酸[11]。长的CDR3环使VHH形成一个凸的结合面, 有利于结合裂缝或空腔[12, 13]。而FR2变小及VHH亲水氨基酸的存在, 有助于提高纳米抗体的稳定性, 这可能是弥补轻链缺失而衍生出的新属性[14]。在软骨鱼体内也存在由两条重链组成的抗体, 称为Ig-NAR (immunoglobulin new antigen receptor), 其重链可变区为V-NAR[15, 16]。尽管V-NAR与VHH的可变序列存在很大的差别, 但软体鱼类和骆驼类的单域抗体在结构(不含轻链且带电和极性氨基酸频率增加) 及功能(靶向相似的抗原决定簇) 上均表现出惊人的趋同进化[17, 18]。
|
Figure 1 The structures of conventional antibody and nanobody. a: Conventional antibodies are composed of two heavy chains and two light chains. Fab (~55 kDa) constitutes the antigen-binding fragment of conventional antibody, VH and VL could be linked by peptide linker to form scFv (~28 kDa); b: Heavy chain antibodies consist of two heavy chains. VHH (~15 kDa) constitutes the antigen-binding fragment of nanobody, in which several highly conserved hydrophobic amino acids in conventional antibodies are replaced by hydrophilic amino acids (Phe, Gly, Arg, Glu). Some nanobodies have an extra disulfide bond (pink) between CDR3 (red) and CDR1 (blue), limiting the flexibility of CDR3 and CDR1. Fab: Fragment antigen-binding; scFv: Single-chain antibody fragment; VHH: Variable region of heavy chain of heavy chain antibody; Nb: Nanobody; CDR: Complementarity determining region |
与常规抗体相比, 纳米抗体具有良好的生化属性, 目前已有多种纳米抗体类药物被批准上市或处在临床试验的不同阶段(表 1), 其优势如下: ①分子质量小: 与Fab (~55 kDa) 和scFv (~28 kDa) 相比, 纳米抗体分子质量更小(~15 kDa)。纳米抗体可穿过血脑屏障(blood brain barrier, BBB), 应用于中枢神经系统疾病的治疗[19]; 也可被肾小球过滤迅速排出体外, 将有毒药物与纳米抗体偶联后, 应用于肿瘤疾病的治疗[2]; ②结合模式特殊: 与常规抗体相比, 纳米抗体的CDR3环更长, 形成一个凸的结合面[20], 可与蛋白质的一些“隐蔽”区域(如酶活性位点、病毒糖蛋白的凹陷区等) 结合[21]; ③易溶解, 稳定性高: 与常规抗体相比, 纳米抗体中几个高度保守的疏水氨基酸替换为亲水氨基酸, 提高了纳米抗体的可溶性和稳定性[12, 17]。纳米抗体可在高温和极端化学条件下保持结构和功能活性[22, 23]。此外, 纳米抗体还具有抗蛋白酶水解属性, 能抵抗胃蛋白酶、胰蛋白酶和糜蛋白酶的消化作用, 使口服纳米抗体类药物成为可能[20]; ④体内作用时间可人工干预: 因纳米抗体分子质量小, 可被肾小球过滤迅速排出体外, 致其血清半衰期短, 体内作用时间短[24]。可将纳米抗体与人血清白蛋白(human serum albumin, HSA) 结合或与人Fc结合, 提高纳米抗体血清半衰期, 延长其在人体内的作用时间[25]; ⑤免疫原性低: VHH与人类的VH表现出高度序列和结构同源性, 因此具有较低的免疫原性[12, 26]; ⑥可由大肠杆菌表达生产, 生产成本较低: 目前临床使用的抗体多通过大规模动物细胞培养生产, 成本较高。小尺寸和单域结构使纳米抗体更易通过细菌进行批量生产[27]。
| Table 1 Nanodody drugs in different phases of clinical trials. vWF: Von willebrand factor; aTTP: Acquired thrombotic thrombocytopenic purpura; TNF: Tumor necrosis factor; HSA: Human serum albumin; IL: Interleukin; VEGF: Vascular endothelial growth factor; Ang 2: Angiopoietin 2; TfR1: Transferrin receptor 1; FDA: Food and Drug Administration |
GPCRs由7个跨膜α螺旋(transmembrane, TM1-7)、3个细胞内环(intracellular loop, ICL1-3)、3个细胞外环(extracellular loop, ECL1-3) 和1个氨基末端(N端)、1个羧基末端(C端) 组成[28]。根据氨基酸序列同源性可将GPCRs分为5类: 视紫红质样家族(rhodopsin-like family, A类受体)、分泌素受体(secretin receptors, B1类受体)、黏附受体(adhesion receptors, B2类受体)、代谢型谷氨酸受体(glutamate receptors, C类受体) 和frizzled/taste 2受体(F类受体)[29]。A类受体是最大的GPCRs亚家族, 通常含有较短的N端, 该家族可进一步分为α、β、γ和δ亚类。B1类受体含有1个大的胞外肽结合域, 且ECL1、ECL2含有3个半胱氨酸残基, 其间的二硫键在N端形成了半胱氨酸桥网络, 这对于稳定N端结构域及增强N端与配体之间的相互作用至关重要。B2类受体是第二大GPCRs亚家族, 富含功能域, 大多数受体含有长的N端进而呈现不同的识别功能。C类受体具有大的胞外激动剂结合域, 可形成稳定的二聚体进行信号转导。F类受体也有长的N端[28, 30]。
GPCRs可识别多种细胞外信号如离子、小分子、酶、激素、神经递质和脂质等。与配体结合后, GPCRs构象发生变化, 触发下游级联信号, 从而调节不同生理过程[31]。对于构象灵活的GPCRs来说, 常规抗体因分子质量较大, 致其组织穿透力受限, 而分子质量小的纳米抗体则更有利于成为锁定GPCRs特定构象的药物。常规抗体的抗原结合部位会形成凹槽或腔与肽或半抗原相互作用, 不利于与“裂缝”结合, 而纳米抗体具有长的CDR3环, 可形成凸的结合面, 与GPCRs表面的“裂缝”高效结合[21]。特别对于A类GPCRs来说, 因其大部分结构嵌于膜中, 胞外仅暴露小部分天然配体结合位点且具有短的N端, 致使常规抗体的识别具有一定的难度。纳米抗体可靠近细胞膜并识别隐藏表位, 利用长的CDR3环高效地插入配体结合口袋(ligand binding pocket) 中[32, 33]。现就纳米抗体在GPCRs研究中的应用作简要介绍(表 2)。
| Table 2 Applications of nanobodies in G protein-coupled receptors research. β2AR: β2 Adrenoreceptor; M2R: M2 muscarinic acetylcholine receptor; μOR: μ-Opioid receptor; κOR: Kappa-opioid receptor; ORs: Opioid receptors; APJ: Apelin receptor |
GPCRs结晶存在两大障碍: 一是GPCRs构象的灵活摆动性; 二是晶格接触的极性表面积极小。例如β2肾上腺素受体(β2 adrenoreceptor, β2AR) 在TM5和TM6之间具有高度灵活的ICL3环。Rasmussen等[34]鉴定出一种单克隆抗体(Mab5) 与β2AR的ICL3结合, 并在bicelles中生长出分辨率为3.4 Å的β2AR-Mab5复合物晶体结构。Rosenbaum等[35]利用T4溶菌酶(T4 lysozyme, T4L) 取代了ICL3, 获得第一张高分辨率(2.4 Å) 的非活性态β2AR三维晶体结构图。Mab5和T4L虽有助于理解GPCRs跨膜信号转导, 但非活性态的GPCRs三维晶体图无法呈现GPCRs与G蛋白(Gs)、激动剂等的相互作用。为更好地观察β2AR的活性态结构, 通过参考人β2AR非活性态结构, 设计了一种β2AR共价激动剂, 其可通过二硫键共价连接到β2AR的特定位点, 由此成功获得了高分辨率(3.5Å) 的β2AR-共价激动剂复合物的晶体, 但受体的胞质面与非活性态结构难以区分[36]。荧光和核磁共振实验表明, GPCRs需与Gs或其他蛋白形成复合物, 其活性构象才能稳定。分子质量小且特异性高的纳米抗体具有长的CDR3环, 可作为GPCRs结晶的潜在促进剂。Rasmussen等[37]鉴定出一种Gs替代物纳米抗体Nb80, 其变构作用使β2AR与激动剂BI-167107的亲和力提高了100倍, 并借此获得第一张高分辨率(3.5 Å) 活性态β2AR三维晶体图(图 2a)。为进一步研究β2AR与Gs的相互作用, 在Gα亚基和Gβ亚基界面插入纳米抗体Nb35。BI-167107与β2AR-Gs复合物结合后, 在脂质立方相(lipidic cubic phase, LC) 介质中生长出分辨率为3.2 Å的晶体, 借助Nb35巧妙揭示了激动剂结合GPCRs后, 如何通过脂质双分子层引起GPCRs结构变化, 进而激活Gs的过程[38]。利用纳米抗体作为GPCRs结晶辅助因子从而获得高分辨率晶体结构, 揭示了GPCRs信号转导过程。目前已开发出几种功能性纳米抗体作为结晶辅助因子替代Gs, 以获得GPCRs高分辨率晶体结构并揭示信号转导过程[39]。此外, 为了更好地理解不同功效的激动剂如何调节GPCRs, Staus等[40]还开发了负变构纳米抗体, 以稳定GPCRs的非活性构象。
|
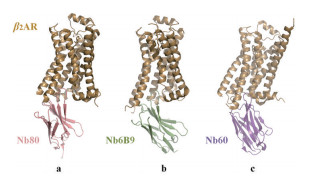
Figure 2 The crystal structures of nanobody-β2AR complexes. a: Side view of Nb80 (red) and β2AR (brown) complex (PDB ID: 3P0G); b: Side view of the complex of Nb80 variant Nb6B9 (green) and β2AR (brown) complex (PDB ID: 4LDE, 4LDL, 4LDO); c: Side view of negative allosteric nanobody Nb60 (purple) and β2AR (brown) complex (PDB ID: 5JQH) |
β2AR属A类GPCRs, 是人体在紧张状态下调节交感神经系统的膜蛋白受体, 也是治疗哮喘、慢性阻塞性肺气肿、心力衰竭和高血压等疾病的重要靶点[41, 42]。Rasmussen等[37]成功开发出Gs替代物Nb80 (图 2a)。β2AR-Nb80复合物结晶条件较为严苛, 仅可由BI-167107激活获得, 这对于理解不同亲和力激动剂如何激活GPCRs具有一定的局限性。Ring等[43]选用了3种亲和力不同的β2AR激动剂: BI-167107、高亲和力激动剂羟基苯异丙肾上腺素(hydroxybenzyl isoproterenol, HBI) 和低亲和力内源性激动剂肾上腺素。通过酵母展示技术和构象特异性遴选策略, 筛选出Nb80变异体Nb6B9。竞争结合实验显示, Nb6B9 (100 nmol·L-1) 展现出与Gs相当的亲和力。利用Nb6B9充当Gs替代物, 促使BI-167107、HBI、肾上腺素与β2AR结晶, 分别生长出X射线衍射率为2.8、3.1和3.2 Å的晶体(图 2b)。通过比较这些晶体结构, 可更好地了解不同亲和力激动剂对GPCRs构象改变的差异。为研究不同功效的配体对β2AR的影响, Staus等[40]鉴定出另一种纳米抗体Nb60, 其优先结合β2AR的反向激动剂(inverse agonist) 复合物, 并可大幅降低激动剂对β2AR的亲和力(图 2c)。因此, 将Nb60作为负变构纳米抗体, Nb80作为正变构纳米抗体分别稳定β2AR的非活性构象和活性构象。结果显示, Nb80/Nb60通过稳定β2AR活性构象/非活性构象, 促进激动剂/反向激动剂与β2AR的结合; 而Nb80和Nb60也可通过分别调节β2AR活性构象和非活性构象之间的平衡, 即“活性”和“非活性”状态的平均值, 进而调节部分激动剂激活受体的能力。此部分结果表明配体通过调节不同的受体状态进而控制受体激活, 改变传统观念认为配体依赖性GPCRs激活是单一非活性受体向活性受体转换的结果, 为靶向GPCRs药物的开发提供了重要信息。
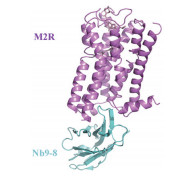
2.1.2 纳米抗体Nb9-8作为M2R的结晶辅助因子M2毒蕈碱型乙酰胆碱受体(M2 muscarinic acetylcholine receptor, M2R) 属A类GPCRs, 在调节神经系统和心脏生理活动中起关键作用[44], 是理解GPCRs信号变构调节的重要模型。Kruse等[45]将M2R与其高亲和力激动剂iperoxo组成的复合物免疫美洲驼, 生成免疫文库。利用酵母展示技术展示纳米抗体文库并结合流式细胞术筛选出Gs替代物纳米抗体Nb9-1、Nb9-8和Nb9-20, 均可增强M2R与激动剂的亲和力。其中Nb9-8可在高浓度下产生与Gs几乎相同的作用。M2R与Nb9-8结合后, 细胞内侧的TM6明显外移, 同时TM5有较小外移, TM7在NPxxY基序附近重排, 此过程为M2R活化的一个关键特征。利用Nb9-8成功获得了M2R与iperoxo单独结合, 以及与iperoxo和正向变构调节剂LY2119620同时结合的高分辨率X射线衍射结构(图 3), 经Phenix.refine后得到清晰的电子密度图, 这为理解M2R的激活机制和变构调节机制提供了大量信息。
|
Figure 3 The crystal structures of Nb9-8-M2R complex. Side view of Nb9-8 (blue) and M2R (purple) complex (PDB ID: 4MQT) |
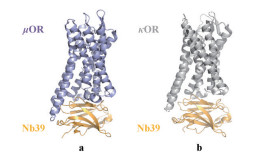
μ阿片受体(μ-opioid receptor, μOR) 属A类GPCRs, 是内源性阿片肽受体之一, 也是鸦片的主要受体。μOR激活后可产生镇静和镇痛的效果[46]。Huang等[47]借鉴β2AR、M2R获得活性构象晶体的经验, 将纳米抗体Nb39作为μOR的Gs替代物。Nb39的作用范围不仅限于吗啡喃激动剂BU72, 还可提高μOR对激动剂DAMGO和endomorphin-2的亲和力。利用Nb39成功在单油酸甘油酯脂质中间相(monoolein lipidic mesophase) 中获得BU72-μOR-Nb39复合物的晶体结构, 其分辨率可达2.1 Å (图 4a)。将此复合物晶体结构分别与失活的μOR、δ阿片受体(delta-opioid receptor, δOR) 及失活或活化的β2AR、M2R、视紫红质的晶体结构进行比较, 结果发现Nb39在复合物结构形成过程起到了很好的晶核聚集作用, 这为理解GPCRs激活的共性机制提供了新的见解。
|
Figure 4 The crystal structures of Nb39 with μOR or κOR complex. a: Side view of Nb39 (orange) and μOR (blue) complex (PDB ID: 5C1M); b: Side view of the complex of Nb39 (orange) and κOR (gray) (PDB ID: 6B73) |
κ阿片受体(kappa-opioid receptor, κOR) 属A类GPCRs, 也是内源性阿片肽受体之一。κOR可介导具有致幻、焦虑和镇痛效果的阿片类药物。由于μOR激动剂的不良反应大, 使κOR成为安全镇痛剂的替代靶点[48]。为更好地了解κOR激活的分子机制, Che等[49]将κOR与激动剂SalA (salvinorin A) 的复合物对美洲驼进行注射, 生成免疫文库, 利用噬菌体展示技术筛选κOR的Gs替代物。但筛选出的纳米抗体Nb6、Nb7仅与κOR结合, 不能稳定κOR的活性构象。在生物发光共振能量转移(bioluminescence resonance energy transfer, BRET) 实验中, Nb39可被κOR完全激动剂SalA及部分激动剂双丙诺啡以剂量依赖性方式募集到κOR, 而κOR拮抗剂JDTic不能募集Nb39, 表明Nb39也可稳定κOR的活性构象。环氧吗啡啉MP1104是κOR、μOR和δOR的强效激动剂。κOR-MP1104-Nb39三元复合物可在LCP中生长出3.1 Å的晶体(图 4b), 将此晶体结构与非活化κOR-JDTic晶体结构进行比较, 揭示了一种A类GPCRs的普遍激活机制, 即细胞外侧螺旋束的收缩促进细胞内侧螺旋束的开放, 从而调节转导蛋白结合。将此结构进一步与其他阿片类受体结构比较, 确定了κOR激活的关键氨基酸残基, 阐明了κOR亚型选择性和信号偏倚关键分子的决定因素。
2.2 纳米抗体作为GPCRs的生物传感器纳米抗体可在哺乳动物细胞内识别抗原, 且纳米抗体分子质量小, 更易对其序列进行简单的基因操作, 如将纳米抗体与荧光蛋白融合, 使其作为一种生化标签, 结合特异受体(GPCRs), 实现对细胞内生物学过程的实时动态监控[21]。
2.2.1 纳米抗体Nb80、Nb37作为β2AR的生物传感器Irannejad等[50]利用β2AR的Gs替代物Nb80, 开发了一种活化β2AR的生物传感器。Nb80选择性地在胞内结合激活状态的β2AR, 将Nb80与绿色荧光蛋白(green fluorescent protein, GFP) 融合表达后, 可在活细胞中示踪活化的β2AR。当β2AR被内源性激动剂异丙肾上腺素活化后, Nb80-GFP迅速富集到细胞膜上, 之后又与内化的β2AR共定位于胞质中。此过程揭示了内化后的β2AR仍处于活化状态并继续介导信号转导。而过去观点一直认为GPCRs与Gs介导的经典信号转导仅限于细胞膜。为探测GPCRs信号通路, 利用Nb37-GFP复合物开发了一种独特的生物传感器, 该传感器特异性地识别Gα无鸟嘌呤核苷酸的形式。当β2AR被内化至内体(endosome) 后, Nb37-GFP也被募集到内体上。在Gs过表达的细胞中, Nb37-GFP会均匀地定位于内化的β2AR上; 在Gs正常表达的细胞中, Nb37-GFP会不均匀地定位于内化的β2AR上。观察Nb37-GFP募集到内体的实时图像, 发现Gs激活动态地发生在内体膜的限定区域内。通过对细胞中cAMP水平的实时监测, 证明cAMP活化需内化的β2AR, 且内化的β2AR将有助于整个细胞中cAMP的循环。此研究利用GPCRs构象特异性纳米抗体Nb80、Nb37, 在哺乳动物活细胞中直接探测β2AR及Gs激活机制, 为GPCRs信号来自质膜和内体这一假说提供了直接支持。
2.2.2 纳米抗体VUN400作为CXCR4的生物传感器CXCR4属A类GPCRs, 与趋化因子配体CXCL12结合, 在艾滋病病毒(human immunodeficiency virus, HIV) 感染和肿瘤进程中起关键作用。CXCL12与CXCR4的N端相互作用, 然后与CXCR4的ECL2和跨膜螺旋内的结合口袋结合, 激活CXCR4[51]。纳米抗体VUN400是CXCR4的拮抗剂, 与CXCR4的ECL2结合, 作为特定CXCR4构象的探针。Soave等[52]在VUN400的C端融合了由11个氨基酸组成的HiBiT标签(VUN400-HiBiT), 该标签可与LgBiT-CXCR4结合形成完整功能性NanoLuc荧光素酶。通过互补发光(complemented luminescence) 效应来检测VUN400与CXCR4的相互作用, 正构(orthosteric) 和变构(allosteric) 配体与CXCR4结合会阻碍VUN400-HiBiT的结合, 从而使VUN400-HiBiT用作CXCR4变构作用的探针。此探针可用于受体药理学研究。
2.2.3 纳米抗体Nb33作为阿片受体的生物传感器鸦片类生物碱药物是目前已知最有效的镇痛药物之一。这类药物毒性较大, 人体容易对药物产生依赖性。阿片类药物与阿片受体(opioid receptors, ORs) 结合后, 产生下游生物学效应。如果能发现天然配体和镇痛药物在激活ORs过程的不同, 可能会揭示镇痛药物成瘾性的分子机制。Stoeber等[53]开发了一种基于纳米抗体Nb33的生物传感器, 可检测配体诱导ORs激活。荧光成像结果显示, 在无配体结合的情况下, Nb33较少与ORs结合; 当肽类激动剂DAMGO与ORs结合时, 大量Nb33与ORs结合, 而继续加入激动剂洗脱剂或拮抗剂后, 这种结合快速解除, 且ORs传感器可在不影响ORs功能的情况下对活细胞中ORs的活化状态进行直接特异性检测。通过此传感器揭示了肽类激动剂的激活模式, 即起始于质膜, 经受体内化后传至内体。而非肽类药物激活的ORs则起始于高尔基体(Golgi apparatus), 进而产生不同激活模式, 这为理解镇痛药物成瘾性提供了一个新视角。
2.3 纳米抗体作为GPCRs的拮抗剂/反向激动剂趋化因子是一类能趋化细胞定向移动的分泌蛋白, 可分为4个家族: CXC、CC、CX3C和XC, 与趋化因子受体结合[54]。趋化因子受体属A类GPCRs, 许多疾病都与趋化因子受体有关, 如类风湿关节炎、多发性硬化、肿瘤和HIV等疾病[55, 56]。靶向趋化因子受体作为重要的药物靶点, 相应的拮抗剂和反向激动剂也成为研究热点。拮抗剂和反向激动剂的实质均为拮抗受体, 但其作用机制不同。拮抗剂通过拮抗激动剂而抑制GPCRs活性; 反向激动剂通过拮抗GPCRs的固有活性而抑制GPCRs活性[57]。
2.3.1 纳米抗体BI 655088作为CX3CR1的拮抗剂CX3CR1是一种在单核细胞、巨噬细胞、树突状细胞和T细胞表面表达的趋化因子受体, 其表达与动脉粥样硬化密切相关[58]。Fractalkine (CX3CL1) 是已知唯一一个激活CX3CR1的配体。研究发现, 利用拮抗剂拮抗CX3CL1与CX3CR1的结合可阻碍CX3CR1激活, 进而降低CX3CR1的表达。Low等[59]发现纳米抗体BI 655088可有效抑制CX3CL1与CX3CR1的结合, 是CX3CR1的强效生物拮抗剂, 且可有效抑制小鼠体内动脉粥样硬化斑块的形成。现针对BI 655088的临床研究已进入I期阶段, 未来可能成为治疗慢性肾病的新型靶向药物。
2.3.2 纳米抗体238D2、238D4作为CXCR4的拮抗剂/反向激动剂CXCR4/CXCL12是研究最多的趋化因子受体/趋化因子对, 其对细胞的生长、修复再生、炎症反应及癌细胞的扩散有重要作用, CXCR4还是HIV的共受体[60, 61]。Jahnichen等[62]利用噬菌体展示技术筛选出CXCR4的两个竞争性拮抗剂纳米抗体238D2和238D4, 两者结合位点不同但存在部分重叠。目前认为使用短肽连接可很容易地生成具有更强效能的多价纳米抗体[63]。单价和双互补位纳米抗体(monovalent and biparatopic nanobodies) 会表现出不同作用模式。单价纳米体238D2和238D4表现为中性拮抗剂。而短肽连接的双互补位纳米抗体(238D2-15GS-238D4和238D2-20GS-238D4) 表现出更强的抑制效果并可作为CXCR4持续激活型突变体N3.35A的反向激动剂。CXCR4在多种肿瘤中过表达, 其基础活性的增加可促进肿瘤的进展, 反向激动剂可有效抑制其基础活性, 用于肿瘤治疗。在人MT-4细胞和外周血单个核细胞(peripheral blood mononuclear cells, PBMCs) 中, 238D2、238D4可抑制HIV-1毒株NL4.3的复制, 且238D2-15GS-238D4和238D2-20GS-238D4的抑制能力更强。238D2-20GS-238D4还可有效诱导模型动物食蟹猴CD34+干细胞的活性。这些研究为靶向CXCR4相关疾病药物的开发开辟了新途径。
2.3.3 纳米抗体Nb4作为CXCR7的拮抗剂/反向激动剂CXCR7在乳腺癌、肺癌和头颈癌等多种肿瘤中过表达。在一些肿瘤模型中, 利用小分子或小干扰RNA (siRNA) 抑制CXCR7已获得良好的治疗效果[64, 65]。CXCL12和CXCL11是CXCR7的天然配体, 与CXCR7具有高亲和力。Maussang等[66]构建了纳米抗体Nb4。Nb4由具有不同结合特性的纳米抗体Nb1和Nb3偶联形成, 可拮抗CXCL12与CXCR7的结合进而减少小鼠头颈癌肿瘤模型的生长。CXCR7是非典型趋化因子受体, 不通过Gs发出信号, 而是通过β-arrestin介导信号转导, 能以非配体依赖方式促进血管的生成和信号转导[67]。CXCL1在黑色素瘤和非小细胞肺癌等多种肿瘤血管生成中发挥重要作用。Nb4在抑制CXCR7的同时会减少血管生成因子CXCL1的分泌, 表明Nb4还可作为CXCR7的反向激动剂, 抑制CXCR7的基础活性, 进而抑制肿瘤血管生成, 减缓肿瘤生长速度, 使其成为头颈癌治疗的潜在纳米抗体类药物。
2.3.4 纳米抗体127D1-35GS-163E3作为CXCR2的拮抗剂CXCR2是最复杂的趋化因子受体, 可与CXCL1、CXCL8、CXCL5高亲和力结合, 也可与CXCL2、CXCL3、CXCL6、CXCL7低亲和力结合。CXCR2在慢性阻塞性肺气肿、哮喘和肿瘤转移等多种疾病中发挥重要作用[68]。Bradley等[69]详细分析了可结合CXCR2的纳米抗体2B2、54B12、127D1、97A9、163E3和163D2, 并将2B2、54B12和127D1划分为1类纳米抗体(N末端结合), 与CXCR2的前19个残基(1~19氨基酸) 高亲和力结合, 却不能完全抑制CXCL1与CXCR2结合, 仅表现出部分竞争性抑制; 将97A9、163E3和163D2划分为2类纳米抗体(胞外环结合), 能完全抑制CXCL1与CXCR2结合, 而其本身却不能高效地与CXCR2结合。1类和2类纳米抗体结合CXCR2的不同表位, 且在与CXCR2结合时, 同一类别的纳米抗体之间存在竞争, 而不同类别的纳米抗体不存在竞争。由此构建了1类和2类纳米抗体相结合的双互补位纳米抗体(127D1-35GS-163E3), 其可与CXCR2的两个不同表位相结合, 既保证了其与CXCR2高效结合, 又可有效抑制CXCL1与CXCR2的结合, 进而抑制CXCR2活性。
2.3.5 纳米抗体CA4910和CA5183作为ChemR23的拮抗剂ChemR23属A类GPCRs, 其结构与趋化因子受体相似, 可在多种白细胞中表达。ChemR23介导单核细胞、巨噬细胞、NK细胞和树突状细胞的趋化, 调节脂肪生成和代谢。ChemR23通过与chemerin结合介导趋化作用。Peyrassol等[70]利用噬菌体展示技术筛选出2种ChemR23的拮抗剂纳米抗体CA4910和CA5183。两者可特异性识别ChemR23, 与chemerin结合位点有所重叠但不完全相同。CA4910和CA518仅可部分抑制chemerin的激动剂效应; 但可完全抑制九肽chemerin (149~157) 的激动剂效应。此外, 与单价CA4910相比, 二价(bivalent) CA4910可更有效地抑制chemerin或chemerin (149~157) 诱导的胞内钙释放, 从而抑制chemerin诱导的人未成熟树突状细胞趋化, 表明其可作为ChemR23更强效的拮抗剂。这为研究chemerin/ChemR23系统在生理和病理条件下的作用提供了新工具。
2.3.6 纳米抗体US28-Nb作为US28的拮抗剂/反向激动剂US28属A类GPCRs, 由人巨细胞病毒(human cytomegalovirus, HCMV) 编码, 与人趋化因子受体家族具有同源性, 可与CX3CL1和CCL5等趋化因子结合。胶质母细胞瘤(glioblastoma, GBM) 是一种侵袭性很强的脑癌。在GBM中可检测出HCMV的DNA及其编码的蛋白质US28。US28已被证明具有致癌性[71], 且在小鼠原位颅内GBM模型中, US28过表达可加速GBM的生长。Heukers等[72]开发了靶向US28的纳米抗体US28-Nb, 可结合US28的N端从而特异性识别GBM组织。单价US28-Nb可作为US28的竞争性拮抗剂, 以剂量依赖方式有效抑制CX3CL1、CCL5与US28的结合, 并具有高度特异性, 不会影响CX3CL1与CX3CR1的结合。而二价US28-Nb不仅可作为US28的拮抗剂拮抗趋化因子, 还可在US28信号转导中起部分反向激动剂的作用。体内实验表明, 二价US28-Nb可降低US28介导的信号传导, 显著抑制HCMV/US28介导的肿瘤生长, 成为治疗HCMV肿瘤的潜在工具。光动力疗法(photodynamic therapy, PDT) 是一种有效治疗肿瘤的微创疗法, 通过近红外光局部激活光敏剂来根除肿瘤。De Groof等[73]在此实验基础上, 通过噬菌体展示技术, 筛选出新型结合US28的纳米抗体VUN100。VUN100与US28-Nb结合US28的表位相同, 即与US28的N端和ECL3环结合, 但与US28-Nb相比, 其结合US28的亲和力大幅提高。将水溶性光敏剂IRDye700DX与VUN100 C端标签上未配对半胱氨酸结合后, 可特异性杀死表达US28的GBM细胞。此研究首次将GPCR作为纳米抗体光动力靶标, 这为利用纳米抗体在过表达GPCRs肿瘤中进行靶向治疗提供了新思路。
2.4 纳米抗体(JN241-9、JN300) 作为GPCRs (APJ) 的激动剂APJ (apelin receptor) 属A类GPCRs, 在心肌细胞、血管内皮细胞和平滑肌细胞中表达。Aplien是APJ的内源性配体, 由Tatemoto等[74]在1998年从牛胃中提取并鉴定。Aplien激活APJ可治疗心力衰竭、心肌梗死和肺动脉高压等多种疾病[75]。
开发APJ激动剂是慢性心力衰竭一种潜在的治疗方法。Ma等[76]通过nanodiscs重建构象稳定的APJ蛋白, 将其作为免疫原免疫骆驼, 生成免疫文库, 通过噬菌体展示筛选出APJ的特异性纳米抗体。该方法共产生186种纳米抗体, 然而没有一种纳米抗体带有激动剂活性。通过分析强效纳米抗体拮抗剂JN241与APJ的晶体结构及其产生拮抗特性的关键残基, 并将此复合物晶体结构与APJ肽类强效激动剂AMG3054与APJ复合物晶体结构比较, 并进一步分析AMG3054与APJ的作用模式, 完成JN241向激动剂的转化, 即在JN241的CDR3的E104和S105之间插入一个酪氨酸, 将其转化为完全激动剂JN241-9。Ren等[77]在上述基础上, 将APJ特异性纳米抗体库亚克隆至含有衰变加速因子(decay accelerating factor, DAF) 的慢病毒载体上, 对稳定表达人APJ的U20S细胞系进行转染, 利用糖基磷脂酰肌醇(glycosylphosphatidylinositol, GPI) 将抗体锚定并展示于细胞表面, 通过β-arrestin报告系统对纳米抗体库进行筛选。利用流式细胞术对细胞进行三轮分选后, 成功鉴定出APJ激动剂JN300。氨基酸突变实验结果显示: APJ的N端和ECL1-3参与了激动剂JN300与APJ的结合。此系统可快速识别和筛选GPCRs的激动剂和拮抗剂, 也可用作为GPCRs抗体类药物的开发平台。
3 靶向GPCRs纳米抗体的开发纳米抗体可在原核、真核系统中重组表达, 并完全保留抗原结合特异性, 且纳米抗体的单结构域及良好的生化特性使其成为体外展示进行高亲和力选择的理想候选抗体[78]。靶向GPCRs的纳米抗体可通过多种途径获得, 如以噬菌体展示技术为代表的各种展示技术, 以及新发展的哺乳动物细胞定向进化平台VEGAS (viral evolution of genetically actuating sequences) 技术等。
3.1 噬菌体展示技术噬菌体展示技术由Smith[79]开发, 将编码外源肽的DNA序列插到M13噬菌体的基因组中, 使外源肽与M13噬菌体的G3P外壳融合表达并展示于噬菌体表面, 现已被广泛应用于抗体的筛选和改进中。目前获得纳米抗体最常见的流程是对驼科动物进行主动免疫后, 获取候选抗体序列文库并利用噬菌体展示技术进行筛选[80]。利用此技术已成功筛选出β2AR (Nb80[37]和Nb60[40])、μOR和κOR (Nb39[47, 49]) 的结晶伴侣; CX3CR1 (BI 655088[59])、CXCR4 (238D2、238D4[62])、CXCR7 (Nb4[66])、CXCR2 (127D1-35GS-163E3[69])、ChemR23 (CA4910、CA5183[70])、US28 (US28-Nb[72]) 的拮抗剂/反向激动剂; APJ (JN241-9[76]) 的激动剂等。
3.2 酵母展示技术酵母展示技术是一种基于真核细胞的展示技术。酵母细胞具有和哺乳动物相似的蛋白合成和翻译后修饰, 可更好地维持蛋白三维结构。虽然噬菌体展示技术能分离高亲和力的结合物, 但对于功能克隆(如构象选择性纳米体) 的鉴定仍存在短板, 而酵母展示技术可很好地解决这一短板[81]。α-凝集素和a-凝集素是酵母细胞壁上的两种甘露糖蛋白。a-凝集素的糖蛋白亚单位由Aga1p和Aga2p组成。Aga1p通过与β-葡萄糖共价连接锚定在酿酒酵母的细胞壁上; Aga2p通过两对二硫键与Aga1p相连, 其C端是a-凝集素的结合活性部位, 可用来展示异源蛋白。Boder等[82]通过将目的蛋白与酿酒酵母细胞壁蛋白Aga2p融合, 筛选出高亲和力抗体。利用此技术分别成功地筛选出β2AR、M2R的结晶伴侣即Nb6B9[43]和Nb9-8[45]。与噬菌体展示技术相比, 酵母展示技术可正确表达和展示多种功能的蛋白分子; 且酵母颗粒较大, 可利用流式细胞术进行大规模筛选。不足之处是酵母转化效率低, 抗体库的库容会受到限制。
3.3 哺乳动物细胞定向进化平台English等[83]利用VEGAS筛选靶向GPCRs的纳米抗体[84]。此系统利用α病毒Sindbis作为遗传、诱变和选择的载体, 可在转录因子(tetracycline transactivator, tTA)、GPCR (MRGPRX2) 和纳米抗体3个不同的应用水平实现定向进化。利用VEGAS在1周内获得了8个针对3种GPCRs (5-HT2A、DRD2、GPR68) 的纳米抗体, 并成功筛选出5-HT2A的正变构调节剂纳米抗体VGS-Nb2。这3个GPCRs分别偶联不同的Gα蛋白(Gαq、Gαi、Gαs), 表明VEGAS系统具有广泛适用性。VEGAS可在宿主细胞的框架内进化, 完全依赖于宿主细胞的翻译, 且具有高度诱变性, 简捷高效。
3.4 合成生物学合成生物学利用工程原理为生物医学重新设计生物系统。目前将高丰度的纳米抗体库引入哺乳动物细胞内具有一定挑战性。本课题组基于合成生物学原理致力于在哺乳动物细胞内建立基于线性双链DNA (linear double-stranded DNA) 的“与门”逻辑基因线路的纳米抗体库。通过PCR反应将完整基因表达模块拆分成两个线性双链DNA (double-stranded DNA, dsDNA) 分子, 在哺乳动物细胞内经非同源重组或同源重组途径重新形成完整的基因表达模块, 实现“与门”运算及基因表达[85]。利用“与门”运算, 尝试通过非同源重组、同源重组及体外连接3种途径在哺乳动物细胞内建立高丰度的纳米抗体库。在转染到HEK293T细胞后, 经非同源重组途径实现“与门”运算的dsDNA核苷酸末端会发生额外突变, 致使纳米抗体阅读框破坏, 仅鉴定出少量独特的纳米抗体序列; 而同源重组和体外连接两种方法均获得了超过100万种不同的纳米抗体序列, 成功在哺乳动物细胞内建立高丰度的纳米抗体库, 这为靶向GPCRs纳米抗体类药物的发展奠定坚实的基础[86, 87]。
4 总结与展望纳米抗体因其分子质量小、生化性能良好和生产成本低等特性成为下一代抗体类治疗药物。对于构象灵活的GPCRs, 特别是具有短N端的A类GPCRs来说, 纳米抗体长而暴露的CDR3环可识别GPCRs表面并与GPCRs的配体结合口袋结合。靶向GPCRs的纳米抗体现已得到广泛应用, 可作为GPCRs的结晶伴侣、生物传感器、拮抗剂/反向激动剂、激动剂等, 在结构生物学、生物技术、基础医学和分子成像等多领域发挥重要作用, 使纳米抗体成为靶向GPCRs抗体类药物的研究热点之一。但是目前还没有一种靶向GPCRs的纳米抗体药物上市, 此类药物的开发任重而道远。
作者贡献: 王怡和李帅为综述撰写思路; 李帅和赵雁杰对综述内容进行评估; 王怡执笔; 李帅和赵雁杰审校。
利益冲突: 所有作者均声明不存在利益冲突。
| [1] |
Hauser AS, Attwood MM, Rask-Andersen M, et al. Trends in GPCR drug discovery: new agents, targets and indications[J]. Nat Rev Drug Discov, 2017, 16: 829-842. DOI:10.1038/nrd.2017.178 |
| [2] |
Bannas P, Hambach J, Koch-Nolte F. Nanobodies and nanobody-based human heavy chain antibodies as antitumor therapeutics[J]. Front Immunol, 2017, 8: 1603. DOI:10.3389/fimmu.2017.01603 |
| [3] |
Chames P, Rothbauer U. Special issue: nanobody[J]. Antibodies (Basel), 2020, 9: 6. DOI:10.3390/antib9010006 |
| [4] |
Morrison C. Nanobody approval gives domain antibodies a boost[J]. Nat Rev Drug Discov, 2019, 18: 485-487. DOI:10.1038/d41573-019-00104-w |
| [5] |
Hamers-Casterman C, Atarhouch T, Muyldermans S, et al. Naturally occurring antibodies devoid of light chains[J]. Nature, 1993, 363: 446-448. DOI:10.1038/363446a0 |
| [6] |
Narciso JE, Uy ID, Cabang AB, et al. Analysis of the antibody structure based on high-resolution crystallographic studies[J]. N Biotechnol, 2011, 28: 435-447. DOI:10.1016/j.nbt.2011.03.012 |
| [7] |
Padlan EA. Anatomy of the antibody molecule[J]. Mol Immunol, 1994, 31: 169-217. DOI:10.1016/0161-5890(94)90001-9 |
| [8] |
Sheriff S, Constantine KL. Redefining the minimal antigen-binding fragment[J]. Nat Struct Biol, 1996, 3: 733-736. DOI:10.1038/nsb0996-733 |
| [9] |
Muyldermans S, Baral TN, Retamozzo VC, et al. Camelid immunoglobulins and nanobody technology[J]. Vet Immunol Immunopathol, 2009, 128: 178-183. DOI:10.1016/j.vetimm.2008.10.299 |
| [10] |
Govaert J, Pellis M, Deschacht N, et al. Dual beneficial effect of interloop disulfide bond for single domain antibody fragments[J]. J Biol Chem, 2012, 287: 1970-1979. DOI:10.1074/jbc.M111.242818 |
| [11] |
De Genst E, Saerens D, Muyldermans S, et al. Antibody repertoire development in camelids[J]. Dev Comp Immunol, 2006, 30: 187-198. DOI:10.1016/j.dci.2005.06.010 |
| [12] |
Mujic-Delic A, de Wit RH, Verkaar F, et al. GPCR-targeting nanobodies: attractive research tools, diagnostics, and therapeutics[J]. Trends Pharmacol Sci, 2014, 35: 247-255. DOI:10.1016/j.tips.2014.03.003 |
| [13] |
Yang EY, Shah K. Nanobodies: next generation of cancer diagnostics and therapeutics[J]. Front Oncol, 2020, 10: 1182. DOI:10.3389/fonc.2020.01182 |
| [14] |
Nguyen VK, Hamers R, Wyns L, et al. Camel heavy-chain antibodies: diverse germline VHH and specific mechanisms enlarge the antigen-binding repertoire[J]. EMBO J, 2000, 19: 921-930. DOI:10.1093/emboj/19.5.921 |
| [15] |
Greenberg AS, Avila D, Hughes M, et al. A new antigen receptor gene family that undergoes rearrangement and extensive somatic diversification in sharks[J]. Nature, 1995, 374: 168-173. DOI:10.1038/374168a0 |
| [16] |
Roux KH, Greenberg AS, Greene L, et al. Structural analysis of the nurse shark (new) antigen receptor (NAR): molecular convergence of NAR and unusual mammalian immunoglobulins[J]. Proc Natl Acad Sci U S A, 1998, 95: 11804-11809. DOI:10.1073/pnas.95.20.11804 |
| [17] |
Muyldermans S. Nanobodies: natural single-domain antibodies[J]. Annu Rev Biochem, 2013, 82: 775-797. DOI:10.1146/annurev-biochem-063011-092449 |
| [18] |
Flajnik MF, Deschacht N, Muyldermans S. A case of convergence: why did a simple alternative to canonical antibodies arise in sharks and camels?[J]. PLoS Biol, 2011, 9: e1001120. DOI:10.1371/journal.pbio.1001120 |
| [19] |
Jank L, Pinto-Espinoza C, Duan Y, et al. Current approaches and future perspectives for nanobodies in stroke diagnostic and therapy[J]. Antibodies (Basel), 2019, 8: 5. DOI:10.3390/antib8010005 |
| [20] |
Nanobodies - from llamas to therapeutic proteins [J]. Drug Discov Today Technol, 2010, 7: e95-e146.
|
| [21] |
De Genst E, Silence K, Decanniere K, et al. Molecular basis for the preferential cleft recognition by dromedary heavy-chain antibodies[J]. Proc Natl Acad Sci U S A, 2006, 103: 4586-4591. DOI:10.1073/pnas.0505379103 |
| [22] |
Tsuda M, Shigemoto-Mogami Y, Koizumi S, et al. P2X4 receptors induced in spinal microglia gate tactile allodynia after nerve injury[J]. Nature, 2003, 424: 778-783. DOI:10.1038/nature01786 |
| [23] |
Perez JM, Renisio JG, Prompers JJ, et al. Thermal unfolding of a llama antibody fragment: a two-state reversible process[J]. Biochemistry, 2001, 40: 74-83. DOI:10.1021/bi0009082 |
| [24] |
Harmsen MM, De Haard HJ. Properties, production, and applications of camelid single-domain antibody fragments[J]. Appl Microbiol Biotechnol, 2007, 77: 13-22. DOI:10.1007/s00253-007-1142-2 |
| [25] |
Chanier T, Chames P. Nanobody engineering: toward next generation immunotherapies and immunoimaging of cancer[J]. Antibodies (Basel), 2019, 8: 13. DOI:10.3390/antib8010013 |
| [26] |
Muyldermans S, Cambillau C, Wyns L. Recognition of antigens by single-domain antibody fragments: the superfluous luxury of paired domains[J]. Trends Biochem Sci, 2001, 26: 230-235. DOI:10.1016/S0968-0004(01)01790-X |
| [27] |
Ghannam A, Kumari S, Muyldermans S, et al. Camelid nanobodies with high affinity for broad bean mottle virus: a possible promising tool to immunomodulate plant resistance against viruses[J]. Plant Mol Biol, 2015, 87: 355-369. DOI:10.1007/s11103-015-0282-5 |
| [28] |
Lagerstrom MC, Schioth HB. Structural diversity of G protein-coupled receptors and significance for drug discovery[J]. Nat Rev Drug Discov, 2008, 7: 339-357. DOI:10.1038/nrd2518 |
| [29] |
Stevens RC, Cherezov V, Katritch V, et al. The GPCR network: a large-scale collaboration to determine human GPCR structure and function[J]. Nat Rev Drug Discov, 2013, 12: 25-34. DOI:10.1038/nrd3859 |
| [30] |
Congreve M, de Graaf C, Swain NA, et al. Impact of GPCR structures on drug discovery[J]. Cell, 2020, 181: 81-91. DOI:10.1016/j.cell.2020.03.003 |
| [31] |
Venkatakrishnan AJ, Deupi X, Lebon G, et al. Molecular signatures of G-protein-coupled receptors[J]. Nature, 2013, 494: 185-194. DOI:10.1038/nature11896 |
| [32] |
Ingram JR, Schmidt FI, Ploegh HL. Exploiting nanobodies' singular traits[J]. Annu Rev Immunol, 2018, 36: 695-715. DOI:10.1146/annurev-immunol-042617-053327 |
| [33] |
Heukers R, De Groof TWM, Smit MJ. Nanobodies detecting and modulating GPCRs outside in and inside out[J]. Curr Opin Cell Biol, 2019, 57: 115-122. DOI:10.1016/j.ceb.2019.01.003 |
| [34] |
Rasmussen SG, Choi HJ, Rosenbaum DM, et al. Crystal structure of the human beta2 adrenergic G-protein-coupled receptor[J]. Nature, 2007, 450: 383-387. DOI:10.1038/nature06325 |
| [35] |
Rosenbaum DM, Cherezov V, Hanson MA, et al. GPCR engineering yields high-resolution structural insights into beta2-adrenergic receptor function[J]. Science, 2007, 318: 1266-1273. DOI:10.1126/science.1150609 |
| [36] |
Rosenbaum DM, Zhang C, Lyons JA, et al. Structure and function of an irreversible agonist-β2 adrenoceptor complex[J]. Nature, 2011, 469: 236-240. DOI:10.1038/nature09665 |
| [37] |
Rasmussen SG, Choi HJ, Fung JJ, et al. Structure of a nanobody-stabilized active state of the β2 adrenoceptor[J]. Nature, 2011, 469: 175-180. DOI:10.1038/nature09648 |
| [38] |
Rasmussen SG, DeVree BT, Zou Y, et al. Crystal structure of the beta2 adrenergic receptor-Gs protein complex[J]. Nature, 2011, 477: 549-555. DOI:10.1038/nature10361 |
| [39] |
Shukla AK, Gupta C, Srivastava A, et al. Antibody fragments for stabilization and crystallization of G protein-coupled receptors and their signaling complexes[J]. Methods Enzymol, 2015, 557: 247-258. |
| [40] |
Staus DP, Strachan RT, Manglik A, et al. Allosteric nanobodies reveal the dynamic range and diverse mechanisms of G-protein-coupled receptor activation[J]. Nature, 2016, 535: 448-452. DOI:10.1038/nature18636 |
| [41] |
Spadari RC, Cavadas C, de Carvalho A, et al. Role of beta-adrenergic receptors and sirtuin signaling in the heart during aging, heart failure, and adaptation to stress[J]. Cell Mol Neurobiol, 2018, 38: 109-120. DOI:10.1007/s10571-017-0557-2 |
| [42] |
Yang A, Yu G, Wu Y, et al. Role of beta2-adrenergic receptors in chronic obstructive pulmonary disease[J]. Life Sci, 2021, 265: 118864. DOI:10.1016/j.lfs.2020.118864 |
| [43] |
Ring AM, Manglik A, Kruse AC, et al. Adrenaline-activated structure of beta2-adrenoceptor stabilized by an engineered nanobody[J]. Nature, 2013, 502: 575-579. DOI:10.1038/nature12572 |
| [44] |
Wess J, Eglen RM, Gautam D. Muscarinic acetylcholine receptors: mutant mice provide new insights for drug development[J]. Nat Rev Drug Discov, 2007, 6: 721-733. DOI:10.1038/nrd2379 |
| [45] |
Kruse AC, Ring AM, Manglik A, et al. Activation and allosteric modulation of a muscarinic acetylcholine receptor[J]. Nature, 2013, 504: 101-106. DOI:10.1038/nature12735 |
| [46] |
Chan HCS, McCarthy D, Li J, et al. Designing safer analgesics via μ-opioid receptor pathways[J]. Trends Pharmacol Sci, 2017, 38: 1016-1037. DOI:10.1016/j.tips.2017.08.004 |
| [47] |
Huang W, Manglik A, Venkatakrishnan AJ, et al. Structural insights into µ-opioid receptor activation[J]. Nature, 2015, 524: 315-321. DOI:10.1038/nature14886 |
| [48] |
Brust TF, Morgenweck J, Kim SA, et al. Biased agonists of the kappa opioid receptor suppress pain and itch without causing sedation or dysphoria[J]. Sci Signal, 2016, 9: ra117. |
| [49] |
Che T, Majumdar S, Zaidi SA, et al. Structure of the nanobody-stabilized active state of the kappa opioid receptor[J]. Cell, 2018, 172: 55-67.e15. DOI:10.1016/j.cell.2017.12.011 |
| [50] |
Irannejad R, Tomshine JC, Tomshine JR, et al. Conformational biosensors reveal GPCR signalling from endosomes[J]. Nature, 2013, 495: 534-538. DOI:10.1038/nature12000 |
| [51] |
Kofuku Y, Yoshiura C, Ueda T, et al. Structural basis of the interaction between chemokine stromal cell-derived factor-1/CXCL12 and its G-protein-coupled receptor CXCR4[J]. J Biol Chem, 2009, 284: 35240-35250. DOI:10.1074/jbc.M109.024851 |
| [52] |
Soave M, Heukers R, Kellam B, et al. Monitoring allosteric interactions with CXCR4 using NanoBiT conjugated nanobodies[J]. Cell Chem Biol, 2020, 27: 1250-1261.e5. DOI:10.1016/j.chembiol.2020.06.006 |
| [53] |
Stoeber M, Jullié D, Lobingier BT, et al. A genetically encoded biosensor reveals location bias of opioid drug action[J]. Neuron, 2018, 98: 963-976.e5. DOI:10.1016/j.neuron.2018.04.021 |
| [54] |
Wang WY, Lv XX, Hu ZW, et al. Research progress of chemokines and their receptors in breast cancer[J]. Acta Pharm Sin (药学学报), 2021, 56: 1521-1531. |
| [55] |
Balkwill FR. The chemokine system and cancer[J]. J Pathol, 2012, 226: 148-157. DOI:10.1002/path.3029 |
| [56] |
Scholten DJ, Canals M, Maussang D, et al. Pharmacological modulation of chemokine receptor function[J]. Br J Pharmacol, 2012, 165: 1617-1643. DOI:10.1111/j.1476-5381.2011.01551.x |
| [57] |
Kenakin T. Efficacy at G-protein-coupled receptors[J]. Nat Rev Drug Discov, 2002, 1: 103-110. DOI:10.1038/nrd722 |
| [58] |
Stolla M, Pelisek J, von Bruhl ML, et al. Fractalkine is expressed in early and advanced atherosclerotic lesions and supports monocyte recruitment via CX3CR1[J]. PLoS One, 2012, 7: e43572. DOI:10.1371/journal.pone.0043572 |
| [59] |
Low S, Wu H, Jerath K, et al. VHH antibody targeting the chemokine receptor CX3CR1 inhibits progression of atherosclerosis[J]. MAbs, 2020, 12: 1709322. DOI:10.1080/19420862.2019.1709322 |
| [60] |
Burger JA, Kipps TJ. CXCR4: a key receptor in the crosstalk between tumor cells and their microenvironment[J]. Blood, 2006, 107: 1761-1767. DOI:10.1182/blood-2005-08-3182 |
| [61] |
Feng Y, Broder CC, Kennedy PE, et al. HIV-1 entry cofactor: functional cDNA cloning of a seven-transmembrane, G protein-coupled receptor[J]. Science, 1996, 272: 872-877. DOI:10.1126/science.272.5263.872 |
| [62] |
Jahnichen S, Blanchetot C, Maussang D, et al. CXCR4 nanobodies (VHH-based single variable domains) potently inhibit chemotaxis and HIV-1 replication and mobilize stem cells[J]. Proc Natl Acad Sci U S A, 2010, 107: 20565-20570. DOI:10.1073/pnas.1012865107 |
| [63] |
Van Bockstaele F, Holz JB, Revets H. The development of nanobodies for therapeutic applications[J]. Curr Opin Investig Drugs, 2009, 10: 1212-1224. |
| [64] |
Burns JM, Summers BC, Wang Y, et al. A novel chemokine receptor for SDF-1 and I-TAC involved in cell survival, cell adhesion, and tumor development[J]. J Exp Med, 2006, 203: 2201-2213. DOI:10.1084/jem.20052144 |
| [65] |
Miao Z, Luker KE, Summers BC, et al. CXCR7 (RDC1) promotes breast and lung tumor growth in vivo and is expressed on tumor-associated vasculature[J]. Proc Natl Acad Sci U S A, 2007, 104: 15735-15740. DOI:10.1073/pnas.0610444104 |
| [66] |
Maussang D, Mujić-Delić A, Descamps FJ, et al. Llama-derived single variable domains (nanobodies) directed against chemokine receptor CXCR7 reduce head and neck cancer cell growth in vivo[J]. J Biol Chem, 2013, 288: 29562-29572. DOI:10.1074/jbc.M113.498436 |
| [67] |
Rajagopal S, Kim J, Ahn S, et al. β-Arrestin- but not G protein-mediated signaling by the "decoy" receptor CXCR7[J]. Proc Natl Acad Sci U S A, 2009, 107: 628-632. |
| [68] |
Cheng Y, Ma XL, Wei YQ, et al. Potential roles and targeted therapy of the CXCLs/CXCR2 axis in cancer and inflammatory diseases[J]. Biochim Biophys Acta Rev Cancer, 2019, 1871: 289-312. |
| [69] |
Bradley ME, Dombrecht B, Manini J, et al. Potent and efficacious inhibition of CXCR2 signaling by biparatopic nanobodies combining two distinct modes of action[J]. Mol Pharmacol, 2015, 87: 251-262. DOI:10.1124/mol.114.094821 |
| [70] |
Peyrassol X, Laeremans T, Gouwy M, et al. Development by genetic immunization of monovalent antibodies (nanobodies) behaving as antagonists of the human ChemR23 receptor[J]. J Immunol, 2016, 196: 2893-2901. DOI:10.4049/jimmunol.1500888 |
| [71] |
Maussang D, Verzijl D, Van Walsum M, et al. Human cytomegalovirus-encoded chemokine receptor US28 promotes tumorigenesis[J]. Proc Natl Acad Sci U S A, 2006, 196: 2893-2901. |
| [72] |
Heukers R, Fan TS, de Wit RH, et al. The constitutive activity of the virally encoded chemokine receptor US28 accelerates glioblastoma growth[J]. Oncogene, 2018, 37: 4110-4121. DOI:10.1038/s41388-018-0255-7 |
| [73] |
De Groof TWM, Mashayekhi V, Fan TS, et al. Nanobody-targeted photodynamic therapy selectively kills viral GPCR-expressing glioblastoma cells[J]. Mol Pharm, 2019, 16: 3145-3156. DOI:10.1021/acs.molpharmaceut.9b00360 |
| [74] |
Tatemoto K, Hosoya M, Habata Y, et al. Isolation and characterization of a novel endogenous peptide ligand for the human APJ receptor[J]. Biochem Biophys Res Commun, 1998, 251: 471-476. DOI:10.1006/bbrc.1998.9489 |
| [75] |
Kuba K, Sato T, Imai Y, et al. Apelin and elabela/toddler; double ligands for APJ/apelin receptor in heart development, physiology, and pathology[J]. Peptides, 2019, 111: 62-70. DOI:10.1016/j.peptides.2018.04.011 |
| [76] |
Ma Y, Ding Y, Song X, et al. Structure-guided discovery of a single-domain antibody agonist against human apelin receptor[J]. Sci Adv, 2020, 6: eaax7379. DOI:10.1126/sciadv.aax7379 |
| [77] |
Ren H, Li J, Zhang N, et al. Function-based high-throughput screening for antibody antagonists and agonists against G protein-coupled receptors[J]. Commun Biol, 2020, 3: 146. DOI:10.1038/s42003-020-0867-7 |
| [78] |
Ledsgaard L, Kilstrup M, Karatt-Vellatt A, et al. Basics of antibody phage display technology[J]. Toxins (Basel), 2018, 10: 236. DOI:10.3390/toxins10060236 |
| [79] |
Smith GP. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface[J]. Science, 1985, 228: 1315-1317. DOI:10.1126/science.4001944 |
| [80] |
Bobkov V, van der Woning B, de Haard H. Display technologies for generation of Ig single variable domains[J]. Methods Mol Biol, 2018, 1827: 129-144. |
| [81] |
Gao J, Sidhu SS, Wells JA. Two-state selection of conformationspecific antibodies[J]. Proc Natl Acad Sci U S A, 2009, 106: 3071-3076. DOI:10.1073/pnas.0812952106 |
| [82] |
Boder ET, Wittrup D. Yeast surface display for screening combinatorial polypeptide libraries[J]. Nat Biotechnol, 1997, 15: 553-557. DOI:10.1038/nbt0697-553 |
| [83] |
English JG, Olsen RHJ, Lansu K, et al. VEGAS as a platform for facile directed evolution in mammalian cells[J]. Cell, 2019, 178: 748-761.e17. DOI:10.1016/j.cell.2019.05.051 |
| [84] |
Hendel SJ, Shoulders MD. Directed evolution in mammalian cells[J]. Nat Methods, 2021, 18: 346-357. DOI:10.1038/s41592-021-01090-x |
| [85] |
Li S, Su W, Zhang C. Linear double-stranded DNAs as innovative biological parts to implement genetic circuits in mammalian cells[J]. FEBS J, 2019, 286: 2341-2354. DOI:10.1111/febs.14816 |
| [86] |
Zhao Y, Wang Y, Su W, et al. Construction of synthetic nanobody library in mammalian cells by dsDNA-based strategies[J]. Chembiochem, 2021, 22: 2957-2965. DOI:10.1002/cbic.202100286 |
| [87] |
Zhao Y, Tong X, Zhang C, et al. Construction of nanobody library in mammalian cells by linear-double-stranded DNA based AND gate genetic circuit [J]. bioRxiv, 2020. DOI: 10.1101/2020.01.21.912907.
|
 2022, Vol. 57
2022, Vol. 57


