病毒是一种个体微小、结构简单, 以DNA或RNA为遗传物质, 必须在活细胞内寄生并以复制方式增殖的非细胞型生物[1]。病毒侵入机体并在易感的宿主细胞中增殖的过程称为病毒感染。按照病毒在机体内滞留的时间, 可分为急性病毒感染[如流感病毒、新型冠状肺炎病毒(severe acute respiratory syndrome-coronavirus 2, SARS-CoV-2) 引发的感染] 和持续性病毒感染[如人类免疫缺陷病毒(human immunodeficiency virus, HIV)、乙肝病毒(hepatitis B virus, HBV) 引发的感染], 后者又可分为慢性感染、潜伏感染和慢发病毒感染[2-4]。
纵观人类历史, 不断涌现的新发病毒已经夺走了数千万人的生命。现代最致命的病毒事件当属1918年的西班牙大流感, 它造成世界三分之一的人口被感染, 约5 000万人死亡[5, 6]; 第二致命的是HIV的流行, 它自1981年首次被确认以来累计造成3 500万人死亡[7, 8]; SARS-CoV-2引起的新冠肺炎病毒疫情(corona virus disease 2019, COVID-19) 也被认为是近100年来最致命的病毒大流行之一[5, 9, 10], 截至2022年2月14日, 确诊病例已达41 056万例, 死亡病例已超过581万例。可见, 病毒感染一直是严重威胁着人类健康的重大公共安全卫生问题。随着环境和气候的变化, 人类可能面临更多新发与再现的病毒性传染病的爆发。药物是抗击病毒感染疾病的有力武器, 目前已针对9个病毒家族研发出百余种抗病毒药物[11, 12]。但慢性难治性病毒感染患者长期服用药物易产生耐药性导致原有抗病毒药物失效以及人们面对新发病毒肆虐时的束手无策, 提醒研究人员应该确证更多能够干扰高致病性病毒复制的新靶标, 同时运用多种新的药物设计策略来发现广谱抗耐药性的抗病毒药物[13-17]。
2 抗病毒药物研究新靶标由于病毒的复制周期与宿主正常细胞的代谢过程密切相关, 抑制病毒的繁殖势必会导致未感染细胞的损伤, 这也是抗病毒药物发展缓慢的原因。同时, 现有的抗病毒药物存在毒副作用大、易产生耐药性、不能彻底清除病毒等缺陷[18]。因此, 开发出更多新型靶标是药物研发的关键。
2.1 复制共性环节病毒的生命周期有几个共同的阶段, 包括侵入(受体识别、融合)、生物合成、装配和释放, 来自同一属病毒的蛋白序列和结构也非常相似[19]。在病毒侵入宿主细胞的过程中, 受体识别不仅需要病毒表面蛋白结合亚单位和宿主细胞表面受体的参与, 而且还需要细胞蛋白酶来启动表面蛋白; 病毒与宿主细胞膜融合需要病毒表面蛋白跨膜亚单位的参与; 进入后, RNA病毒借助RNA依赖性RNA或DNA聚合酶(RNA-dependent RNA polymerase, RdRp和RNA-dependent DNA polymerase, RdDp) 进行复制; 然后, 在组装过程中包膜病毒的表面蛋白表达并插入质膜或内质网; 病毒颗粒经细胞膜包裹后通过出芽或胞吐方式释放到细胞外, 其在释放前后还需借助病毒蛋白酶进行成熟转化(图 1)。这些复制的共性环节通常作为抗病毒药物研发的有效靶标。
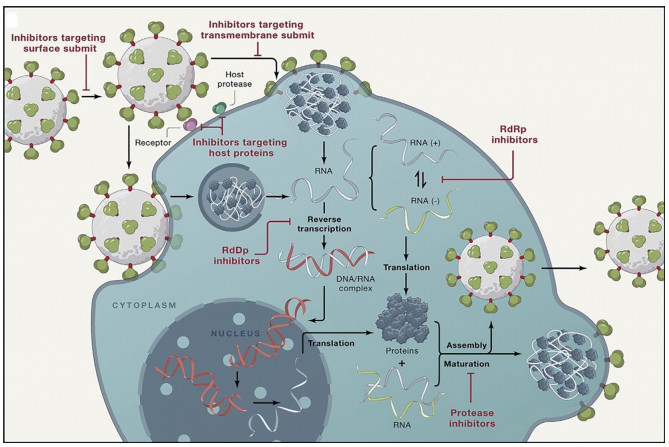
|
Figure 1 Brief life cycle of most enveloped RNA viruses[19] |
病毒表面蛋白(surface protein, SP) 表面亚基(surface subunit, SfS) 是针对病毒侵入环节的有效靶点。小分子化合物、多肽及抗体可以通过靶向病毒受体结合域或者靶向非结合域引起SP构象变化以阻断SP与受体的结合[20]。其中代表药物是抗HIV药物rukobia (1, 图 2), 它可以与融合前的HIV SfS结合并诱导构象发生改变以阻断HIV包膜蛋白与CD4 (cluster of differentiation 4) 的结合[21]。但是病毒SfS易发生突变, 所以针对该靶点的抑制剂很难开发成广谱抗病毒药物。因此, 为应对新发病毒暴发的紧急情况, 需要建立起靶向SfS的抗病毒药物的快速筛选和安全性评估体系。
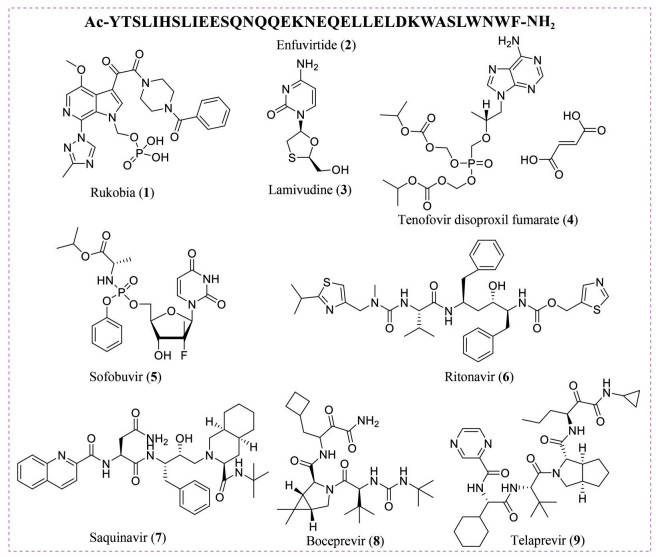
|
Figure 2 The chemical structures of compounds 1-9 |
与SfS相比, Ⅰ类融合蛋白的病毒表面跨膜亚基(transmembrane subunit, TmS) 在同属病毒中更保守, 可作为开发针对同属病毒的广谱抑制剂的靶点。Ⅰ类融合蛋白大致分为融合肽(fusion peptide, FP)、七肽重复序列1 (heptad repeat-1, HR1)、七肽重复序列2 (heptad repeat-2, HR2)、近膜区域(membrane proximal external region, MPER)、跨膜区域(transmembrane region, TM) 和胞内区(cyto-plasmic tail, CT)。在病毒进入宿主细胞的过程中, FP负责插入细胞膜, HR1与HR2相互作用形成六螺旋束(six-he-lix bundle, 6-HB)[22]。因此, 靶向病毒TmS的抑制剂可以包括针对FP、HR1及HR2、MPER及CT的小分子化合物、多肽以及抗体[23-25]。其中代表药物为HIV药物恩夫韦肽(enfuvirtide, 2, 图 2), 它通过结合HIV的HR1区阻断病毒与靶细胞的膜融合[26]。
2.1.2 核酸复制RdRp在大多数RNA病毒的基因组复制和转录过程中发挥关键作用, 且在RNA病毒中高度保守, 是开发广谱抗病毒药物的有效靶标[27, 28]。此外, 逆转录酶(reverse transcriptase, RT) 也是逆转录病毒复制过程中的一个关键酶, 它可以合成与病毒RNA模板互补的DNA。p66/p51异质体是艾滋病毒RT的一部分, 在p66的N端有一个RNA和DNA依赖的DNA聚合酶域及处理RNA的核糖核酸酶(ribonuclease H, RNase H) 域[29], 靶向RT的抗艾滋病毒药物拉米夫定(lamivudine, 3) 和富马酸泰诺福韦酯(tenofovir disoproxil fumarate, 4) 等能够有效地抑制乙肝病毒的复制[11]。另外抗丙型肝炎病毒(hepatitis C virus, HCV) 药物索非布韦(sofobuvir, 5) 也可以有效抑制寨卡病毒(Zika virus, ZIKV)、登革热病毒(dengue virus, DENV) 和SARS-CoV-2引起的感染[30]。这说明RNA和DNA聚合酶的催化域是高度保守的, 针对该结构域的抑制剂可作为广谱抗病毒药物。
2.1.3 蛋白水解病毒蛋白酶通过参与病毒多蛋白前体的裂解来释放功能性蛋白。在HIV病毒粒子的释放阶段, HIV蛋白酶负责在多个位点处理Gag和Gag-Pol多聚蛋白以产生新病毒颗粒的结构蛋白。随后, 病毒粒子内部结构重排成成熟的颗粒。通过对HIV蛋白酶底物结合口袋的结构进行分析, 研究人员设计出一些具有纳摩尔活性的HIV蛋白酶抑制剂[31]。如HIV治疗药物利托那韦(ritonavir, 6)、沙奎那韦(saquinavir, 7), 以及抗HCV药物波普瑞韦(boceprevir, 8)、特拉匹韦(telaprevir, 9, 图 2) 等均为针对相应病毒蛋白酶的小分子抑制剂[32]。由于不同病毒的蛋白酶不同, 所以病毒蛋白酶类抑制剂没有RNA/DNA聚合酶抑制剂广谱, 但是也有可能开发成针对同属病毒的广谱抑制剂。
2.2 蛋白复合物蛋白复合物是一组通过非共价的蛋白质-蛋白质相互作用(protein-protein interaction, PPI) 联系在一起的两个或多个相关联的多肽链, 不同的多肽链可以具有不同的功能[33]。靶向蛋白复合物中的关键PPI存在诸多优势, 主要表现在: ①相对于酶的活性位点, PPI具有很大的特异性; ②参与相互作用的两种蛋白上至少一个残基的同时突变才能产生耐药性。因此, 靶向蛋白复合物为开发更具选择性、不良反应更少和抗耐药性的药物提供了新选择[34]。PPI在流感病毒聚合酶复合体的形成中起着关键作用。流感病毒RNA聚合酶是由酸性蛋白(acidic polymerase, PA)、碱性蛋白1 (basic polymerase 1, PB1) 和碱性蛋白2 (basic polymerase 2, PB2) 3个亚基组成的异源三聚体复合物(也称为3P复合体, 图 3), 是开发新型抗流感药物最有前景的靶点之一[35, 36]。PB1位于3P复合体的核心, 其N端与PA亚基的C端相连, C端与PB2亚基的N端相连, 三者形成了稳定的蛋白复合物。PA亚基内存在核酸内切酶活性位点, 且PA亚基为磷酸化蛋白, 其水解活性与聚合酶活性呈正相关。PB1亚基主要参与病毒基因组的复制过程, 而PB2亚基主要负责与宿主前信使RNA帽状结构结合, 协助完成内切酶的剪切过程[37, 38]。由于流感RNA聚合酶的关键功能和高度保守的蛋白质序列, 干扰聚合酶亚基的蛋白相互作用可影响聚合酶活性和病毒复制[39]。
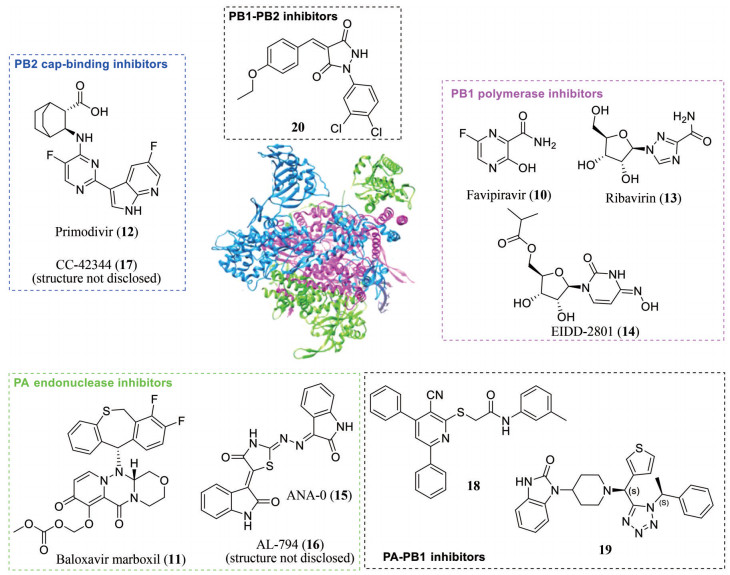
|
Figure 3 Crystal structure of flu A RdRP determined from bat flu A H17N10 strain (PDB code: 4WSB) and chemical structures of inhibitors of PA (green), PB1 (magenta), PB2 (blue), PA-PB1 and PB1-PB2 (black) |
近年来, 一些靶向流感病毒RNA聚合酶的化合物相继被报道, 如PB1抑制剂法匹拉韦(favipiravir, 10)[40]、PA核酸内切酶抑制剂巴洛沙韦玛波西酯(baloxavir marboxil, 11)[41]以及PB2帽结合抑制剂吡莫地韦(pimodivir, 12)[42]。此外, 还包括广谱抗病毒药物利巴韦林(ribavirin, 13) 和对季节性和高致病性流感A和B病毒有广谱抑制作用的EIDD-2801 (14)[43]; PA核酸内切酶抑制剂ANA-0 (15)[44] (正在进行临床前评估) 和AL-794 (16)[45], 以及对季节性和大流行甲型流感病毒株表现出高效广谱抗病毒活性(EC50 = 0.1~9 nmol·L-1) 的PB2抑制剂CC-42344 (17, 图 3)[46]。
2018年, D'agostino等[47]报道了一类3-氰基-4, 6二苯基吡啶类PA-PB1抑制剂, 其中化合物18 (图 3) 能抑制PA-PB1的相互作用(IC50 = 3.5 μmol·L-1) 和流感病毒复制(EC50 = 9.2 μmol·L-1), 在测试浓度下无细胞毒性。Zhang等[48]首先通过对2 000个化合物进行虚拟筛选, 再利用酶联免疫吸附剂测定法(enzyme-linked immunosorbnent assay, ELASA) 检测优选分子对PA-PB1亚基相互作用的抑制能力, 最后对苗头化合物优化得到了具有良好抗病毒活性的(S, S) 非对映异构体19 (图 3), 其对PA-PB1的抑制呈剂量依赖性, IC50为7.6 μmol·L-1。化合物19不仅对流感A/WSN/1933 (H1N1) 株有较强的抑制活性(EC50 = 0.7 μmol·L-1), 而且对6株甲型流感和5株乙型流感的临床分离株也有较好的抑制活性(EC50 = 0.6~2.7 μmol·L-1)。2017年, Yuan等[49]发现具有良好抗病毒活性(EC50 = 1.4 μmol·L-1) 的吡唑烷-3, 5-二酮衍生物20 (图 3) 能够抑制PB1-PB2相互作用(IC50 = 8.6 μmol·L-1), 并且20对H1N1、H7N9和H9N2毒株的复制具有剂量依赖性的抑制作用。
因此, 流感病毒-聚合酶复合物是开发下一代抗流感病毒药物的重要靶点, 同时也表明靶向蛋白复合物可能是发现广谱抗耐药性抗病毒药物的有效途径。
2.3 多功能蛋白耐药性逐渐成为抗艾滋病药物研究面临的最大挑战[50], 靶向HIV-1复制周期关键多功能蛋白, 是克服现有药物耐药性的一个重要途径。HIV-1衣壳蛋白(capsid protein, CA) 是构成一个具有感染性的病毒颗粒所必需的结构蛋白, 其内包裹着对病毒复制至关重要的遗传物质和关键酶[51]。成熟的HIV-1 CA结构为圆锥形“富勒烯”样晶格外壳, 每个晶格约含1 500个CA单体, 包括约250个六聚体和12个五聚体。CA单体由N端结构域(N-terminal domain, NTD)、C端结构域(C-terminal domain, CTD) 以及中间的柔性连接链组成。HIV-1 CA在病毒复制的早期(逆转录、整合) 和晚期(装配、成熟) 阶段中均发挥着非常关键的调控作用(图 4), CA的正常组装及结构稳定性对保持病毒的感染力至关重要[29, 52]。

|
Figure 4 The roles of CA in the HIV-1 replication cycle[29] |
靶向HIV-1 CA的多肽和小分子化合物如CAI (21)、CAP-1 (22)、BD1 (23)、BM1 (24)、BI-2 (25)、PF74 (26)、11l (27) 和GS-6207 (lenacapavir, 28, 图 5) 等可以在病毒复制的早期、晚期或在这两个阶段均有抑制作用[51]。作为第一个与HIV-1 CA共结晶的小分子, 26受到了广泛的关注。然而, 26的抗HIV-1活性低且代谢稳定性较差。鉴于此, 本课题组以HIV-1CA六聚体相邻亚基蛋白界面为靶标, 以26为先导化合物构建了基于苯丙氨酸优势片段的化合物库, 经细胞水平的抗病毒活性筛选及机制研究, 发现了一系列结构新颖的苯丙氨酸类衣壳蛋白抑制剂[53]。其中, 化合物27抗HIV-1活性最佳(EC50 = 90 nmol·L-1)。机制实验表明在病毒复制早期, 27通过选择性结合到HIV-1 CA (IC50 = 0.24 μmol·L-1) 来加速病毒脱壳, 而在复制晚期, 27通过干扰HIV-1 CA组装产生传染性较小的病毒颗粒来发挥作用(IC50 = 8.96 nmol·L-1)。另外, 吉利德科学公司研发的HIV-1 CA抑制剂28具有皮摩尔的抗病毒活性(EC50 = 100 pmol·L-1), 其在体外显示出比目前批准的抗HIV药物更强的效力, 单次皮下注射给药后可迅速降低病毒载量, 并且注射6个月后在体内仍具有抗病毒作用。体外机制实验表明28可阻断病毒生命周期的多个不同阶段, 有阻止病毒感染和接触未感染细胞的潜力[54]。28目前正处于Ⅱ/Ⅲ期临床试验中, 用于与其他抗逆转录病毒药物联合治疗感染HIV-1的患者[55]。
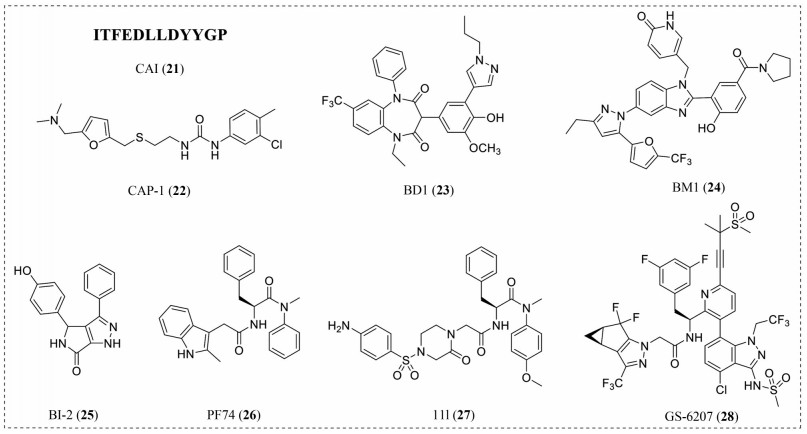
|
Figure 5 The chemical structures of compounds 21-28 |
病毒是寄生生物, 必须依赖宿主细胞的原料进行核酸及蛋白质的生产和加工以完成复制, 靶向宿主细胞(host-targeting antiviral, HTA) 的药物不仅可以有效地抑制病毒核酸的快速复制, 而且能对抗病毒耐药突变, 做到“以不变应万变”[56]。因此, 开发具有广谱抗病毒疗效的HTA药物一直是抗病毒药物研发领域追求的目标。
2.4.1 靶向宿主细胞激酶宿主细胞激酶在许多病毒的复制过程中发挥至关重要的作用, 已成为抗病毒药物开发的潜在靶点[57]。Meertens等[58, 59]发现DENV和ZIKV利用受体酪氨酸激酶Ax1作为感染宿主的进入因子。表皮生长因子受体(epidermal growth factor receptor, EGFR) 可作为腺相关病毒血清6型和人巨细胞病毒进入宿主的共同受体[60, 61]。Coleman等[62]指出, Abelson酪氨酸蛋白激酶2 (Abelson tyrosine-protein kinase 2, ABL2) 在严重急性呼吸综合征冠状病毒(severe acute respiratory syndrome-coronavirus, SARS-CoV) 和中东呼吸综合征冠状病毒(middle east respiratory syndrome-coronavirus, MERS-CoV) 的复制中发挥非常关键的作用。衔接蛋白相关激酶1 (adaptor-associated kinase 1, AAK1) 是一种细胞丝氨酸-苏氨酸蛋白激酶, 可调节多种病毒在细胞内的转运。2019年, Verdonck等[63]报道了一个具有高AAK1亲和力(IC50 = 0.0042 μmol·L-1) 的新型吡咯并[2, 3-b]吡啶类化合物29 (图 6), 其在人原代树突状细胞(EC50 = 0.043 μmol·L-1) 中表现出优异的抗DENV活性, 同时还发现其具有较好的抗埃博拉病毒(Ebola virus, EBOV) 活性(EC50 = 1.59 μmol·L-1), 这表明靶向细胞AAK1是开发广谱抗病毒药物的理想靶标。与传统的、直接作用的抗病毒药物相比, 激酶靶向药物提供了更高的耐药性屏障, 覆盖了多种病毒基因型或血清型[64]。因此, 靶向宿主细胞激酶是发现广谱抗病毒药物的重要治疗途径。
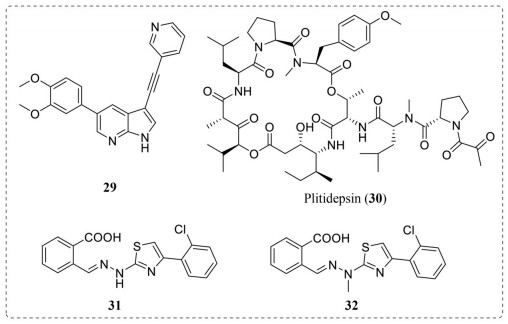
|
Figure 6 The chemical structures of compounds 29-32 |
真核细胞翻译延伸因子(eukaryotic translation elongation factor 1A, eEF1A) 是一个在蛋白质合成过程中发挥肽链延伸作用的重要因子。SARS-CoV-2如何感染细胞的研究表明, 抑制病毒病原体复制时使用的宿主翻译复合物可以显著地抑制病毒的复制过程[65]。在此基础上, 研究人员评估了一种已知的参与宿主蛋白翻译的抑制剂plitidepsin (30, 又称aplidin, 图 6), 它已被澳大利亚监管局批准用于治疗多发性骨髓瘤。体外抗病毒活性结果表明, plitidepsin在体外对SARS-CoV-2的抑制活性(EC50 = 0.88 nmol·L-1) 比瑞德西韦(remdesivir) 强27.5倍, 并且它在两种SARS-CoV-2感染的小鼠模型体内均有效。同时机制实验证明plitidepsin通过抑制eEF1A来发挥抗SARS-CoV-2活性[66]。总之, plitidepsin是一种有前途的COVID-19治疗候选药物。
2.4.2.2 二氢乳清酸脱氢酶DHODH (dihydroorotate dehydrogenase) 位于线粒体内膜, 是催化嘧啶核苷酸从头合成途径第四步氧化还原反应的关键酶。病毒复制需要依赖从头合成途径获得的嘧啶核苷酸以满足病毒复制对核酸的需求[67]。因此, 可以通过抑制DHODH的活性以抑制嘧啶碱基的从头合成, 从而抑制病毒的DNA和RNA的生物合成, 起到抑制病毒复制的作用[68]。2020年8月, 中国科学家报道了一类靶向宿主细胞的DHODH抑制剂, 其能够通过抑制病毒基因组复制和调节免疫两种途径发挥广谱抗病毒作用, 代表化合物31 (EC50 = 0.017 μmol·L-1) 和32 (EC50 = 1.56 μmol·L-1) (图 6) 在细胞水平均表现出高效的抗SARS-CoV-2活性[69], 且有望成为抗RNA病毒的候选新药。这一工作揭示了DHODH可以作为一个极具吸引力的抗病毒靶点并通过“两手抓”策略(抗病毒+免疫调节) 实现广谱抗病毒的目的, 为SARS-CoV-2和其他RNA病毒提供更多的治疗方案。
2.5 靶向核酸核酸是脱氧核糖核酸和核糖核酸的总称, 其中G-四链体(G-quadruplexes, G4s) 是由G-quartet层叠而形成的DNA或RNA四链构象, 是最重要的非典型核酸二级结构之一, 因其独特的构象和重要的基因功能而受到广泛的关注[70]。G4s被认为是调节病毒生命周期的关键要素, 一些靶向G4s的化合物已被证实具有有效的抗病毒作用[71]。
近日, 中国科学家利用多种生物物理技术和分子生物学分析验证了SARS-CoV-2 RNA中G4s的存在, 并最终选取位于编码区核衣壳蛋白(nucleocapsid protein, N) 上能够在活细胞中折叠成稳定单分子RNA G4s结构的RG-1用于后续的研究[72]。SARS-CoV-2 N蛋白通过与病毒基因组和膜蛋白(membrane protein, M) 的相互作用, 在病毒组装过程中发挥关键作用, RG-1位于N蛋白的编码(coding sequences, CDS) 区域, 很可能通过抑制其mRNA的翻译来调节N蛋白的水平。于是研究人员通过建立增强绿色荧光蛋白(enhanced green fluorescent protein, EGFP) 系统发现在转染pLV-NEGFP的HeLa细胞中加入G4s特异结合试剂PDP (33, 图 7) 可以稳定RG-1, 抑制mRNA的翻译, 显著降低N蛋白水平, 证明了靶向G4s的化合物有可能抑制SARS-CoV-2的组装和复制。因此, G4s有望成为开发COVID-19治疗药物的新靶点, 为寻找有效的抗新冠病毒药物提供了新思路。
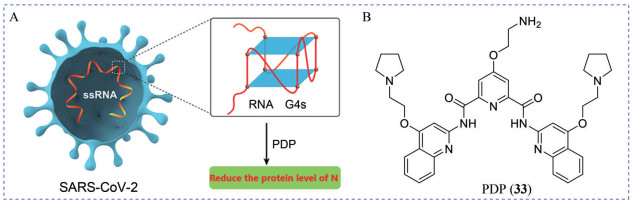
|
Figure 7 (A) Schematic drawing of G4-specific stabilizer PDP binding RNA G4 in SARS-CoV-2 genome and reducing N protein expression; (B) The chemical structure of PDP (33) |
由于药物研发的成本不断增加, 传统的药物随机筛选策略和盲目优化先导化合物的药物发现策略会消耗大量的资源和时间。因此, 药物设计新策略的应用就显得尤为重要。
3.1 共价结合药物靶点大多是蛋白质, 由于其结构中富含羟基、巯基、氨基等基团, 本质上可作为亲核试剂, 而共价结合化合物含有的亲电性基团(如迈克尔受体、卤素、羰基、异氰等) 是优异的亲电试剂。两者相互作用可以形成新的共价键导致蛋白质构象发生变化, 从而起到抑制蛋白质活性的作用。共价抑制剂与靶标的共价结合可分为两个过程: 第一步是抑制剂可逆地与靶标结合, 使弱亲电配体上的官能团靠近蛋白质上的特定亲核残基; 第二步是配体与蛋白质中涉及的官能团反应形成共价键[73] (图 8)。影响共价抑制剂靶标特异性的关键因素是弹头部分的反应性。理想的弹头部分应在生理条件下保持稳定, 并尽可能消除脱靶反应[74]。共价抑制剂因其在疗效、选择性和安全性方面具有潜在优势, 近年来逐渐受到抗病毒领域的关注。

|
Figure 8 The process of covalent inhibitors binding |
HIV-1逆转录酶(RT) 中Y181C耐药突变一直是发现非核苷类RT抑制剂(non-nucleoside reverse transcriptase inhibitors, NNRTIs) 的研究难点。鉴于此, 2017年, Chan等[75]报道了一系列能够有效抑制Y181C耐药突变株活性的RT共价抑制剂(covalent inhibitors of Tyr181Cys RT, CRTIs)。研究者根据化合物34 (图 9A) 与野生株逆转录酶的共晶结构发现, 34中苯环上的氯原子朝向Tyr181, 表明可在此位点引入亲电弹头以期与突变体Cys181共价结合。最终以丙烯酰胺为弹头得到的化合物35 (IC50 = 0.5 μmol·L-1) 抗病毒活性最为优异, 它能够与Cys181的巯基形成共价键, 并且通过体外实验和质谱证明化合物35是Y181C RT的共价抑制剂。此外, 2020年, 本课题组还报道了一类针对耐药突变株Y181C的新型吲哚芳砜类HIV-1 RT共价抑制剂, 其中化合物36和37 (图 9B) 对Y181C突变株具有选择性抑制作用[76]。
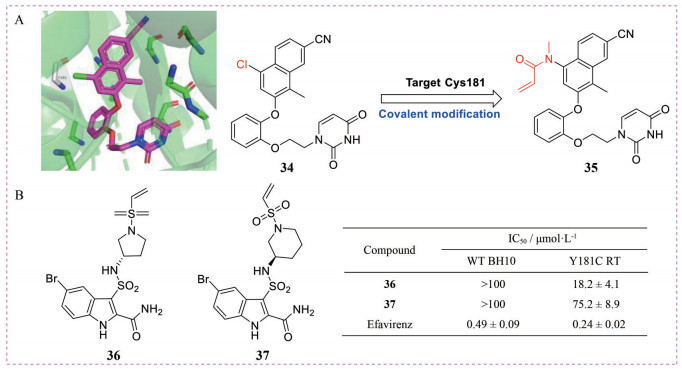
|
Figure 9 (A) The crystal structure of 34 in complex with RT (PDB code: 5TER) and the chemical structure of compounds 34 and 35. The figure is generated in PyMOL (www.pymol.org); (B) The chemical structure and activity data of compounds 36 and 37 |
虽然共价抑制剂可能存在潜在的毒性和脱靶效应, 但是其作用较强并且持久, 可以降低给药剂量和给药频率, 因此具有很好的发展前景。
3.2 蛋白降解蛋白的靶向降解可以通过开发靶向蛋白降解嵌合体(proteolysis-targeting chimera, PROTAC) 分子来实现[77]。PROTAC是一种双功能小分子, 它由靶蛋白特异性配体和E3泛素连接酶募集配体通过linker连接组成。PROTAC分子可以将靶蛋白和E3泛素连接酶拉近, 从而诱导靶蛋白的泛素化和随后通过泛素-蛋白酶体途径降解(图 10)[78]。该原理已成功地应用到多个靶标, 例如激酶(RIPK2、BTK、BCR-ABL、CDK9)[79-81]和转录酶(BRD4、BRD9、TRIM24)[82, 83]等。靶标特异性与抗耐药性是抗肿瘤及抗病毒药物的迫切要求, 在药物设计策略方面有相通之处。因此, 靶向蛋白降解也有望成为开发抗病毒药物的新策略。
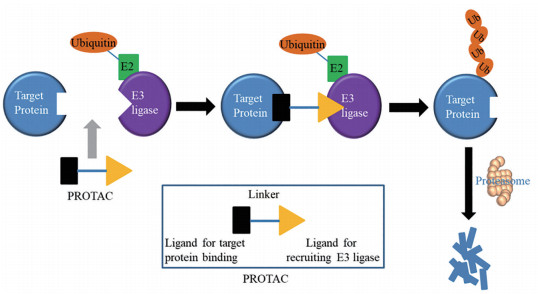
|
Figure 10 A mechanistic overview of PROTAC-mediated protein degradation |
哈佛医学院的Yang课题组开发出一类新的PROTAC抗丙肝病毒分子, 其可诱导病毒蛋白酶体降解[84]。Telaprevir是一种与HCV蛋白酶活性位点结合的可逆共价抑制剂, 与CRL4CRBN的配体组合的PROTAC分子既能抑制又能诱导HCV的NS3/4A蛋白酶降解。优化后的PROTAC分子DGY-08-097 (38, 图 11) 在细胞感染模型中能够有效抑制HCV (EC50 = 748 nmol·L-1), 证明了蛋白质降解有助于其抗病毒活性。最后, 研究者还证明这种新型抗病毒PROTAC分子可以克服病毒变异, 解决了对传统蛋白抑制剂如telaprevir的耐药性问题。总之, 该研究工作验证了靶向降解蛋白可能作为开发抗耐药性抗病毒药物的新途径。

|
Figure 11 The chemical structure of DGY - 08 -097 (38) |
RNA结合蛋白(RNA binding proteins) 功能缺陷是许多疾病的起源, 然而用常规药物来靶向RNA结合蛋白却比较困难。受靶向蛋白降解技术的启发, 研究人员开发了一种靶向降解RNA结合蛋白的新技术, 即RNA-PROTAC[85]。其在抗肿瘤领域得到成功应用, 有望为抗病毒药物设计开拓新思路。
3.3 核酸降解核糖核酸酶靶向嵌合体(ribonuclease targeting chimera, RIBOTAC) 是一个有前景的靶向RNA的新策略。RIBOTAC的关键创新是将RNA结合分子转化为RNA降解分子, 即通过将RNA结合分子与一个小分子结合并激活核糖核酸酶L (ribonuclease L, RNase L) 以实现降解病毒基因组的目的[86, 87]。
Haniff等[86]通过对SARS-CoV-2基因组的结构进行分析, 在移码元件(frameshifting element, FSE) 中确定了结构趋同的位点, 该位点控制着pp1a和pp1ab多聚蛋白的翻译, 这对于病毒复制和发病机制至关重要。增强FSE的热力学稳定性会降低移码效率, 研究人员通过一系列实验验证了化合物C5 (39, 图 12) 能够使FSE稳定从而显著地抑制SARS-CoV-2 FSE的移码能力。为了增强39的疗效和选择性, 研究人员试图通过对39进行结构修饰以发展RIBOTAC的思路, 即通过合适长度的连接链将39与可以招募RNase L的小分子连接以实现定向降解SARS-CoV-2 RNA的目的。改造之后得到的C5-RIBOTAC (40) 可以降低SARS CoV-2 FSE的移码效率, 与39的简单结合不同, 40可以通过募集细胞内的RNase L来降解病毒基因组, 从而彻底清除新冠病毒。这一策略为研发新的抗SARS CoV-2药物提供了启发。
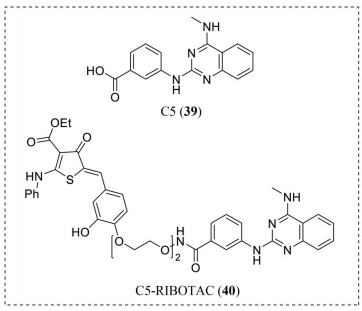
|
Figure 12 The chemical structures of compounds 39 and 40 |
HIV-1包膜糖蛋白三聚体复合物(envelope glycoprotein trimeric complex, Env) 是一种由gp120和gp41蛋白亚基通过非共价相互作用连接的亚稳态三聚体, 其在病毒颗粒与宿主细胞的融合中发挥着关键的作用[88]。研究表明通过改变Env的构象可以使成熟病毒粒子裂解的同时抑制病毒对细胞的感染[89]。因此, 可设计能够劫持Env的小分子化合物从而特异性地和不可逆转地破坏成熟病毒粒子以及杀灭被HIV-1感染的细胞。
近期, Gaffney等[90]报道了一类双功能HIV-1抑制剂, 它们能够同时与HIV-1包膜糖蛋白中的两个亚单元结合, 并通过改变包膜糖蛋白的内部构象诱导HIV-1病毒裂解, 同时抑制HIV-1病毒对细胞的感染。双功能抑制剂一部分源自可与HIV-1包膜糖蛋白中的gp120亚单元结合的小分子CD4模拟物BNM-III-170; 另一部分源自位于HIV-1病毒gp41膜近侧区域的N端并能够与gp41亚单元结合的一段带3个色氨酸的多肽(Trp3) (图 13)。实验结果表明, 这类双功能抑制剂能够有效抑制HIV-1 BaL.01假病毒对HOS.T4.R5细胞的感染, 并通过与病毒膜结构发生相互作用对其产生一定的破坏作用。但是它只能选择性地杀死呈递包膜糖蛋白的细胞, 而对不呈递包膜糖蛋白的细胞没有影响。以上说明双功能抑制剂能够通过有效清除被HIV-1感染的细胞发挥作用。
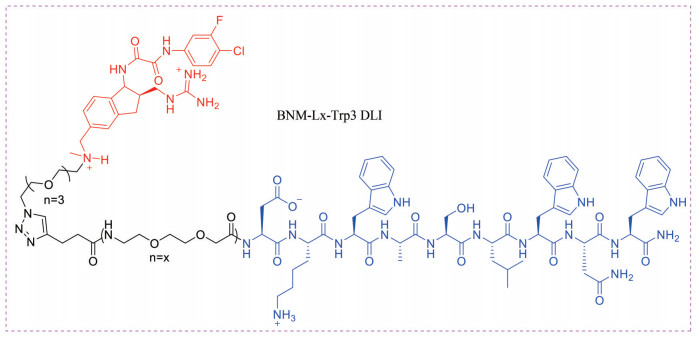
|
Figure 13 The chemical structure of BNM-Lx-Trp3 DLI |
合成致死包括两个基因或两个基因所在的信号通路同时因功能异常(如基因突变、过度表达等) 而导致的细胞无法存活的情况[91]。虽然突变是病毒进化的驱动力, 但大多数突变都是有害的, 因此, 提高病毒的突变率使其超过病毒生存或致死性突变的阈值是一种有效的抗病毒策略。
在病毒培养模型中使用化学诱变剂会导致病毒变异率增加, 从而产生有缺陷或不具感染性的颗粒。小分子诱导病毒超突变有两种基本策略, 一是使用与病毒聚合酶或核酸相互作用的试剂以降低复制准确度进行直接突变; 二是使用影响涉及核酸合成或分解代谢等生化过程的试剂进行间接突变[92, 93]。
广谱抗病毒药物利巴韦林被证明是一种具有诱变性的核苷[94]。它的致突变性是由于围绕C3羰基键旋转而产生s-顺式和s-反式构象(图 14A)。这导致非特异性碱基与类似腺苷的s-顺式构象配对并能够与尿苷配对, 以及与类似鸟苷的s-反式构象配对并能够与胞苷配对, 这种碱基对错配会导致病毒突变率增加从而使药物发挥抗病毒活性[95]。此外, 能够对流感和其他多种RNA病毒有活性的β-D-N4-羟基胞苷(β-D-N4-hydroxycytidine, NHC) 也可诱导突变。通过产生互变异构和旋转异构体, NHC可以同时替代嘧啶核苷尿嘧啶和胞嘧啶, 从而诱导致死性突变(图 14B)。值得注意的是, NHC及其可口服的前药5′-异丙酯EIDD-2801还对SARS-CoV-2等多种冠状病毒有抑制活性[96]。因此, 抑制核苷酸生物合成的新途径是一种广谱抗病毒策略。
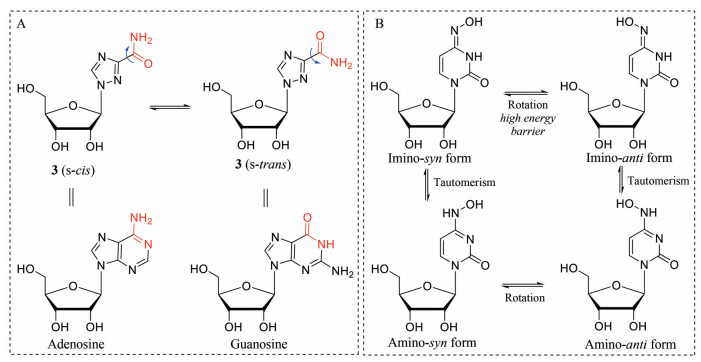
|
Figure 14 (A) The chemical structures of s-cis and s-trans conformer of ribavirin; (B) The chemical structures of the imino- and amino-forms of the mutagenic nucleoside analogue β-D-N4-hydroxycytidine (NHC) |
临床治疗慢性病毒感染的药物存在严重的交叉耐药、多重耐药和严重的毒副作用等问题, 当前面对新疫情, 仍无有效药物可用。因此, 广谱抗耐药性抗病毒药物研究是当前及未来的前沿及热点领域。本综述从药物化学的角度总结了近几年在抗病毒药物领域出现的新靶标和新策略, 包括靶向多种病毒的复制共性环节、靶向宿主细胞、靶向核酸及合成致死策略以开发广谱抗病毒药物来应对新发病毒疫情的暴发, 以及靶向蛋白复合物和多功能蛋白等新靶点和利用共价结合、蛋白降解、核酸降解和杀灭细胞等新策略解决现有药物耐药性的难题(图 15)。因此, 新靶点与新策略的不断发现将是研发新型抗病毒药物的有效途径。

|
Figure 15 Novel targets and strategies in antiviral drug discovery |
此外, 将药物化学与生物信息学和人工智能技术相结合, 对已上市的药物进行高通量表型筛选、反向药效团匹配虚拟筛选以探索药物新适应症, 以及建立快速的药物筛选系统和可靠的安全性和有效性评估体系也会加快抗病毒药物的研发进程。
作者贡献: 徐淑静负责全文的撰写; 丁当和张续杰负责全文的格式修改; 刘新泳和展鹏对论文进行整体的指导和修改。
利益冲突: 所有作者均声明不存在利益冲突。
| [1] |
Woolhouse M, Scott F, Hudson Z, et al. Human viruses: discovery and emergence[J]. Philos Trans R Soc Lond B Biol Sci, 2012, 367: 2864-2871. DOI:10.1098/rstb.2011.0354 |
| [2] |
Sun YY, Kang DW, Gao SH, et al. Recent advances in the research of nucleoside antiviral agents[J]. Chin J Med Chem (中国药物化学杂志), 2021, 31: 55-75. |
| [3] |
Ma Y, Wei FJ, Yu J, et al. Advances in research on HBV inhibitors based in new targets (1): capsid protein inhibitors[J]. Acta Pharm Sin (药学学报), 2020, 55: 554-565. |
| [4] |
Wei FJ, Ma Y, Yu J, et al. Advances in research on HBV inhibitors based on new targets (2): RNase H and others[J]. Acta Pharm Sin (药学学报), 2020, 55: 566-574. |
| [5] |
Morens DM, Fauci AS. Emerging pandemic diseases: how we got to COVID-19[J]. Cell, 2020, 182: 1077-1092. DOI:10.1016/j.cell.2020.08.021 |
| [6] |
Xiu SY, Zhang J, Ju H, et al. Progress on IFV drug targets and small molecule inhibitors[J]. Acta Pharm Sin (药学学报), 2020, 55: 611-626. |
| [7] |
Cohn LB, Chomont N, Deeks SG. The biology of the HIV-1 latent reservoir and implications for cure strategies[J]. Cell Host Microbe, 2020, 27: 519-530. DOI:10.1016/j.chom.2020.03.014 |
| [8] |
Wang Z, Samuel D, Kang DW, et al. The progress in the development of HIV inhibitors targeting protein-protein interactions[J]. Acta Pharm Sin (药学学报), 2019, 54: 620-628. |
| [9] |
Qi WT, Ju H, Hou LX, et al. Basic pharmaceutical knowledge in SARS-CoV-2 detection and drug discovery[J]. Univ Chem (大学化学), 2020, 35: 21-28. |
| [10] |
Li J, Jiang XY, Xu SJ, et al. Medicinal chemistry strategies in seeking coronavirus inhibitors[J]. Acta Pharm Sin (药学学报), 2020, 55: 537-553. |
| [11] |
De Clercq E, Li G. Approved antiviral drugs over the past 50 years[J]. Clin Microbiol Rev, 2016, 29: 695-747. DOI:10.1128/CMR.00102-15 |
| [12] |
Chaudhuri S, Symons JA, Deval J. Innovation and trends in the development and approval of antiviral medicines: 1987-2017 and beyond[J]. Antiviral Res, 2018, 155: 76-88. DOI:10.1016/j.antiviral.2018.05.005 |
| [13] |
Agnello S, Brand M, Chellat MF, et al. A structural view on medicinal chemistry strategies against drug resistance[J]. Angew Chem Int Ed Engl, 2019, 58: 3300-3345. DOI:10.1002/anie.201802416 |
| [14] |
Matthew AN, Leidner F, Lockbaum GJ, et al. Drug design strategies to avoid resistance in direct-acting antivirals and beyond[J]. Chem Rev, 2021, 121: 3238-3270. DOI:10.1021/acs.chemrev.0c00648 |
| [15] |
Xu SJ, Liu XY, Zhan P. New progress in research on respiratory syncytial virus inhibitors[J]. Acta Pharm Sin (药学学报), 2020, 55: 597-610. |
| [16] |
Fu ZP, Zhou ZX, Liu XY, et al. Advances in the study of antiviral natural products[J]. Acta Pharm Sin (药学学报), 2020, 55: 703-719. |
| [17] |
Huang TG, Sun L, Zhan P, et al. Recent advances in the research of broad-spectrum antiviral agents[J]. Acta Pharm Sin (药学学报), 2020, 55: 679-693. |
| [18] |
Fu ZP, Kang DW, Liu XY, et al. Advances of research on target-based anti-AIDS drugs[J]. Prog Pharm Sci (药学进展), 2020, 44: 681-697. |
| [19] |
Lu L, Su S, Yang H, et al. Antivirals with common targets against highly pathogenic viruses[J]. Cell, 2021, 184: 1604-1620. DOI:10.1016/j.cell.2021.02.013 |
| [20] |
Su X, Wang Q, Wen Y, et al. Protein- and peptide-based virus inactivators: inactivating viruses before their entry into cells[J]. Front Microbiol, 2020, 11: 1063. DOI:10.3389/fmicb.2020.01063 |
| [21] |
Pancera M, Lai YT, Bylund T, et al. Crystal structures of trimeric HIV envelope with entry inhibitors BMS-378806 and BMS-626529[J]. Nat Chem Biol, 2017, 13: 1115-1122. DOI:10.1038/nchembio.2460 |
| [22] |
Mercer J, Lee JE, Saphire EO, et al. SnapShot: enveloped virus entry[J]. Cell, 2020, 182: 786-786.e1. DOI:10.1016/j.cell.2020.06.033 |
| [23] |
Forssmann WG, The YH, Stoll M, et al. Short-term monotherapy in HIV-infected patients with a virus entry inhibitor against the gp41 fusion peptide[J]. SciTransl Med, 2010, 2: 63re3. |
| [24] |
Nishimura Y, Francis JN, Donau OK, et al. Prevention and treatment of SHIVAD8 infection in rhesus macaques by a potent d-peptide HIV entry inhibitor[J]. Proc Natl Acad Sci U S A, 2020, 117: 22436-22442. DOI:10.1073/pnas.2009700117 |
| [25] |
Holthausen DJ, Lee SH, Kumar VT, et al. An amphibian host defense peptide is virucidal for human H1 hemagglutinin-bearing influenza viruses[J]. Immunity, 2017, 46: 587-595. DOI:10.1016/j.immuni.2017.03.018 |
| [26] |
Xie D, Yao C, Wang L, et al. An albumin-conjugated peptide exhibits potent anti-HIV activity and long in vivo half-life[J]. Antimicrob Agents Chemother, 2010, 54: 191-196. DOI:10.1128/AAC.00976-09 |
| [27] |
Jia H, Gong P. A structure-function diversity survey of the RNA-dependent RNA polymerases from the positive-strand RNA viruses[J]. Front Microbiol, 2019, 10: 1945. DOI:10.3389/fmicb.2019.01945 |
| [28] |
Venkataraman S, Prasad B, Selvarajan R. RNA dependent RNA polymerases: insights from structure, function and evolution[J]. Viruses, 2018, 10: 1945. |
| [29] |
Engelman A, Cherepanov P. The structural biology of HIV-1: mechanistic and therapeutic insights[J]. Nat Rev Microbiol, 2012, 10: 279-290. DOI:10.1038/nrmicro2747 |
| [30] |
Jacome R, Campillo-Balderas JA, Ponce De Leon S, et al. Sofosbuvir as a potential alternative to treat the SARS-CoV-2 epidemic[J]. Sci Rep, 2020, 10: 9294. DOI:10.1038/s41598-020-66440-9 |
| [31] |
Ghosh AK, Osswald HL, Prato G. Recent progress in the development of HIV-1 protease inhibitors for the treatment of HIV/AIDS[J]. J Med Chem, 2016, 59: 5172-5208. DOI:10.1021/acs.jmedchem.5b01697 |
| [32] |
Kurt Yilmaz N, Swanstrom R, Schiffer CA. Improving viral protease inhibitors to counter drug resistance[J]. Trends Microbiol, 2016, 24: 547-557. DOI:10.1016/j.tim.2016.03.010 |
| [33] |
Rosenbaum MI, Clemmensen LS, Bredt DS, et al. Targeting receptor complexes: a new dimension in drug discovery[J]. Nat Rev Drug Discov, 2020, 19: 884-901. DOI:10.1038/s41573-020-0086-4 |
| [34] |
Massari S, Desantis J, Nizi MG, et al. Inhibition of influenza virus polymerase by interfering with its protein-protein interactions[J]. ACS Infect Dis, 2021, 7: 1332-1350. DOI:10.1021/acsinfecdis.0c00552 |
| [35] |
Toots M, Plemper RK. Next-generation direct-acting influenza therapeutics[J]. Transl Res, 2020, 220: 33-42. DOI:10.1016/j.trsl.2020.01.005 |
| [36] |
Mifsud EJ, Hayden FG, Hurt AC. Antivirals targeting the polymerase complex of influenza viruses[J]. Antiviral Res, 2019, 169: 104545. DOI:10.1016/j.antiviral.2019.104545 |
| [37] |
Zhang J, Hu Y, Musharrafieh R, et al. Focusing on the influenza virus polymerase complex: recent progress in drug discovery and assay development[J]. Curr Med Chem, 2019, 26: 2243-2263. DOI:10.2174/0929867325666180706112940 |
| [38] |
Lo CY, Tang YS, Shaw PC. Structure and function of influenza virus ribonucleoprotein[J]. Subcell Biochem, 2018, 88: 95-128. |
| [39] |
Fodor E, Te Velthuis AJW. Structure and function of the influenza virus transcription and replication machinery[J]. Cold Spring Harb Perspect Med, 2020, 10: a038398. DOI:10.1101/cshperspect.a038398 |
| [40] |
Furuta Y, Gowen BB, Takahashi K, et al. Favipiravir (T-705), a novel viral RNA polymerase inhibitor[J]. Antiviral Res, 2013, 100: 446-454. DOI:10.1016/j.antiviral.2013.09.015 |
| [41] |
Jones JC, Marathe BM, Lerner C, et al. A novel endonuclease inhibitor exhibits broad-spectrum anti-influenza virus activity in vitro[J]. Antimicrob Agents Chemother, 2016, 60: 5504-5514. DOI:10.1128/AAC.00888-16 |
| [42] |
Clark MP, Ledeboer MW, Davies I, et al. Discovery of a novel, first-in-class, orally bioavailable azaindole inhibitor (VX-787) of influenza PB2[J]. J Med Chem, 2014, 57: 6668-6678. DOI:10.1021/jm5007275 |
| [43] |
Toots M, Yoon JJ, Hart M, et al. Quantitative efficacy paradigms of the influenza clinical drug candidate EIDD-2801 in the ferret model[J]. Transl Res, 2020, 218: 16-28. DOI:10.1016/j.trsl.2019.12.002 |
| [44] |
Yuan S, Chu H, Singh K, et al. A novel small-molecule inhibitor of influenza A virus acts by suppressing PA endonuclease activity of the viral polymerase[J]. Sci Rep, 2016, 6: 22880. DOI:10.1038/srep22880 |
| [45] |
Yogaratnam J, Rito J, Kakuda TN, et al. Antiviral activity, safety, and pharmacokinetics of AL-794, a novel oral influenza endonuclease inhibitor: results of an influenza human challenge study[J]. J Infect Dis, 2019, 219: 177-185. DOI:10.1093/infdis/jiy410 |
| [46] |
Beigel JH, Nam HH, Adams PL, et al. Advances in respiratory virus therapeutics - a meeting report from the 6th isirv Antiviral Group conference[J]. Antiviral Res, 2019, 167: 45-67. DOI:10.1016/j.antiviral.2019.04.006 |
| [47] |
D'agostino I, Giacchello I, Nannetti G, et al. Synthesis and biological evaluation of a library of hybrid derivatives as inhibitors of influenza virus PA-PB1 interaction[J]. Eur J Med Chem, 2018, 157: 743-758. DOI:10.1016/j.ejmech.2018.08.032 |
| [48] |
Zhang J, Hu Y, Foley C, et al. Exploring ugi-azide four-component reaction products for broad-spectrum influenza antivirals with a high genetic barrier to drug resistance[J]. Sci Rep, 2018, 8: 4653. DOI:10.1038/s41598-018-22875-9 |
| [49] |
Yuan S, Chu H, Ye J, et al. Identification of a novel small-molecule compound targeting the influenza A virus polymerase PB1-PB2 interface[J]. Antiviral Res, 2017, 137: 58-66. DOI:10.1016/j.antiviral.2016.11.005 |
| [50] |
Zhan P, Pannecouque C, De Clercq E, et al. Anti-HIV drug discovery and development: current innovations and future trends[J]. J Med Chem, 2016, 59: 2849-2878. DOI:10.1021/acs.jmedchem.5b00497 |
| [51] |
Sun L, Zhang X, Xu S, et al. An insight on medicinal aspects of novel HIV-1 capsid protein inhibitors[J]. Eur J Med Chem, 2021, 217: 113380. DOI:10.1016/j.ejmech.2021.113380 |
| [52] |
Xu S, Sun L, Huang B, et al. Medicinal chemistry strategies of targeting HIV-1 capsid protein for antiviral treatment[J]. Future Med Chem, 2020, 12: 1281-1284. DOI:10.4155/fmc-2020-0084 |
| [53] |
Sun L, Dick A, Meuser ME, et al. Design, synthesis, and mechanism study of benzenesulfonamide-containing phenylalanine derivatives as novel HIV-1 capsid inhibitors with improved antiviral activities[J]. J Med Chem, 2020, 63: 4790-4810. DOI:10.1021/acs.jmedchem.0c00015 |
| [54] |
Margot N, Ram R, Rhee M, et al. Absence of lenacapavir (GS-6207) phenotypic resistance in HIV Gag cleavage site mutants and in isolates with resistance to existing drug classes[J]. Antimicrob Agents Chemother, 2021, 65: e02057-20. |
| [55] |
Link JO, Rhee MS, Tse WC, et al. Clinical targeting of HIV capsid protein with a long-acting small molecule[J]. Nature, 2020, 584: 614-618. DOI:10.1038/s41586-020-2443-1 |
| [56] |
Jordan PC, Stevens SK, Deval J. Nucleosides for the treatment of respiratory RNA virus infections[J]. Antivir Chem Chemother, 2018, 26: 2040206618764483. |
| [57] |
García-Cárceles J, Caballero E, Gil C, et al. Kinase inhibitors as underexplored antiviral agents[J]. J Med Chem, 2022, 65: 935-954. DOI:10.1021/acs.jmedchem.1c00302 |
| [58] |
Meertens L, Carnec X, Lecoin MP, et al. The TIM and TAM families of phosphatidylserine receptors mediate dengue virus entry[J]. Cell Host Microbe, 2012, 12: 544-557. DOI:10.1016/j.chom.2012.08.009 |
| [59] |
Meertens L, Labeau A, Dejarnac O, et al. Axl Mediates ZIKA virus entry in human glial cells and modulates innate immune responses[J]. Cell Rep, 2017, 18: 324-333. DOI:10.1016/j.celrep.2016.12.045 |
| [60] |
Weller ML, Amornphimoltham P, Schmidt M, et al. Epidermal growth factor receptor is a co-receptor for adeno-associated virus serotype 6[J]. Nat Med, 2010, 16: 662-664. DOI:10.1038/nm.2145 |
| [61] |
Wang X, Huong SM, Chiu ML, et al. Epidermal growth factor receptor is a cellular receptor for human cytomegalovirus[J]. Nature, 2003, 424: 456-461. DOI:10.1038/nature01818 |
| [62] |
Coleman CM, Sisk JM, Mingo RM, et al. Abelson kinase inhibitors are potent inhibitors of severe acute respiratory syndrome coronavirus and middle east respiratory syndrome coronavirus fusion[J]. J Virol, 2016, 90: 8924-8933. DOI:10.1128/JVI.01429-16 |
| [63] |
Verdonck S, Pu SY, Sorrell FJ, et al. Synthesis and structure-activity relationships of 3, 5-disubstituted-pyrrolo[2, 3-b]pyridines as inhibitors of adaptor-associated kinase 1 with antiviral activity[J]. J Med Chem, 2019, 62: 5810-5831. DOI:10.1021/acs.jmedchem.9b00136 |
| [64] |
Raghuvanshi R, Bharate SB. Recent developments in the use of kinase inhibitors for management of viral infections[J]. J Med Chem, 2022, 65: 893-921. DOI:10.1021/acs.jmedchem.0c01467 |
| [65] |
Papapanou M, Papoutsi E, Giannakas T, et al. Plitidepsin: mechanisms and clinical profile of a promising antiviral agent against COVID-19[J]. J Pers Med, 2021, 11: 668. DOI:10.3390/jpm11070668 |
| [66] |
White KM, Rosales R, Yildiz S, et al. Plitidepsin has potent preclinical efficacy against SARS-CoV-2 by targeting the host protein eEF1A[J]. Science, 2021, 371: 926-931. DOI:10.1126/science.abf4058 |
| [67] |
Xu Y, Jiang H. Potential treatment of COVID-19 by inhibitors of human dihydroorotate dehydrogenase[J]. Protein Cell, 2020, 11: 699-702. DOI:10.1007/s13238-020-00769-9 |
| [68] |
Diao Y, Lu W, Jin H, et al. Discovery of diverse human dihydroorotate dehydrogenase inhibitors as immunosuppressive agents by structure-based virtual screening[J]. J Med Chem, 2012, 55: 8341-8349. DOI:10.1021/jm300630p |
| [69] |
Xiong R, Zhang L, Li S, et al. Novel and potent inhibitors targeting DHODH are broad-spectrum antivirals against RNA viruses including newly-emerged coronavirus SARS-CoV-2[J]. Protein Cell, 2020, 11: 723-739. DOI:10.1007/s13238-020-00768-w |
| [70] |
Cammas A, Millevoi S. RNA G-quadruplexes: emerging mechanisms in disease[J]. Nucleic Acids Res, 2017, 45: 1584-1595. |
| [71] |
Hänsel-Hertsch R, Di Antonio M, Balasubramanian S. DNA G-quadruplexes in the human genome: detection, functions and therapeutic potential[J]. Nat Rev Mol Cell Biol, 2017, 18: 279-284. DOI:10.1038/nrm.2017.3 |
| [72] |
Zhao C, Qin G, Niu J, et al. Targeting RNA G-quadruplex in SARS-CoV-2: a promising therapeutic target for COVID-19?[J]. Angew Chem Int Ed Engl, 2021, 60: 432-438. DOI:10.1002/anie.202011419 |
| [73] |
De Cesco S, Kurian J, Dufresne C, et al. Covalent inhibitors design and discovery[J]. Eur J Med Chem, 2017, 138: 96-114. DOI:10.1016/j.ejmech.2017.06.019 |
| [74] |
Lonsdale R, Ward RA. Structure-based design of targeted covalent inhibitors[J]. Chem Soc Rev, 2018, 47: 3816-3830. DOI:10.1039/C7CS00220C |
| [75] |
Chan AH, Lee WG, Spasov KA, et al. Covalent inhibitors for eradication of drug-resistant HIV-1 reverse transcriptase: From design to protein crystallography[J]. Proc Natl Acad Sci U S A, 2017, 114: 9725-9730. DOI:10.1073/pnas.1711463114 |
| [76] |
Gao P, Song S, Frutos-Beltrán E, et al. Novel indolylarylsulfone derivatives as covalent HIV-1 reverse transcriptase inhibitors specifically targeting the drug-resistant mutant Y181C[J]. Bioorg Med Chem, 2021, 30: 115927. DOI:10.1016/j.bmc.2020.115927 |
| [77] |
Cromm PM, Crews CM. Targeted protein degradation: from chemical biology to drug discovery[J]. Cell Chem Biol, 2017, 24: 1181-1190. DOI:10.1016/j.chembiol.2017.05.024 |
| [78] |
Schneekloth JS, Fonseca FN, Koldobskiy M, et al. Chemical genetic control of protein levels: selective in vivo targeted degradation[J]. J Am Chem Soc, 2004, 126: 3748-3754. DOI:10.1021/ja039025z |
| [79] |
Bondeson DP, Mares A, Smith IE, et al. Catalytic in vivo protein knockdown by small-molecule PROTACs[J]. Nat Chem Biol, 2015, 11: 611-617. DOI:10.1038/nchembio.1858 |
| [80] |
Lai AC, Toure M, Hellerschmied D, et al. Modular PROTAC design for the degradation of oncogenic BCR-ABL[J]. Angew Chem Int Ed Engl, 2016, 55: 807-810. DOI:10.1002/anie.201507634 |
| [81] |
Olson CM, Jiang B, Erb MA, et al. Pharmacological perturbation of CDK9 using selective CDK9 inhibition or degradation[J]. Nat Chem Biol, 2018, 14: 163-170. DOI:10.1038/nchembio.2538 |
| [82] |
Remillard D, Buckley DL, Paulk J, et al. Degradation of the BAF complex factor BRD9 by heterobifunctional ligands[J]. Angew Chem Int Ed Engl, 2017, 56: 5738-5743. DOI:10.1002/anie.201611281 |
| [83] |
Gechijian LN, Buckley DL, Lawlor MA, et al. Functional TRIM24 degrader via conjugation of ineffectual bromodomain and VHL ligands[J]. Nat Chem Biol, 2018, 14: 405-412. DOI:10.1038/s41589-018-0010-y |
| [84] |
De Wispelaere M, Du G, Donovan KA, et al. Small molecule degraders of the hepatitis C virus protease reduce susceptibility to resistance mutations[J]. Nat Commun, 2019, 10: 3468. DOI:10.1038/s41467-019-11429-w |
| [85] |
Ghidini A, Cléry A, Halloy F, et al. RNA-PROTACs: degraders of RNA-binding proteins[J]. Angew Chem Int Ed Engl, 2021, 60: 3163-3169. DOI:10.1002/anie.202012330 |
| [86] |
Haniff HS, Tong Y, Liu X, et al. Targeting the SARS-CoV-2 RNA genome with small molecule binders and ribonuclease targeting chimera (RIBOTAC) degraders[J]. ACS Cent Sci, 2020, 6: 1713-1721. DOI:10.1021/acscentsci.0c00984 |
| [87] |
Costales MG, Matsumoto Y, Velagapudi SP, et al. Small molecule targeted recruitment of a nuclease to RNA[J]. J Am Chem Soc, 2018, 140: 6741-6744. DOI:10.1021/jacs.8b01233 |
| [88] |
Herschhorn A, Gu C, Espy N, et al. A broad HIV-1 inhibitor blocks envelope glycoprotein transitions critical for entry[J]. Nat Chem Biol, 2014, 10: 845-852. DOI:10.1038/nchembio.1623 |
| [89] |
Contarino M, Bastian AR, Kalyana Sundaram RV, et al. Chimeric cyanovirin-MPER recombinantly engineered proteins cause cell-free virolysis of HIV-1[J]. Antimicrob Agents Chemother, 2013, 57: 4743-4750. DOI:10.1128/AAC.00309-13 |
| [90] |
Gaffney A, Nangarlia A, Ang CG, et al. HIV-1 Env-dependent cell killing by bifunctional small-molecule/peptide conjugates[J]. ACS Chem Biol, 2021, 16: 193-204. DOI:10.1021/acschembio.0c00888 |
| [91] |
Vignuzzi M, Stone JK, Arnold JJ, et al. Quasispecies diversity determines pathogenesis through cooperative interactions in a viral population[J]. Nature, 2006, 439: 344-348. DOI:10.1038/nature04388 |
| [92] |
Benigni R, Bossa C. Mechanisms of chemical carcinogenicity and mutagenicity: a review with implications for predictive toxicology[J]. Chem Rev, 2011, 111: 2507-2536. DOI:10.1021/cr100222q |
| [93] |
Bonnac LF, Mansky LM, Patterson SE. Structure-activity relationships and design of viral mutagens and application to lethal mutagenesis[J]. J Med Chem, 2013, 56: 9403-9414. DOI:10.1021/jm400653j |
| [94] |
Crotty S, Maag D, Arnold JJ, et al. The broad-spectrum antiviral ribonucleoside ribavirin is an RNA virus mutagen[J]. Nat Med, 2000, 6: 1375-1379. DOI:10.1038/82191 |
| [95] |
Parker WB. Metabolism and antiviral activity of ribavirin[J]. Virus Res, 2005, 107: 165-171. DOI:10.1016/j.virusres.2004.11.006 |
| [96] |
Huchting J. Targeting viral genome synthesis as broad-spectrum approach against RNA virus infections[J]. Antivir Chem Chemother, 2020, 28: 2040206620976786. |
 2022, Vol. 57
2022, Vol. 57


