甘草(Glycyrrhiza uralensis Fisch.) 为豆科多年生草本植物, 主要分布在宁夏、甘肃、新疆和内蒙古等干旱、半干旱区域[1]。甘草药用部位为根及根茎, 具有补脾益气, 清热解毒, 祛痰止咳, 缓急止痛, 调和诸药的功效[2], 为我国最常用的大宗药材之一, 素有“十方九草”之说, 被称为“国老”, 在美国、日本等国家又被称为“仙草”、“神草”[3]。现代药理研究发现甘草具有抗炎、抗癌、抗病毒、保肝等多种功效[4-7]。除了药用, 甘草还作为添加剂和矫味剂应用于食品、烟草、日化以及畜牧等行业中。此外, 甘草是国家重点保护、管理的野生固沙植物, 在保护生态环境、防止沙漠化方面起着重要作用[8]。因甘草用途广泛, 自然资源匮乏, 被科技部列为首批中药现代化建设中药材生产质量管理规范(GAP) 的中药材之一。因此, 人工栽培甘草成为市场需求的主要来源[9]。但是, 由于干旱等环境因子和甘草遗传因素的影响, 栽培甘草和野生甘草的活性成分含量存在显著差异, 栽培甘草的甘草酸含量普遍偏低, 达不到药典标准, 制约了甘草资源的可持续发展[10]。
脱落酸(abscisic acid, ABA) 属于倍半萜类激素, 在植物生长过程中起到重要的作用。ABA除了促使叶片脱落外, 还能够通过内源合成、信号转导以及与其他激素的互作等多方面调控次生代谢物质的合成[11]。有研究用ABA处理一年生甘草后, 甘草的活性成分含量明显增高, 这表明ABA与甘草活性成分的积累密切相关[12]。ABA信号调控途径是一个复杂的生物学过程, 需要多种信号因子、转录因子和功能基因的参与。有研究报道碱性亮氨酸拉链(basic leucine zipper, bZIP) 转录因子能够通过参与ABA信号转导通路来调控次生代谢产物的合成[13]。Zhang等[14]为了阐明ABA介导的青蒿素生物合成机制, 采用100 μmol·L-1的ABA处理黄花蒿(Artemisia annua) 植株, 双荧光素酶检测结果显示ABA能增强黄花蒿bZIP转录因子AabZIP1对青蒿素合成途径的关键酶基因ADS、CYP71AV1启动子的转录活性, 而且在AabZIP1转录因子基因过表达且青蒿素含量明显增加的黄花蒿植株中分离出ABA受体蛋白AaPYL9, 表明AabZIP1可在ABA诱导合成青蒿素过程中起到关键的作用。短角蒲公英(Taraxacum brevicorniculatum) 中的小橡胶粒子蛋白(SRPP) 可在橡胶粒子结构稳定和天然橡胶合成中发挥重要作用[15]。Fricke等[16]研究发现短角蒲公英bZIP转录因子A亚家族成员TbbZIP1可与TbSRPP1基因启动子区域的顺式作用元件特异性结合, ABA能促进TbbZIP1和TbSRPP基因的表达, TbbZIP1转录因子在ABA信号转导途径中正向调控TbSRPP1基因的转录, 从而影响天然橡胶的合成。bZIP转录因子由碱性(basic) 区和亮氨酸拉链(leucine zipper) 区两部分构成。其碱性区高度保守, 位于bZIP结构域的N端, 亮氨酸拉链区保守度较低, 位于bZIP结构域的C端[17]。根据碱性区及其他保守序列的相似性, Jakoby等[18]将拟南芥的75个bZIP转录因子(AtbZIP) 分为A~I和S共10个亚家族。TGA是拟南芥中抗病相关的bZIP转录因子, 当病原菌侵染植物后, 植物体内水杨酸含量上升, 使得中介蛋白NPR1从多聚体转化成单体同TGA蛋白作用, 以此来调控致病相关基因PR的表达, 从而增强植物的抗病性[19]。Zhang等[20]对拟南芥的研究发现, 其bZIP转录因子HY5和HYH通过上调花色素苷生物合成途径下游的关键基因DFR, 促进花色素苷的积累。bZIP转录因子在真核生物中分布广泛、相对保守[17], 随着全基因组测序技术的进步, 目前已在多种高等植物中发现了bZIP转录因子, 如在玉米(Zea mays) 基因组中鉴定出125个bZIP转录因子[21]、水稻(Oryza sativa) 89个[22]、大豆(Glycine max) 131个[23]、阳芋(Solanum tuberosum) 65个[24]、烟草(Nicotiana tabacum) 132个[25]、紫苜蓿(Medicago sativa) 57个[26]。bZIP转录因子在植物生物或非生物胁迫响应[27]、生长发育[28]、次生代谢产物合成[14]等方面具有重要的调控作用。
本研究测定不同浓度ABA处理的甘草转录组数据并基于Mochida等[29]发表的甘草全基因组数据, 以拟南芥基因组中发现的bZIP氨基酸序列为参照, 鉴定出甘草bZIP转录因子并进行分析, 为进一步开展甘草bZIP转录因子功能研究, 阐明其调控机制奠定了基础。
材料与方法实验材料 材料采自于甘肃省武威市民勤县, 经中国医学科学院药用植物研究所林余霖研究员鉴定为豆科植物甘草Glycyrrhiza uralensis, 凭证标本保存于中国医学科学院药用植物研究所。样品分别用0、25、50 mg·L-1的ABA对两年生甘草的根部进行浸泡处理, 并在处理后3, 6, 12 h收集根样品, 立刻放入液氮中冷冻, 并将其储存在-80 ℃冰箱备用。
总RNA提取及转录组测序 总RNA采用RNAprep Pure Plant Plus Kit试剂盒(TIANGEN, DP441, 中国) 法提取, DNase I处理去除基因组DNA后, 根据操作流程利用Illumina高通量测序平台进行RNA测序。
甘草bZIP转录因子鉴定与分析 以拟南芥基因组中发现的bZIP氨基酸序列为参照, 利用BLASTP (the protein basic local alignment search tool) 对甘草基因组中的bZIP基因家族进行了E值阈值为1×10-5的鉴定, 并用DNAMAN软件去除冗余基因, 用Apollo软件对编码序列、DNA序列和蛋白质序列信息进行了校正。利用Gene Structure Display Server (GSDS 2.0) 在线工具(http://gsds.gao-lab.org/) 分析基因结构。利用ExPaSy网站(http://gsds.gao-lab.org/) 测定bZIPs的氨基酸数量、分子质量(MW)、理论等电点(PI)、不稳定指数(II)、脂肪族指数(AI) 和亲水性总平均数(GRAVY) 等性能。Multiple Em for Motif Elicitation (MEME, http://memesuite.org/) 被用来识别motifs, E值小于1×10-30。应用DNAStar Protean软件提供的Garnier-Robson法预测bZIP编码蛋白的二级结构, SWISS-MODEL在线工具(https://swissmodel.expasy.org/interactive) 预测蛋白三级结构, Pfam网站(http://pfam.xfam.org/search/batch) 鉴定bZIP编码蛋白结构域。
bZIPs的系统发育分析 为了确定bZIP家族的进化关系, 利用MAFFT软件对拟南芥和甘草的bZIP全长蛋白序列进行比对, 并利用IQ-TREE软件构建了最大似然(ML) 系统发育树, Bootstrap重复次数为1 000。
qRT-PCR验证bZIP转录因子基因表达 按照PrimeScript™ II 1st Strand cDNA Synthesis Kit试剂盒中(TaKaRa, 6210A, 中国) 的说明, 将RNA反转录得到cDNA第1链。使用Primer Premier 6软件设计荧光定量分析的引物(表 1)。以甘草cDNA为模板, actin作为内参基因, 反应体系按照TB Green® Premix Ex Taq™试剂盒(TaKaRa, RR420A, 中国) 说明书配置, 每个反应重复3次, 在CFX96 Real-Time PCR Detection System中进行。根据得到的Ct值, 采用2-ΔΔCt方法计算基因的相对表达。
| Table 1 The primers of six GubZIPs genes of G. uralensis used for qRT-PCR |
本研究以75个拟南芥bZIP转录因子基因AtbZIPs为参考, 对甘草bZIP家族基因组序列数据进行检测。基于甘草基因组和转录组数据, 共鉴定到69个甘草bZIP转录因子家族基因, 命名为GubZIP1-69。根据它们与拟南芥AtbZIPs的同源相似性, 分为10个亚家族, 分别为A~I以及S亚家族, 每个亚家族基因的结构和功能各不相同(图 1)。其中S亚家族基因数目最多, 为16个; B亚家族数目最少, 只有1个。GubZIP44和GubZIP66分别与拟南芥中未分组的AtbZIP60和AtbZIP62序列聚为一支, 归为U亚家族。
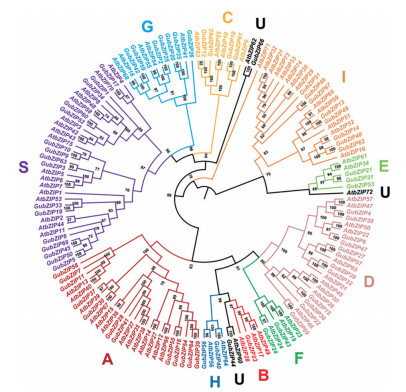
|
Figure 1 Phylogenetic relationships of bZIP genes of G. uralensis and A. thaliana. A total of 69 GubZIPs from G. uralensis and 75 AtbZIPs from A. thaliana were included to construct maximum likelihood (ML) tree. Node labels represent values for bootstrap support |
对甘草GubZIPs基因的基因结构进行分析。结果显示, 这些亚家族基因之间内含子的数量和长度上存在差异(图 2)。GubZIPs基因的内含子数量为0~15个。G亚家族平均内含子数量最多, S亚家族最少。G亚家族的GubZIP59基因含有的内含子数量最多, 为15个。S亚家族中除了GubZIP45有1个内含子外, 其他基因都没有内含子, F亚家族的GubZIP24也没有内含子。同一亚家族的大多数成员具有相似的内含子/外显子结构, 这与系统发育树中描述的进化关系一致。
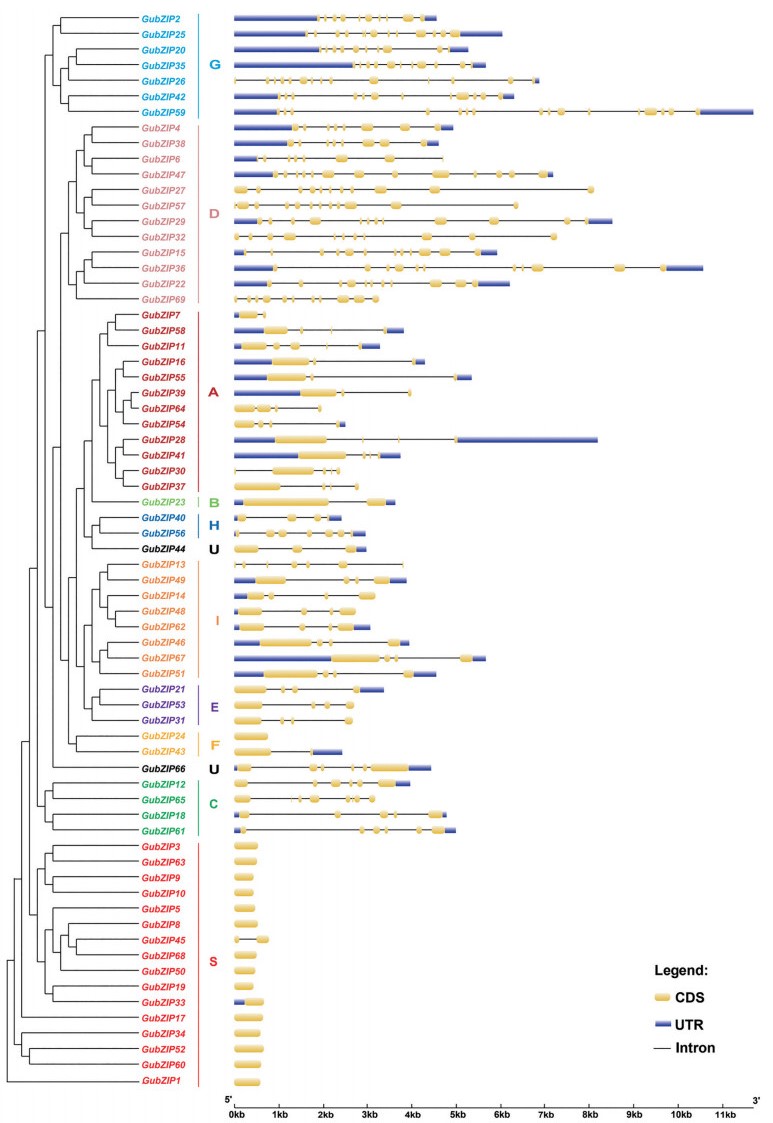
|
Figure 2 Exon-intron structure of GubZIPs genes based on their phylogenetic relationships |
首先, 对69个GubZIPs基因的物理特性进行了分析。结果显示, 基因长度为432~11 690 bp, 蛋白质编码区CDS长度为396~2 367 bp, GubZIPs蛋白序列包含132~788个氨基酸, 蛋白的分子质量MW为15.68~85.39 kDa。理论等电点PI值在4.97到10.18之间, 平均为7.26, 有39个蛋白的PI值小于7.00, 30个蛋白的PI值大于7.00, 说明GubZIPs蛋白氨基酸酸碱性较为平衡。只有GubZIP24、GubZIP43和GubZIP56蛋白的不稳定指数II值小于40, 为稳定蛋白, 其余为不稳定蛋白。脂肪族指数AI值在46.20~92.93之间。这些GubZIPs蛋白家族成员的总平均亲水性指数GRAVY均为负值, 均为亲水性蛋白(表 2)。
| Table 2 List of GubZIPs genes and their basic characterizations. CDS: Coding sequence; AA: Amino acids; MW: Molecular weight; PI: Theoretical pI; II: Instability index; AI: Aliphatic index; GRAVY: Grand average of hydropathicity |
利用MEME在线预测甘草bZIP基因家族的保守motif, 结果预测了15个motif, 分别为motif1~motif15 (表 3)。除GubZIP20外, 所有的bZIP蛋白质都含有motif1, 说明motif1在甘草bZIP蛋白质中分布最广, 保守性最强。不同motif在蛋白质序列中的分布可为研究不同亚家族基因功能差异作为参考, 一些motif只存在特定的基因家族中, motif14是G家族特有的, motif2、motif5、motif6、motif7和motif8是D家族特有的, motif9和motif11是A家族特有的, motif15是I家族特有的, motif10只存在于I家族和E家族, motif12只存在于G家族、B家族和S家族。同一个亚家族所含有的motif高度相似, 支持了它们的密切进化关系(图 3)。
| Table 3 The protein motifs and its composition of GubZIP of G. uralensis |
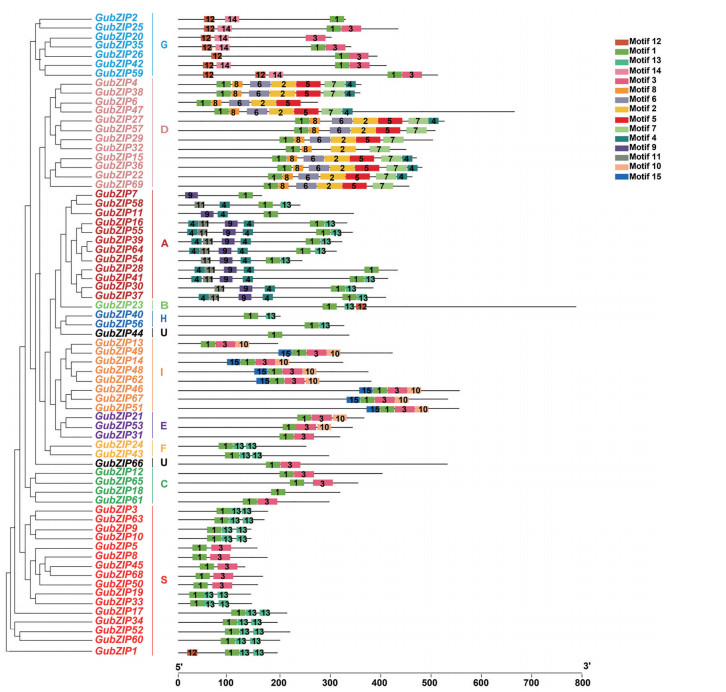
|
Figure 3 Classification of G. uralensis bZIP family and conversed motifs |
为了研究GubZIPs基因在不同外源ABA胁迫下的表达模式, 本研究测定了它们的转录组数据, 并计算其FPKM值。69个GubZIPs基因在不同外源ABA胁迫下的相对表达量如图 4所示。有30个GubZIPs基因表达量相比于对照组在所有条件下均升高, 其中GubZIP1、GubZIP5、GubZIP8、GubZIP30、GubZIP33和GubZIP56这6个基因表达量是对照组的至少3倍, 说明这些基因对ABA胁迫更敏感, 可能参与了ABA信号通路的调控, 可作为候选基因继续研究。此外, GubZIP26、GubZIP45、GubZIP48、GubZIP53和GubZIP55基因表达量相比于对照组在所有条件下均下降。GubZIP54在所有条件下均检测不到或没有表达。

|
Figure 4 Expression profiles analysis of GubZIPs genes under various ABA stresses at different times. CK indicated the plants without soaking and was used as control. The 3, 6 and 12 h labels indicated the time after treatment. The 0, 25, and 50 mg·L-1 labels indicated the different ABA concentrations. The bar on the top right corner represented the FPKM values, and different colors denote various expression levels |
应用DNAStar Protean软件提供的Garnier-Robson法预测GubZIP1、GubZIP5、GubZIP8、GubZIP30、GubZIP33和GubZIP56蛋白的二级结构, 二级结构以α螺旋为主, 除GubZIP33没有β折叠外, 都伴有β折叠、转角(turn)、无规则卷曲(coil) 结构。应用SWISS-MODLE同源建模预测蛋白三级结构, 结果显示GubZIP1和GubZIP30蛋白为同源二聚体结构, 可以与镁离子结合; GubZIP8和GubZIP33蛋白为异源二聚体结构, 可以与鸟苷二磷酸、鸟苷三磷酸和镁离子结合; GubZIP56蛋白为单体结构, 可以与锌离子结合; GubZIP5蛋白为同源二聚体结构(图 5)。
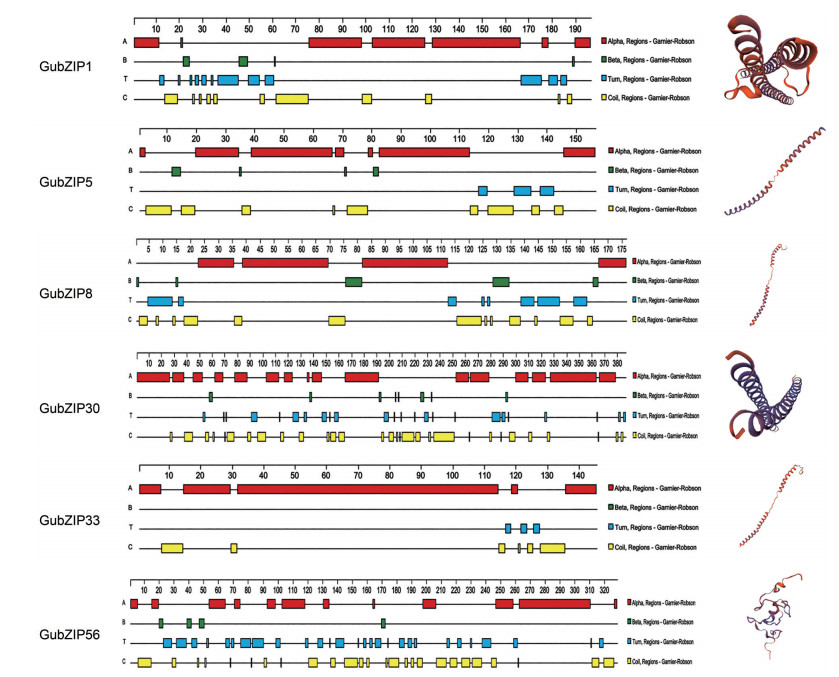
|
Figure 5 Secondary and tertiary structures of the encoded proteins |
不同结构域组合所产生的蛋白质本质上是不同的, 蛋白质结构域的识别对于分析蛋白质功能尤为重要。图 6列出了GubZIP1、GubZIP5、GubZIP8、GubZIP30、GubZIP33和GubZIP56的蛋白结构域。结果显示所有的候选基因都含有bZIP_1结构域, GubZIP56除了含有bZIP_1结构域还含有锌指蛋白结构域(zf_UDP)。
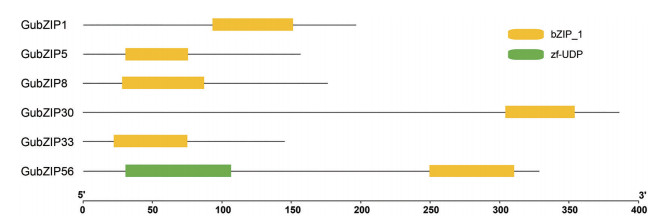
|
Figure 6 Protein functional domains of GubZIPs genes |
转录组数据结果表明, 不同浓度的外源ABA浸泡甘草根时, GubZIP1、GubZIP5、GubZIP8、GubZIP30、GubZIP33和GubZIP56基因优先表达, 说明这些基因可能参与了ABA信号通路的调控。为进一步验证这些基因的表达模式, 分别用0、25、50 mg·L-1的ABA对两年生甘草的根部进行浸泡处理3、6和12 h后, 采用qRT-PCR检测GubZIP1、GubZIP5、GubZIP8、GubZIP30、GubZIP33和GubZIP56基因的表达情况(图 7)。结果显示这些基因的表达模式与ABA的浓度相关, 在0和25 mg·L-1浓度ABA处理下表达模式相同, 除了GubZIP5和GubZIP30在0 mg·L-1浓度ABA处理下相对表达量变化不显著外, 其他基因相对表达量均在处理后3 h显著上升, 6 h之后逐渐下降, 12 h后基因相对表达量较0 h均显著下降。在50 mg·L-1浓度ABA处理下, 基因的表达模式与0和25 mg·L-1浓度ABA处理下相反, 基因相对表达量均在处理后3 h下降, 6 h之后逐渐上升, 除了GubZIP8, 12 h后基因相对表达量较0 h均显著上升。
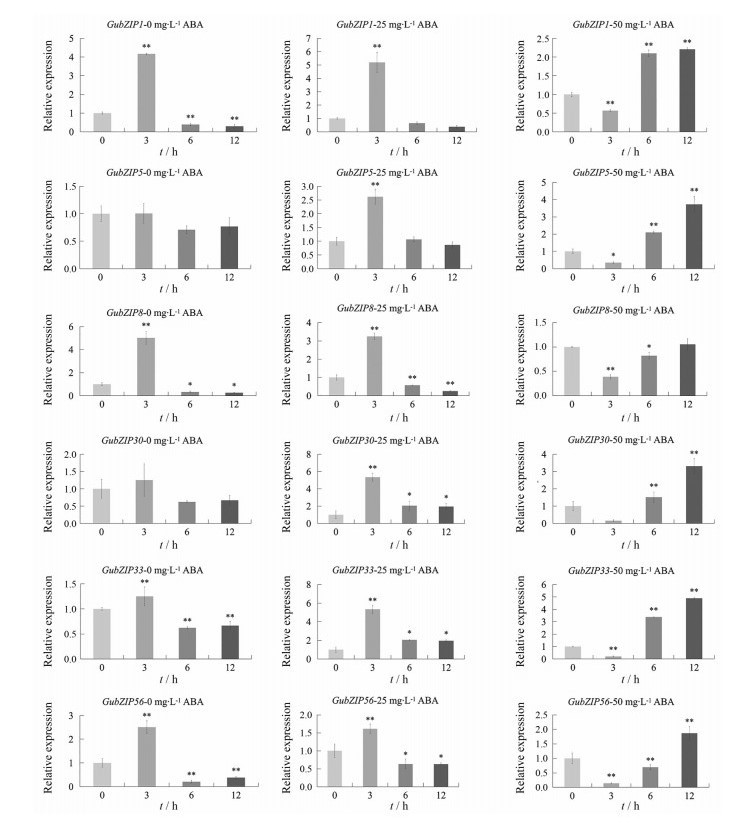
|
Figure 7 Expression patterns of GubZIPs genes in response to ABA. The relative expression of GubZIPs was examined after treatments with diverse concentrations of exogenous ABA for the indicated period. The actin gene in G. uralensis was used as internal control. The gene expression value at 0 h was set as 1. The values are expressed as means ± SD (n = 3). *P < 0.05, **P < 0.01 vs expression value at 0 h |
ABA信号调控途径是一个复杂的生物学过程, 需要多种信号因子、转录因子和功能基因的参与, 其过程大致如下: ABA可与其受体PYR/PYL/RCAR (pyrabactin resistance/PYR-like/regulatory components of ABA receptors, PYLs) 蛋白家族结合[30]。ABA的共受体A类型的蛋白磷酸酶2C (PP2Cs) 如ABI1、ABI2、HAB1和PP2CA可使PYL与ABA的亲和力增加近100倍[31]。ABA进入PYL的中央疏水区域, 诱导疏水区域处于关闭状态, 并创建一个PP2Cs结合表面[32]。在PYL-ABA-PP2C复合物的里面, PP2C中一个色氨酸残基插入到ABA结合区域, 将ABA锁定在该区域, 复合物中PP2C的蛋白磷酸酶活性被ABA-PYL复合体所抑制[30]。ABA-PYL对PP2Cs的结合和抑制导致蔗糖非酵解型蛋白激酶SnRK2s (sucrose non-fermenting 1-related protein kinase 2s) 从PP2Cs的结合中释放出来。释放的SnRK2s通过自身磷酸化激活, 并磷酸化许多下游的效应蛋白包括转录因子、SLAC1 (slow anion channel-associated 1)、RbohD/F (respiratory burst oxidase homologs), 以此来实现信号的传递以及细胞对ABA的响应[33]。ABA激活的SnRK2s也可以使质膜NADPH氧化酶RbohF磷酸化, 在质外体空间产生O2-, O2-随后形成H2O2, 作为信号分子调节包括气孔关闭在内的各种ABA应答过程[34]。H2O2也可以调节Ca2+信号从而影响ABA的应答[35]。除了诱导H2O2和Ca2+信号, ABA也引发一氧化氮(NO) 和磷脂分子如PA (磷脂酸) 的合成[36], NO抑制SnRK2s和PYLs, PA调节Rbohs蛋白活性[36], 继而进行信号转导, 通过调节蛋白与转录因子等特异结合, 激活干旱胁迫下ABA依赖基因的表达, 使植物对干旱胁迫做出响应[37, 38]。甘蓝型油菜中参与干旱胁迫下的ABA信号转导途径的相关转录因子BnMYC2、BnNAC485[39]和蛋白激酶BnCIPK6、BnCBL1和BnGA1[40, 41]基因也被鉴定或分离出来, 这些基因在ABA的处理下其表达会上调。
bZIP转录因子是普遍存在于植物中的一类转录因子, 本研究以75个拟南芥AtbZIPs基因为参考, 基于甘草基因组和转录组数据, 共鉴定到69个甘草bZIP转录因子家族基因, 与拟南芥AtbZIPs序列构建系统发育树, 根据同源相似性, 将甘草bZIP转录因子(GubZIPs) 分为A~I以及S这10个亚家族。拟南芥AtbZIPs中有3个基因AtbZIP60、AtbZIP62和AtbZIP72并未分到这10个亚家族中, 甘草的GubZIP44和GubZIP66分别与拟南芥中的AtbZIP60和AtbZIP62序列聚为一支, 将其归为U亚家族。每个亚家族基因都在植物的生长发育过程中起到重要的作用, A亚家族主要参与ABA和各种胁迫信号; B和F亚家族在盐胁迫中起到重要的作用; C亚家族较为保守, 可以响应环境胁迫和病原侵染; D亚家族的作用体现在抗击病原和植物发育两个方面; E亚家族与花粉壁的形成有关, 且调控花粉发育的几个代谢途径; G和H亚家族主要参与光信号的感应和传导; I和S亚家族都参与微管系统的调节, 但S亚家族功能更为广泛, 受干旱、低温、无氧和机械损伤处理激活, 还参与花发育等[42]。其中, A亚族bZIP基因的信号传递主要依赖于ABA信号途径, 如拟南芥A亚族的AREB/ABF (ABA-response element binding protein/ABA-responsive element binding factor) 类bZIP转录因子, 可激活非生物逆境胁迫下的ABA依赖基因的表达[43]。其中, ABF1主要参与低温、ABA胁迫应答, ABF2与ABF4主要参与干旱、高盐、高温及氧化胁迫应答, 而ABF3则受到ABA、低温胁迫的诱导[43-45]。
有研究表明bZIP转录因子对盐和干旱胁迫有很强的响应功能, 其基因的高表达分别使拟南芥[23]、棉花[46]、水稻[47]和玉米[48]具有良好的耐旱性和耐盐性。此外, bZIP转录因子能够通过参与ABA信号转导通路来调控次生代谢产物的合成。植物次生代谢产物种类繁多、结构迥异, 一般可分为萜类、酚类(如黄酮类) 和含氮化合物(如生物碱等) 3大类, bZIP转录因子对这3类化合物生物合成的调控均有报道[13]。Zhang等[14]首先从黄花蒿腺毛高表达的64个bZIP转录因子中鉴定出6个A亚家族成员, 并将这6个转录因子及青蒿素合成途径的关键酶基因ADS和CYP71AV1转化烟草叶片, 双荧光素酶检测结果预测AabZIP1可激活这2个关键酶基因的启动子, 酵母单杂交实验结果显示AabZIP1可与ADS和CYP71AV1基因启动子元件ABRE直接结合, 并激活2个关键酶基因的表达; 之后构建黄花蒿AabZIP1过表达载体转化黄花蒿植株, 可使青蒿素含量明显增加, 说明转录因子AabZIP1正向调控青蒿素的合成; 最后又通过实验阐明了ABA介导的青蒿素生物合成机制。OsbZIP23转录因子在水稻抗旱性中起重要作用, 有研究发现OsbZIP23可以正向调控OsPP2C49, 而在水稻中过表达OsPP2C49会导致ABA反应敏感性显著降低和快速脱水, ABA生物合成的关键基因OsNCED4也受到OsbZIP23的正调控, 表明OsbZIP23在水稻ABA信号转导、生物合成和抗旱性中起中枢调节作用[49]。Okada等[50]构建了水稻bZIP转录因子OsTGAP1基因过表达载体转化水稻, 使萜类植保素生物合成途径中的5个关键酶基因表达量大幅提高, 萜类植保素含量明显增加; 当敲除OsTGAP1基因时, 上述5个关键酶基因几乎不表达, 萜类植保素的合成受到显著抑制, 说明OsTGAP1是萜类植保素生物合成途径的核心调控因子。覆盆子果实的颜色有红色、金色和黑色, 黄酮类次生代谢产物花青素是影响其果实颜色的重要因素, 研究表明bZIP转录因子可影响红色覆盆子中花青素的合成[51]。柿子中MYB转录因子DkMyb4可正向调控原花青素合成途径基因的表达, 进而影响其含量[52]。Akagi等[53]从柿子果实中分离出一个高表达的bZIP转录因子基因DkbZIP5, 构建过表达载体转化柿子叶片, 检测发现DkMyb4基因表达量增加, 愈伤组织中积累大量的原花青素。bZIP调控次生代谢产物合成的机制较为复杂, 有1个转录因子调控一种次生代谢产物生物合成的情况[16], 也有2个或2种不同转录因子共同调节的现象[53, 54]。Zhang等[55]发现丹参bZIP基因家族中的SmbZIP7和SmbZIP20可能参与丹参酮生物合成的调控。刘宝玲等[56]发现在干旱和盐胁迫条件下, 多数谷子SibZIPs基因不同程度地被诱导表达, 预示着部分SibZIP成员在谷子干旱和盐胁迫响应中起重要作用, 共表达关联性分析进一步揭示19个谷子SibZIP转录因子可通过与蛋白激酶或NPR1相关调节蛋白等互作介导谷子胁迫响应。bZIP转录因子识别核心序列为ACGT的顺式作用元件。ABRE (ABA-responsive element) 在依赖ABA的基因表达中作为顺式作用元件, 在很多ABA响应基因中均存在, 能被很多bZIP类转录因子识别, 调控逆境诱导表达基因的表达[57]。Nieva等[58]研究发现玉米的两个bZIP转录因子EmBP-2和ZmBZ-1都是与ABREs结合并参与了ABA诱导基因rab28的表达, 它们的活性受ABA和磷酸化的调节。Dröge-Laser等[59]用无毒的丁香假单胞菌处理大豆bZIP转录因子G/HBF-1, 其被快速磷酸化, 并与关键酶CHS基因启动子区顺式作用元件H-box和G-box相结合, 激活CHS基因的表达, 黄酮类植保素的含量大幅增加。有的bZIP转录因子在化合物生物合成的过程中起到负调控作用。所有萜类吲哚生物碱的合成都起源于中心分子异胡豆苷, 而异胡豆苷合酶(STR) 是催化其生成的关键酶。Sibéril等[54]发现长春花bZIP转录因子CrGBF1和CrGBF2能特异性结合G-box区; 将Crgbf1和Crgbf2融合基因转化长春花细胞, 检测到STR启动子被显著抑制, 说明GBF1和GBF2负调控关键酶STR基因的表达, 减少萜类吲哚生物碱的合成与积累。植物次生代谢产物生物合成的途径烦琐, 且受到多种酶催化, 但转录因子可以激活多个次生代谢产物合成酶基因协同表达, 从而有效调控次生代谢途径, 因此寻找调控酶基因表达的转录因子对植物次生代谢研究有着重要的意义, 已有越来越多的转录因子也在不同的药用植物中被鉴定[60-63]。
本研究基于不同浓度ABA处理的甘草转录组数据, 计算了69个GubZIPs基因在不同外源ABA胁迫下的相对表达量, 并以此筛选出6个可能参与ABA信号通路的调控的基因, 即GubZIP1、GubZIP5、GubZIP8、GubZIP30、GubZIP33和GubZIP56, 作为候选基因继续研究。为验证候选基因的表达模式, 采用qRT-PCR检测不同浓度ABA处理下基因的表达情况。本研究实验过程中将两年生甘草浸泡在ABA溶液中, 与传统的甘草种植相比, 实验过程中甘草根所处的环境主要为水, 故设计0 mg·L-1的ABA处理作为参照。结果显示基因在25 mg·L-1浓度ABA处理下的表达模式与0 mg·L-1浓度ABA处理下相同, 说明25 mg·L-1的ABA浓度过低, 没有引起基因的表达发生变化, 而在50 mg·L-1浓度ABA处理下的表达模式与0 mg·L-1浓度ABA处理下相比发生了明显的变化, 说明这些基因的表达响应50 mg·L-1浓度的ABA。本研究不仅有利于对甘草bZIP转录因子分类、性质和结构的认识与了解, 还为进一步开展甘草bZIP转录因子功能研究, 阐明其调控机制奠定了基础。
作者贡献: 武立伟负责样品制备、实验操作、数据分析和论文撰写; 徐志超负责研究课题总体设计生信分析和论文修改; 王清负责实验操作; 聂丽萍负责数据分析; 崔英贤负责样品制备; 王瑀负责提供实验材料; 宋经元负责实验指导; 姚辉负责研究课题总体设计、实验指导、论文修改及论文终稿审查。
利益冲突: 本文所有作者均声明不存在利益冲突。
| [1] |
Li S, Xu L, Chen D, et al. Flora of China. Vol. 10 (Fabaceae)[M]. Beijing: Science Press, 2010.
|
| [2] |
Chinese Pharmacopoeia Commission. Pharmacopoeia of the People's Republic of China (中华人民共和国药典)[S]. Beijing: China Medical Science Press, 2020.
|
| [3] |
Yang R, Wang L, Liu Y. Research progress on tissue culture of Glycyrrhizae Radix et Rhizoma[J]. Chin Tradit Herb Drugs (中草药), 2014, 45: 1796-1802. |
| [4] |
Hawthorne S, Gallagher S. Effects of glycyrrhetinic acid and liquorice extract on cell proliferation and prostate-specific antigen secretion in LNCaP prostate cancer cells[J]. J Pharm Pharmacol, 2008, 60: 661-666. |
| [5] |
Ming LJ, Yin AC. Therapeutic effects of glycyrrhizic acid[J]. Nat Prod Commun, 2013, 8: 415-418. |
| [6] |
Shin EM, Zhou HY, Guo LY, et al. Anti-inflammatory effects of glycyrol isolated from Glycyrrhiza uralensis in LPS-stimulated RAW264.7 macrophages[J]. Int Immunopharmacol, 2008, 8: 1524-1532. DOI:10.1016/j.intimp.2008.06.008 |
| [7] |
Shin YW, Bae EA, Lee B, et al. In vitro and in vivo antiallergic effects of Glycyrrhiza glabra and its components[J]. Planta Med, 2007, 73: 257-261. DOI:10.1055/s-2007-967126 |
| [8] |
Ye X, Xue J, Xie X, et al. Effects of different disturbances on plant growth and content of main medicinal ingredients of rhizomatous clonal plant Glycyrrhiza uralensis in a natural population[J]. Chin J Plant Ecol (植物生态学报), 2020, 44: 951-961. DOI:10.17521/cjpe.2020.0188 |
| [9] |
Huang M, Wang W, Wei S. Investigation on medicinal plant resources of Glycyrrhiza uralensis in China and chemical assessment of its underground part[J]. China J Chin Mater Med (中国中药杂志), 2010, 35: 947-952. |
| [10] |
Wei S, Wang W, Wang J, et al. Preliminary study in glycyrrhizin content and its influencing factors of wild and cultivated in different region of China[J]. China J Chin Mater Med (中国中药杂志), 2012, 37: 1341-1345. |
| [11] |
Zhang R, Wang X, Gao W. Regulation mechanism of plant hormones on secondary metabolites[J]. China J Chin Mater Med (中国中药杂志), 2020, 45: 4205-4210. |
| [12] |
Qiao J, Luo Z, Li Y, et al. Effect of abscisic acid on accumulation of five active components in root of Glycyrrhiza uralensis[J]. Molecules, 2017, 22: 1982. DOI:10.3390/molecules22111982 |
| [13] |
Zhang Y, Xu Z, Ji A, et al. Regulation of secondary metabolite biosynthesis by bZPl transcription factors in plants[J]. Plant Sci J (植物科学学报), 2017, 35: 128-137. |
| [14] |
Zhang F, Fu X, Lv Z, et al. A basic leucine zipper transcription factor, AabZIP1, connects abscisic acid signaling with artemisinin biosynthesis in Artemisia annua[J]. Mol Plant, 2015, 8: 163-175. DOI:10.1016/j.molp.2014.12.004 |
| [15] |
Collins-Silva J, Nural AT, Skaggs A, et al. Altered levels of the Taraxacum kok-saghyz (Russian dandelion) small rubber particle protein, TkSRPP3, result in qualitative and quantitative changes in rubber metabolism[J]. Phytochemistry, 2012, 79: 46-56. DOI:10.1016/j.phytochem.2012.04.015 |
| [16] |
Fricke J, Hillebrand A, Twyman RM, et al. Abscisic acid-dependent regulation of small rubber particle protein gene expression in Taraxacum brevicorniculatum is mediated by TbbZIP1[J]. Plant Cell Physiol, 2013, 54: 448-464. DOI:10.1093/pcp/pcs182 |
| [17] |
Hurst HC. Transcription factors. 1: bZIP proteins[J]. Protein Profile, 1994, 1: 123-168. |
| [18] |
Jakoby M, Weisshaar B, Dröge-Laser W, et al. bZIP transcription factors in Arabidopsis[J]. Trends Plant Sci, 2002, 7: 106-111. DOI:10.1016/S1360-1385(01)02223-3 |
| [19] |
Johnson C, Boden E, Arias J. Salicylic acid and NPR1 induce the recruitment of trans-activating TGA factors to a defense gene promoter in Arabidopsis[J]. Plant Cell, 2003, 15: 1846-1858. DOI:10.1105/tpc.012211 |
| [20] |
Zhang Y, Zheng S, Liu Z, et al. Both HY5 and HYH are necessary regulators for low temperature-induced anthocyanin accumulation in Arabidopsis seedlings[J]. J Plant Physiol, 2011, 168: 367-374. DOI:10.1016/j.jplph.2010.07.025 |
| [21] |
Wei K, Chen J, Wang Y, et al. Genome-wide analysis of bZIP-encoding genes in maize[J]. DNA Res, 2012, 19: 463-476. DOI:10.1093/dnares/dss026 |
| [22] |
Nijhawan A, Jain M, Tyagi AK, et al. Genomic survey and gene expression analysis of the basic leucine zipper transcription factor family in rice[J]. Plant Physiol, 2008, 146: 333-350. |
| [23] |
Liao Y, Zou HF, Wei W, et al. Soybean GmbZIP44, GmbZIP62 and GmbZIP78 genes function as negative regulator of ABA signaling and confer salt and freezing tolerance in transgenic Arabidopsis[J]. Planta, 2008, 228: 225-240. DOI:10.1007/s00425-008-0731-3 |
| [24] |
Zhao P, Ye M, Wang R, et al. Systematic identification and functional analysis of potato (Solanum tuberosum L.) bZIP transcription factors and overexpression of potato bZIP transcription factor StbZIP-65 enhances salt tolerance[J]. Int J Biol Macromol, 2020, 161: 155-167. DOI:10.1016/j.ijbiomac.2020.06.032 |
| [25] |
Li Z, Chao J, Li X, et al. Systematic analysis of the bZIP family in tobacco and functional characterization of NtbZIP62 involvement in salt stress[J]. Agronomy, 2021, 11: 148. DOI:10.3390/agronomy11010148 |
| [26] |
Liu S, Qin B, Fang Q, et al. Genome-wide identification, phylogeny and expression analysis of the bZIP gene family in alfalfa (Medicago Sativa)[J]. Biotechnol Biotechnol Equip, 2021, 35: 905-916. DOI:10.1080/13102818.2021.1938674 |
| [27] |
Shearer HL, Cheng YT, Wang L, et al. Arabidopsis clade I TGA transcription factors regulate plant defenses in an NPR1-independent fashion[J]. Mol Plant Microbe Interact, 2012, 25: 1459-1468. DOI:10.1094/MPMI-09-11-0256 |
| [28] |
Hossain MA, Cho JI, Han M, et al. The ABRE-binding bZIP transcription factor OsABF2 is a positive regulator of abiotic stress and ABA signaling in rice[J]. J Plant Physiol, 2010, 167: 1512-1520. DOI:10.1016/j.jplph.2010.05.008 |
| [29] |
Mochida K, Sakurai T, Seki H, et al. Draft genome assembly and annotation of Glycyrrhiza uralensis, a medicinal legume[J]. Plant J, 2017, 89: 181-194. DOI:10.1111/tpj.13385 |
| [30] |
Park SY, Fung P, Nishimura N, et al. Abscisic acid inhibits type 2C protein phosphatases via the PYR/PYL family of START proteins[J]. Science, 2009, 324: 1068-1071. DOI:10.1126/science.1173041 |
| [31] |
Ma Y, Szostkiewicz I, Korte A, et al. Regulators of PP2C phosphatase activity function as abscisic acid sensors[J]. Science, 2009, 324: 1064-1068. DOI:10.1126/science.1172408 |
| [32] |
Melcher K, Ng LM, Zhou XE, et al. A gate-latch-lock mechanism for hormone signalling by abscisic acid receptors[J]. Nature, 2009, 462: 602-608. DOI:10.1038/nature08613 |
| [33] |
Fujii H, Chinnusamy V, Rodrigues A, et al. In vitro reconstitution of an abscisic acid signalling pathway[J]. Nature, 2009, 462: 660-664. DOI:10.1038/nature08599 |
| [34] |
Sirichandra C, Gu D, Hu HC, et al. Phosphorylation of the Arabidopsis AtrbohF NADPH oxidase by OST1 protein kinase[J]. FEBS Lett, 2009, 583: 2982-2986. DOI:10.1016/j.febslet.2009.08.033 |
| [35] |
Hua D, Wang C, He J, et al. A plasma membrane receptor kinase, GHR1, mediates abscisic acid- and hydrogen peroxide-regulated stomatal movement in Arabidopsis[J]. Plant Cell, 2012, 24: 2546-2561. DOI:10.1105/tpc.112.100107 |
| [36] |
Hou Q, Ufer G, Bartels D. Lipid signalling in plant responses to abiotic stress[J]. Plant Cell Environ, 2016, 39: 1029-1048. DOI:10.1111/pce.12666 |
| [37] |
Shulaev V, Cortes D, Miller G, et al. Metabolomics for plant stress response[J]. Physiol Plant, 2008, 132: 199-208. DOI:10.1111/j.1399-3054.2007.01025.x |
| [38] |
Xiong L, Schumaker KS, Zhu JK. Cell signaling during cold, drought, and salt stress[J]. Plant Cell, 2002, 14Suppl(Suppl): S165-183. |
| [39] |
Ying L, Chen H, Cai W. BnNAC485 is involved in abiotic stress responses and flowering time in Brassica napus[J]. Plant Physiol Biochem, 2014, 79: 77-87. DOI:10.1016/j.plaphy.2014.03.004 |
| [40] |
Chen L, Ren F, Zhou L, et al. The Brassica napus calcineurin B-Like 1/CBL-interacting protein kinase 6 (CBL1/CIPK6) component is involved in the plant response to abiotic stress and ABA signalling[J]. J Exp Bot, 2012, 63: 6211-6222. DOI:10.1093/jxb/ers273 |
| [41] |
Gao Y, Li T, Liu Y, et al. Isolation and characterization of gene encoding G protein α subunit protein responsive to plant hormones and abiotic stresses in Brassica napus[J]. Mol Biol Rep, 2010, 37: 3957-3965. DOI:10.1007/s11033-010-0054-x |
| [42] |
Qi X, Wang X. Analysis of bZIP transcription factor family in grape and the response to sugar and ABA signals[J]. Mol Plant Breed (分子植物育种), 2021. https://kns.cnki.net/kcms/detail/46.1068.S.20210602.1613.012.html.
|
| [43] |
Bai Y, Zhu W, Hu X, et al. Genome-wide analysis of the bZIP gene family identifies two ABI5-Like bZIP transcription factors, BrABI5a and BrABI5b, as positive modulators of ABA signalling in Chinese cabbage[J]. PLoS One, 2016, 11: e0158966. DOI:10.1371/journal.pone.0158966 |
| [44] |
Noman A, Liu Z, Aqeel M, et al. Basic leucine zipper domain transcription factors: the vanguards in plant immunity[J]. Biotechnol Lett, 2017, 39: 1779-1791. DOI:10.1007/s10529-017-2431-1 |
| [45] |
Uno Y, Furihata T, Abe H, et al. Arabidopsis basic leucine zipper transcription factors involved in an abscisic acid-dependent signal transduction pathway under drought and high-salinity conditions[J]. Proc Natl Acad Sci U S A, 2000, 97: 11632-11637. DOI:10.1073/pnas.190309197 |
| [46] |
Liang C, Meng Z, Meng Z, et al. GhABF2, a bZIP transcription factor, confers drought and salinity tolerance in cotton (Gossypium hirsutum L.)[J]. Sci Rep, 2016, 6: 35040. DOI:10.1038/srep35040 |
| [47] |
Liu C, Mao B, Ou S, et al. OsbZIP71, a bZIP transcription factor, confers salinity and drought tolerance in rice[J]. Plant Mol Biol, 2014, 84: 19-36. DOI:10.1007/s11103-013-0115-3 |
| [48] |
Ying S, Zhang DF, Fu J, et al. Cloning and characterization of a maize bZIP transcription factor, ZmbZIP72, confers drought and salt tolerance in transgenic Arabidopsis[J]. Planta, 2012, 235: 253-266. DOI:10.1007/s00425-011-1496-7 |
| [49] |
Zong W, Tang N, Yang J, et al. Feedback regulation of ABA signaling and biosynthesis by a bZIP transcription factor targets drought-resistance-related genes[J]. Plant Physiol, 2016, 171: 2810-2825. DOI:10.1104/pp.16.00469 |
| [50] |
Okada A, Okada K, Miyamoto K, et al. OsTGAP1, a bZIP transcription factor, coordinately regulates the inductive production of diterpenoid phytoalexins in rice[J]. J Biol Chem, 2009, 284: 26510-26518. DOI:10.1074/jbc.M109.036871 |
| [51] |
Mccallum S, Woodhead M, Hackett CA, et al. Genetic and environmental effects influencing fruit colour and QTL analysis in raspberry[J]. Theor Appl Genet, 2010, 121: 611-627. DOI:10.1007/s00122-010-1334-5 |
| [52] |
Akagi T, Ikegami A, Tsujimoto T, et al. DkMyb4 is a Myb transcription factor involved in proanthocyanidin biosynthesis in persimmon fruit[J]. Plant Physiol, 2009, 151: 2028-2045. DOI:10.1104/pp.109.146985 |
| [53] |
Akagi T, Katayama-Ikegami A, Kobayashi S, et al. Seasonal abscisic acid signal and a basic leucine zipper transcription factor, DkbZIP5, regulate proanthocyanidin biosynthesis in persimmon fruit[J]. Plant Physiol, 2012, 158: 1089-1102. DOI:10.1104/pp.111.191205 |
| [54] |
Sibéril Y, Benhamron S, Memelink J, et al. Catharanthus roseus G-box binding factors 1 and 2 act as repressors of strictosidine synthase gene expression in cell cultures[J]. Plant Mol Biol, 2001, 45: 477-488. DOI:10.1023/A:1010650906695 |
| [55] |
Zhang Y, Xu Z, Ji A, et al. Genomic survey of bZIP transcription factor genes related to tanshinone biosynthesis in Salvia miltiorrhiza[J]. Acta Pharm Sin B, 2018, 8: 295-305. DOI:10.1016/j.apsb.2017.09.002 |
| [56] |
Liu B, Zhang L, Sun Y, et al. Genome-wide characterization of bZIP transcription factors in foxtail millet and their expression profiles in response to drought and salt stresses[J]. Chin Bull Bot (植物学报), 2016, 51: 473-487. |
| [57] |
Kim SY. The role of ABF family bZIP class transcription factors in stress response[J]. Physiol Plant, 2006, 126: 519-527. |
| [58] |
Nieva C, Busk PK, Domínguez-Puigjaner E, et al. Isolation and functional characterisation of two new bZIP maize regulators of the ABA responsive gene rab28[J]. Plant Mol Biol, 2005, 58: 899-914. DOI:10.1007/s11103-005-8407-x |
| [59] |
Dröge-Laser W, Kaiser A, Lindsay WP, et al. Rapid stimulation of a soybean protein-serine kinase that phosphorylates a novel bZIP DNA-binding protein, G/HBF-1, during the induction of early transcription-dependent defenses[J]. EMBO J, 1997, 16: 726-738. DOI:10.1093/emboj/16.4.726 |
| [60] |
Li Y, Zhang N, Li H, et al. Cloning and expression characteristics of a transcription factor gene RpMYB4 in Rheum palmatum L.[J]. Acta Pharm Sin (药学学报), 2021, 56: 1170-1177. |
| [61] |
Zhan Z, Li X. Biosynthesis and transcriptional regulation of tanshinones and salvianolic acids[J]. Acta Pharm Sin (药学学报), 2020, 55: 2110-2121. |
| [62] |
Feng F, Lv R, Li Y, et al. Characterization and expression analysis of the transcription factor BcWRKY70 in Bupleurum chinense DC[J]. Acta Pharm Sin (药学学报), 2021, 56: 3184-3189. |
| [63] |
Xu J, Hu Z, He H, et al. Identification and expression analysis of the DaNAC transcription factor family in Dipsacus asper[J]. Acta Pharm Sin (药学学报), 2021, 56: 3325-3330. |
 2022, Vol. 57
2022, Vol. 57


