2. 浙江大学药学院, 浙江 杭州 310058
2. College of Pharmaceutical Sciences, Zhejiang University, Hangzhou 310058, China
蛋白质稳态的维持已被证明是细胞健康的关键因素[1], 其主要通过蛋白质稳态系统精准协调细胞活动来实现, 该系统由蛋白质合成机制(核糖体)、蛋白质折叠修饰(分子伴侣等) 和两个蛋白质水解系统(溶酶体和泛素-蛋白酶体) 组成[2]。蛋白质经合成后, 通过折叠后修饰, 成熟为具有完整空间结构的有活性蛋白质而发挥作用, 而具有无法修复损伤的错误蛋白质则通过蛋白质水解途径被消除, 以避免其聚集而引发各种疾病。体内约80%的蛋白质通过蛋白酶体进行降解, 因此蛋白酶体水平的稳定, 对于机体健康具有重要意义。
蛋白酶体通过降解错误折叠、氧化损伤和其他不被机体所需的蛋白, 从而调节各细胞的生理功能。研究证实蛋白酶体功能障碍与各种疾病存在密切联系, 包括癌症、传染性疾病、免疫性疾病和神经退行性疾病等, 因此其作为理想药物靶点具有广泛发展前景[3]。
近年来, 非选择性蛋白酶体抑制剂硼替佐米、卡非佐米和伊沙佐米已被批准用于临床, 治疗多发性骨髓瘤和套细胞淋巴瘤[4]。对免疫蛋白酶体的选择性抑制在自身免疫性疾病上展现了良好的应用前景, 选择性抑制剂KZR-616对系统性红斑狼疮、自身免疫性溶血性贫血等的治疗正在进行临床II期试验[5]。此外, 对疟原虫、结合分枝杆菌蛋白酶体的选择性抑制有望为感染性疾病的治疗带来新的选择。相比之下, 蛋白酶体激动剂的研究相对落后[6], 但也已被证明与包括神经退行性疾病在内的多种疾病存在相关性。随着年龄的增长, 蛋白酶体功能和含量普遍下降, 而蛋白酶体功能降低使得各种受损、错误折叠或聚集的蛋白质积累, 产生蛋白毒性, 从而引发各种神经退行性疾病[7]。蛋白酶体激动剂旨在通过增强蛋白酶体活性, 促进毒性蛋白降解, 起到治疗疾病的目的。本综述将讨论已报道小分子蛋白酶体激动剂的结构特点、构效关系及药理应用, 并展望该领域所面临的机遇和挑战。
1 蛋白酶体的结构和功能 1.1 蛋白酶体的结构蛋白酶体是一种大型的酶复合物, 在体内有多种存在形式, 最常见的有20S和26S蛋白酶体。26S蛋白酶体由一个20S核心颗粒(CP) 和两个19S调节颗粒(RP)组合而成[8] (图 1)。除最常见的组成型蛋白酶体外, 人体内还存在两种特殊的蛋白酶体, 即免疫蛋白酶体和胸腺蛋白酶体。组成型蛋白酶体的β1、β2、β5亚基替换成β1i、β2i、β5i后转变为免疫蛋白酶体, 参与细胞凋亡和免疫应答; 胸腺蛋白酶体的β5亚基转变为β5t, 在胸腺中表达, 参与T细胞的发育调控。
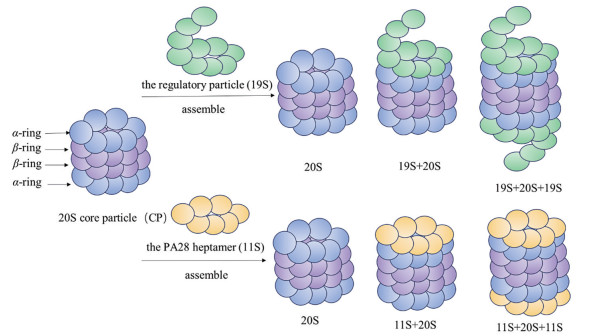
|
Figure 1 Different forms of proteasome and their assemblies |
20S核心颗粒由4个堆叠的环组成, 包括两端的两个α环和中间的两个β环, 每个环都由7个亚基组成, 形成α1-7β1-7β1-7α1-7的桶状结构[9]。两个β环分别含有3个催化位点(β1、β2、β5), β1上活性位点为半胱天冬酶样(Casp-L) 位点, 主要在酸性残基后酶切底物, β2上活性位点为胰蛋白酶样(Tryp-L) 位点, 主要在碱性残基后酶切底物, β5上活性位点为糜蛋白酶样(CT-L) 位点, 主要在疏水性残基后酶切底物(图 1)[10]。4个环围绕同一中心轴排列形成空腔, 外α环作为门控通道, 可通过变构作用决定蛋白质能否进入内部催化核心。
1.1.2 19S调节颗粒19S调节颗粒由与20S中的α环直接作用的基部和外周盖部两部分组成。基部由六聚体AAA家族的ATP酶亚单位Rpt1-6和四聚体非ATP酶亚单位Rpn1、Rpn2、Rpn10和Rpn13组成, 其中ATP酶亚基对蛋白质底物去折叠、α门控打开和底物向20S核心颗粒移动具有至关重要的作用[11]。盖部由9个非ATP酶亚单位Rpn3、Rpn5-9、Rpn11、Rpn12和Sem1组成, 主要作用是加工泛素链, 其中Rpn11作为去泛素化酶发挥作用[12]。19S调节颗粒与20S核心颗粒组装, 使得识别的蛋白质去折叠进入催化部位。组装过程中, 基部ATP酶亚单位(Rpt1-6) 将其C末端疏水肽插入α亚基间口袋, 使门控打开[13]。当然, 除了19S调节颗粒外, 体内还存在11S调节颗粒(AP28), 其作用和结合方式与19S相似, 对降解短肽具有重要作用(图 1)[14]。
细胞内蛋白酶体的形态主要是20S和26S, 且两者的数量处于平衡状态, 这对细胞维持内稳态具有重要作用[15]。这种平衡是由19S调节颗粒和20S核心颗粒可逆对接形成的, 19S帽子结构赋予26S蛋白酶体识别并展开泛素化蛋白的作用, 而20S蛋白酶体缺乏这种帽子结构, 外α环形成动态闭环, 不具备蛋白解折叠功能, 一般不能降解结构蛋白[16]。同时26S蛋白酶体容易受到氧化应激的影响而失活, 而20S蛋白酶体相对稳定, 对氧化应激具有较好抗性, 因此在氧化条件下, 20S蛋白酶体发挥主要作用[17]。而体内20S蛋白酶体水平的提高, 主要由26S蛋白酶体解体来实现, 这也为药物机制研究提供了新思路[18]。
1.2 蛋白酶体参与的蛋白降解途径在真核细胞中, 泛素-蛋白酶体系统(ubiquitin-proteasome system, UPS) 是主要的蛋白质降解途径, 主要降解各种寿命较短和错误折叠蛋白质, 而多数非结构蛋白更多通过非泛素依赖系统(ubiquitin-independent proteasome system, UIPS) 进行降解(图 2)。
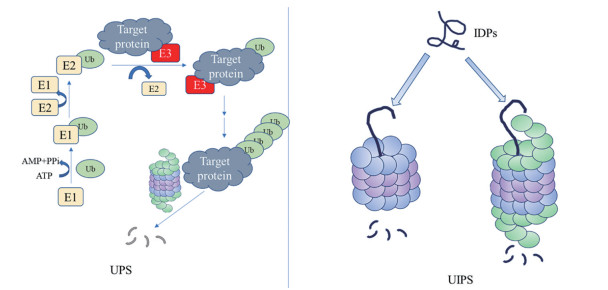
|
Figure 2 Ubiquitin proteasome system (UPS) and ubiquitin-independent proteasome system (UIPS) protein degradation pathways |
26S蛋白酶体参与的泛素依赖系统主要降解结构蛋白, 也有部分内在无序蛋白(intrinsically disordered proteins, IDPs) 会被26S以非泛素依赖方式降解[19]。泛素-蛋白酶体降解蛋白质的基本过程是, 首先, 泛素活化酶(E1) 活化泛素分子, 此过程需要消耗ATP; 活化的泛素转移到泛素结合酶(E2) 上; 然后, 泛素连接酶(E3) 识别被降解靶蛋白, E2与E3相连, 并将泛素分子转移到靶蛋白上, 释放被泛素分子标记的靶蛋白; 重复上述步骤, 直至靶蛋白上连接一条泛素短链[20]。最后, 复合物被26S蛋白酶体识别, 靶蛋白被降解成短肽或氨基酸, 泛素分子被去泛素化酶(DUB) 作用并循环利用。在这个过程中, E3连接酶作为特异性识别分子, 在蛋白降解过程中发挥着至关重要的作用[21]。
在26S蛋白酶体降解靶蛋白过程中, Rpn10和Rpn13作为泛素受体识别多泛素化蛋白, 随后泛素标记被去泛素化酶Rpn11、USP14、UCH37去除[22], 然后19S基部的ATP酶将蛋白解折叠并进入核心颗粒进行催化降解。其中去泛素化酶会对蛋白降解进行反向调节, 已经成为药物设计中的重要靶点[23]。最新研究发现, 20S蛋白酶体同样可以降解泛素标记的蛋白, 但与26S蛋白酶体在底物选择性和降解方式上存在差异[24]。
1.2.2 20S蛋白酶体介导的UIPS20S蛋白酶体参与的非泛素依赖系统主要降解氧化损伤和内在无序蛋白[25], 此过程不需要泛素参与, 也不消耗ATP。独立存在的20S蛋白酶体主要以潜在的闭门构象存在, 外α亚基N末端汇聚而阻断进入催化室的通路, 在其间歇开门构象时可接受肽或IDPs进入。当蛋白底物与20S蛋白酶体接近时, 蛋白底物与α亚基直接相互作用, 诱导构象变化, 进而进入催化部位。因此在设计蛋白酶体激动剂时, 可模拟这一过程, 促使α环打开, 促进蛋白质降解。同时, UIPS也可通过26S蛋白酶体介导进行, 此过程同样不需要泛素和ATP的参与, 即可实现底物的选择性降解, 但要求底物存在非结构化区域[26]。事实上, 两种蛋白酶体的降解机制, 即UPS和UIPS通路, 并不是相互对立的, 而是相互补充, 同一种蛋白即可通过UPS降解, 也可通过UIPS进行降解[27], 两条通路的相互补充, 实现体内蛋白稳态。
1.3 蛋白酶体活性与疾病之间的关系蛋白酶体在体内承担的主要作用包括: 将无活性的蛋白前体加工活化为有活性蛋白; 维持蛋白水平恒定, 尤其是各种信号转导蛋白, 控制生长、分化过程; 降解错误折叠、损坏和不需要蛋白。过去几十年, 研究人员逐步破解了蛋白酶体在细胞内各项生命活动中承担的作用, 包括细胞周期调节、转录调节、凋亡、免疫反应和神经可塑性等。而当蛋白质平衡系统受到干扰时, 会诱发产生各种疾病。例如许多癌细胞依赖于强大的蛋白酶体活性来降解肿瘤抑制因子和细胞周期检查点抑制因子, 从而使细胞异常增殖。因此, 蛋白酶体抑制剂[28]已在临床用于治疗各种肿瘤, 尤其是多发性骨髓瘤。此外, 蛋白酶体抑制剂还在自身免疫性疾病、动脉粥样硬化、囊状纤维化等疾病的治疗中表现出巨大优势, 但蛋白酶体激动剂的研究较少。
IDPs是一类缺乏稳定三级结构的蛋白质[29], 如α-突触核蛋白(α-syn)、β-淀粉样蛋白(Aβ)、Tau蛋白和朊病毒蛋白等, 其内部的无序结构能和多种信号转导途径发生相互作用, 起到调节信号转导的作用[30]。同时由于IDPs缺乏稳定的三级结构, 其很难成为传统药物设计的靶标。IDPs的聚集是各种神经退行性疾病的主要特征[31], 包括帕金森病[32] (PD)、阿尔茨海默症[33] (AD)、亨廷顿病(HD) 和肌萎缩性侧索硬化(ALS) 等, 例如在PD和AD患者中, α-syn和Aβ水平明显提高[34]。体内IDPs主要通过UIPS系统降解, 也有部分通过UPS系统降解, 但随着机体的衰老, 来自外源、线粒体呼吸链和其他细胞活动产生的活性氧使蛋白质发生损伤, 伴随着蛋白酶体活性降低, 损伤蛋白的产生大于消耗, IDPs堆积产生蛋白毒性[35, 36]。因此, 提高蛋白酶体活性, 增强蛋白酶体介导的蛋白水解有望成为治疗神经退行性疾病的潜在策略。近些年来, 研究人员将靶向蛋白降解技术(proteolysis targeting chimeras, PROTAC) 应用于神经退行性疾病中, 设计合成的双功能分子能很好实现靶向蛋白降解, 在动物水平降低这些蛋白的神经毒性[37], 也侧面佐证了这一方案的可行性。除此之外, IDPs还存在于其他疾病中, 包括癌症[38]、糖尿病[39]等, 但目前在这些疾病中的研究还较少。
2 蛋白酶体激动剂的研究进展蛋白酶体激动剂被认为是神经退行性疾病的潜在治疗手段, 目前已发现一些具有蛋白酶体激动活性的小分子化合物[40]。蛋白酶体抑制剂通过与β亚基活性位点相互结合来抑制蛋白降解能力[41], 而蛋白酶体激动剂的作用机制目前尚未完全阐明, 主流观点认为该类分子通过变构作用于α环的亚基之间, 促使20S蛋白酶体的α环打开, 从而使底物进入催化部位(图 3)。此类激动剂选择性作用于20S蛋白酶体[42], 通过UIPS通路促进无序化蛋白降解。还有一些化合物通过间接的方式起到激活蛋白酶体促进蛋白降解的作用, 如去泛素化酶抑制剂、激酶上游调控、转录因子激活剂等。
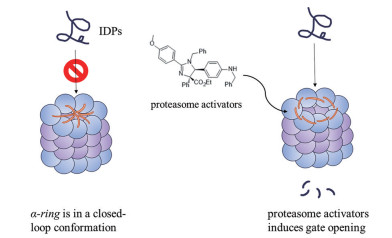
|
Figure 3 The main mechanism of proteasome activators |
十二烷基硫酸钠(SDS) 是最早报道的蛋白酶体激动剂[43], 具有低浓度促进, 高浓度抑制的特性, 与其相似的还有聚阳离子、聚阴离子脂质[44]、脂肪酸和天然产物橄榄苦苷[45]等。但这些结构作为去污剂发挥作用, 只能狭窄地打开α门, 在动物实验中未见蛋白酶体激动剂活性, 为了寻找新的激活剂, 国内外学者进行了多方面的探索, 找到了几类具有活性的分子结构, 包括吩噻嗪类、咪唑啉类、二氢喹唑啉类和喹唑啉酮类等。
2.1 直接激动剂 2.1.1 吩噻嗪类Jones等[46]通过筛选NCC和Prestwick文库, 确定吩噻嗪类药物氯丙嗪(CPZ) 可以激动20S蛋白酶体。对其进行结构修饰(图 4) 发现的化合物CJ-7-42对多巴胺D2受体(D2R) 的抑制活性明显下降, 提高了对蛋白酶体的选择性。化合物CJ-7-42对蛋白酶体激动的EC50达到了15.6 μmol·L-1 (表 1), 研究表明, 其可能作用于α环中的α1/α2亚基之间, 促使门控开启, 进而促进IDPs的降解。对接研究显示, 该分子能与蛋白酶体α1/α2亚基多个氨基酸残基形成氢键结合作用, 如Arg83。Western blot研究显示, 与对照相比, 化合物1以浓度依赖的方式显著提高了20S蛋白酶体介导的α-syn降解, 而CPZ、1和CJ-7-42则能促进20S蛋白酶体介导的tau蛋白降解。并且, 在AD模型小鼠中, 氯丙嗪结构类似物亚甲基蓝已被证实可以降低Aβ水平[47], 这进一步证明了吩噻嗪类在治疗神经退行性疾病上具有重大潜力。
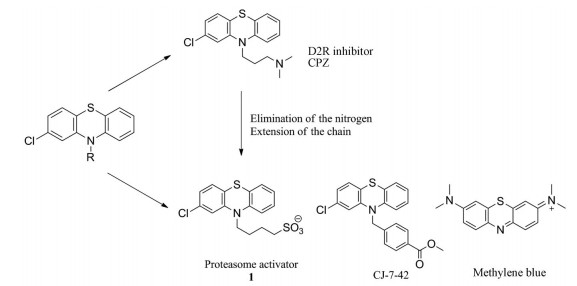
|
Figure 4 Phenothiazine proteasome activators and the derivatives |
| Table 1 20S proteasome agonistic activities and inhibitory activities against D2R of phenothiazine derivatives |
后续Fiolek课题组通过高通量筛选, 确定二苯丁哌啶类小分子抗精神病药物氟司比林可作为开发20S蛋白酶体激动剂的前体结构[48]。与吩噻嗪类化合物改造思路类似, 通过降低化合物碱性来抑制其与多巴胺受体结合(图 5), 衍生物N-酰化氟司比林具有比氟司比林更强的蛋白酶体激动活性, 较高浓度条件下, 在T-L和Casp-L位点均能达到活性加倍(表 2)。与吩噻嗪类不同的是, 氟司比林优先结合于α2/3亚基口袋之间, 维持门控开启。进一步探索发现, 酰胺键的N-H与各氨基酸残基之间形成的氢键以及二苯基尾部与残基之间的π-π相互作用是其能结合于α2/3亚基之间的关键。在体外实验中, 这两个结构已被证明能以浓度依赖性的方式预防IDP低聚物对20S蛋白酶体的损伤, 促进α-syn的降解。

|
Figure 5 Structures of fluspirilene and its analogs |
| Table 2 The AC200 and max fold increases in activity of fluspirilene and its analog. AC200: Drug concentration at doubling of activity |
吡唑啉酮类化合物CMB-087229 (图 6) 被证实具有蛋白酶体激动活性, 其EC50值达到了0.07 μmol·L-1, 但具体机制尚不明确[49]。为了进一步获得具有药用价值的蛋白酶体激动小分子并阐述药理作用, 后续通过筛选一个吡唑啉酮文库[50], 发现了氨基比林和尼芬那宗也具有激活20S蛋白酶体活性的作用。尼芬那宗在0.5 μmol·L-1时达到最大活性, 药物浓度大于1 μmol·L-1后, 氨基比林活性最佳。分子对接和NMR实验证实了吡唑啉酮类化合物通过氢键和π-π相互作用与蛋白酶体α环结合, 从而起到门控作用, 其主要作用位点为α1/α2亚基和α5/α6亚基之间。在随后构效关系研究中发现, 此类激动剂具有两部分重要结构特征, 首先R4位苯环参与了与α1亚基中许多非极性基团和芳香基团的疏水相互作用, 也可与α5、α6作用; 其次R1基团与亚基之间形成疏水相互作用及氢键。通过ESI-MS实验证实, 氨基比林可以促进蛋白酶体对Aβ的降解, 有望通过结构改造, 用于治疗AD。
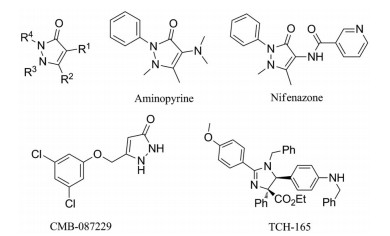
|
Figure 6 Structures of pyrazolone and imidazoline proteasome activators |
Njomen等[51]通过原子力显微镜(AFM) 发现一种咪唑啉类化合物TCH-165 (图 6) 同样可以结合于α1/α2亚基之间, 诱导蛋白酶体产生构象变化, 促使α环打开, 促进无序蛋白降解。并且, 其可以同时增强CT-L、Casp-L、Tryp-L三个位点的活性(表 3)。同时, TCH-165可以阻止19S帽子结构与20S蛋白酶体结合, 从而使20S-26S平衡向20S移动。由于单端蛋白酶体(19S-20S) 能够有效降解泛素化蛋白, 因此TCH-165所引起的完全组装蛋白酶体(19S-20S-19S) 的减少并不会引起泛素化蛋白的显著堆积。这一发现也为本研究领域提供了新思路, 即通过小分子调节不同蛋白酶体复合物之间的动态平衡来调控蛋白质降解。在后续研究中发现, 其可在细胞层面促进IDPs的降解, 并且具有剂量依赖性。如在某些癌症中过表达的蛋白鸟氨酸脱羧酶(ODC) 和c-Fos蛋白, 并且这种增强蛋白降解的效果能被蛋白酶体抑制剂阻断。
| Table 3 EC50 values of TCH-165 and maximum fold enhancement against 20S proteasome |
为了探索其他可能的分子骨架, Fiolek实验室进一步筛选了一系列二氢喹唑啉结构, 发现部分结构具有明显的20S蛋白酶体激动作用[52] (图 7)。在随后构效关系中发现, C-7位(R1) 上的取代基团对二氢喹唑啉类化合物活性有较大影响, 烷氧基或烷基取代时, 化合物能维持较好活性, 当为长链烷基时活性降低, 如R1为C5H11时, 活性最大增加倍数仅为2.8。C-2位(R2) 为苯环时, 活性较好; 若用其他烷基替代, 活性降低, 如R2为甲基时, 活性最大增加倍数为1.8倍。C-3位(R3) 为较大疏水基团如苯环时, 活性较好; R3中引入杂原子, 会阻断疏水相互作用, 而使活性减弱, 如R3为N-甲基哌啶时, 无蛋白酶体激动剂活性(表 4)。通过结构优化, 化合物3、4、5显示了较好的蛋白酶体激动活性。此外, 化合物5在体外还能促进20S蛋白酶体降解α-syn, 且呈剂量依赖性, 显示了在神经退行性疾病上的应用前景。

|
Figure 7 Structures of dihydroquinazolines |
| Table 4 AC200 values of dihydroquinazolines and maximum fold enhancement against 20S proteasome |
此外, 一些天然产物也被证实具有蛋白酶体激动活性, 如熊果酸、白桦脂酸和齐墩果酸[53]等(图 8)。早期较为系统报道的20S蛋白酶体激动剂就是一类三萜类化合物白桦脂酸(EC50 = 5.5 μmol·L-1), 它能特异性增强20S蛋白酶体的CT-L活性, 但其部分修饰产物对蛋白酶体有抑制作用, 具有复杂的构效关系[54]。Coleman等[55]经筛选发现了3个具有蛋白酶体激动剂活性的化合物, 熊果酸(EC50 = 14 μmol·L-1)、维生素E琥珀酸酯(EC50 = 7 μmol·L-1) 和一类具有三嗪支架结构的金雀花碱衍生物(EC50 = 10 μmol·L-1)。机制研究发现, 金雀花碱衍生物与门控调节剂熊果酸和维生素E琥珀酸酯不同, 其可以与20S CP的一个活性位点发生特定的相互作用, 而激活蛋白酶体。由于这种新机制的发现, 该课题组认为三嗪支架结构具有进一步的研究价值。
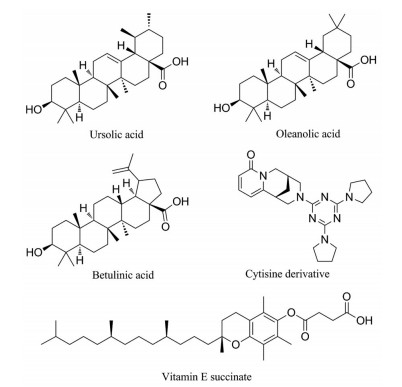
|
Figure 8 Structures of natural products with proteasome activation activities |
Trader实验室[56]认为SDS等去污剂不能作为真正的蛋白酶体激动剂, 为了获得更有效的激动分子, 该课题组筛选了NCC文库(the NIH clinical collection), 确定了12个潜在结构。通过LCMS定量分析确定每个化合物对蛋白酶体降解底物的能力, 并利用绿色荧光蛋白测定化合物激动HEK细胞蛋白酶体活性的能力, 最终发现MK-866和AM-404具有较好蛋白酶体激动活性(图 9), EC50均达到32 μmol·L-1。后续研究发现, AM404会抑制脂肪酸酰胺水解酶活性, 产生细胞毒性, 因此Coleman实验室对花生四烯酸衍生物AM-404进行一系列结构改造, 包括改变链长、取代基和不饱和度, 最后证明了C8顺式烯烃对增强20S蛋白酶体活性是必需的, 并发现了两个结构(化合物6、7) 可以在保持活性的基础下, 高浓度下降低毒性[57]。后续还需要利用各种生物生理技术来判断AM404与20SCP相互作用的核心部分, 以便设计更合理结构。

|
Figure 9 Structures of MK-866, AM404, 6 and 7 |
除了直接作用于蛋白酶体, 针对去泛素化酶设计蛋白酶体激动剂也成为一种新的策略。例如, Lee等[58]研究发现, USP14可将泛素化链从基部末端释放, 从而达到缩短链长而不是完全移除的作用, 这种链修剪会抑制泛素-蛋白酶体复合物的降解, 因此USP14的小分子抑制剂可以增强蛋白酶体活性。通过高通量筛选发现, 化合物IU1 (图 10) 可作为去泛素化酶抑制剂发挥蛋白酶体激动作用, 其可抑制USP14所导致的链修剪, 从而促进蛋白降解[59]。而UCH37与USP14具有相似的作用, 其抑制剂是否也有激活蛋白酶体的作用还需要进一步的研究。有趣的是, UCH37和USP14的小分子抑制剂b-AP15可以导致多泛素化蛋白的积累, 并增加多发性骨髓瘤细胞的凋亡, 在肿瘤治疗方面也具有重要意义[60, 61]。

|
Figure 10 Structures of representative indirect proteasome activators |
蛋白酶体的激活也可以通过上游激酶的调节来实现。26S蛋白酶体中存在很多磷酸化位点, 在调节26S蛋白酶体组装、稳定性和活性方面各自发挥着重要作用。前期研究已经证明, p38 MAPK可以通过Rpn2亚基上的Thr-273磷酸化抑制蛋白酶体活性[62], 其他具有相似或相反作用的还有, ASK1磷酸化Rpt5[63], PKA磷酸化Rpn6亚基上的Ser-14[64], CaMKⅡ磷酸化Rpt6[65], DYRK2磷酸化Rpt3亚基上的Thr-25[66]。因此, Leestemaker等[67]鉴定了几种能够间接激活蛋白酶体活性的p38 MAPK抑制剂, 如PD169316 (图 10), 它们能提高过度表达α-syn的细胞存活率。相似的, cAMP依赖性蛋白激酶A (PKA) 磷酸化Rpn6亚基而上调26S蛋白酶体组装及活性[68]。据报道, 一种磷酸二酯酶4型(PDE4) 抑制剂咯利普兰(Rolipram) 可以提高cAMP水平, 激活PKA[69], 进而提高蛋白酶体活性并抑制tau蛋白聚集, 改善认知能力[70]。此外, 内源性物质胰高血糖素、肾上腺素和氟司比林也可通过刺激Rpn6磷酸化, 使蛋白酶体活性增强[71]。由此可见, 体内cAMP-PKA介导的蛋白酶体活性增加是一种常见的细胞对各种内分泌刺激的反应, 并能迅速提高目标组织降解调节蛋白和错误折叠蛋白的能力。
转录因子Nrf2的激活是另一种蛋白酶体活性上调的方式。Nrf2是一类含有高度保守碱性亮氨酸拉链结构的转录因子家族成员, 其激活后可启动下游众多抗氧化酶的合成, 也可以导致蛋白酶体的过表达[72]。研究发现, 多个Nrf2激活剂可实现蛋白酶体水平的上调, 例如抗氧剂D3T (图 10) 可以在Nrf2阳性成纤维细胞中上调19S和20S蛋白酶体水平, 激活蛋白酶体活性[73]。AD模型小鼠体内研究发现, D3T可以显著降低Aβ水平, 改善AD小鼠认知能力[74]。
3 展望虽然近些年来, 蛋白酶体激动剂的研究正在稳步推进, 也发现了很多具有开发价值的潜力化合物, 但是该领域的发展仍然面临着巨大的挑战。①研究方法的局限性, 目前获得蛋白酶体激动剂的主要方法是化合物库的筛选, 缺少理性设计的生物学研究基础; ②标准化评价手段的缺乏, 目前蛋白酶体活性的测定方法主要是使用针对每个催化位点的特异性底物, 观测底物的降解程度[75], 但是对于研究蛋白酶体门控调节剂而言, 无法定量小分子激动剂的变构效应是否足以实现20S蛋白酶体真正的闸门开启[76], 如SDS等去污剂可以狭窄地打开20S蛋白酶体的α环, 表现出激动剂活性, 但在动物实验中并未见活性; ③潜在的毒性, 蛋白酶体抑制剂已被成功开发为抗肿瘤药物[77], 因此蛋白酶体激动剂是否会致癌, 将成为一个广泛关注的问题; ④实现靶向的困难性, 神经退行性疾病的毒性蛋白主要集中于脑内, 如何通过血脑屏障会成为一个难关; ⑤机制的复杂性, 目前的研究仅仅阐述了部分可能的机制, 详细准确的机制还有待进一步研究, 尤其是还需要大量的工作来破译门控机制。
蛋白酶体作为调控体内蛋白稳态的大分子复合物, 其功能异常会导致各种疾病的产生, 蛋白酶体抑制剂已被用于癌症、免疫性疾病等的治疗。而蛋白酶体活性的降低, 则是多种神经退行性疾病的特征, 蛋白酶体激动剂有希望成为多种神经退行性疾病的治疗选择。本文讨论了小分子蛋白酶体激动剂的研究进展, 包括各类结构的蛋白酶体激动活性、其构效关系、作用机制和药理作用, 为未来研究提供借鉴依据。通过进一步结构筛选、优化和评价, 更精准阐述作用机制和药理作用, 蛋白酶体激动剂将在多种疾病的治疗中开启新的篇章。
作者贡献: 余倩论文撰写与修改; 曹宇图片制作; 祝华建文献检索及整理; 邵加安论文修改; 张建康确定选题, 修订论文并定稿。
利益冲突: 本文内容没有利益冲突。
| [1] |
Hetz C, Glimcher LH. Protein homeostasis networks in physiology and disease[J]. Curr Opin Cell Biol, 2011, 23: 123-125. DOI:10.1016/j.ceb.2011.01.004 |
| [2] |
Kaushik S, Cuervo AM. Proteostasis and aging[J]. Nat Med, 2015, 21: 1406-1415. DOI:10.1038/nm.4001 |
| [3] |
Shen M, Chan TH, Dou QP. Targeting tumor ubiquitin-proteasome pathway with polyphenols for chemosensitization[J]. Anti-cancer Agent Med Chem, 2012, 12: 891-901. DOI:10.2174/187152012802649978 |
| [4] |
Romaniuk W, Oldziej AE, Zinczuk J, et al. Proteasome inhibitors in cancer therapy[J]. Postepy Hig Med Dosw (Online), 2015, 69: 1443-1450. |
| [5] |
Kisselev AF, van der Linden WA, Overkleeft HS. Proteasome inhibitors: an expanding army attacking a unique target[J]. Chem Biol, 2012, 19: 99-115. DOI:10.1016/j.chembiol.2012.01.003 |
| [6] |
Jones CL, Tepe JJ. Proteasome activation to combat proteotoxicity[J]. Molecules, 2019, 24: 2841. DOI:10.3390/molecules24152841 |
| [7] |
Chondrogianni N, Voutetakis K, Kapetanou M, et al. Proteasome activation: an innovative promising approach for delaying aging and retarding age-related diseases[J]. Ageing Res Rev, 2015, 23: 37-55. DOI:10.1016/j.arr.2014.12.003 |
| [8] |
Xie Y. Structure, assembly and homeostatic regulation of the 26S proteasome[J]. J Mol Cell Biol, 2010, 2: 308-317. DOI:10.1093/jmcb/mjq030 |
| [9] |
Bard JAM, Goodall EA, Greene ER, et al. Structure and function of the 26S proteasome[J]. Annu Rev Biochem, 2018, 87: 697-724. DOI:10.1146/annurev-biochem-062917-011931 |
| [10] |
Groll M, Heinemeyer W, Jager S, et al. The catalytic sites of 20S proteasomes and their role in subunit maturation: a mutational and crystallographic study[J]. Proc Natl Acad Sci U S A, 1999, 96: 10976-10983. DOI:10.1073/pnas.96.20.10976 |
| [11] |
He J, Kulkarni K, Da Fonseca PC, et al. The structure of the 26S proteasome subunit Rpn2 reveals its PC repeat domain as a closed toroid of two concentric alpha-helical rings[J]. Structure, 2012, 20: 513-521. DOI:10.1016/j.str.2011.12.015 |
| [12] |
Schweitzer A, Aufderheide A, Rudack T, et al. Structure of the human 26S proteasome at a resolution of 3.9 A[J]. Proc Natl Acad Sci U S A, 2016, 113: 7816-7821. DOI:10.1073/pnas.1608050113 |
| [13] |
Smith DM, Chang SC, Park S, et al. Docking of the proteasomal ATPases' carboxyl termini in the 20S proteasome's alpha ring opens the gate for substrate entry[J]. Mol Cell, 2007, 27: 731-744. DOI:10.1016/j.molcel.2007.06.033 |
| [14] |
Hill CP, Masters EI, Whitby FG. The 11S regulators of 20S proteasome activity[J]. Curr Top Microbiol, 2002, 268: 73-89. |
| [15] |
Kumar Deshmukh F, Yaffe D, Olshina MA, et al. The contribution of the 20S proteasome to proteostasis[J]. Biomolecules, 2019, 9: 190. DOI:10.3390/biom9050190 |
| [16] |
Finley D, Chen X, Walters KJ. Gates, channels, and switches: elements of the proteasome machine[J]. Trends Biochem Sci, 2016, 41: 77-93. DOI:10.1016/j.tibs.2015.10.009 |
| [17] |
Grune T, Catalgol B, Licht A, et al. HSP70 mediates dissociation and reassociation of the 26S proteasome during adaptation to oxidative stress[J]. Free Radical Bio Med, 2011, 51: 1355-1364. DOI:10.1016/j.freeradbiomed.2011.06.015 |
| [18] |
Wang X, Yen J, Kaiser P, et al. Regulation of the 26S proteasome complex during oxidative stress[J]. Sci Signal, 2010, 3: rs8. |
| [19] |
Orlowski M, Wilk S. Ubiquitin-independent proteolytic functions of the proteasome[J]. Arch Biochem Biophys, 2003, 415: 1-5. DOI:10.1016/S0003-9861(03)00197-8 |
| [20] |
Pickart CM. Mechanisms underlying ubiquitination[J]. Annu Rev Biochem, 2001, 70: 503-533. DOI:10.1146/annurev.biochem.70.1.503 |
| [21] |
Schapira M, Calabrese MF, Bullock AN, et al. Targeted protein degradation: expanding the toolbox[J]. Nat Rev Drug Discov, 2019, 18: 949-963. DOI:10.1038/s41573-019-0047-y |
| [22] |
Lee MJ, Lee BH, Hanna J, et al. Trimming of ubiquitin chains by proteasome-associated deubiquitinating enzymes[J]. Mol Cell Proteomics, 2011, 10: R110 003871. DOI:10.1074/mcp.R110.003871 |
| [23] |
Hu M, Li P, Song L, et al. Structure and mechanisms of the proteasome-associated deubiquitinating enzyme USP14[J]. EMBO J, 2005, 24: 3747-3756. DOI:10.1038/sj.emboj.7600832 |
| [24] |
Sahu I, Mali SM, Sulkshane P, et al. The 20S as a stand-alone proteasome in cells can degrade the ubiquitin tag[J]. Nat Commun, 2021, 12: 6173. DOI:10.1038/s41467-021-26427-0 |
| [25] |
Ben-Nissan G, Sharon M. Regulating the 20S proteasome ubiquitin-independent degradation pathway[J]. Biomolecules, 2014, 4: 862-884. DOI:10.3390/biom4030862 |
| [26] |
Erales J, Coffino P. Ubiquitin-independent proteasomal degradation[J]. Biochim Biophys Acta, 2014, 1843: 216-221. DOI:10.1016/j.bbamcr.2013.05.008 |
| [27] |
Asher G, Tsvetkov P, Kahana C, et al. A mechanism of ubiquitin-independent proteasomal degradation of the tumor suppressors p53 and p73[J]. Genes Dev, 2005, 19: 316-321. DOI:10.1101/gad.319905 |
| [28] |
Thibaudeau TA, Smith DM. A practical review of proteasome pharmacology[J]. Pharmacol Rev, 2019, 71: 170-197. DOI:10.1124/pr.117.015370 |
| [29] |
Dunker AK, Lawson JD, Brown CJ, et al. Intrinsically disordered protein[J]. J Mol Graph Model, 2001, 19: 26-59. |
| [30] |
Deforte S, Uversky VN. Order, disorder, and everything in between[J]. Molecules, 2016, 21: 1090. DOI:10.3390/molecules21081090 |
| [31] |
Ross CA, Poirier MA. Protein aggregation and neurodegenerative disease[J]. Nat Med, 2004, 10 Suppl: S10-S17. |
| [32] |
Mcnaught KS, Olanow CW, Halliwell B, et al. Failure of the ubiquitin-proteasome system in Parkinson's disease[J]. Nat Rev Neurosci, 2001, 2: 589-594. DOI:10.1038/35086067 |
| [33] |
Lam YA, Pickart CM, Alban A, et al. Inhibition of the ubiquitin-proteasome system in Alzheimer's disease[J]. Proc Natl Acad Sci U S A, 2000, 97: 9902-9906. DOI:10.1073/pnas.170173897 |
| [34] |
Uversky VN, Oldfield CJ, Dunker AK. Intrinsically disordered proteins in human diseases: introducing the D2 concept[J]. Annu Rev Biophys, 2008, 37: 215-246. DOI:10.1146/annurev.biophys.37.032807.125924 |
| [35] |
Ding Q, Dimayuga E, Keller JN. Proteasome regulation of oxidative stress in aging and age-related diseases of the CNS[J]. Antioxid Redox Sign, 2006, 8: 163-172. DOI:10.1089/ars.2006.8.163 |
| [36] |
Klaips CL, Jayaraj GG, Hartl FU. Pathways of cellular proteostasis in aging and disease[J]. J Cell Biol, 2018, 217: 51-63. DOI:10.1083/jcb.201709072 |
| [37] |
Hyun S, Shin D. Chemical-mediated targeted protein degradation in neurodegenerative diseases[J]. Life-Basel, 2021, 11: 607. DOI:10.3390/life11070607 |
| [38] |
Kumar D, Sharma N, Giri R. Therapeutic interventions of cancers using intrinsically disordered proteins as drug targets: c-Myc as model system[J]. Cancer Inform, 2017, 16: 1176935117699408. |
| [39] |
Du Z, Uversky VN. A comprehensive survey of the roles of highly disordered proteins in type 2 diabetes[J]. Int J Mol Sci, 2017, 18: 2010. DOI:10.3390/ijms18102010 |
| [40] |
Njomen E, Tepe JJ. Proteasome activation as a new therapeutic approach to target proteotoxic disorders[J]. J Med Chem, 2019, 62: 6469-6481. DOI:10.1021/acs.jmedchem.9b00101 |
| [41] |
Li Y, Liang JY, Liu XL, et al. Research progress on 26S proteasome inhibitors[J]. Acta Pharm Sin (药学学报), 2017, 52: 524-530. |
| [42] |
Latham MP, Sekhar A, Kay LE. Understanding the mechanism of proteasome 20S core particle gating[J]. Proc Natl Acad Sci U S A, 2014, 111: 5532-5537. DOI:10.1073/pnas.1322079111 |
| [43] |
Shibatani T, Ward WF. Sodium dodecyl sulfate (SDS) activation of the 20S proteasome in rat liver[J]. Arch Biochem Biophys, 1995, 321: 160-166. DOI:10.1006/abbi.1995.1381 |
| [44] |
Ruiz De Mena I, Mahillo E, Arribas J, et al. Kinetic mechanism of activation by cardiolipin (diphosphatidylglycerol) of the rat liver multicatalytic proteinase[J]. Biochem J, 1993, 296: 93-97. DOI:10.1042/bj2960093 |
| [45] |
Katsiki M, Chondrogianni N, Chinou I, et al. The olive constituent oleuropein exhibits proteasome stimulatory properties in vitro and confers life span extension of human embryonic fibroblasts[J]. Rejuv Res, 2007, 10: 157-172. DOI:10.1089/rej.2006.0513 |
| [46] |
Jones CL, Njomen E, Sjogren B, et al. Small molecule enhancement of 20S proteasome activity targets intrinsically disordered proteins[J]. ACS Chem Biol, 2017, 12: 2240-2247. DOI:10.1021/acschembio.7b00489 |
| [47] |
Medina DX, Caccamo A, Oddo S. Methylene blue reduces Aβ levels and rescues early cognitive deficit by increasing proteasome activity[J]. Brain Pathol, 2011, 21: 140-149. DOI:10.1111/j.1750-3639.2010.00430.x |
| [48] |
Fiolek TJ, Keel KL, Tepe JJ. Fluspirilene analogs activate the 20S proteasome and overcome proteasome impairment by intrinsically disordered protein oligomers[J]. ACS Chem Neurosci, 2021, 12: 1438-1448. DOI:10.1021/acschemneuro.1c00099 |
| [49] |
Trippier PC, Zhao KT, Fox SG, et al. Proteasome activation is a mechanism for pyrazolone small molecules displaying therapeutic potential in amyotrophic lateral sclerosis[J]. ACS Chem Neurosci, 2014, 5: 823-829. DOI:10.1021/cn500147v |
| [50] |
Santoro AM, Lanza V, Bellia F, et al. Pyrazolones activate the proteasome by gating mechanisms and protect neuronal cells from beta-amyloid toxicity[J]. ChemMedChem, 2020, 15: 302-316. DOI:10.1002/cmdc.201900612 |
| [51] |
Njomen E, Osmulski PA, Jones CL, et al. Small molecule modulation of proteasome assembly[J]. Biochemistry, 2018, 57: 4214-4224. DOI:10.1021/acs.biochem.8b00579 |
| [52] |
Fiolek TJ, Magyar CL, Wall TJ, et al. Dihydroquinazolines enhance 20S proteasome activity and induce degradation of alpha-synuclein, an intrinsically disordered protein associated with neurodegeneration[J]. Bioorg Med Chem Lett, 2021, 36: 127821. DOI:10.1016/j.bmcl.2021.127821 |
| [53] |
Maresh ME, Chen P, Hazbun TR, et al. A yeast chronological lifespan assay to assess activity of proteasome stimulators[J]. Chembiochem, 2021, 22: 2553-2560. DOI:10.1002/cbic.202100117 |
| [54] |
Huang L, Ho P, Chen CH. Activation and inhibition of the proteasome by betulinic acid and its derivatives[J]. FEBS Lett, 2007, 581: 4955-4959. DOI:10.1016/j.febslet.2007.09.031 |
| [55] |
Coleman RA, Trader DJ. Development and application of a sensitive peptide reporter to discover 20S proteasome stimulators[J]. ACS Comb Sci, 2018, 20: 269-276. DOI:10.1021/acscombsci.7b00193 |
| [56] |
Trader DJ, Simanski S, Dickson P, et al. Establishment of a suite of assays that support the discovery of proteasome stimulators[J]. BBA-Gen Subjects, 2017, 1861: 892-899. DOI:10.1016/j.bbagen.2017.01.003 |
| [57] |
Coleman RA, Muli CS, Zhao Y, et al. Analysis of chain length, substitution patterns, and unsaturation of AM-404 derivatives as 20S proteasome stimulators[J]. Bioorg Med Chem Lett, 2019, 29: 420-423. DOI:10.1016/j.bmcl.2018.12.030 |
| [58] |
Lam YA, Xu W, Demartino GN, et al. Editing of ubiquitin conjugates by an isopeptidase in the 26S proteasome[J]. Nature, 1997, 385: 737-740. DOI:10.1038/385737a0 |
| [59] |
Lee BH, Lee MJ, Park S, et al. Enhancement of proteasome activity by a small-molecule inhibitor of USP14[J]. Nature, 2010, 467: 179-184. DOI:10.1038/nature09299 |
| [60] |
Tian Z, Darcy P, Wang X, et al. A novel small molecule inhibitor of deubiquitylating enzyme USP14 and UCHL5 induces apoptosis in multiple myeloma and overcomes bortezomib resistance[J]. Blood, 2014, 123: 706-716. DOI:10.1182/blood-2013-05-500033 |
| [61] |
Wang K, Zhang Y, Wang J, et al. Self-assembled IR780-loaded transferrin nanoparticles as an imaging, targeting and PDT/PTT agent for cancer therapy[J]. Sci Rep, 2016, 6: 27421. DOI:10.1038/srep27421 |
| [62] |
Lee SH, Park Y, Yoon SK, et al. Osmotic stress inhibits proteasome by p38 MAPK-dependent phosphorylation[J]. J Biol Chem, 2010, 285: 41280-41289. DOI:10.1074/jbc.M110.182188 |
| [63] |
Um JW, Im E, Park J, et al. ASK1 negatively regulates the 26S proteasome[J]. J Biol Chem, 2010, 285: 36434-36446. DOI:10.1074/jbc.M110.133777 |
| [64] |
Lokireddy S, Kukushkin NV, Goldberg AL. cAMP-induced phosphorylation of 26S proteasomes on Rpn6/PSMD11 enhances their activity and the degradation of misfolded proteins[J]. Proc Natl Acad Sci U S A, 2015, 112: E7176-7185. |
| [65] |
Djakovic SN, Schwarz LA, Barylko B, et al. Regulation of the proteasome by neuronal activity and calcium/calmodulin-dependent protein kinase Ⅱ[J]. J Biol Chem, 2009, 284: 26655-26665. DOI:10.1074/jbc.M109.021956 |
| [66] |
Guo X, Wang X, Wang Z, et al. Site-specific proteasome phosphorylation controls cell proliferation and tumorigenesis[J]. Nat Cell Biol, 2016, 18: 202-212. DOI:10.1038/ncb3289 |
| [67] |
Leestemaker Y, de Jong A, Witting KF, et al. Proteasome activation by small molecules[J]. Cell Chem Biol, 2017, 24: 725-736.e7. DOI:10.1016/j.chembiol.2017.05.010 |
| [68] |
Zhang F, Hu Y, Huang P, et al. Proteasome function is regulated by cyclic AMP-dependent protein kinase through phosphorylation of Rpt6[J]. J Biol Chem, 2007, 282: 22460-22471. DOI:10.1074/jbc.M702439200 |
| [69] |
Park SJ, Ahmad F, Philp A, et al. Resveratrol ameliorates aging-related metabolic phenotypes by inhibiting cAMP phosphodiesterases[J]. Cell, 2012, 148: 421-433. DOI:10.1016/j.cell.2012.01.017 |
| [70] |
Myeku N, Clelland CL, Emrani S, et al. Tau-driven 26S proteasome impairment and cognitive dysfunction can be prevented early in disease by activating cAMP-PKA signaling[J]. Nat Med, 2016, 22: 46-53. DOI:10.1038/nm.4011 |
| [71] |
Verplank JJS, Lokireddy S, Zhao J, et al. 26S proteasomes are rapidly activated by diverse hormones and physiological states that raise cAMP and cause Rpn6 phosphorylation[J]. Proc Natl Acad Sci U S A, 2019, 116: 4228-4237. DOI:10.1073/pnas.1809254116 |
| [72] |
Kwak MK, Itoh K, Yamamoto M, et al. Role of transcription factor Nrf2 in the induction of hepatic phase 2 and antioxidative enzymes in vivo by the cancer chemoprotective agent, 3H-1, 2-dimethiole-3-thione[J]. Mol Med, 2001, 7: 135-145. DOI:10.1007/BF03401947 |
| [73] |
Kwak MK, Wakabayashi N, Greenlaw JL, et al. Antioxidants enhance mammalian proteasome expression through the Keap1-Nrf2 signaling pathway[J]. Mol Cell Biol, 2003, 23: 8786-8794. DOI:10.1128/MCB.23.23.8786-8794.2003 |
| [74] |
Cui Y, Ma S, Zhang C, et al. Pharmacological activation of the Nrf2 pathway by 3H-1, 2-dithiole-3-thione is neuroprotective in a mouse model of Alzheimer disease[J]. Behav Brain Res, 2018, 336: 219-226. DOI:10.1016/j.bbr.2017.09.011 |
| [75] |
Kisselev AF, Goldberg AL. Monitoring activity and inhibition of 26S proteasomes with fluorogenic peptide substrates[J]. Method Enzymol, 2005, 398: 364-378. |
| [76] |
Lazaro DF, Bellucci A, Brundin P, et al. Editorial: protein misfolding and spreading pathology in neurodegenerative diseases[J]. Front Mol Neurosci, 2019, 12: 312. |
| [77] |
Sherman DJ, Li J. Proteasome inhibitors: harnessing proteostasis to combat disease[J]. Molecules, 2020, 25: 671. DOI:10.3390/molecules25030671 |
 2022, Vol. 57
2022, Vol. 57


