2. 山东大学附属山东省妇幼保健院, 山东 济南 250014;
3. 中国中医科学院, 北京 100700
2. Shandong Provincial Maternal and Child Health Care Hospital, Shandong University, Jinan 250014, China;
3. China Academy of Chinese Medical Sciences, Beijing 100700, China
枸杞是我国历史悠久的药食同源品种, 具有广泛的药用和经济价值。2020版《中国药典》收录枸杞来源为茄科植物宁夏枸杞Lycium barbarum L.的干燥成熟果实, 具有滋补肝肾、益精明目的功效[1]。现代药理研究表明枸杞具有抗氧化、抗衰老、神经保护、保肝明目等多种活性[2], 药用价值广泛。此外, 枸杞常作为保健品、食品的优质天然原料及普通群众的滋补茶蔬, 具有显著的经济价值。枸杞原植物来源于枸杞属, 枸杞属在世界范围内广泛分布, 仅在我国作为经济植物广泛栽培于西北和华北地区[3]。
核糖体DNA (nuclear ribosomal DNA, nrDNA), 是由一些高度重复序列组成的多基因家族, 在高等植物中有成千上万的拷贝, 这些拷贝在基因组内串联重复, 分布于一对或多对染色体上[4]。每个重复单元由3个rRNA (18S rRNA、5.8S rRNA、26S rRNA) 共同构成, 基因间由转录间隔区(internal transcribed spacer, ITS1和ITS2) 隔开[5-7]。nrDNA (包括ITS) 的进化模式通常为致同进化(concerted evolution), 拷贝之间趋于均一化, 几乎没有差异, 为ITS序列的PCR扩增产物直接测序奠定了基础。此外, ITS序列进化速率快, 片段长度适中, 能够提供较多的变异信息, 已经成为现今植物系统发育和DNA条形码鉴定研究中常用的分子标记之一[4, 8, 9]。
随着研究的不断深入和测序技术的突飞猛进, 植物类群中ITS多态性逐渐受到学者们的关注。有研究发现, ITS序列在植物基因组中存在致同进化不完全的现象, 拷贝之间存在明显的差异。此外, 一些ITS拷贝可能会由于功能退化而变成假基因(pseudogenes)。已有研究证实, 在许多植物类群中存在ITS假基因现象, 如苏铁属(Cycas)[10]、梨属(Pyrus)[11]、栎属(Quercus)[12]、落叶松属(Larix)[13]、山茶属(Camellia)[14-16]、樟属(Cinnamomum)[17]、胡枝子属(Lespedeza)[18]、乳突球属(Mammillaria)[19, 20]、茜草科(Rubiaceae)[21, 22]等。药用植物中ITS多态性也逐渐引起重视, 如紫堇属(Corydalis)[23]、人参属(Panax)[24]等植物ITS多态性已被证明, 但是, 对药用植物ITS假基因的现象尚未展开深入研究。
中国枸杞属植物有7种3变种, 目前枸杞属ITS的研究主要集中于物种鉴定和遗传关系分析。2017年首次有报道提及枸杞品种及个体内的ITS多态性和假基因现象[25], 但尚未展开系统而深入的研究。本研究以枸杞属植物为研究对象, 通过直接测序、克隆测序DNA和cDNA的ITS序列, 评估枸杞种内和种间的ITS多态性, 在此基础上进行ITS假基因的鉴定, 同时推测ITS致同进化不完全和假基因的成因, 以期为枸杞属系统进化和物种形成研究提供一定的参考。
材料与方法材料 本研究以枸杞属栽培品种为研究对象, 采集新鲜叶片, 通过变色硅胶干燥和液氮低温保存两种方式储存, 具体情况见表 1。所有样品由中国医学科学院药用植物研究所姚入宇博士和中国中医科学院医学实验中心钱丹副研究员鉴定。同时, 从GenBank下载44条枸杞属ITS序列(登录号: JQ320139~JQ320172; KJ189761~KJ189770), 具体情况见表 2。
| Table 1 The information of the Lycium materials |
| Table 2 The information of the GenBank download |
植物总DNA提取和PCR扩增 采用天根生化科技(北京) 有限公司的Plant Genomics DNA Kit (Cat No. DP305-03) 提取植物总DNA。使用通用引物ITS1 (5'-TCCGTAGGTGAACCTGCGG-3') 和ITS4 (5'-TCCTC CGCTTATTGATATGC-3')[26]进行ITS序列扩增。
PCR扩增使用全式金生物技术(北京) 有限公司的EasyTaq酶。PCR扩增体系为50 μL: 2×SuperMix 25 μL、上下游引物各2 μL、模板DNA 2 μL、ddH2O 19 μL。扩增程序如下: 94 ℃, 预变性5 min; 94 ℃变性30 s, 56 ℃退火30 s, 72 ℃延伸45 s, 38个循环; 72 ℃延伸10 min。
植物总RNA提取和RT-PCR 为了更好地判断基因组5.8S序列是否具有功能, 将相关参数与RNA反转录合成的cDNA ITS进行比较[19]。使用天根生化科技(北京) 有限公司的RNA Easy Fast Plant Tissue Kit (Cat No.DP452) 提取植物总RNA。用FastKing RT Kit(With gDNase) 试剂盒合成cDNA第一链, 再以与PCR扩增相同的引物和条件进行RT-PCR获得cDNA ITS。
克隆和测序 PCR和RT-PCR产物经1.0%琼脂糖凝胶电泳后, 切下目的片段并用Thermo Scientific Gene JET Gel Extraction Kit凝胶回收试剂盒回收、纯化产物, 使用天根生化科技(北京) 有限公司Lethal Based Fast Cloning Kit试剂盒进行克隆。然后每个样品挑选11个克隆产物, 经PCR鉴定阳性克隆后送往北京诺赛基因组研究中心有限公司测序, 同时, 对PCR产物也进行了直接测序。
序列处理与分析 使用Seqman (DNAStar, Madison, WI) 软件, 通过将测序序列与相应峰图对比完成单向序列的校正, 然后将正向和反向互补序列进行对比拼接, 以保证所得序列的准确性。根据GenBank中已有的枸杞属植物ITS序列, 确定ITS1、5.8S和ITS2的边界。
为了确定枸杞属植物是否存在ITS假基因, 进行如下分析: ①使用MEGA 7计算ITS序列长度和GC含量, 并基于K2P双参数模型(Kimura 2-parameter model) 构建NJ无根进化树, 以bootstrap方法2 000次重复取样分析分支支持度; ②检测是否存在种子植物5.8S特有的3个保守基序(motif1: CGATGAAGAACGTAGC; motif2: GAATTGCAGAATCC; motif3: TTTGAACGCA)[27]; ③通过在线软件Mfold[28]预测5.8S和ITS2区的最小自由能(37 ℃时的ΔG)。在初步确定假基因的基础上, 再用DnaSP计算功能序列和假基因的核苷酸差异平均值(average number of nucleotide differences, K)、核苷酸多样性(nucleotide diversity, π) 并进行中性检验(Tajima's D或Fu and Li's D), 进一步对前面的分析结果加以验证。
结果与分析 1 ITS序列长度、GC含量、最小自由能分析及假基因的鉴定本研究共得到100条不同的ITS序列, 其中包括10条直接测序ITS序列和90条克隆ITS序列(57条基因组DNA ITS序列和33条cDNA ITS序列)。直接测序ITS序列长度变异范围为601~648 bp, 基因组DNA ITS序列长度变异范围为535~694 bp, cDNA ITS序列长度变异范围为536~648 bp。与功能基因相比, 假基因具有明显的长度变异、较低的GC含量及较高的二级结构最小自由能等特征, 通过对144条ITS序列(本研究100条和GenBank 44条) 的碱基组成、长度变异、5.8S和ITS2二级结构最小自由能进行统计分析(表 3), 发现83条序列符合假基因特征, 因此鉴定为假基因。
| Table 3 Characteristic information of ITS regions of Lycium. F: Functional ITS sequences; P: ITS pseudogene sequences; SD: Standard deviation; Δ: Direct sequencing succeeded |
宁杞5号克隆测序ITS序列中(N5、QH-N5-1和QH-N5-5) 既有功能序列, 也存在假基因序列。而所有样品的直接测序结果和其他材料(N1、N7和ZH) 的克隆序列得到的全部为假基因序列。GenBank下载的44条ITS序列分析发现功能序列和假基因序列各占一半, 均为22条。假基因序列的ITS1、5.8S和ITS2三个区域GC含量显著低于功能序列, 表明其甲基化C位点碱基发生频繁突变, 而5.8S和ITS2的最小自由能却显著高于功能序列, 表明假基因序列的二级结构稳定性较功能序列低。
种子植物5.8S特有的3个保守基序(motif1、motif2和motif3) 的碱基突变是假基因判定的主要标准。本研究的144条ITS序列中, motif3保守型最高, 95%的序列中均含有该基序。其中, 28条序列含有5.8S全部的3个保守基序且没有发生碱基变异, 被鉴定为功能序列。33条cDNA功能序列的3个保守基序中均以motif1第13位T→C和motif2第13位C→T组合变异。83条序列的5.8S不含有保守基序或3个保守基序中至少有一个存在碱基变异, 被鉴定为假基因。假基因的3个保守基序碱基变异呈现一定的规律性, 其中, 以motif1第12位G→A、16位C→A和motif2第3位A→G组合变异为主, 占总体变异的67.5%。
2 核苷酸多样性及中性检测核苷酸多样性分析显示, 假基因序列5.8S区的核苷酸多样性(π) 和平均核苷酸差异数(K) 相对于功能序列高(表 4), 其π值和K值均是功能序列的1.34倍, 说明5.8S假基因序列具有较多的变异。对于所有假基因序列, 3个区域的核苷酸多样性和平均核苷酸差异数数值高低排列顺序不同, 核苷酸多样性排列顺序为ITS1 > 5.8S > ITS2, 平均核苷酸差异数排列顺序为ITS2 > ITS1 > 5.8S。
| Table 4 Nucleotide diversity of the ITS regions in Lycium. F: Functional ITS sequences; P: ITS pseudogene sequences; Nucleotide diversity (π); Average number of nucleotide differences (K) |
GenBank的44条序列和本研究的100条序列因测序引物的不同分别进行中性检验。通过Tajima's D或Fu and Li's D检验结果发现(表 5), ITS功能序列D值的绝对值较大, 且5.8S区呈现显著性差异(P < 0.001或P < 0.02), 表明ITS功能序列偏离中性进化; 而假基因序列D值的绝对值较小, 且未呈现显著差异(P > 0.10), 表明假基因序列倾向于中性进化[29, 30]。
| Table 5 Neutrality test of the ITS regions in Lycium. F: Functional ITS sequences; P: ITS pseudogene sequences. Significant difference at ***P < 0.001 in Tajima's D analysis and ***P < 0.02 in Fu and Li's D analysis |
基于所有ITS序列的遗传距离构建的NJ树中, 除宁杞5号的部分cDNA (cDN5-2、cDN5-4、cDN5-9和cDN5-10) 外, 所有cDNA功能序列单独聚为一支; 除JQ320148、JQ320154、JQ320163、JQ320172外, 枸杞属植物ITS功能序列和假基因序列全部分离出来(图 1), 但不同品种的功能序列并没有完全聚为一支, 而是以较高的支持度聚为3支(Ⅰ、Ⅱ、Ⅲ), 其中第Ⅰ支多为不同品种的宁夏枸杞及其杂交种; 第Ⅱ支为黑果枸杞; 第Ⅲ支为宁杞5号(N5、QH-N5-1和QH-N5-5) 的6条克隆序列。
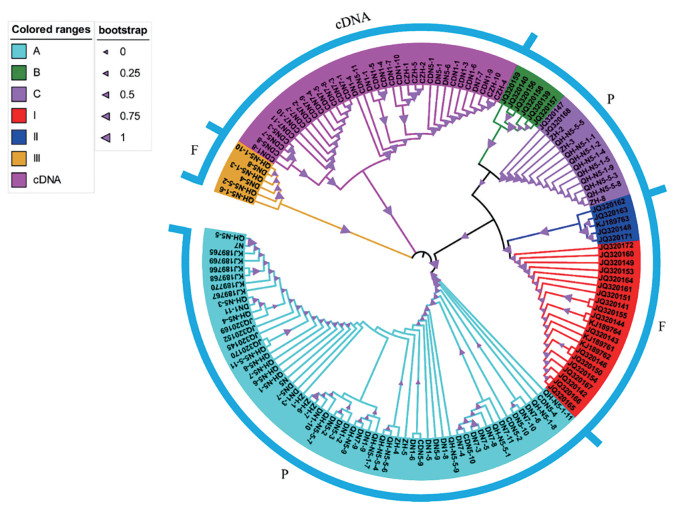
|
Figure 1 Neighbor-joining tree topology constructed on Kimura two-parameter distance matrix using ITS entire region of all paralogs in Lycium. A, B and C represent the three different types of pseudogenes, respectively. Ⅰ, Ⅱ and Ⅲ represent the three different types of functional gene, respectively. cDNA represents cDNA ITS sequence |
本研究鉴定的83条假基因序列, 同样以较高的支持度聚为3支(A、B、C), 其中A支主要为宁夏枸杞和栽培品种(N1、N5、N7、ZH、QH-N5) 的直接测序和DNA克隆序列, 均只有motif3保守基序; B支为枸杞属其他种和变种(包括红枝枸杞、新疆枸杞、韩国枸杞、蔓生枸杞、云南枸杞和常熟枸杞), 均只有motif2保守基序; C支主要为QH-N5-1、QH-N5-5和ZH克隆序列, 均含有motif2和motif3两个保守基序。
N1、N7和ZH的cDNA ITS克隆序列全部分布在同一支, 而N5的cDNA ITS克隆序列及N1、N5、N7的DNA ITS克隆序列均分布在cDNA支和A支两支上。这两支序列长度相差约100 bp, 且cDNA支ITS1、5.8S、ITS2三个区域的GC含量均低于A支, 说明枸杞属植物DNA和cDNA的ITS序列存在明显的差异。此外, 本研究的系统发育聚类结果与其他两个研究团队的报道一致[31, 32], 推测假基因可能对枸杞属植物种属系统构建影响不大。从假基因的角度分析聚类形成, 能更好地解释前人研究中聚类分支形成的原因。
讨论nrDNA ITS序列在高等植物中通常为致同进化, 不同的ITS序列基本趋于一致或完全一致, 这为其作为PCR扩增产物直接测序奠定了基础。同时, ITS能够提供较为合适的变异和信息位点, 加之双亲遗传的特点, 已经广泛地应用于属内种间或种内群体的系统进化研究, 成为DNA条形码鉴定的核心序列。在应用ITS序列进行枸杞属植物的鉴定研究中, 有报道因ITS序列PCR直接测序合格率低, 无法获得理想结果, 不纳入统计[33]; 也有研究结果[25]显示直接测序合格率仅为64%; 本研究中直接测序合格率为71%。为提高合格率, 在PCR扩增中加入DMSO进行改善[16], 筛选了7个不同的浓度梯度(0%、1%、2%、4%、6%、8%、10%), 但直接测序合格率并未得到改善。由此可见, ITS在枸杞属存在较低的扩增和鉴定成功率。
本研究对ITS序列在枸杞属的应用开展了进一步研究, 通过多次重复试验, 排除PCR过程污染、测序错误等可能性。发现枸杞属植物不同种间及个体间的基因组内、cDNA组内的ITS序列均存在广泛的多态性, 不仅GC含量有一定的变化, 不同序列长度之间也存在变化。枸杞属ITS序列总长度为535~694 bp, GC含量为46.17%~59.03%。不同序列长度间存在超过100 bp的差异且主要在ITS1区。基因组内不同克隆序列GC含量变化最大, 为47.29%~59.03%, 而cDNA组GC含量变化范围为46.17%~55.40%。此外, 枸杞属植物存在多种ITS变异类型, 如DN5检测到3种类型, 包括1种功能基因和2种假基因, 而其他样品均检测到一到两种变异类型。
高度重复的nrDNA ITS有成千上万的拷贝, 其基因位点在进化过程中易发生功能缺失突变, 如插入、缺失或移码, 从而形成假基因[34]。本研究发现枸杞属植物种间和个体内均存在大量的ITS假基因。与功能基因相比, 假基因的GC含量明显降低, 表明其甲基化C位点碱基发生频繁突变, 低GC含量可能会导致基因功能的丧失, 也可能是假基因的起源[35]; 而5.8S和ITS2区二级结构最小自由能明显偏高, 表明其二级结构稳定性较功能序列显著降低。进一步分析发现, 假基因序列在高度保守的5.8S区的核苷酸多态性和平均核苷酸差异数明显偏大, 说明其序列发生了较多的变异, 包括碱基替换和长度变异。同时, 由于假基因不受选择压力的约束, 在进化过程中核苷酸置换脱离了功能限制, 倾向于中性进化, 因此, 比功能基因的进化速率要快。
5.8S序列通常被认为高度保守, 有报道在系统发育分析时往往将其排除不加以考虑[31, 32]。然而在ITS假基因的鉴定研究中, 5.8S是ITS拷贝中极具价值的功能指标。通过检测5.8S区是否含有种子植物特有的3个保守基序或是否发生碱基变异及GC含量、二级结构最小自由能等可对假基因进行判定。本研究通过5.8S序列的变异进行了相关评估, 判定方法简便有效, 同时发现了前人在枸杞属系统发育研究中使用的ITS序列部分为假基因。
基于系统发育树分析, 本研究中枸杞属植物共存在3种类型的假基因且彼此间存在较大的差异, 类型A以宁夏枸杞和栽培品种的直接测序和DNA克隆序列为主; 类型B以枸杞属其他种和变种的克隆序列为主; 而类型C以QH-N5-1、QH-N5-5和ZH克隆序列为主。此外, ZH、QH-N5-1和QH-N5-5三个个体中存在两种类型的ITS假基因, 在系统发育树中分布于A和C两支, 其中, A支仅含有motif1保守基序, C支含有motif2和motif3两个保守基序。这些结果表明, 枸杞属植物不同种间及个体基因组内均存在显著的致同进化不完全现象。
基因家族成员以协同的方式进化, 这种进化模式称为“致同进化”, 即当某个成员发生突变时, 它就能够通过反复的基因交换或不等交换传播到其他成员中去[36]。以往的研究显示, 物种致同进化不完全受多种因素的影响, 既有物种进化和生活史方面的因素, 如多倍体化[37]、异源多倍体化[38]、孤雌生殖[39]、世代周期长[40]和杂交[12]等, 也有基因家族在染色体上、基因组中组织结构方面的因素。枸杞在中国的栽培历史悠久, 存在很多的农家品种和品系。杂交是其主要的育种手段之一[41]。如雄性不育种质“宁杞5号”的样品种内和个体内均存在明显的多态性, 克隆测序后序列均为功能序列和假基因序列两种类型。甚至其cDNA功能序列也分为两类, 在系统发育树上分布于cDNA支和A支, 且这两类cDNA长度存在100 bp的差异。此外, 枸杞属植物虽存在多种变异类型, 但同一个体的不同克隆序列大多被置于一到两个不同的分支, 并且位于同一分支上的序列间具有很高的相似度, 仅存在少数碱基的差异。因此, 推测基因组内串联重复序列之间的近期分化、物种间的杂交可能是导致枸杞属植物个体内ITS相似序列的变异及致同进化不完全的主要原因。
与四照花属[42]、苏铁属[10]的结果相似, 枸杞属绝大部分假基因序列与功能序列在基因树上各自独立形成分支, 表明假基因与功能基因起源时间存在明显的差异, 又因假基因较功能基因进化速率快, 倾向于中性进化, 因此推测枸杞属ITS假基因起源时间可能相对较早, 并且具备了较多的遗传变异, 但假基因没有形成一个独立的单系, 说明他们可能不是一次起源, 或者一次起源后变异分化的速度差异较大。物种的进化起源对物种的分类鉴定、生物多样性保护及遗传育种具有重要意义, ITS多态性对枸杞属植物起源进化和系统发育具有重要影响, 但是克隆测序结果往往偏向于获取高丰度的序列, 而低丰度的序列难以获取[24], 因此, 后继研究可采用高通量测序技术, 更深层次地揭示ITS多态性对物种进化形成的影响。
本文首次对枸杞属植物ITS序列致同进化不完全和假基因现象进行了较为系统地报道, 发现枸杞属种间、种内及个体内均存在ITS多态性和假基因现象。对枸杞属致同进化不完全进行探讨, 发现枸杞属ITS致同进化不完全或许是导致枸杞属植物DNA条形码研究中测序成功率和鉴定率较低的重要原因之一。初步推测杂交可能是导致枸杞属ITS多态性的主要原因, 后续研究将进一步扩大样品量, 采用高通量测序进行相关研究, 为假基因在枸杞属的应用提供更多DNA序列的分子数据。本研究为ITS序列在枸杞属内亲缘关系、系统发育重建和DNA条形码鉴定的研究提供了一定的参考和科学依据。
致谢: 感谢中国医学科学院药用植物研究所姚入宇博士为本研究提供部分材料。
作者贡献: 黄璐琦构思并设计了实验方案; 钱丹完成样品采集, 数据分析和文章修改; 张娇完成样品采集, 实验并撰写论文初稿; 聂颖兰和郭威参与数据分析; 所有作者均阅读并参与了文章的修改。
利益冲突: 本文无相关利益冲突。
| [1] |
Chinese Pharmacopoeia Committee. Pharmacopoeia of the People's Republic of China (中华人民共和国药典)[S]. Beijing: China Medical Science Press, 2020: 260.
|
| [2] |
Wang SS, Zhang Z, Chen NH. Research progress on main active components and pharmacological effects of wolfberry[J]. Acta Neuropharmcol (神经药理学报), 2018, 8: 53. |
| [3] |
Qian D, Ji R, Guo W, et al. Advances in research on relationships among Lycium species and origin of cultivated Lycium in China[J]. China J Clin Mater Med (中国中药杂志), 2017, 42: 3282-3285. |
| [4] |
Eickbush TH, Eickbush DG. Finely orchestrated movements: evolution of the ribosomal RNA genes[J]. Genetics, 2007, 175: 477-485. DOI:10.1534/genetics.107.071399 |
| [5] |
Tian X, Li DZ. Application of DNA sequences in plant phylogentic study[J]. Acta Bot Yunnan (云南植物研究), 2002, 24: 170-184. |
| [6] |
Álvarez I. Ribosomal ITS sequences and plant phylogenetic inference[J]. Mol Phylogenet Evol, 2003, 29: 417-434. |
| [7] |
Li DZ, Gao LM, Li HT, et al. Comparative analysis of a large dataset indicates that internal transcribed spacer (ITS) should be incorporated into the core barcode for seed plants[J]. Proc Natl Acad Sci U S A, 2011, 108: 19641-19646. DOI:10.1073/pnas.1104551108 |
| [8] |
Bailey C. Characterization of angiosperm nrDNA polymorphism paralogy, and pseudogenes[J]. Mol Phylogenet Evol, 2003, 29: 435-455. DOI:10.1016/j.ympev.2003.08.021 |
| [9] |
Kovarik A, Pires JC, Leitch AR, et al. Rapid concerted evolution of nuclear ribosomal DNA in two tragopogon allopolyploids of recent and recurrent origin sequence data from this article have been deposited with the EMBL/GenBank Data Libraries under accession nos. AY458586, AY458588, AY458589, and AY458587[J]. Genetics, 2005, 169: 931-944. DOI:10.1534/genetics.104.032839 |
| [10] |
Xiao LQ, Möller M, Zhu H. High nrDNA ITS polymorphism in the ancient extant seed plant Cycas: incomplete concerted evolution and the origin of pseudogenes[J]. Mol Phylogenet Evol, 2010, 55: 168-177. DOI:10.1016/j.ympev.2009.11.020 |
| [11] |
Zheng XY, Cai DY, Yao LH, et al. Non-concerted ITS evolution, early origin and phylogenetic utility of ITS pseudogenes in Pyrus[J]. Mol Phylogenet Evol, 2008, 48: 892-903. DOI:10.1016/j.ympev.2008.05.039 |
| [12] |
Muir G, Fleming CC, Schlötterer C. Three divergent rDNA clusters predate the species divergence in Quercus petraea (Matt. ) Liebl. and Quercus robur L. [J]. Mol Biol Evol, 2001, 18: 112-119.
|
| [13] |
Weu XX, Wang XQ. Recolonization and radiation in Larix (Pinaceae): evidence from nuclear ribosomal DNA paralogues[J]. Mol Ecol, 2004, 13: 3115-3123. DOI:10.1111/j.1365-294X.2004.02299.x |
| [14] |
Xu Y, Xu J, Gao JY, et al. Polymorphism of ITS in Camellia: an example of widespread escape from consistent evolution[J]. Chin Bull Bot (植物学报), 2011, 46: 162-169. |
| [15] |
You H, Zhou AT, Yue LL, et al. Amplification and sequence characterization of ITS in Camellia[J]. Bull Bot Res (植物研究), 2014, 34: 403-408. |
| [16] |
Zhou AT, Yue LL, Li M, et al. Intra-genomic polymorphism in the nrDNA ITS sequence of Camellia reticulata[J]. Plant Sci J (植物科学学报), 2013, 31: 1-10. DOI:10.3724/SP.J.1142.2013.10001 |
| [17] |
Huang JF, Li L, van der Werff H, et al. Origins and evolution of cinnamon and camphor: a phylogenetic and historical biogeographical analysis of the Cinnamomum group (Lauraceae)[J]. Mol Phylogenet Evol, 2016, 96: 33-44. DOI:10.1016/j.ympev.2015.12.007 |
| [18] |
Xu B, Zeng XM, Gao XF, et al. ITS non-concerted evolution and rampant hybridization in the legume genus Lespedeza (Fabaceae)[J]. Sci Rep, 2017, 7: 40057. DOI:10.1038/srep40057 |
| [19] |
Harpke D, Peterson A. Non-concerted ITS evolution in Mammillaria (Cactaceae)[J]. Mol Phylogenet Evol, 2006, 41: 579-593. DOI:10.1016/j.ympev.2006.05.036 |
| [20] |
Harpke D, Peterson A. Extensive 5.8S nrDNA polymorphism in Mammillaria (Cactaceae) with special reference to the identification of pseudogenic internal transcribed spacer regions[J]. J Plant Res, 2008, 121: 261-270. DOI:10.1007/s10265-008-0156-x |
| [21] |
Razafimandimbison SG, Kellogg EA, Bremer B. Recent origin and phylogenetic utility of divergent ITS putative pseudogenes: a case study from Naucleeae (Rubiaceae)[J]. Syst Biol, 2004, 53: 177-192. DOI:10.1080/10635150490423278 |
| [22] |
de Sousa Queiroz C, de Carvalho Batista FR, de Oliveira LO. Evolution of the 5.8S nrDNA gene and internal transcribed spacers in Carapichea ipecacuanha (Rubiaceae) within a phylogeographic context[J]. Mol Phylogenet Evol, 2011, 59: 293-302. DOI:10.1016/j.ympev.2011.01.013 |
| [23] |
Li M, Zhao H, Zhao F, et al. Alternative analyses of compensatory base changes in an ITS2 phylogeny of Corydalis (Papaveraceae)[J]. Ann Bot, 2019, 124: 233-243. DOI:10.1093/aob/mcz062 |
| [24] |
Song J, Shi L, Li D, et al. Extensive pyrosequencing reveals frequent intra-genomic variations of internal transcribed spacer regions of nuclear ribosomal DNA[J]. PLoS One, 2012, 7: e43971. DOI:10.1371/journal.pone.0043971 |
| [25] |
Chen LL, Zhao MX, Jiang S, et al. Identification, phylogenetic relationship analysis of Lycium based on rbcL-a and ITS sequences and the discovery of ITS pseudogene[J]. Biotechnol Bull (生物技术通报), 2017, 33: 123-130. |
| [26] |
White T, Bruns T, Lee S, et al. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics[M]//Innis M, Gel-fand J, Sainsky J, et al, editors. PCR Protocols, a Guide to Methods and Applications. San Diego: Academic Press, Inc., 1990: 315-321
|
| [27] |
Harpke D, Peterson A. 5.8S motifs for the identification of pseudogenic ITS regions[J]. Botany, 2008, 86: 300-305. DOI:10.1139/B07-134 |
| [28] |
Zuker M. Mfold web server for nucleic acid folding and hybridization prediction[J]. Nucleic Acids Res, 2003, 31: 3406-3415. DOI:10.1093/nar/gkg595 |
| [29] |
Fu YX. New statistical tests of neutrality for DNA samples from a population[J]. Genetics, 1996, 143: 557-570. DOI:10.1093/genetics/143.1.557 |
| [30] |
Fu YX. Statistical tests of neutrality of mutations against population growth, hitchhiking and background selection[J]. Genetics, 1997, 147: 915-925. DOI:10.1093/genetics/147.2.915 |
| [31] |
Shi ZG, Wan R, Li YL, et al. Identification of Lycium L. germplasm resources based on nrDNA ITS sequence[J]. Huibei Agric Sci (湖北农业科学), 2016, 55: 5966-5968. |
| [32] |
Li XG, Wang YK, Li J, et al. Study on 10 species of Lycium L. genetic relationship based on nrDNA-ITS sequence[J]. Chin Agric Sci Bull (中国农学通报), 2014, 30: 128-135. |
| [33] |
Xin TY, Yao H, Gao HH, et al. Super food Lycium barbarum (Solanaceae) traceability via an internal transcribed spacer 2 barcode[J]. Food Res Int, 2013, 54: 1699-1704. DOI:10.1016/j.foodres.2013.10.007 |
| [34] |
You H, Zhou AT, Yue LL, et al. Research on nrDNA ITS pseudogenes in plants[J]. Biotechnol Bull (生物技术通报), 2013, 11: 14-18. |
| [35] |
Bailey JA, Liu G, Eichler EE. An alu transposition model for the origin and expansion of human segmental duplications[J]. Am J Hum Genet, 2003, 73: 823-834. DOI:10.1086/378594 |
| [36] |
Nei M, Rooney AP. Concerted and birth-and-death evolution of multigene families[J]. Annu Rev Genet, 2005, 39: 121-152. DOI:10.1146/annurev.genet.39.073003.112240 |
| [37] |
Suh Y, Thien LB, Zimmer EA. Nucleotide sequences of the internal transcribed spacers and 5.8S rRNA gene in Canella winterana (Magnoliales; Canellaceae)[J]. Nucleic Acids Res, 1992, 20: 6101-6102. DOI:10.1093/nar/20.22.6101 |
| [38] |
Karvonen P, Savolainen O. Variation and inheritance of ribosomal DNA in Pinus sylvestris L. (Scots pine)[J]. Heredity, 1993, 71: 614-622. DOI:10.1038/hdy.1993.186 |
| [39] |
Campbell CS, Wright WA, Cox M, et al. Nuclear ribosomal DNA internal transcribed spacer 1(ITS1) in Picea (Pinaceae): sequence divergence and structure[J]. Mol Phylogenet Evol, 2005, 35: 165-185. DOI:10.1016/j.ympev.2004.11.010 |
| [40] |
Sang T, Crawford DJ, Stuessy TF. Documentation of reticulate evolution in peonies (Paeonia) using internal transcribed spacer sequences of nuclear ribosomal DNA: implications for biogeography and concerted evolution[J]. Proc Natl Acad Sci U S A, 1995, 92: 6813-6817. DOI:10.1073/pnas.92.15.6813 |
| [41] |
An W, Zhan HX, He J, et al. Progress and prospects of wolfberry breeding[J]. Nor Hort (北方园艺), 2009, 33: 125-128. |
| [42] |
Hu ZF, Xiang QY, Huang HW. Incomplete concerted evolution of nrDNA ITS in Cornus subg. Syncarpea (Cornaceae)[J]. J Trop Subtrop Bot (热带亚热带植物学报), 2013, 21: 29-37. |
 2022, Vol. 57
2022, Vol. 57


