2. 上海中医药大学教学实验中心, 上海 201203;
3. 上海上药杏灵科技药业股份有限公司, 上海 201703
2. Experiment Center for Teaching and Learning, Shanghai University of Traditional Chinese Medicine, Shanghai 201203, China;
3. Shanghai Xingling Science and Technology Pharmaceutical Co., Ltd., Shanghai 201703, China
miRNA (microRNA) 是一类长度约21nt的内源性的非编码小RNA, 在真核细胞中广泛存在, 大部分来源于基因间隔区或者基因的内含子区, 具有自己独立的转录单元[1]。一个成熟的miRNA通常有多个靶基因, 植物miRNA与靶基因序列几乎完全互补配对, 可在转录后剪切或者抑制其翻译来负调控靶基因的表达水平[2]。1993年, Lee和Wightman等[3, 4]分别在秀丽隐杆线虫(Caenorhabditis elegans) 中发现了第一个动物miRNA—lin-4。植物miRNA的研究相对较晚, 直到2002年, 人们才在拟南芥中分离出首批植物miRNA并鉴定了第一个植物miRNA—miR171[5]。随后, 植物miRNA的发展十分迅速, 截至目前, miRBase22数据库中收录了48 860条miRNA成熟序列, 其中仅拟南芥就收录了428条miRNA成熟序列[6]。
植物miRNA的生物合成主要是在细胞核和细胞质中进行(图 1), 首先, 在细胞核中, 大多数编码miRNA基因由RNA聚合酶II转录出pri-miRNA (primary miRNA)[7, 8]; 接着pri-miRNA在DCL1 (dicer-like 1)、HYL1 (hyponastic leaves 1) 和SE (serrate) 酶的共同作用下形成pre-miRNA (precursor miRNA)[9]; 然后经过第二次剪切加工形成不完全配对的双链miRNA: miRNA*; 随后在HENI (HUA enhancer1) 作用下进行甲基化修饰[10], 甲基化的miRNA: miRNA*在HASTY蛋白作用下转运到细胞质中, 其中一条成熟的miRNA则与Ago (argonaute) 蛋白组成沉默复合体(RNA induced silencing complex, RISC), miRNA*大部分会被降解, 只有少数有功能的被保留下来。然而, 近期Bologna等[11]的研究表明, RISC主要是在细胞核内通过CRM1/EXPORTIN1 (EXPO1) 组装完成后再运送到细胞质中。RISC通过碱基互补配对与靶基因结合, 介导靶基因的剪切或者翻译抑制[1]。在植物中, 关于miRNA翻译抑制现象报道较少, 可能是由于剪切现象较易确认及植物中高质量的抗体缺乏而难以确定蛋白水平[12]。此外, 被miRNA剪切的靶基因, 还有可能产生相位siRNA (phased small interfering RNA, phasiRNA), 这是植物中广泛存在的一种保守机制, 即miRNA对一类特殊靶基因的剪切可以触发产生具有一定相位的siRNA[13]。

|
Figure 1 The biogenesis of miRNA in plants |
近年来miRNA已成为生物学研究领域的热点, 众多研究表明植物miRNA在生长发育、激素信号转导及胁迫响应等方面的都发挥着重要的调控作用[14-20], 甚至可以跨界调控其他物种体内基因的表达[21]。此外, 研究发现, miRNA对植物次生代谢产物合成也发挥了重要调控作用[18, 22-24]。药用植物药效活性成分多为次生代谢产物, 通常在原植物体内含量都很低, 限制了其开发利用。近年来, 借助计算机辅助的高通量测序技术, 大量药用植物miRNA被发现与挖掘[25-30]。miRNA调控研究一直是中药资源领域的研究热点问题, 它对药用植物优良品质的形成及对具有重要药用价值的药用活性成分的资源开发具有非常重要的意义, 也有不少文献对此进行了总结。Zhu等[22]总结了近年来药用植物miRNA调控次生代谢的研究情况及miRNA靶基因预测及验证方法; Lu等[23]主要综述了药用植物miRNA的鉴定情况及与次生代谢产物生物合成相关miRNA; Lu等[24]综述了miRNA调控药用植物次生代谢产物生物合成情况, 重点综述了miRNA对萜类、生物碱和黄酮类生物合成的影响。本文在前人基础上对当前植物miRNA一般研究方法及近年来药用植物miRNA的研究进展做一综述, 重点介绍了miRNA对具有药用活性的次生代谢产物形成特别是萜类化合物的调控作用, 并对药用植物miRNA发展趋势做了展望。
1 植物miRNA研究方法miRNA研究一般包括miRNA的发现与生物学功能解析两个方面。miRNA的发现有遗传筛选、直接克隆、生物信息学预测和高通量测序等方法。计算机辅助的高通量测序技术是当前植物miRNA发现的最主要方法, 被广泛应用于药用植物miRNA发现与挖掘。如何验证miRNA的靶基因及阐明其生物学功能是miRNA研究的难点与重点。miRNA靶基因预测和验证是解析miRNA生物学功能的首要环节, 靶基因预测有多种软件可以使用, 如psRNATarget、C-mii、TargetFinder及psROBOT等[31-34]; 靶基因验证常用方法有基于cDNA末端快速扩增(rapid amplification of cDNA ends, RACE) 技术的5′RLM-RACE和3′PPM-RACE、基于高通量测序技术的降解组测序(degradome sequencing)、基于靶基因表达水平检测的烟草瞬间表达系统、荧光定量PCR及蛋白质免疫印迹(Western blot) 等[35-38]。另一方面, miRNA的过表达、靶基因突变体表达或抑制表达等, 可用于探索miRNA表达水平改变而引起的植物基因型与表型变化, 进而研究miRNA调控的生理活动过程及相关的分子机制。下面就植物miRNA功能解析主要实验方法(图 2) 进行介绍。
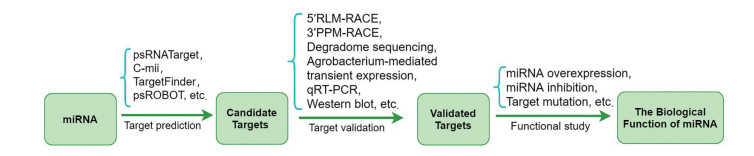
|
Figure 2 General methods for functional study of plant miRNAs |
利用T4 RNA连接酶介导的5′RLM-RACE是目前最可靠的验证植物miRNA靶基因的方法。靶基因被miRNA诱导的沉默复合体在靶位点切断后, 产生一个3′剪切产物和一个5′剪切产物, 其中3′剪切产物含有一个5′端磷酸基团和3′poly A尾巴, 5′剪切产物含有3′羟基和5′帽子结构。5′RLM-RACE是利用T4 RNA连接酶在3′剪切产物的5′端连上一个人工合成的RNA接头, 由于完整mRNA的5′端有帽子结构, 因此不能与RNA接头相连。然后用随机引物进行反转录, 接着用接头上的内外两层锚定引物和基因下游的两个特异性引物进行巢氏PCR, 最后将扩增产物连接到克隆载体上进行测序, 将测得片段与靶基因序列比对即可确定miRNA是否剪切靶基因及剪切位点。
3′PPM-RACE的原理与5′RLM-RACE相似, 它选取靶基因的5′端剪切产物, 在ploy A聚合酶的作用下在其3′端连一个poly A尾巴。然后用特异的逆转录引物进行反转录, 再用接头上的通用引物和基因上游的特异性引物进行巢氏PCR, 最后将扩增产物与载体连接后进行测序[35]。由于RNA一般从3′端开始降解, 因此对更稳定的3′剪切产物的5′端进行RACE更合适。3′PPM-RACE和5′RLM-RACE不仅可以验证miRNA对靶基因的切割, 还可以精确鉴定miRNA的切割位点。研究结果表明, RISC通常在miRNA的5′端的第10和第11碱基的位置剪切靶基因[7, 31]。Song等[35]同时运用5′RLM-RACE和3′PPM-RACE鉴定甜橙(Citrus sinensis) 中miRNA相应的6个靶基因, 并结合荧光定量PCR技术定量研究不同生长时期以及不同器官组织中剪切产物的表达水平, 发现靶基因5′剪切产物和3′剪切产物在相对表达量的趋势上是一致的。
1.1.2 降解组测序降解组测序也称为平行末端分析技术(parallel analysis of RNA end, PARE), 它的原理与5′RLM-RACE相似, 由于miRNA的3′剪切产物的5′末端带磷酸基团, 3′末端有poly A尾巴, 测定这种序列, 可以发现所有被剪切的mRNA序列[37]。首先也要利用RNA连接酶在3′剪切产物的5′端连上一个RNA接头, 然后用特异性引物进行反转录, RNA接头上有一个II型限制性内切酶MmeI识别位点, MmeI酶可对识别位点下游20个核苷酸处进行酶切, 酶切后用DNA连接酶在3′端连一个双链DNA接头, 根据两侧接头设计引物PCR后进行高通量测序, 测序后通过生物信息学方法与原mRNA进行序列比对分析, 即可确定miRNA对靶基因的剪切[37]。Garg等[39]利用小RNA测序鉴定了鹰嘴豆(Cicer arietinum L.) 中651个miRNA, 联合降解组与转录组测序数据鉴定了其中629个miRNA的2 131个靶基因。Shao等[40]通过降解组测序鉴定了灵芝(Ganoderma lucidum) 中168个miRNA的靶基因。
1.1.3 农杆菌介导的烟草瞬时表达系统农杆菌介导的烟草(Nicotiana tabacum) 瞬时表达体系可以为miRNA调控靶基因提供简单、快速和直观的证据。分别构建miRNA表达载体和靶基因与GFP报告基因融合的表达载体, 借助农杆菌侵染烟草表皮细胞, 使2种表达载体在烟草活体组织内共表达, 一段时间后通过观察GFP融合蛋白的荧光强弱, 验证miRNA对靶基因的切割作用。如果miRNA可以切割靶基因, 那么融合荧光的强度应该减弱或消失。Llave等[41]将构建好的含miR171的表达载体和靶基因SCL的表达载体借助农杆菌注射到烟草叶片中共表达, 报告基因表达减弱, 表明miR171可以剪切SCL基因。Hu等[42]建立了基于瞬时表达系统的水稻miRNA靶基因快速验证系统。
1.1.4 荧光定量PCR与蛋白质免疫印迹miRNA负调控靶基因的结果是靶基因mRNA表达水平和蛋白水平的下降。因此, 可以利用荧光定量PCR和蛋白质免疫印迹进一步验证通过降解组测序、基因芯片或生物信息学等手段发现的靶基因, 检测过表达或抑制miRNA表达前后植物体内靶基因的mRNA和其编码蛋白表达量的变化, 以此来确定miRNA与靶基因的调控关系。这种方法可以准确鉴定miRNA的靶基因, 方法简单, 但是需要构建miRNA的过表达或抑制表达转基因株系, 耗时较长, 无法鉴定miRNA的剪切位点, 也难以发现miRNA的所有靶基因。另外, 在植物中, 每一个组织存在不同的细胞类型, 而miRNA和靶基因可能表达在同一个组织不同类型的细胞中, 因此有可能出现某种情况下miRNA和靶基因的表达量同时上调或下调的情况。由于植物中高质量的抗体制备比较困难, 蛋白质免疫印迹应用较少, 这可能也是植物中miRNA抑制现象报道较少的原因之一。
上述靶基因验证方法各有优缺点, 为获得准确的实验结果, 通常会选择2种或3种方法对miRNA靶基因进行实验验证。
1.2 miRNA调控生理活动研究方法 1.2.1 miRNA的过表达技术miRNA的过表达是利用转基因的方法过表达miRNA, 引起miRNA靶基因的表达发生变化, 从而研究miRNA的生物学功能。根据所导入miRNA结构的不同, 主要有以pri-miRNA和pre-miRNA形式构建。由于pri-miRNA和pre-miRNA都包含形成成熟miRNA的完整序列, 因此都可以在体内被加工成正确的miRNA成熟序列。其中, pre-miRNA需要构建到含H1或U6启动子的载体上才能转录, 以这种形式构建的miRNA过表达载体是目前应用最广泛的。但是近年来研究发现, miRNA的双臂对于miRNA的成熟非常重要。以pri-miRNA形式构建的载体可以更好的模拟miRNA在体内的加工过程, 因此, 在植物中应用得越来越多。Mao等[43]以pri-miRNA的形式分别在大豆(Glycine max) 和蒺藜苜蓿(Medicago truncatula) 中过表达miR164和miR393来研究它们对根瘤结节形成的影响。
1.2.2 miRNA靶基因的同义突变或缺失突变突变靶基因也是miRNA功能研究的常用方法。在miRNA靶基因确定的情况下, 可以通过靶基因的同义突变或者靶基因功能缺失突变体, 间接研究miRNA功能。靶基因的同义突变, 即在靶基因的miRNA作用位点处引入碱基突变, 使miRNA被整合住而不能剪切靶基因, 同时又不改变靶基因所编码蛋白质的氨基酸序列。靶基因功能缺失突变体则由于氨基酸种类和数目发生改变, 靶基因原有生理功能缺失, 相当于过量miRNA抑制其表达。
Debernardi等[44]通过构建拟南芥(Arabidopsis thaliana) 中miR396靶基因bHLH74同义突变体转基因系发现, 转基因植株中bHLH74表达水平明显升高, 并且叶片与对照相比较小, 叶脉分支数减少, 表明拟南芥中miR396通过bHLH74调控叶片边缘及叶脉的形成。Jin等[45]利用miRNA缺失突变体对miR159的功能开展了深入研究。需要注意的是, 由于一个miRNA通常有多个靶基因, 因此, miRNA某个特定靶基因的功能往往并不能体现miRNA完整的生物学功能。
1.2.3 miRNA的抑制表达除miRNA的过表达外, 抑制miRNA表达也是解析miRNA生物功能的常用方法。TM (target mimicry) 和STTM (short tandem target mimic) 是目前常用的抑制miRNA表达的技术手段, 可以在植物体内特异地诱导miRNA的降解, 降低内源miRNA的表达水平。
2007年, Franco-Zorrilla等[46]发现拟南芥中一个非编码基因IPS1 (induced by phosphate starvation 1) 含有miR399结合域, 但是其与miR399的5′端第10和11位碱基之间有3个不配对碱基, 从而阻止了miR399-RISC复合体对IPS1的切割。研究还发现, IPS1作用原理同样可以运用于抑制其他miRNA的表达。Todesco等[47]通过定向改造拟南芥中IPS1基因构建TM对miR156、miR157和miR159等多个miRNA实现了下调表达。Li等[48]通过构建MIM162成功对水稻(Oryza sativa) miR162a实现了下调表达。
Yan等[49]在TM的基础上进行了改进, 开创了一种新的下调miRNA的方法—STTM。该方法通过人工设计合成一段短DNA序列, 该序列通过一个48碱基的间隔区(spacer) 连接2个与miRNA互补配对的基序, 但是在miRNA剪切位点处有3个不配对碱基, 导致该DNA序列不能被剪切, 但是螯合了目标miRNA, 最终导致目标miRNA的表达量降低, 从而减弱miRNA对靶基因的剪切或翻译抑制, 是目前报道的一种非常有效的抑制miRNA活性的方法。此外, SPONGEs(SPs) 在动物中用于抑制miRNA活性, Reichel等[50]为了测试它在植物中有没有效果, 首次在拟南芥用SPs技术构建了多个表达载体并与TM和STTM做比较, 结果发现SP165/166和STTM165/166抑制miRNA效果明显强于MIM165/166, 而MIM159的效果却优于SP159和STTM159。总之, 没有一种技术可以保证对每个miRNA都有很好的抑制作用, 在实际应用中建议同时选取多种方法以达到理想的实验效果。
在实际应用中, 应同时选取多个方法共同研究miRNA的生物学功能。
2 药用植物miRNA研究进展众所周知, 药用植物可产生种类繁多、结构独特的次生代谢产物, 这些次生代谢产物广泛应用于医药、食品、农业等领域。miRNA在植物生长发育、胁迫响应、次生产物代谢等生理活动中的重要调控作用, 也使药用植物miRNA备受重视。近年来, 随着高通量测序技术和生物信息学的飞速发展, 大量药用植物miRNA被发现和鉴定[51], 药用植物miRNA研究逐渐开展, 下面以几种药用植物为例, 概述药用植物miRNA研究进展情况及其对具有药用活性的次生代谢产物特别是萜类化合物合成的调控作用。
2.1 药用植物miRNA的发现与功能预测动物、人和模式植物miRNA的研究成果为药用植物miRNA研究奠定了基础, 近年来借助于高通量测序与生物信息学分析等手段, 药用植物miRNA被批量发现和注释, 如人参[52, 53]、小蔓长春花[54]、三七[30, 55]、丹参[56, 57]、杜仲[58]、金银花[59]、薄荷[27]、罗勒[60]、姜黄[61]、黄芪[25]、石斛[62]、金丝桃[63]和姜[64]等。但是, 目前关于药用植物miRNA功能的研究大部分仅停留在miRNA的发现和靶基因预测阶段, 关于miRNA的生物学功能研究较少。
Verma等[54]利用小蔓长春花(Vinca minor) 转录组数据鉴定了37个miRNA并进行了靶基因预测, 靶基因功能分析显示这些miRNA与小蔓长春花的生长发育和其有效成分萜类吲哚类生物碱的次生代谢高度相关。
基于杜仲(Eucommia ulmoides) 的基因组数据, Wang等[58]预测到33个保守的和92个新的miRNA及相应的202个和306个潜在靶基因, 这些靶基因主要是富集于代谢途径和次级代谢产物的生物合成过程中的转录因子和功能基因; 对miRNA和预测的靶基因的转录表达特性及杜仲Eu-rubber产物的积累模式分析发现, 6个miRNA (Eu-miR14、Eu-miR91、miR162a、miR166a、miR172c和miR396a) 与靶基因和目标化合物积累具有负相关性, 由此推测这些miRNA可能是杜仲Eu-rubber产物生物合成潜在的调控因子。
在转录组高通量测序基础上, Singh等[27]使用C-mii (http://www.biotec.or.th/isl/c-mii) 计算方法从薄荷(Mentha spp) 中鉴定出11个miRNA家族, 包括5个唇形科中首次报道的miRNA家族, 并预测到8个miRNA家族的130个不同的靶基因, 这些靶基因涉及发育与繁殖、信号传导、刺激响应及代谢过程。结合KAAS (KEGG automatic annotation server, http://www.genome.jp/tools/kaas) 在线注释及以往的研究结果, 推测薄荷家族中miR5021、miR414和miR156可能通过调控萜类骨架生物合成途径包括2-C-甲基-D-赤藓糖醇4-磷酸途径(MEP) 和1-脱氧-D-木酮糖5-磷酸途径(DOXP) 中酶的表达来控制薄荷精油的生物合成, 也预测到miR156、miR5021和miR5015b也参与毛状体发育。
Patel等[60]采用基于同源搜索的计算方法, 从NCBI中已发布的罗勒(Ocimum basilicum) EST数据中鉴定到10个可能的miRNA, 分属于8个miRNA家族, 接着以人类转录组为靶基因库, 用psRNAtarget预测对应的靶基因, 结果显示其共靶向87个人类基因, 其中56个基因与人类疾病及代谢相关。
温度是影响黄芪(Astragalus membranaceus) 产量和质量的重要因素[25]。Abla等[25]首次通过高通量测序鉴定得到黄芪中168个保守和14个非保守的miRNA, 并对这些miRNA进行了靶基因预测, 同时还分析了低温和正常温度下黄芪miRNA表达特征, 并通过荧光定量PCR鉴定了具有显著低温响应的部分miRNA及靶基因的表达情况, 发现同一家族的miRNA在低温下通常具有相同的表达模式。
金丝桃属Hypericum (Hypericaceae) 植物在世界各地广泛分布, 它的主要药用成分金丝桃素在肿瘤和自身免疫性疾病的光敏治疗和诊断中得到了广泛的应用[63]。Petijová等[63]用5个典型金丝桃属植物的叶子进行转录组测序, 通过计算机预测其miRNA和对应的靶基因, 并进行了miRNA-mRNA交互分析, 共鉴定得到881个miRNA家族及对应的靶基因, 并发现了一些新的miRNA-mRNA相互作用, 如miR1171可能在褪黑激素的生物合成中发挥调控作用。
生姜(Zingiber officinale) 和芒果姜(Curcuma amada) 同属于姜科(Zingiberaceae), 后者对青枯菌(Ralstonia solanacearum) 具有较好的抗性[64]。为了研究miRNA在应对青枯病中的作用, Snigdha等[64]利用前期已有的转录组数据分别分析了感染了青枯菌和未感染的生姜及芒果姜中的差异表达基因, 共鉴定得到4 736和4 485个差异基因, 接着运用psRNATarget预测生姜和芒果姜中miRNA对应的靶基因(miRTG), 并用Cytoscape 3.7.1构建miRNA-miRTG调控网络, 最终通过荧光定量PCR, 确定了9对miRNA-miRTG可能是姜科植物参与响应青枯菌感染的调控因子。
2.2 药用植物miRNA调控具有药用活性的次生代谢产物合成药用植物一般通过药用活性成分发挥临床疗效, 其活性成分大多是次生代谢产物, 比如丹参中的丹参酮、红豆杉中的紫杉醇和人参中的人参皂苷等。植物次生代谢产物是相对于初生代谢产物的概念, 指植物在特定的时期或者特定的阶段, 在一定的或者特殊的环境条件, 对初生代谢产物进行结构修饰、合成或分解而来的衍生物, 包括萜类、生物碱、酚类、皂苷类和黄酮类等化合物[65]。近年来研究发现, miRNA是植物次生代谢的重要调控因子[18, 66, 67], 且miRNA在药用植物次生代谢中的调控作用非常广泛(图 3), 它可以通过控制基因(结构基因或者调控基因) 的表达直接或间接影响次生代谢产物的生成。
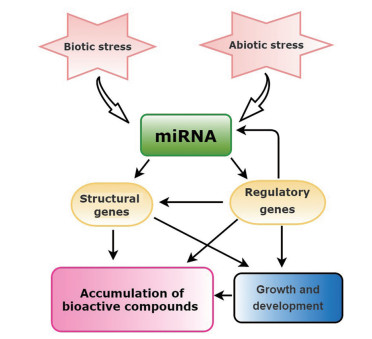
|
Figure 3 The regulatory roles of miRNA in plant secondary metabolism of medicinal plants |
当前, 有关miRNA调控药用植物中具有生物活性次生代谢产物合成(表 1) 的报道还较少, 其中主要是miRNA调控萜类次生代谢产物。萜类化合物是指由两个或两个以上异戊二烯结构单元头尾相连构成的天然产物[68], 在自然界中广泛存在, 几乎存在于所有植物中, 很多都具有较高的药用价值, 临床上应用广泛。
| Table 1 miRNA regulates the synthesis of bioactive compounds in medicinal plants |
青蒿素属于倍半萜类化合物, 是青蒿(Artemisia annua) 的药效成分, 具有非常好的抗疟作用[69]。Khan等[70]利用小RNA测序技术鉴定了青蒿叶子中80个保守和41个新的miRNA, 靶基因预测显示很多miRNA靶向与青蒿素合成相关的关键酶基因和转录因子, 荧光定量PCR验证结果显示miR858在青蒿生长的不同时期和青蒿素合成关键酶基因紫穗槐-4, 11-二烯合酶(amorpha-4, 11-diene synthase, ADS) 及青蒿素合成相关基因MYB1的表达水平呈负相关, 表明青蒿miR858可能通过抑制ADS和MYB1的表达调控青蒿素的合成[71]。
紫杉醇为二萜化合物, 由红豆杉属植物产生, 具有非常好的抗癌活性[72]。Hao等[73]通过南方红豆杉(Taxus mairei) 降解组测序发现2个紫杉醇生物合成途径中的关键酶基因—紫杉烷13α-羟基化酶(taxane 13α-hydroxylase) 和紫杉二烯2α-O-苯甲酰转移酶(taxane 2α-O-benzoyltransferase) 基因, 分别被miR164和miR171切割, 表明miR164和miR171可能参与了紫杉醇的生物合成。Zhang等[74]通过高通量测序揭示miRNA对长期继代培养的红豆杉(Taxus chinensis) 细胞中初生和次生代谢产物生成的影响, 进一步研究发现在红豆杉细胞中过表达miR8154和miR5298可以上调编码紫杉醇、苯丙烷和黄酮类化合物的关键酶基因的表达水平, 表明miR8154和miR5298可能和紫杉醇的合成相关。
丹参酮为二萜类化合物, 是丹参(Salvia miltiorrhiza) 的主要活性成分之一, 临床上广泛用于心脑血管疾病的治疗[75]。Xu等[56]在高通量测序获得的丹参452个保守和40个新的miRNA基础上, 通过降解组测序鉴定了25个miRNA的69个潜在靶基因。其中, 参与丹参酮生物合成的乙酰辅酶A酰基转移酶(acetyl-CoA C-acetyltransferase, AACT) 被miR5072剪切[56]。丹参酮主要由甲羟戊酸(MVA) 途径和丙酮酸/磷酸甘油醛(DXP) 途径合成, 乙酰辅酶A酰基转移酶在MVA途径起始阶段发挥重要作用[76], 由此推测miR5072可能参与丹参酮的生物合成。Zhang[77]为了研究丹参中miRNA与连作障碍及丹参次生代谢产物合成的关系, 通过高通量测序建立了丹参连作和非连作2个miRNA库, 筛选出差异表达miRNA共41个, 同时对丹参酮和丹酚酸含量进行了检测, 推测miR164a和miR160a可能与丹参连作障碍及丹参次生代谢产物的生物合成相关。进一步研究发现, miR164a过表达毛状根中丹参酮IIA含量降低, 丹酚酸B的含量升高, 研究表明miR164a可能通过对靶基因NAC2和NAC3的调控参与丹参酮和丹酚酸的生物合成, 其中NAC3可能发挥了主要作用[77]。由于没有丹参MIR160a基因序列信息, Zhang等[78]通过过表达成熟序列相同的拟南芥pre-MIR160b研究其功能, 联合转录组和降解组数据, 鉴定了miR160a的三个靶基因SmARF10、SmARF16和SmARF17。过表达miR160a丹参毛状根中丹参酮含量降低, 荧光定量PCR结果显示SmARF10、SmARF16和SmARF17的表达水平也显著降低, 并且丹参酮合成途径中一些关键酶基因如AACT1、DXS3及KSL2等基因的表达水平也显著降低, 表明丹参miR160a和丹参酮的生物合成相关[78]。本课题组通过小RNA高通量测序鉴定了丹参中152个保守的miRNA, 采用降解组测序和5′RLM-RACE发现miR396b靶向SmGRF1-7、SmHDT1和SmMYB37/4等13个靶基因, 转录组测序、小RNA测序及荧光定量PCR结果证实miR396b靶向SmGRFs、SmHDT1和SmMYB37/4介导植物激素信号通路, 特别是赤霉素信号通路, 而miR396b过表达毛状根中丹参酮含量升高, 丹酚酸含量下调, 表明miR396b可能通过激素信号通路参与丹参酮和丹酚酸的生物合成[79]。
胡黄连苷-I是胡黄连(Picrorhiza kurroa) 中环烯醚萜苷类的重要活性成分, 具有抗炎和抗肿瘤的功效[28]。Vashisht等[28]在胡黄连中预测到18个保守的miRNA及其30个靶基因, 5′RLM-RACE和荧光定量PCR结果显示miR-4995靶向苯丙烷代谢途径中的一个关键酶基因3-脱氧-7-磷庚酸合酶(3-deoxy-7-phosphoheptulonate synthase), 该酶是苯丙烷代谢途径中决定肉桂酸含量的第一个关键酶, 可以调控胡黄连苷-I生物合成, 由此表明miR-4995是胡黄连苷-I生物合成的重要调控因子。
萜类内酯是银杏叶(Ginkgo biloba) 提取物的主要活性成分, 对心脑血管疾病具有很好的疗效[80]。然而萜类内脂在银杏叶中含量很低, 难以满足市场需求, 并且它的化学结构复杂, 很难通过化学途径合成。Ye等[80]通过高通量测序技术鉴定了银杏中1 197个保守的miRNA和2 117个新的miRNA, 结合5′RLM-RACE和荧光定量PCR, 证明9个miRNA靶向萜类内脂生物合成途径的多个结构基因(AACT、HMGS、HMGR和MK等), 可能参与银杏叶中萜类内脂的合成; 此外, 还有25个miRNA靶向萜类内脂生物合成的候选调控基因(bHLH、WRKY和AP2), 可能在萜类内脂生物合成中发挥重要调控作用。
人参皂苷属于三萜苷类, 是人参(Panax ginseng C. A. Meyer) 的主要活性成分, 是评价人参药材质量的重要指标, 具有抗氧化、抗炎、抗肿瘤等功效[81]。低温下, 人参皂苷含量增加[81]。对低温和常温下的人参miRNA进行测序, 共鉴定得到482个新的miRNA和265个保守的miRNA, 此外, 鉴定出差异表达miRNA共16个, 荧光定量PCR结果显示pgi-MIR6135h-p3_1ss22TC和pgi-MIR6135d-p3_1ss23CT与预测的靶基因DNA-3-甲基腺嘌呤糖基化酶(DNA-3-methyladenine glycosylase) 的表达量呈负相关, 表明低温条件下pgi-MIR6135h-p3_1ss22TC和pgi-MIR6135d-p3_1ss23CT可能通过降解DNA-3-甲基腺嘌呤糖基化酶调控人参皂苷合成[81]。
三七皂苷也是典型的萜类化合物, 是三七(Panax notoginseng) 的主要活性成分, 对血液系统、心血管系统及神经系统疾病都具有较好的疗效[82]。研究发现, 干旱胁迫后三七叶子和根中总皂苷含量显著增加, 对干旱胁迫后的转录组和miRNA表达进行分析, 发现4对表达量呈显著负相关且与萜类合成相关的miRNA-靶基因对(MIR156aaai-GGPS、MIR482g-ispF、t15589037-ispH和t49562107-HMGCS), 接着5′RLM-RACE证明t49562107剪切萜类骨架合成基因HMGCS参与三七皂苷的合成[83]。
广藿香油是广藿香(Pogostemon cablin) 的主要药用成分, 主要包括单萜、倍半萜和小分子醇等次生代谢产物, 具有免疫调节、抗炎、抗氧化等功效[84]。miR156是植物中高度保守的miRNA, 可以靶向SPL转录因子影响植物营养阶段的转变和开花[85, 86]。Yu等[87]研究表明广藿香中miR156靶向SPL转录因子间接调控负责广藿香油生物合成的关键倍半萜合成酶基因PatPTS的表达, 最终影响广藿香油的生物合成。
类胡萝卜素是枸杞(Lycium barbarum) 中的主要有效成分之一, 属四萜类, 具有抗氧化和视觉保护的功效[88]。Zeng等[89]通过高通量测序鉴定了枸杞中50个新的miRNA和38个保守的miRNA, 结合降解组测序与荧光定量PCR, 发现miR156靶向1-脱氧-D-木糖-5-磷酸酯合成酶(1-deoxy-D-xylulose-5-phosphate synthase, DXS) 参与枸杞中类胡萝卜素合成。
除了萜类外, miRNA也可调控多酚类、生物碱和木脂素等次生代谢产物的合成。
丹酚酸B是丹参中有效成分之一, 具有抗氧化、活血化瘀和通经活络等功效[90]。研究表明, 丹参多酚氧化酶基因(SmPPOs) 可以正向调控丹酚酸B的生物合成[91]。Li等[92]通过降解组测序发现smi-miR12112可以剪切丹参中15个SmPPOs基因, 表明miR12112可能和丹酚酸B的合成相关。研究表明Sm-miR858过表达植株中Sm-miR858通过负调控靶基因PAP1的表达, 抑制丹酚酸B和迷迭香酸的合成, 表明Sm-miR858对丹参酚酸类成分合成具有重要的调控作用[93, 94]。Zou等[95]通过5′RLM-RACE和荧光定量PCR证明丹参中miR408靶向SmLAC3, 过表达SmLAC3和抑制miR408后丹参植株中丹酚酸B和迷迭香酸含量均明显提高, 表明miR408靶向SmLAC3调控酚酸类的生物合成。
萜类吲哚生物碱是长春花(Catharanthus roseus) 的主要活性成分, 如长春碱和长春新碱, 具有很好的抗肿瘤作用[96]。Shen等[96]运用小RNA测序技术鉴定了长春花中181个保守的和173个新的miRNA, 靶基因预测结果显示这些miRNA共有519个潜在的靶基因。PPM-RACE实验表明miR160可以剪切靶基因CrARF16, 而CrARF16可以结合到萜类吲哚生物碱合成途径关键酶基因(TDC、G10H和STR) 的启动子上抑制其表达, 表明miR160可能参与了长春花中萜类吲哚生物碱的合成[96]。
苄基异喹啉生物碱是药用植物罂粟(Papaver somniferum) 的主要活性产物之一[97]。Boke等[97]研究表明罂粟中pso-miR13、pso-miR2161和pso-miR408分别靶向编码7-O-甲基转移酶(7-O-methyltransferase, 7-OMT)、4'-O-甲基转移酶(4'-O-methyltransferase, 4-OMT) 和与FAD结合且含BBE结构域蛋白的基因(a gene encoding FAD-binding and BBE domain-containing protein), 可能参与BIAs的合成。
脂肪酸是金银花(Lonicera japonica) 的主要活性成分之一[59], 不同产地之间有效成分含量的高低往往导致了疗效的差异。鉴于miRNA在次生代谢中发挥的重要作用, Liu等[59]利用小RNA高通量测序和直接克隆技术鉴定两个产地来源的3个金银花样品的差异表达miRNA。结果显示, 在3个样本中, 共有37个miRNA在两个地点表达差异。5′RLM-RACE和荧光定量PCR结果表示U436803、U977315、U805963、U3938865和U4351355分别靶向LACS、ACP、FAH、MYB34和MYB34, 可能参与脂肪酸生成。
鬼臼毒素是桃儿七(Podophyllum hexandrum) 的主要活性成分, 属于木脂素类, 可用于制备抗癌药物, 在原植物中含量仅占0.3%左右[98]。Kumar等[98]把桃儿七转录组数据和植物microRNA数据库PMRD (Plant microRNA database) 进行Blast比对, 首次获得桃儿七潜在miRNA共279个并选取其中6个miRNA进一步进行靶基因预测及功能分析, 荧光定量PCR验证显示miR396b、miR2673a、miR828b和miR2910表达水平和它们的靶基因(UDP糖基转移酶、WRKY37和MYB F1、磷酸烯醇丙酮酸羧化酶、苹果酸脱氢酶) 呈负相关, 这些靶基因通过三羧酸循环、糖酵解、莽草酸途径和苯丙烷途径不同程度地参与了鬼臼毒素的生成, 表明这4个miRNA可能参与调控鬼臼毒素的生物合成。
3 展望我国药用植物资源丰富, 其有效成分多为植物次生代谢产物, 药用植物中有效成分含量是衡量药用植物品质优劣的重要性状之一, 但在野生药用植物体内含量往往很低。目前对药用植物有效成分的调控研究大多集中在合成途径上的关键酶基因及转录因子上, 也取得了一定成效。现有药用植物miRNA研究结果表明, 除参与调控生长发育等生理活动外, miRNA还可以通过靶向结构基因或调控基因调控植物体内萜类、苯丙烷类、生物碱等次生代谢产物的生物合成与积累[24, 99, 100]。因此, 对miRNA的调控将对药用植物优良品质形成具有至关重要的意义。另一方面, 利用功能明确的miRNA作为基因工程工具, 用以调控目标化合物在药用植物中的生物合成过程[66], 提高目标化合物在药用植物中的产量, 也是药用植物资源可持续发展的重要途径。
Liao等[101]构建了全球药典草药基因组数据库(Global Pharmacopoeia Genome Database), 该数据库(http://www.gpgenome.com) 将全球多地草药基因组数据统一整合, 并提供不同层面的草药基因组学相关数据及分析工具, 将为药用植物miRNA的发现和功能研究提供坚实的基础。此外, Yu等[51]构建了药用植物miRNA数据库MepmiRDB (medicinal plant microRNA database), 包含了29种药用植物miRNA (http://mepmirdb.cn/mepmirdb/index.html), 为药用植物miRNA的序列、生物学功能和调控机制研究提供了一定的基础。目前, 虽然人们对一些药用植物miRNA及其靶基因有了一定的了解, 但是大部分药用植物miRNA的研究停留在miRNA的发现及其靶基因预测阶段, 对miRNA的生物学功能研究相对较少, 并且已有的功能研究多集中在miRNA参与药用植物的生长发育和胁迫等方面, 对miRNA参与药用植物次生代谢的研究相对较少。此外, 对部分功能已知的miRNA在药用植物体内具体的调控机制和参与的调控网络还不完全明了。未来需要综合运用组学、生物信息学、分子生物学等技术手段, 对药用植物miRNA开展更深入的研究, 才能在阐明miRNA生物学功能基础上, 使其成为调控药用植物次生代谢产物生物合成、调控药用植物优良品质性状的有力工具。今后对miRNA调控药用植物次生代谢的研究可以从以下几方面进行: ①进一步挖掘药用植物miRNA及其靶基因, 完善药用植物miRNA数据库。②通过实验明确miRNA的靶基因, 通过生物学功能研究掌握miRNA参与药用植物性状和次生代谢的调控机制和调控网络。③重点研究miRNA和已有明确临床应用的药用植物有效成分的调控关系, 着力解决临床应用问题。④结合分子鉴定技术手段, 筛选出具有特异性表达的miRNA作为药用植物产地、品种与用药部位等性状鉴别的候选标记[23]。
作者贡献: 本文主要由郑小宇负责撰写, 陈敏与李航负责修改, 赵淑娟负责对论文进行整体指导和修改。
利益冲突: 所有作者无任何利益冲突。
| [1] |
Pashkovskiy PP, Ryazansky SS. Biogenesis, evolution, and functions of plant microRNAs[J]. Biochemistry (Mosc), 2013, 78: 627-637. DOI:10.1134/S0006297913060084 |
| [2] |
Flynt AS, Lai EC. Biological principles of microRNA-mediated regulation: shared themes amid diversity[J]. Nat Rev Genet, 2008, 9: 831-842. |
| [3] |
Lee RC, Feinbaum RL, Ambros V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14.[J]. Cell, 1993, 75: 843-854. DOI:10.1016/0092-8674(93)90529-Y |
| [4] |
Wightman B, Ha I, Ruvkun G. Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans[J]. Cell, 1993, 75: 855-862. DOI:10.1016/0092-8674(93)90530-4 |
| [5] |
Llave C. Endogenous and silencing-associated small RNAs in plants[J]. Plant Cell Online, 2002, 14: 1605-1619. DOI:10.1105/tpc.003210 |
| [6] |
Kozomara A, Birgaoanu M, Griffiths-Jones S. MiRBase: from microRNA sequences to function[J]. Nucleic Acids Res, 2019, 47: 155-162. DOI:10.1093/nar/gky1141 |
| [7] |
Lee Y, Kim M, Han J, et al. MicroRNA genes are transcribed by RNA polymerase Ⅱ[J]. EMBO J, 2004, 23: 4051-4060. DOI:10.1038/sj.emboj.7600385 |
| [8] |
Wang JL, Mei J, Ren GD. Plant microRNAs: biogenesis, homeostasis, and degradation[J]. Front Plant Sci, 2019, 10: 360. DOI:10.3389/fpls.2019.00360 |
| [9] |
Jones-Rhoades MW, Bartel DP, Bartel B. MicroRNAS and their regulatory roles in plants[J]. Annu Rev Plant Biol, 2006, 57: 19-53. DOI:10.1146/annurev.arplant.57.032905.105218 |
| [10] |
Yu B, Yang ZY, Li JJ, et al. Methylation as a crucial step in plant microRNA biogenesis[J]. Science, 2005, 307: 932-935. DOI:10.1126/science.1107130 |
| [11] |
Bologna NG, Iselin R, Abriata LA, et al. Nucleo-cytosolic shuttling of ARGONAUTE1 prompts a revised model of the plant microRNA pathway[J]. Mol Cell, 2018, 69: 709-719. DOI:10.1016/j.molcel.2018.01.007 |
| [12] |
Yu Y, Jia TR, Chen XM. The 'how' and 'where' of plant microRNAs[J]. New Phytol, 2017, 216: 1002-1017. DOI:10.1111/nph.14834 |
| [13] |
Yu Y, Zhang YC, Chen XM, et al. Plant noncoding RNAs: hidden players in development and stress responses[J]. Annu Rev Cell Dev Biol, 2019, 35: 407-431. DOI:10.1146/annurev-cellbio-100818-125218 |
| [14] |
Yuan SR, Zhao JM, Li ZG, et al. MicroRNA396-mediated alteration in plant development and salinity stress response in creeping bentgrass[J]. Hortic Res, 2019, 6: 48. DOI:10.1038/s41438-019-0130-x |
| [15] |
Curaba J, Singh MB, Bhalla PL. MiRNAs in the crosstalk between phytohormone signalling pathways[J]. J Exp Bot, 2014, 65: 1425-1438. DOI:10.1093/jxb/eru002 |
| [16] |
Jiang N, Meng J, Cui J, et al. Function identification of miR482b, a negative regulator during tomato resistance to Phytophthora infestans[J]. Hortic Res, 2018, 5: 9. DOI:10.1038/s41438-018-0017-2 |
| [17] |
Tirumalai V, Swetha C, Nair A, et al. miR828 and miR858 regulate VvMYB114 to promote anthocyanin and flavonol accumulation in grapes[J]. J Exp Bot, 2019, 70: 4775-4791. DOI:10.1093/jxb/erz264 |
| [18] |
Li CL, Wang MZ, Qiu XX, et al. Noncoding RNAs in medicinal plants and their regulatory roles in bioactive compound production[J]. Curr Pharm Biotechnol, 2020, 22: 341-359. |
| [19] |
Ding J, Ruan CJ, Guan Y, et al. Identification of microRNAs involved in lipid biosynthesis and seed size in developing sea buckthorn seeds using high-throughput sequencing[J]. Sci Rep, 2018, 8: 1-15. |
| [20] |
Zhu FJ, Liu J, Yuan Y, et al. Expression analysis of Hsp70-related miRNA in Lonicera japonica Thunb. during different flowering stages[J]. Acta Pharm Sin (药学学报), 2018, 53: 1202-1208. |
| [21] |
Tian XM, Zhang J, Rong H, et al. Advances in mechanism and application for plant microRNA in cross-kingdom regulation[J]. Acta Pharm Sin (药学学报), 2020, 55: 1137-1146. |
| [22] |
Zhu FJ, Liu J, Yu NJ, et al. Progress on miRNA-based secondary metabolism regulation in medicinal plants[J]. Chem Life (生命的化学), 2017, 37: 1056-1064. |
| [23] |
Lu BW, An FX, Yang YJ, et al. MiRNA and secondary metabolism of medicinal plants[J]. Plant Physiol J (植物生理学报), 2019, 55: 569-576. |
| [24] |
Lu Y, Gao W, Huang LQ. Synthesis and regulation of secondary metabolites by microRNA in medicinal plants[J]. China J Chin Meter Med (中国中药杂志), 2018, 43: 1806-1811. |
| [25] |
Abla M, Sun HG, Li ZY, et al. Identification of miRNAs and their response to cold stress in astragalus membranaceus[J]. Biomolecules, 2019, 9: 182. DOI:10.3390/biom9050182 |
| [26] |
Najafabadi AS, Naghavi MR. Mining Ferula gummosa transcriptome to identify miRNAs involved in the regulation and biosynthesis of terpenes[J]. Gene, 2018, 645: 41-47. DOI:10.1016/j.gene.2017.12.035 |
| [27] |
Singh N, Srivastava S, Shasany AK, et al. Identification of miRNAs and their targets involved in the secondary metabolic pathways of Mentha spp.[J]. Comput Biol Chem, 2016, 64: 154-162. DOI:10.1016/j.compbiolchem.2016.06.004 |
| [28] |
Vashisht I, Mishra P, Pal T, et al. Mining NGS transcriptomes for miRNAs and dissecting their role in regulating growth, development, and secondary metabolites production in different organs of a medicinal herb, Picrorhiza kurroa[J]. Planta, 2015, 241: 1255-1268. DOI:10.1007/s00425-015-2255-y |
| [29] |
Zinati Z, Shamloo-Dashtpagerdi R, Behpouri A. In silico identification of miRNAs and their target genes and analysis of gene co-expression network in saffron (Crocus sativus L.) stigma.[J]. Mol Biol Res Commun, 2016, 5: 233-246. |
| [30] |
Wei RC, Qiu DY, Wilson IW, et al. Identification of novel and conserved microRNAs in Panax notoginseng roots by high-throughput sequencing[J]. BMC Genom, 2015, 16: 1-10. DOI:10.1186/1471-2164-16-1 |
| [31] |
Dai XB, Zhuang, Zhao PX. PsRNATarget: a plant small RNA target analysis server[J]. Nucleic Acids Res, 2018, 46: 49-54. |
| [32] |
Numnark S, Mhuantong W, Ingsriswang S, et al. C-mii: a tool for plant miRNA and target identification[J]. BMC Genom, 2012, 13: S16. |
| [33] |
Bo XC, Wang SQ. TargetFinder: a software for antisense oligonucleotide target site selection based on MAST and secondary structures of target mRNA[J]. Bioinformatics, 2005, 21: 1401-1402. DOI:10.1093/bioinformatics/bti211 |
| [34] |
Wu HJ, Ma YK, Chen T, et al. PsRobot: a web-based plant small RNA meta-analysis toolbox[J]. Nucleic Acids Res, 2012, 40: 22-28. |
| [35] |
Song CN, Yu ML, Han J, et al. Validation and characterization of Citrus sinensis microRNAs and their target genes[J]. BMC Res Notes, 2012, 5: 235. DOI:10.1186/1756-0500-5-235 |
| [36] |
Wang SS, Liu S, Liu L, et al. miR477 targets the phenylalanine ammonia-lyase gene and enhances the susceptibility of the tea plant (Camellia sinensis) to disease during Pseudopestalotiopsis species infection[J]. Planta, 2020, 251: 59. DOI:10.1007/s00425-020-03353-x |
| [37] |
German MA, Pillay M, Jeong DH, et al. Global identification of microRNA-target RNA pairs by parallel analysis of RNA ends[J]. Nat Biotechnol, 2008, 26: 941-946. DOI:10.1038/nbt1417 |
| [38] |
Liu QK, Wang F, Axtell MJ. Analysis of complementarity requirements for plant microRNA targeting using a Nicotiana benthamiana quantitative transient assay[J]. Plant Cell, 2014, 26: 741-753. DOI:10.1105/tpc.113.120972 |
| [39] |
Garg V, Khan AW, Kudapa H, et al. Integrated transcriptome, small RNA and degradome sequencing approaches provide insights into Ascochyta blight resistance in chickpea[J]. Plant Biotechnol J, 2019, 7: 914-931. |
| [40] |
Shao JJ, Wang LQ, Liu Y, et al. Identification of miRNAs and their target genes in Ganoderma lucidum by high-throughput sequencing and degradome analysis[J]. Fungal Genet Biol, 2020, 136: 103313. DOI:10.1016/j.fgb.2019.103313 |
| [41] |
Llave C, Xie ZX, Kasschau KD, et al. Cleavage of scarecrow-likemRNA targets directed by a class of Arabidopsis miRNA[J]. Science, 2002, 297: 2053-2056. DOI:10.1126/science.1076311 |
| [42] |
Hu J, Cao Y, Zhu X, et al. Rapid validation of target rice miRNAs genes in transient expression system[J]. Biotechnol Bull (生物技术通报), 2019, 35: 57-63. |
| [43] |
Mao G, Turner M, Yu O, et al. miR393 and miR164 influence indeterminate but not determinate nodule development[J]. Plant Signal Behav, 2013, 8: e26753. DOI:10.4161/psb.26753 |
| [44] |
Debernardi JM, Rodriguez RE, Mecchia MA, et al. Functional specialization of the plant miR396 regulatory network through distinct microRNA-target interactions[J]. PLoS Genet, 2012, 8: e1002419. DOI:10.1371/journal.pgen.1002419 |
| [45] |
Jin WB, Wu FL. Identification and characterization of cucumber microRNAs in response to Pseudoperonospora cubensis infection[J]. Gene, 2015, 569: 225-232. DOI:10.1016/j.gene.2015.05.064 |
| [46] |
Franco-Zorrilla JM, Valli A, Todesco M, et al. Target mimicry provides a new mechanism for regulation of microRNA activity[J]. Nat Genet, 2007, 39: 1033-1037. DOI:10.1038/ng2079 |
| [47] |
Todesco M, Rubio-Somoza I, Paz-Ares J, et al. A collection of target mimics for comprehensive analysis of microRNA function in Arabidopsis thaliana[J]. PLoS Genet, 2010, 6: e1001031. DOI:10.1371/journal.pgen.1001031 |
| [48] |
Li XP, Ma XC, Wang H, et al. Osa-miR162a fine-tunes rice resistance to Magnaporthe oryzae and yield[J]. Rice, 2020, 13: 38. DOI:10.1186/s12284-020-00396-2 |
| [49] |
Yan J, Gu YY, Jia XY, et al. Effective small RNA destruction by the expression of a short tandem target mimic in Arabidopsis[J]. Plant Cell, 2012, 24: 415-427. DOI:10.1105/tpc.111.094144 |
| [50] |
Reichel M, Li YJ, Li JY, et al. Inhibiting plant microRNA activity: molecular SPONGEs, target MIMICs and STTMs all display variable efficacies against target microRNAs[J]. Plant Biotechnol J, 2015, 13: 915-926. DOI:10.1111/pbi.12327 |
| [51] |
Yu DL, Lu JJ, Shao WS, et al. MepmiRDB: a medicinal plant microRNA database[J]. Database, 2019, 2019: 1-6. |
| [52] |
Li CF, Zhu YJ, Guo X, et al. Transcriptome analysis reveals ginsenosides biosynthetic genes, microRNAs and simple sequence repeats in Panax ginseng C. A. Meyer[J]. BMC Genom, 2013, 14: 245. DOI:10.1186/1471-2164-14-245 |
| [53] |
Wang YF, Peng MY, Chen YL, et al. Analysis of Panax ginseng miRNAs and their target prediction based on high-throughput sequencing[J]. Planta Med, 2019, 85: 1168-1176. DOI:10.1055/a-0989-7302 |
| [54] |
Verma P, Singh N, Khan SA, et al. TIAs pathway genes and associated miRNA identification in Vinca minor: supporting aspidosperma and eburnamine alkaloids linkage via transcriptomic analysis[J]. Physiol Mol Biol Plants, 2020, 26: 1695-1711. DOI:10.1007/s12298-020-00842-x |
| [55] |
Zhang D, Li W, Xia EH, et al. The medicinal herb Panax notoginseng genome provides insights into ginsenoside biosynthesis and genome evolution[J]. Mol Plant, 2017, 10: 903-907. DOI:10.1016/j.molp.2017.02.011 |
| [56] |
Xu XB, Jiang QH, Ma XY, et al. Deep sequencing identifies tissue-specific microRNAs and their target genes involving in the biosynthesis of tanshinones in Salvia miltiorrhiza[J]. PLoS One, 2014, 9: e111679. DOI:10.1371/journal.pone.0111679 |
| [57] |
Lu SF. De novo origination of MIRNAs through generation of short inverted repeats in target genes[J]. RNA Biol, 2019, 16: 846-859. DOI:10.1080/15476286.2019.1593744 |
| [58] |
Wang L, Du HY, Wuyun TN. Genome-wide identification of microRNAs and their targets in the leaves and fruits of Eucommia ulmoides using high-throughput sequencing[J]. Front Plant Sci, 2016, 7: 1632. |
| [59] |
Liu J, Yuan Y, Wang YL, et al. Regulation of fatty acid and flavonoid biosynthesis by miRNAs in: Lonicera japonica[J]. RSC Adv, 2017, 7: 35426-35437. DOI:10.1039/C7RA05800D |
| [60] |
Patel M, Mangukia N, Jha N, et al. Computational identification of miRNA and their cross kingdom targets from expressed sequence tags of Ocimum basilicum[J]. Mol Biol Rep, 2019, 46: 2979-2995. DOI:10.1007/s11033-019-04759-x |
| [61] |
Singh N, Sharma A. Turmeric (Curcuma longa): miRNAs and their regulating targets are involved in development and secondary metabolite pathways[J]. C R Biol, 2017, 340: 481-491. DOI:10.1016/j.crvi.2017.09.009 |
| [62] |
Qian X, Zhu JY, Yuan QJ, et al. Illumina sequencing reveals conserved and novel microRNAs of Dendrobium nobile protocorm involved in synthesizing dendrobine, a potential nanodrug[J]. J Biomed Nanotechnol, 2021, 17: 416-425. DOI:10.1166/jbn.2021.3036 |
| [63] |
Petijová L, Jurčacková Z, Čellárová E. Computational screening of miRNAs and their targets in leaves of Hypericum spp. by transcriptome-mining: a pilot study[J]. Planta, 2020, 251: 49. DOI:10.1007/s00425-020-03342-0 |
| [64] |
Snigdha M, Prasath D. Transcriptomic analysis to reveal the differentially expressed miRNA targets and their miRNAs in response to Ralstonia solanacearum in ginger species[J]. BMC Plant Biol, 2021, 21: 355. DOI:10.1186/s12870-021-03108-0 |
| [65] |
Adjei MO, Zhou XZX, Mao MQ, et al. MicroRNAs roles in plants secondary metabolism[J]. Plant Signal Behav, 2021, 16: e1915590. DOI:10.1080/15592324.2021.1915590 |
| [66] |
Sabzehzari M, Naghavi MR. Phyto-miRNAs-based regulation of metabolites biosynthesis in medicinal plants[J]. Gene, 2019, 682: 13-24. DOI:10.1016/j.gene.2018.09.049 |
| [67] |
Gupta OP, Karkute SG, Banerjee S, et al. Contemporary understanding of miRNA-based regulation of secondary metabolites biosynthesis in plants[J]. Front Plant Sci, 2017, 8: 374. |
| [68] |
Lu X, Tang KX, Li P. Plant metabolic engineering strategies for the production of pharmaceutical terpenoids[J]. Front Plant Sci, 2016, 7: 1647. |
| [69] |
Wani KI, Choudhary S, Zehra A, et al. Enhancing artemisinin content in and delivery from Artemisia annua: a review of alternative, classical, and transgenic approaches[J]. Planta, 2021, 254: 29. DOI:10.1007/s00425-021-03676-3 |
| [70] |
Khan S, Ali A, Saifi M, et al. Identification and the potential involvement of miRNAs in the regulation of artemisinin biosynthesis in A. annua[J]. Sci Rep, 2020, 10: 13614. DOI:10.1038/s41598-020-69707-3 |
| [71] |
Matías-Hernández L, Jiang WM, Yang K, et al. AaMYB1 and its orthologue AtMYB61 affect terpene metabolism and trichome development in Artemisia annua and Arabidopsis thaliana[J]. Plant J, 2017, 90: 520-534. DOI:10.1111/tpj.13509 |
| [72] |
Lenka SK, Boutaoui N, Paulose B, et al. Identification and expression analysis of methyl jasmonate responsive ESTs in paclitaxel producing Taxus cuspidata suspension culture cells[J]. BMC Genom, 2012, 13: 148. DOI:10.1186/1471-2164-13-148 |
| [73] |
Hao DC, Yang L, Xiao PG, et al. Identification of Taxus microRNAs and their targets with high-throughput sequencing and degradome analysis[J]. Physiol Plant, 2012, 146: 388-403. DOI:10.1111/j.1399-3054.2012.01668.x |
| [74] |
Zhang M, Dong YS, Nie L, et al. High-throughput sequencing reveals miRNA effects on the primary and secondary production properties in long-term subcultured Taxus cells[J]. Front Plant Sci, 2015, 6: 604. |
| [75] |
Zhou LM, Zuo Z, Chow MSS. Danshen: an overview of its chemistry, pharmacology, pharmacokinetics, and clinical use[J]. J Clin Pharmacol, 2005, 45: 1345-1359. DOI:10.1177/0091270005282630 |
| [76] |
Lu S. Biosynthesis and regulatory mechanisms of bioactive compounds in Salvia miltiorrhiza, a model system for medicinal plant biology[J]. CRC Crit Rev Plant Sci, 2021, 40: 243-283. DOI:10.1080/07352689.2021.1935719 |
| [77] |
Zhang HH. The Studies on miRNA Responsing to Salvia miltiorrhiza Continuous Cropping Problem and Its Role in Secondary Metabolic Regulation (丹参miRNA对连作障碍的响应及其在次生代谢调控中的作用研究)[D]. Yangling: Northwest A & F University, 2019.
|
| [78] |
Zhang HH, Chen HM, Hou ZN, et al. Overexpression of Ath-MIR160b increased the biomass while reduced the content of tanshinones in Salvia miltiorrhiza hairy roots by targeting ARFs genes[J]. Plant Cell Tissue Organ Cult, 2020, 142: 327-338. DOI:10.1007/s11240-020-01865-8 |
| [79] |
Zheng XY, Li H, Chen M, et al. smi‑miR396b targeted SmGRFs, SmHDT1, and SmMYB37/4 synergistically regulates cell growth and active ingredient accumulation in Salvia miltiorrhiza hairy roots[J]. Plant Cell Rep, 2020, 39: 1263-1283. DOI:10.1007/s00299-020-02562-8 |
| [80] |
Ye JB, Zhang X, Tan JP, et al. Global identification of Ginkgo biloba microRNAs and insight into their role in metabolism regulatory network of terpene trilactones by high-throughput sequencing and degradome analysis[J]. Ind Crops Prod, 2020, 148: 112289. DOI:10.1016/j.indcrop.2020.112289 |
| [81] |
Zhang T. Physiological and Ecological Response Mechanism of Panax ginseng and Its Saponins Biosynthesis to Low Temperature (人参及其皂苷生物合成对低温的生理生态响应机制研究)[D]. Changchun: Jilin Agricultural University, 2019.
|
| [82] |
Ng TB. Pharmacological activity of sanchi ginseng (Panax notoginseng)[J]. J Pharm Pharmacol, 2006, 58: 1007-1019. |
| [83] |
Liao PR. Molecular Mechanisms of Drought Stress Affecting Saponins Synthesis Pathway of Panax notoginseng (干旱胁迫影响三七皂苷合成途径的分子机制研究)[D]. Kunming: Kunming University of Science and Technology, 2019.
|
| [84] |
Chen MW, Zhang JM, Lai YF, et al. Analysis of pogostemon cablin from pharmaceutical research to market performances[J]. Expert Opin Investig Drugs, 2013, 22: 245-257. DOI:10.1517/13543784.2013.754882 |
| [85] |
Wang JW, Czech B, Weigel D. miR156-regulated SPL transcription factors define an endogenous flowering pathway in Arabidopsis thaliana[J]. Cell, 2009, 138: 738-749. DOI:10.1016/j.cell.2009.06.014 |
| [86] |
Wu G, Park MY, Conway SR, et al. The sequential action of miR156 and miR172 regulates developmental timing in Arabidopsis[J]. Cell, 2009, 138: 750-759. DOI:10.1016/j.cell.2009.06.031 |
| [87] |
Yu ZX, Wang LJ, Zhao B, et al. Progressive regulation of sesquiterpene biosynthesis in Arabidopsis and patchouli (Pogostemon cablin) by the miR156-targeted SPL transcription factors[J]. Mol Plant, 2015, 8: 98-110. DOI:10.1016/j.molp.2014.11.002 |
| [88] |
Amagase H, Farnsworth NR. A review of botanical characteristics, phytochemistry, clinical relevance in efficacy and safety of Lycium barbarum fruit (Goji)[J]. Food Res Int, 2011, 44: 1702-1717. DOI:10.1016/j.foodres.2011.03.027 |
| [89] |
Zeng SH, Liu YL, Pan LZ, et al. Identification and characterization of miRNAs in ripening fruit of Lycium barbarum L. using high-throughput sequencing[J]. Front Plant Sci, 2015, 6: 778. |
| [90] |
Cao W, Guo XW, Zheng HZ, et al. Current progress of research on pharmacologic actions of salvianolic acid B[J]. Chin J Integr Med, 2012, 18: 316-320. DOI:10.1007/s11655-012-1052-8 |
| [91] |
Shi WK, Sun YY, Shu ZM, et al. Cloning of polyphenol oxidase gene from Salvia miltiorrhiza hairy roots and its expression analysis[J]. Acta Bot Boreali-occid Sin (西北植物学报), 2016, 36: 1735-1942. |
| [92] |
Li CL, Li DQ, Li J, et al. Characterization of the polyphenol oxidase gene family reveals a novel microRNA involved in posttranscriptional regulation of PPOs in Salvia miltiorrhiza[J]. Sci Rep, 2017, 7: 44622. DOI:10.1038/srep44622 |
| [93] |
Chen F, She TT, Zhang L, et al. Mechanism of Sm-miR858 negatively regulated a R2R3-MYB transcription factor SmPAP1 in Salvia miltiorrhiza[J]. Bull Bot Res (植物研究), 2021, 41: 522-530. |
| [94] |
Chen F, Feng MY, Wang J. Overexpression of a Sm-miR858 caused significant reduction of phenolic acids contents in transgenic Salvia miltiorrhiza Bunge[J]. Chin J Biochem Mol Biol (中国生物化学与分子生物学报), 2020, 36: 1235-1244. |
| [95] |
Zou HL, Guo XR, Yang R, et al. MiR408-SmLAC3 module participates in salvianolic acid b synthesis in salvia miltiorrhiza[J]. Int J Mol Sci, 2021, 22: 7541. DOI:10.3390/ijms22147541 |
| [96] |
Shen EM, Singh SK, Ghosh JS, et al. The miRNAome of Catharanthus roseus: identification, expression analysis, and potential roles of microRNAs in regulation of terpenoid indole alkaloid biosynthesis[J]. Sci Rep, 2017, 7: 43027. DOI:10.1038/srep43027 |
| [97] |
Boke H, Ozhuner E, Turktas M, et al. Regulation of the alkaloid biosynthesis by miRNA in Opium poppy[J]. Plant Biotechnol J, 2015, 13: 409-420. DOI:10.1111/pbi.12346 |
| [98] |
Kumar P, Padhan JK, Kumar A, et al. Transcriptomes of Podophyllum hexandrum unravel candidate miRNAs and their association with the biosynthesis of secondary metabolites[J]. J Plant Biochem Biotechnol, 2018, 27: 46-54. DOI:10.1007/s13562-017-0414-x |
| [99] |
Shi L, Guo YB, Shen Y. Regulation of growth, development and secondary metabolism of meicinal plnats by miRNA[J]. Chin J Biochem Mol Biol (中国生物化学与分子生物学报), 2019, 35: 361-370. |
| [100] |
Zhou FN, Bai ZC, Lu SF. MicroRNAs in medicinal plants[J]. Chin Tradit Herb Drugs (中草药), 2013, 44: 232-237. |
| [101] |
Liao BS, Hu HY, Xiao SM, et al. Global Pharmacopoeia Genome Database is an integrated and mineable genomic database for traditional medicines derived from eight international pharmacopoeias[J]. Sci China Life Sci, 2021. DOI:10.1007/s11427-021-1968-7 |
 2021, Vol. 56
2021, Vol. 56


