2. 甘肃中医药大学和政药用植物园, 甘肃 和政 731299
2. Gansu University of Traditional Chinese Medicine, Hezheng Medicinal Botanical Garden, Hezheng 731299, China
铁棒锤Aconitum pendulum隶属于毛茛科乌头属, 别名雪上一枝蒿、一只箭、三转半, 是我国西南、西北地区特有的多年生药用植物[1], 主要分布于陕西、西藏、青海、四川、宁夏等地区[2]。在藏医药中, 铁棒锤又称“榜那”, 块根有毒供药用, 主要用于治疗跌打损伤、骨折、风湿腰痛、关节炎、疖肿、肿瘤等症[3]。二萜生物碱是铁棒锤主要的药用活性成分, 兼具毒性作用。定量构效关系及毒理学研究证明二萜生物碱毒性与其骨架上C8位乙酰基、C14位苯甲酰基密切相关[4]。乌头碱类化合物显著的药效、剧毒性与其结构之间的关系, 是国内外学者研究的重要方向[2, 4-6]。
课题组前期开展了乌头属7种植物不同部位的代谢组学分析, 确定了铁棒锤中生物碱的数量及种类, 注意到铁棒锤与传统中药乌头Aconitum carmichaelii相比, 其体内生物碱的数量和种类明显减少[5-8], 乌头中鉴定出66个生物碱(包括C18、C19及C20型二萜生物碱), 而铁棒锤仅鉴定了14个生物碱(主要为C19和C20型二萜生物碱), 并以乌头碱和2-羟基乌头碱为其主要成分[7]。铁棒锤相对单一的化学组成为研究乌头碱类化合物生物合成途径的解析提供了新的方向。
目前二萜生物碱已有大量化学合成的报道[9-12], 但乌头属植物体内的生物合成途径研究却进展缓慢。推测的二萜生物碱生物合成途径如下: 首先细胞质中的甲羟戊酸途径(mevalonate pathway, MVA) 和位于质体中的甲基赤藓糖醇磷酸途径(methylerythritol 4-phosphate pathway, MEP) 构建二萜前体牻牛儿基牻牛儿基焦磷酸(geranylgeranyl pyrophosphate GGPP); GGPP经过Ⅱ型萜类合酶柯巴基焦磷酸合酶(copalyl-diphosphate synthase, CPS) 及Ⅰ型合酶类贝壳杉烯合酶(kaurene synthase-like, KSL) 催化, 进一步环化重排形成二萜骨架贝壳杉烷(kaurane) 及阿替生烷(atisane)[8-12]。二萜骨架在L-丝氨酸转氨酶的作用下形成C20二萜生物碱骨架阿替生(atisine)[13], 在此基础上经过Wagnere-Meerwein重排、环化形成C19、C18二萜生物碱骨架; 细胞色素P450、BAHD[13]酰基转移酶(其命名是由该家族中最早完成生化功能鉴定的4类酶的英文首字母组合形成) 等蛋白家族进行结构后修饰, 催化形成结构多样的二萜生物碱[8-15]。
随着转录组测序技术的快速发展, 高通量测序技术广泛应用于药用植物次生代谢物生物合成及功能基因研究。现已报道了乌头A. carmichaeli、异叶乌头A. heterophyllum、黄草乌A. vilmorinianum等乌头属植物的转录组特征, 预测了大量参与二萜生物碱生物合成的候选基因[14-22], 为阐明药材品质形成奠定了重要基础。毛柳英等[23]利用三代全长转录组测序技术筛选了乌头中萜类合酶基因家族并进行功能鉴定, 从转录组水平验证了C18和C19型二萜生物碱来源于C20型贝壳杉烷及阿替生烷的推测。但由于缺乏体内实验证据, 还需要更多乌头属植物转录组信息和相关基因的发掘。为了进一步丰富乌头属植物的基因资源, 科学阐述铁棒锤品质形成的内在机制。本研究利用Illumina高通量测序技术, 首次开展铁棒锤根、叶及花的转录组学研究, 挖掘20种56个乌头碱生物合成关键酶基因, 为后续开展铁棒锤二萜生物碱生物合成途径的解析及比较功能基因组学提供了基因资源及参考数据。
材料与方法植物材料 铁棒锤植株于2019年8月7号采自甘肃中医药大学和政药用植物园, 将其带回中国中医科学院中药资源中心后, 立即将根、叶和花置于液氮中速冻。每个样品两个生物学重复, 用于总RNA提取与文库构建。样品经甘肃中医药大学杜弢教授鉴定为铁棒锤Aconitum pendulum。
RNA提取与文库构建 利用华越洋植物RNA提取试剂盒分别提取铁棒锤花、叶和根的总RNA (中国北京华越洋生物科技有限公司), 操作步骤严格按照说明书进行。采用琼脂糖凝胶电泳、NanoPhotometer spectrophotometer、Agilent 2100 bioanalyzer检测总RNA的纯度及完整性。用带有Oligo (dT) 的磁珠分离mRNA, 加入NEB buffer进行短片段打散, 以片段化的mRNA为模板, 利用随机寡核苷酸和逆转录酶合成双链cNDA。双链cDNA经过纯化、末端修复、加poly (A) 尾、连接测序接头, 进一步PCR扩增得到cDNA文库。Qubit2.0 FluorometerAgilent、2100 bioanalyzer对文库的浓度及大小进行初步定量检测, 质检合格后采用Illumina HiSeqTM2000测序平台进行上机测序。
转录组测序分析及功能注释 原始序列(raw reads) 过滤去除含接头(adaptor)、含N (无法确定碱基的reads) 及低质量reads (Qphred ≤ 20的碱基数占整个read长度的50%以上的reads), 得到高质量序列(clean reads)。由于铁棒锤基因组测序尚未完成, 缺少参考序列, 因此使用Trinity软件对clean reads从头组装(de novo assembly) 成转录本(transcript), 对转录本去冗余, 以最长cluster序列作为非重复序列基因(unigene)。
使用diamond v0.8.22软件将unigene序列与NR (Non-redundant Protein Sequence Database in GenBank)、KOG (cluster of orthologous groups of proteins)、Swiss-Prot (Swiss-Prot Protein Sequence Database) 等数据库比对, 进行功能注释及分类处理; 利用BLAST及HMMER 3.0软件将unigene序列与Nt、Pfam (Protein family) 数据库进行功能注释; 使用KAAS软件与KEGG (Kyoto encyclopedia of genes and genomes) 数据库进行比对, 得到代谢通路注释信息; 基于NR和Pfam两部分的蛋白注释结果, 利用Blast2GO v2.5软件与GO数据库进行比对, 得到GO (gene ontology) 注释信息。使用iTAK软件进行植物转录因子分析, 参考文献中TF的鉴定和分类方法(2017)[24]。
差异基因表达分析 采用DESeq2软件进行样品间差异表达分析。以|log2 (Fold Change)| > 1, q-value < 0.005作为差异基因筛选的标准。利用GOseq和KOBAS软件对差异表达基因(DEGs) 分别进行GO及KEGG等富集分析。
乌头碱类化合物生物合成相关基因的挖掘 根据7大数据库中的注释信息, 结合乌头已鉴定的萜类合酶基因进行本地BLAST, 挖掘本转录组中乌头碱类化合物生物合成相关unigene。利用TBtools软件对12个全长BAHD酰基转移酶基因进行开放读码框(ORF) 筛选, 并进行翻译[25]。使用DNAMAN对氨基酸序列进行比对, MEGA7.0进行系统进化树构建, 进化树中引用的44个序列信息为D'Auria[26]报道的BAHD酰基转移酶家族。
结果与分析 1 转录组测序与组装铁棒锤叶、根及花3个部位分别得到43 473 784、49 317 840、47 336 908条原始序列, 去除接头及低质量序列后, 得到42 340 502、48 205 418、45 755 116条高质量序列。将质控后得到的高质量序列进行de novo拼接, 共得到107 674个转录本, 平均长度为895 bp, N50长度为1 780 bp。在此基础上对转录本进行Corset层次聚类, 得到47 264条unigenes, 平均长度为1 140 bp、N50为1 678 bp用于后续分析(表 1)。
| Table 1 Summary of the sample sequencing data |
将47 264条unigenes与Nr、Nt、KOG、Swiss-Prot、Pfam、KEGG、GO 7大数据库进行比对及功能注释, 30 231 (63.96%) 条unigenes至少在一种数据库中得到注释。结果表明, 27 080条unigenes (57.29%) 在Nr数据库中得到注释, Nt、KEGG、Swiss-Prot、Pfam、GO、KOG数据库获得注释的unigene数目依次为15 140条(32.03%)、9 633条(20.38%)、20 281条(24.91%)、20 330条(43.01%)、20 330条(43.01%) 和6 625条(14.01%)。与Nr数据库比对, 同源性最高的物种为洛矶山耧斗菜Aquilegia coerulea, 其次为博落回Macleaya cordata、莲Nelumbo nucifera。
2.2 KOG分类经过KOG功能分类, 6 625条unigenes注释到25条功能亚类中(图 1)。其中“翻译后修饰, 蛋白质周转, 伴侣” (posttranslational modification, protein turnover, chaperones) 是最大类群, 有928条, 占总数的14.01%; 其次为“一般功能预测” (general function prediction only), 为841条unigenes, 占总数的12.69%; “翻译、核糖体结构、生物进化” (translation, ribosomal structure and biogenesis) unigenes数目为685条, 占总数的10.34%; “RNA加工与修饰” (RNA processing and modification) unigenes数目为486条, 占总数的7.34%。与二萜生物碱生物合成相关的“次生代谢物的生物合成” (specialized metabolite biosynthesis, transport and catabolism) 和“氨基酸转运与代谢” (amino acid transport and metabolism) 亚类则分别有121条(1.82%) 和324条unigenes (4.89%)。

|
Figure 1 KOG annotation distribution of transcriptomic unigenes |
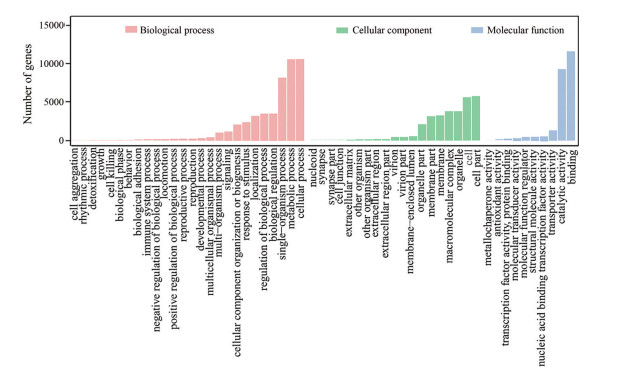
基于GO数据库, 共有20 330条unigenes成功注释到生物学过程(biological process, 50 930条)、细胞组分(cellular component, 30 002条) 和分子功能(molecular function, 24 715条) 三大类55个亚类中(图 2)。生物学过程主要集中在细胞过程(cellular process, 11 341条)、代谢过程(metabolic process, 10 685条) 和单组织过程(single-organism process, 8 273条); 细胞组分中细胞(cell, 5 805条) 和细胞部分(cell part, 5 805条) 相关基因丰度最高, 其次为细胞器(organelle, 3 840条)、复杂大分子(macromolecular complex, 3 830条)、膜(membrane, 3 296条) 和膜部分(membrane part, 3 088条); 分子功能中结合功能(binding, 11 691条) 和催化活性(catalytic activity, 9 365条) 的基因数目最多。
|
Figure 2 GO classification of transcriptomic unigenes |
铁棒锤9 633条unigenes参与KEGG数据库中的129个代谢通路中(图 3)。涉及细胞过程(cellular processes, 407条)、环境信息处理(environmental information processing, 363条)、遗传信息处理(genetic information processing, 1 983条)、代谢(metabolism, 4 086条)、有机系统(organismal systems, 333条) 五大类19个中类129个小类。其中核糖体途径unigenes数量最多, 为355条, 碳代谢和氨基酸生物合成数量次之, 分别为324条、305条。最少的是异黄酮生物合成和β-半乳糖生物合成, 分别仅有3条、2条。

|
Figure 3 KEGG classification of transcriptomic unigenes |
根据铁棒锤KEGG注释结果, 542条unigenes参与苯丙素类、萜类骨架、类胡萝卜素、玉米素、生物碱、黄酮类等生物合成相关的17个次生代谢通路(表 2)。其中, 萜类及聚酮类化合物次生代谢途径(metabolism of terpenoids and polyketides) 的unigenes最多, 有252条, 主要包括萜类骨架生物合成(terpenoid backbone biosynthesis) 75条、二萜生物合成(diterpenoid biosynthesis) 21条、单萜生物合成(monoterpenoid biosynthesis) 9条、倍半萜及三萜生物合成(sesquiterpenoid and triterpenoid biosynthesis) 6条。其次为苯丙素生物合成(phenylpropanoids biosynthesis)、生物碱类生物合成(isoquinoline, tropane, piperidine and pyridine alkaloid biosynthesis)、类胡萝卜素生物合成(carotenoid biosynthesis), 分别含有130、64、51条。这些基因的注释为铁棒锤次生代谢产物生物合成及代谢网络的解析提供了参考。
| Table 2 Secondary metabolism KEGG pathway analysis of transcriptomic unigenes |
转录因子在植物次生代谢生物合成中起到重要作用, 它通过激活通路中多个基因协同表达以提高次生代谢物的产量[27]。铁棒锤中注释到1 538个转录因子, 存在于91个分支中。TOP10家族有C2H2、AP2/ERF-ERF、bHLH、Others、MYB、MYB-related、C3H、NAC、bZIP、mTERF, 基因数量分别为83、70、67、63、58、57、55、52、49、49条。其中C2H2、AP2/ERF-ERF、bHLH、MYB、C3H、WRKY等转录因子可能参与萜类生物合成过程[27]。
4 差异表达基因筛选为了研究铁棒锤根、茎、叶中转录本的丰度及基因表达模式, 将各条样本得到的read count转换为每百万千碱基片段(FPKM), 以评估unigene的表达水平。差异基因以|log2 (Fold Change)| > 1, q-value < 0.005为阈值进行筛选。对铁棒锤根、茎、叶3个样品中差异基因进行综合分析, 在47 264条unigenes中共检测到17 391条差异基因。叶与根组织(TBCLvsTBCR)、根与花组织(TBCRvsTBCF)、叶与花组织(TBCLvsTBCF) 中分别检测到差异基因10 524条(5 496条上调基因, 5 028条下调基因)、10 129条(3 622条上调基因, 6 507条下调基因)、10 393条(4 123条上调基因, 6 270条下调基因)。
根据KEGG注释分析, 在17 391条差异基因中, 叶与根组织中发现2 574条差异表达基因参与197个KEGG次生代谢通路, 根与花组织中发现2 329条差异表达基因参与199个KEGG次生代谢通路, 叶与花组织中发现2 249条差异表达基因参与198个KEGG次生代谢通路。其中淀粉与蔗糖代谢、植物激素信号转导、核糖体、植物病原相互作用和苯丙烷生物合成的基因丰度最高。并且3个样本均检测到萜类骨架生物合成(ko00900)、二萜生物合成(ko00904) 代谢通路, 其中在叶/花和叶/根组织中萜类骨架及二萜生物合成差异基因数量最多, 分别为10、21; 8、23条差异基因。
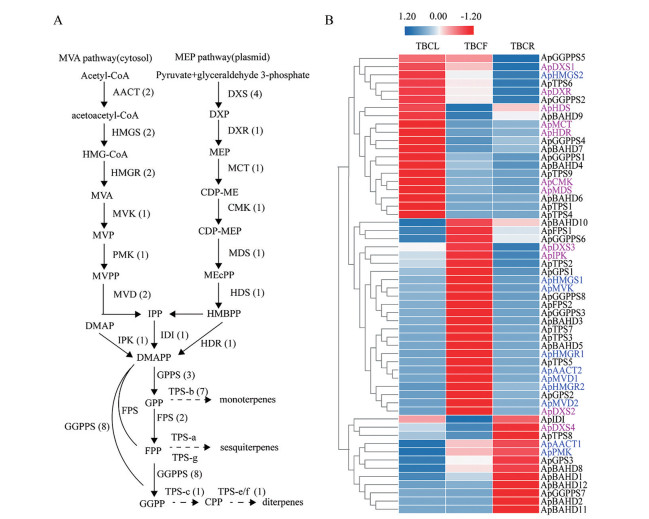
5 二萜生物碱合成相关基因 5.1 萜类骨架合成相关基因基于铁棒锤转录组数据库注释结果, 筛选到66条基因参与萜类骨架生物合成, 其中22个小于500 bp的基因序列不予统计(图 4A)。10条基因编码MVA途径6个关键酶, 包括acetyl-CoA acetyltransferase (AACT)、hydroxy methylglutaryl-CoA synthase (HMGS)、hydroxy methylglutaryl-CoA reductase (HMGR)、mevalonate kinase (MVK)、phosphomevalonat kinase (PMK)、mevalonate diphosphate decarboxylase (MVD); 12条基因编码MEP途径9个关键酶, 1-deoxy-D-xylulose 5-phosphate synthase (DXS)、1-deoxy-D-xylulose 5-phosphate reductoisomerase (DXR)、2-C-methyl-D-erythritol 4-phosphate cytidylyltransferase (MCT)、4-(cytidine 5-diphospho)-2-C-methyl-D-erythritol kinase (CMK)、2-C-methyl-D-erythritol 2, 4-cyclodiphosphate synthase (MDS)、(E)-4-hydroxy-3-methylbut-2-enyl-diphosphate synthase (HDS)、(E)-4-hydroxy-3-methylbut-2-enyl diphosphate reductase (HDR) 以及isopentenyl diphosphate-isomerase (IDI)、isopentenyl phosphate kinase (IPK); 13条异戊烯基转移酶基因分别编码geranyl diphosphate synthase (GPPS)、farnesyl diphosphate synthase (FPS) 和geranylgeranyl diphosphate synthase (GGPPS); 9条terpene synthase (TPS) 基因, 包括7条单萜合酶(mono-TPS)、2条二萜合酶(diTPS)。与乌头中12条二萜合酶基因相比[23], 铁棒锤中二萜合酶基因家族发生了明显的收缩。
|
Figure 4 Schematic diagram of terpenoid biosynthetic pathway and the expression level of genes of Aconitum pendulum. A: The putative terpenoid biosynthetic pathway in A. pendulum. Numbers in the brackets indicates the gene family numbers of each enzyme; B: A clusterted heat map for 44 terpenoid backbone genes and 12 ApBAHDs with log-2 transfored FRKM value in leaf (TBCL), folwer (TBCF), and root (TBCR) in A. pendulum. Blue marked enzymes are involved in MVA pathway, purple marked enzymes are involved in MEP pathway |
根据铁棒锤转录组注释信息, 共筛选到15条ApBAHD酰基转移酶基因, 其中12条基因长度大于500 bp, 均含有保守结构域HXXXD与DFGWG。为进一步预测这些基因的功能, 对铁棒锤12条ApBAHD酰基转移酶基因与已鉴定功能的44条BAHD酰基转移酶基因[24]进行聚类分析(图 5)。结果发现9条基因ApBAHD1 (Cluster-8543.2319)、ApBAHD2 (Cluster-395.0)、ApBAHD3 (Cluster-13967.0)、ApBAHD4 (Cluster-8543.3560)、ApBAHD5 (Cluster-16590.0)、ApBAHD6 (Cluster-20993.0)、ApBAHD7 (Cluster-8543.1781)、ApBAHD8 (Cluster-17474.1)、ApBAHD9 (Cluster-8543.19115) 归属于V-i子家族, 据报道该家族可利用苯甲酰辅酶A作为酰基供体参与苯甲酸酯的生物合成[28, 29], 推测这9条基因可能在生物碱苯甲酰酯生物合成过程中发挥作用。3条基因ApBAHD10 (Cluster-8543.22066)、ApBAHD11 (Cluster-212.0)、ApBAHD12 (Cluster-8543.4464) 归属于Ⅲ亚家族。Ⅲ亚家族酶主要以乙酰辅酶A为酰基供体, 以多种醇类化合物为底物, 参与生物碱生物合成[30, 31], 据此推测这3条基因可能参与生物碱乙酰酯生物合成。与乌头中11条苯甲酰转移酶、34条乙酰转移酶相比[23], 铁棒锤中BAHD酰基转移酶基因家族发生了明显的收缩, 这一结果与铁棒锤的代谢组数据相吻合[6]。

|
Figure 5 Phylogenetic relationships among the 44 functionally characterized BAHD members[24] plus the 12 ApBAHDs acyltransferases of A. pendulum. The neighbour-joining tree was generated using the MEGA 7.0 software with 1 000 bootstrap replicates |
为了进一步预测上述基因的生物学功能, 利用其在根、叶及花中的基因表达量进行聚类分析(图 4B), 结果显示56条基因主要分为三大类。第一大类主要在叶中表达, 包括20条基因。该分支包括MEP途径中的ApDXS1、ApDXR、ApMCT、ApCMK、ApMDS、ApHDS, 二萜途径中的ApTPS9、ApGGPPS1/2/4、ApBAHD4/6/7/9, 其中ApTPS9为二萜合酶基因。MEP途径中的基因在叶中有优势表达, 与二萜途径主要来源于MEP途径认知相符合。第二大类主要在花中表达, 包括24条基因。该分支包括MVA途径中的ApAACT1/2、ApHMGS1、ApHMGR1/2、ApMVK、ApMVD1/2, 单萜途径中的ApGPPS2/3、ApTPS1/2、ApTPS2/3/5/7及倍半萜途径中的ApFPPS1/2。其中单萜合酶主要注释为松油醇、月桂烯、芳樟醇、柠檬烯合酶, 推测这些基因可能与花中萜类物质的生物合成有关。第三大类主要在根中表达, 包括12条基因。该分支包括MEP途径中的ApDXS4, MEP途径中的ApAACT、ApPMK及二萜途径中的ApTPS8、ApBAHD1/2/8、ApBAHD11/12、ApGGPPS7, 其中ApTPS8为对映柯巴基焦磷酸合酶基因, 是二萜骨架形成的第一个关键酶, 因此提示根可能为二萜生物碱类化合物合成起始的部位。综上所述, 单萜前体主要在花中高表达, 二萜前体主要在叶中高表达, 而下游途径酰基转移酶在不同部位均有高表达。
讨论本研究首次报道铁棒锤转录组序列, 填补了铁棒锤转录组信息的空白。Trinity组装得到47 264条unigenes, 平均长度为1 140 bp, N50为1 678 bp, 转录组数据质量优良。对二萜生物碱类生物合成相关基因进行深入分析, 30 231条基因得到注释信息, 鉴定出萜类及酰基转移酶基因56条, 并对其生物学功能进行了预测, 为后续二萜生物碱生物合成途径解析提供支持。
二萜类化合物来源于相同的前体物质GGPP[28], 其生源合成追溯于两个交叉且独立的生物途径, MVA和MEP途径。GGPP在二萜合酶的作用下生成二萜骨架, 为萜类化合物丰富多样性的基础。Ⅱ型萜类合酶柯巴基焦磷酸合酶(CPS) 为形成二萜生物碱前体ent-CPP的关键酶[23], 二萜合酶基因ApTPS8在根中高表达, 推测根是二萜生物碱前体合成的主要部位。虽然铁棒锤中二萜上游途径的基因主要在叶中高表达, 但是部分基因如异戊烯基转移酶ApGGPPS3/6/8、乙酰转移酶ApBAHD10在花中表达量也很高。与其他二萜类成分不同, 如丹参酮类化合物特异积累于丹参根部木栓层[32], 铁棒锤中主要化合物乌头碱及2-羟基乌头碱在根、茎、叶3个部位中含量均较高[6], 没有明显的组织特异性, 这与上述基因在不同组织中均有表达较为一致。而不同部位中的乌头碱类化合物是由不同的酶进行催化, 这些化合物是否存在组织间的运输或转移则需要进行深入的分析。
BAHD酰基转移酶是植物中特有的对次生代谢物进行酰基化修饰的超基因家族[26]。该基因家族复杂且庞大, 主要分为五大分支, 参与生物碱合成的乙酰转移酶及苯甲酰转移酶集中于Ⅲ亚家族及Ⅴ亚家族的V-i子家族[30-33]。目前已从红豆杉Taxus chinensis[33]、长春花Catharanthus roseus[34]、罂粟Papaver somniferum[35]、蛇根木Rauvolfia serpentina[30]中鉴定出了与生物碱生物合成相关的BAHD酰基转移酶。乌头碱类化合物的生物合成涉及广泛的酰基修饰过程, 在不同位置如C8、C3、C14等均存在酰基或苯甲酰基修饰, 其中乌头碱、次乌头碱、新乌头碱在C8位及C14位特征性的乙酰酯及苯甲酰酯是乌头碱类成分毒性形成的重要原因[13]。铁棒锤转录组中共有12条unigenes注释为BAHD酰基转移酶基因, 根中高水平表达的ApBAHD1/2/8在不同组织的表达变化趋势与2-羟基乌头碱、dehydrated 14-benzoylaconitine、8-O-methyl-14-benzoylaconine、benzoyldeoxyaconitine和benzoylaconitine等生物碱含量变化趋势一致[5], 推测ApBAHD1/2/8参与上述二萜生物碱的生物合成。ApBAHD10与含有乙酰基团的乌头碱类化合物如乌头碱、lucidusculine、14-O-acetylneoline、14-O-acetylvirescenine等生物碱的含量变化趋势一致[5], 由此推测ApBAHD10基因参与上述二萜生物碱的生物合成。
二萜生物碱复杂的母核结构为其生物合成途径解析带来了巨大的挑战。因此, 寻找代谢物数量少、代谢网络单一的研究材料是进行特定化合物生物合成途径解析研究的捷径。与乌头相比, 铁棒锤无疑是进行乌头碱生物合成途径解析的理想材料。为获得更丰富的基因信息, 铁棒锤PacBio全长测序联合多组学分析, 将有望实现二萜生物碱生物合成途径研究的重大突破。
作者贡献: 田梅为本文的第一作者, 负责分子生物学实验、数据分析及稿件撰写, 陈灵丽参与实验设计及结果分析, 靳保龙与郭娟参与稿件修改, 葛慧与赵鑫参与样品的收集, 崔光红为本文的通讯作者, 为项目的总负责人, 指导实验设计、数据分析、论文撰写及修改。全体作者均阅读并且同意最终的文本。
利益冲突: 本文研究内容无利益冲突。
| [1] |
Chinese Pharmacopoeia Commission. Pharmacopoeia of the People's Republic of China (中华人民共和国药典)[S]. Beijing: China Medical Science Press, 2015: 424.
|
| [2] |
Ma L, Gu R, Tang L, et al. Important poisonous plants in tibetan ethnomedicine[J]. Toxins, 2015, 7: 138-155. DOI:10.3390/toxins7010138 |
| [3] |
Editorial Committee of flora of China, Chinese Academy of Sciences. Flora of China (中国植物志)[M]. Beijing: Science Press, 1978: 52.
|
| [4] |
Cherney ME, Baran PS. Terpenoid-alkaloids: their biosynthetic twist of fate and total synthesis[J]. Isr J Chem, 2011, 51: 391-405. DOI:10.1002/ijch.201100005 |
| [5] |
Wang B, Dong J, Ji J, et al. Study on the alkaloids in tibetan medicine Aconitum pendulum Busch by HPLC-MS combined with column chromatography[J]. J Chromatogr Sci, 2016, 54: 752-760. DOI:10.1093/chromsci/bmw002 |
| [6] |
Wang YJ, Zeng CJ, Yao Z, et al. Diterpene alkaloids from roots and processed products of Aconitum pendulum[J]. Chin Tradit Herb Drugs (中草药), 2010, 41: 347-352. |
| [7] |
Chen LL, Lai CJS, Mao LY, et al. Chemical constituents in different parts of seven species of Aconitum based on UHPLC-Q-TOF/MS[J]. J Pharm Biomed Anal, 2020, 193: 113713. |
| [8] |
Wang J, Meng XH, Chai T, et al. Diterpenoid alkaloids and one lignan from the roots of Aconitum pendulum Busch[J]. Nat Prod Bioprospect, 2019, 9: 419-423. DOI:10.1007/s13659-019-00227-y |
| [9] |
Devkota KP, Sewald N. Terpenoid alkaloids derived by amination reaction[J]. Nat Prod, 2013, 28: 923-951. |
| [10] |
Nishiyama Y, Yokoshima S, Fukuyama T. Total synthesis of (-)-cardiopetaline[J]. Org Lett, 2016, 18: 2359-2362. DOI:10.1021/acs.orglett.6b00789 |
| [11] |
Nishiyama Y, Yokoshima S, Fukuyama T. Synthesis of cardiopetaline via a Wagner-Meerwein rearrangement without preactivation of the pivotal hydroxy group[J]. Org Lett, 2017, 19: 5833-5835. DOI:10.1021/acs.orglett.7b02812 |
| [12] |
Dank C, Sanichar R, Choo KL, et al. Recent advances towards syntheses of diterpenoid alkaloids[J]. Synthesis, 2019, 51: 3915-3946. DOI:10.1055/s-0037-1611897 |
| [13] |
Zhao PJ, Gao S, Fan LM, et al. Approach to the biosynthesis of atisine-type diterpenoid alkaloids[J]. J Nat Prod, 2009, 72: 645-649. DOI:10.1021/np800657j |
| [14] |
Rai M, Rai A, Kawano N, et al. De Novo RNA sequencing and expression analysis of Aconitum carmichaelii to analyze key genes involved in the biosynthesis of diterpene alkaloids[J]. Molecules, 2017, 22: 2155. DOI:10.3390/molecules22122155 |
| [15] |
Zhao D, Shen Y, Shi Y, et al. Probing the transcriptome of Aconitum carmichaelii reveals the candidate genes associated with the biosynthesis of the toxic aconitine-type C19-diterpenoid alkaloids[J]. Phytochemistry, 2018, 152: 113-124. DOI:10.1016/j.phytochem.2018.04.022 |
| [16] |
Zhang DY, Wen H, Wang W, et al. Transcriptional analysis of terpenoid biosynthesis in Aconitum carmichaelii[J]. Chin J Exp Tradit Med Form (中国实验方剂学杂志), 2017, 23: 45-50. |
| [17] |
Gao JH, Zhang DY, Hou FX, et al. Transcriptome analysis of Aconitum carmichaelii identifies genes involved in terpenoid, alkaloid and phenylpropanoid biosynthesis[J]. Int J Agric Biol, 2019, 22: 710-720. |
| [18] |
Yang Y, Hu P, Zhou X, et al. Transcriptome analysis of Aconitum carmichaelii and exploration of the salsolinol biosynthetic pathway[J]. Fitoterapia, 2020, 140: 104412. DOI:10.1016/j.fitote.2019.104412 |
| [19] |
Zhong S, Yin YP, He YN, et al. Analysis of transcriptome differences in two leaf-type cultivars of Aconitum carmichaelii[J]. China J Chin Mater Med (中国中药杂志), 2020, 45: 1633-1640. |
| [20] |
Pal T, Malhotra N, Chanumolu SK, et al. Next-generation sequencing (NGS) transcriptomes reveal association of multiple genes and pathways contributing to secondary metabolites accumulation in tuberous roots of Aconitum heterophyllum Wall[J]. Planta, 2015, 242: 239-258. DOI:10.1007/s00425-015-2304-6 |
| [21] |
Malhotra N, Kumar V, Sood H, et al. Multiple genes of mevalonate and non-mevalonate pathways contribute to high aconites content in an endangered medicinal herb Aconitum heterophyllum Wall[J]. Phytochemistry, 2014, 108: 26-34. DOI:10.1016/j.phytochem.2014.08.025 |
| [22] |
Kumar V, Malhotra N, Pal T, et al. Molecular dissection of pathway components unravel atisine biosynthesis in a non-toxic Aconitum species A. heterophyllum Wall[J]. 3 Biotech, 2016, 6: 106. |
| [23] |
Mao LY, Jin BL, Chen LL, et al. Functional identification of the terpene synthase family involved in diterpenoid alkaloids biosynthesis in Aconitum carmichaelii[J]. Acta Pharm Sin B, 2021, 10: 3310-3321. |
| [24] |
Jin J, Tian F, Yang DC, et al. Plant TFDB 4.0: toward a central hub for transcription factors and regulatory interactions in plants[J]. Nucleic Acids Res, 2017, 45: D1040-D1045. DOI:10.1093/nar/gkw982 |
| [25] |
Chen C, Chen H, Zhang Y, et al. TBtools: an integrative toolkit developed for interactive analyses of big biological data[J]. Mol Plant, 2020, 13: 1194-1202. DOI:10.1016/j.molp.2020.06.009 |
| [26] |
D'Auria JC. Acyltransferases in plants: a good time to be BAHD[J]. Curr Opin Plant Biol, 2006, 9: 331-340. DOI:10.1016/j.pbi.2006.03.016 |
| [27] |
Zhao HW, Ge F, Sun Y, et al. Transcription factors involved in plant terpenoid biosynthesis and their application prospect[J]. Chin Tradit Herb Drugs (中草药), 2012, 43: 2512-2519. |
| [28] |
Walker K, Croteau R. Taxol biosynthesis: molecular cloning of a benzoyl-CoA: taxane 2α-O-benzoyltransferase cDNA from taxus and functional expression in Escherichia coli[J]. Proc Natl Acad Sci U S A, 2000, 97: 13591-13596. DOI:10.1073/pnas.250491997 |
| [29] |
Liu YY, Mo T, Wang XH, et al. Reasearch progress of plant BAHD acyltransferase family[J]. China J Chin Mater Med (中国中药杂志), 2016, 41: 2175. |
| [30] |
Bayer A, Ma XY, Stockigt J. Acetyltransfer in natural product biosynthesis-functional cloning and molecular analysis of vinorine synthase[J]. Bioorg Med Chem, 2004, 12: 2787-2795. DOI:10.1016/j.bmc.2004.02.029 |
| [31] |
Grothe T, Lenz R, Kutchan TM. Molecular characterization of the salutaridinol 7-O-acetyltransferase involved in morphine biosynthesis in opium poppy Papaver somniferum[J]. J Biol Chem, 2001, 276: 30717-30723. DOI:10.1074/jbc.M102688200 |
| [32] |
Cui GH, Duan L, Jin BL, et al. Functional divergence of diterpene syntheses in the medicinal plant Salvia miltiorrhiza[J]. Plant Physiol, 2015, 169: 1607-1618. |
| [33] |
Nevarez DM, Mengistu YA, Nawarathne IN, et al. An N-aroyltransferase of the BAHD superfamily has broad aroyl CoA specificity in vitro with analogues of N-dearoylpaclitaxel[J]. J Am Chem Soc, 2009, 131: 5994-6002. DOI:10.1021/ja900545m |
| [34] |
Carqueijeiro I, Dugé de Bernonville T, Lanoue A, et al. A BAHD acyltransferase catalyzing 19-O-acetylation of tabersonine derivatives in roots of Catharanthus roseus enables combinatorial synthesis of monoterpene indole alkaloids[J]. Plant J, 2018, 94: 469-484. DOI:10.1111/tpj.13868 |
| [35] |
St-Pierre B, Laflamme P, Alarco AM, et al. The terminal O-acetyltransferase involved in vindoline biosynthesis defines a new class of proteins responsible for coenzyme A-dependent acyl transfer[J]. Plant J, 1998, 14: 703-713. DOI:10.1046/j.1365-313x.1998.00174.x |
 2021, Vol. 56
2021, Vol. 56


