2. 河南中医药大学药学院, 河南 郑州 450046;
3. 安徽中医药大学药学院, 安徽 合肥 230071
2. School of Pharmaceutical Sciences, Henan University of Chinese Medicine, Zhengzhou 450046, China;
3. School of Pharmaceutical Sciences, Anhui University of Chinese Medicine, Hefei 230071, China
植物天然产物作为天然产物的重要组成部分, 具有抗氧化、抗肿瘤、抗炎、镇痛等诸多活性, 广泛应用于医药、化妆品、保健品、调味品、色素等领域, 具有非常高的应用价值[1]。1985年, 世界卫生组织调查显示约有65%的人口依赖植物来源的药物进行医疗保健[2], 其中大部分来源于植物的特殊代谢产物[3], 包括我们熟知的青蒿素、紫杉醇、丹参酮、穿心莲内酯等。
植物天然产物主要从栽培种植或野生植物中提取获得, 但大多数活性成分在植物的特定组织部位或特定生长阶段积累, 且含量较低, 如青蒿素含量仅达到干重(DW) 的0.1%~1%[4]。一方面受限于植物生长周期以及气候、地理等因素, 产量品质不稳定, 难以满足日益增长的需求; 另一方面植物中结构类似物多, 限制了植物天然产物的分离纯化及其深入发掘利用。近年兴起的合成生物学为植物天然产物的开发应用提供了新的获取方式, 通过发掘植物天然产物生物合成关键基因, 采用合成生物学策略设计和改造微生物菌株来生产植物天然产物。改造的人工合成细胞能连续、高效地合成特定的植物天然产物, 具有低碳、经济和环境友好的特点, 是一种极具潜力的资源获取方法[5, 6]。
随着基因组测序、组学分析技术以及基因工程的发展, 植物天然产物生物合成研究取得突破进展。2006年, 《Nature》首次报道了青蒿酸在酿酒酵母中的生物合成[7]; 2013年通过引入植物脱氢酶和细胞色素CYB5, 调节基因表达并采用两相发酵的方法, 最终将青蒿酸产量提升至25 g·L-1 [5]。2015年, 研究人员通过利用来自植物、哺乳动物、细菌和酵母的20余种基因, 首次实现了阿片类化合物在酵母中的从头合成, 完成蒂巴因(6.4 μg·L-1) 和氢可酮(0.3 μg·L-1) 的生物合成[8], 实现复杂代谢途径的微生物转移, 是植物天然产物合成生物学的里程碑事件, 被评为《Science》 2015年度十大科学进展之一。最近由中国科学院天津工业生物技术研究所完成的人工淀粉的从头合成刊登在《Science》上, 基于计算机途径设计, 通过模块化组装和替换, 结合三种瓶颈相关酶的蛋白质工程优化, 实现人工淀粉的快速合成, 其淀粉合成速率是玉米淀粉合成速率的8.5倍, 为创建新功能的生物系统提供科学基础[9]。这些里程碑工作的报道促进了植物天然产物生物合成以及合成生物学生产的研究和应用。
我国是中药资源大国, 据第四次全国中药资源普查显示我国药用植物资源达12 800多种, 丰富的药用植物资源一方面为我国药物研发提供结构多样的小分子库, 另一方面为基础研究、生物合成途径解析、合成生物学和组合生物学提供丰富的酶库。国际植物天然产物合成生物学相关研究以及我国中药资源的传统优势极大地推动了植物来源天然产物的合成途径解析、元件挖掘改造、途径创建和发酵生产的研究与应用。本文在作者前期综述的基础上[10], 进一步对植物天然产物的途径解析和途径创建的整体思路和研究策略进行系统梳理, 并对近年来的重要成果、新方法新技术的应用进行总结和展望, 为植物天然产物的研究开发利用提供参考(图 1)。
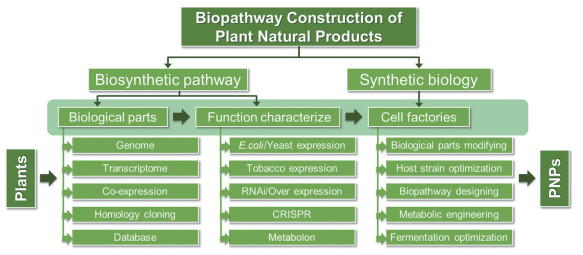
|
Figure 1 Biopathway construction of plant natural products |
植物天然产物主要包括萜类(terpenoids)、生物碱(alkaloids)、苯丙素类化合物(phenylpropanoids) 和聚酮(polyketides) 等。迄今, 超过22 000种萜类及其衍生物的化学结构被鉴定, 占天然产物总数的25%~50%, 其中包括大家熟知的青蒿素、紫杉醇、丹参酮、人参皂苷等。生物碱类化合物包括多数天然有机化合物中的含氮物质, 是天然产物中最大类群化合物之一。目前已知的生物碱有21 000多种[4], 根据其基本骨架主要分为吲哚类生物碱、异喹啉类生物碱、有机胺类生物碱、吡啶类生物碱、莨菪烷类生物碱等。常用于临床研究的生物碱有抗肿瘤化合物长春花碱、镇痛化合物吗啡、可待因、延胡索乙素等[11]。苯丙素类化合物是指基本母核具有一个或几个C6-C3单元的天然有机化合物类群, 通常可分为简单苯丙素类、香豆素类、木脂素和木质素类以及黄酮类等[12]。植物天然产物的生物合成主要以植物初生代谢途径产物为前体, 经过一系列的酶催化形成结构多样的天然产物。在植物中, 不同类型的天然产物均具有较为保守的前体形成过程, 而催化功能多样的结构后修饰酶在植物天然产物的合成中起关键作用[13, 14]。
1.2 结构后修饰酶植物天然产物种类繁多、结构复杂, 其上游合成途径较保守, 在不同的综述中已有较为详细的阐述[15-17]。前体经过结构修饰后能显著提高天然产物的活性和成药性, 植物中具有良好活性的天然产物多是修饰程度较高、结构复杂的次生代谢产物, 在其合成过程中, 多是由后修饰酶催化产生的。常见的后修饰酶包括细胞色素P450 (cytochrome P450, P450)、糖基转移酶(glycosyltransferase, GT)、酰基转移酶(acyltransferase, AT)、甲基转移酶(methyltransferase, MT), 除此之外酮戊二酸依赖双加氧酶(Fe2+/2-oxoglutarate-dependent dioxygenase, 2ODD)、黄素腺嘌呤二核苷酸依赖氧化还原酶(flavin adenine dinucleotide-dependent oxidoreductase, FADOX)、脱氢酶(dehydrogenase)、脱羧酶(decarboxylase) 和异戊烯基转移酶(prenyltransferase) 等在植物天然产物生物合成中亦发挥重要作用。本文主要对广泛参与植物次生代谢途径并且研究较多的P450、糖基转移酶、酰基转移酶、甲基转移酶等进行总结, 为结构后修饰酶的挖掘和功能研究提供参考。
1.2.1 细胞色素P450P450是植物代谢中的最大酶蛋白家族之一, 占植物蛋白编码基因的1%左右[18], P450分化与陆生植物的生存进化紧密相关[19], 在初生代谢和次生代谢中均发挥催化作用。植物中参与激素合成或者上游合成途径的P450相对较为保守, 主要集中在几个家族[19], 而参与次生代谢产物生物合成的P450则分化较大, 催化产生化学结构的多样性[20, 21]。P450参与包括羟基化、碳碳偶联、羧化、脱甲基等各种催化反应[22]。对目前已经报道功能的P450进行系统进化分析(图 2), 结果表明不同植物来源的P450主要以基因家族聚类, 并且通常情况下, 参与结构类似化合物修饰过程的P450具有较高的同源性, 聚在同一个分支, 比如参与紫杉醇生物合成的P450s具有70%同源性[23]; 参与丹参酮及鼠尾草酸等类似位点羟基化或者羧化的P450大多来自于CYP76家族[24-26]。羟基化是P450常参与的催化反应, 在苄基异喹啉类生物碱(benzylisoquinoline alkaloids, BIAs) 生物合成中, P450被报道参与结构异构化和结构偶联反应[14]。例如来源于日本柳杉(Cryptomeria japonica) 的CYP80G2, 可催化S-牛心果碱的C-C苯酚偶联生成S-紫堇块茎碱[27]; CYP719A亚家族被鉴定参与分子内偶联反应形成亚甲基二氧桥[28]。此外, P450还能够催化连续氧化反应、芳香化合物的环氧化反应和胺类及其衍生物的甲基或氨基转移反应等[22]。甘草中的CYP72A154能够进行连续3步氧化反应, 催化甘草酸前体11-羰基-β-香树脂醇的C-30位成羧基, 从而生成甘草次酸[29]。来源于薄荷(Mentha haplocalyx) 的CYP71D18能够催化(+)-(4R)-柠檬烯的C-1, C-2位置发生环氧化反应, 生成顺式-1, 2-环氧化物[30]。虽然不同P450参与的反应不尽相同, 但据目前的研究报道, 参与类似反应的P450大多具有相对较高的同源性, 且随着越来越多的P450被鉴定, 结合系统进化分析, 将为这一重要酶的功能筛选和研究提供更多参考, 促进生物合成途径解析。
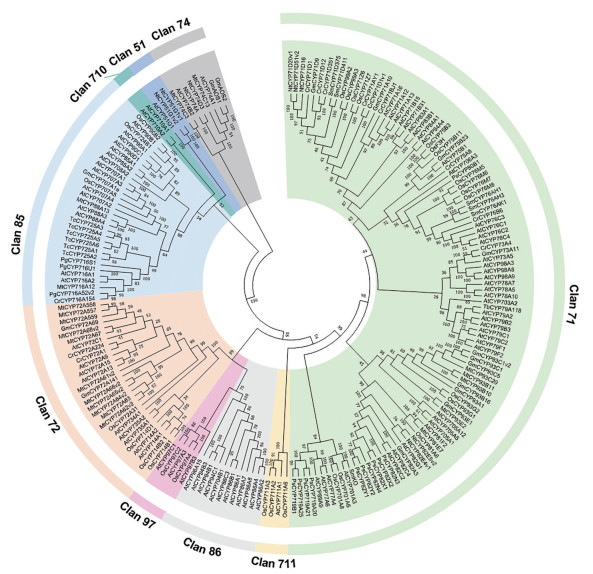
|
Figure 2 Phylogenetic analysis of P450s in plant natural product biosynthesis pathway. P450s from Arabidopsis thaliana were employed as references. Sm: Salviae miltiorrhiae; Pg: Panax ginseng; Mt: Medicago truncatula; Tc: Taxus chinensis; Cr: Catharanthus roseus; Ps: Papaver somniferum; Gm: Glycine max; Os: Oryza sativa; Nt: Nicotiana tabacum; At: Arabidopsis thaliana. Sequences were downloaded from NCBI or Unipro |
糖基转移酶是多基因家族, 其中家族1 (GT1) 最大, 称为尿苷二磷酸糖基转移酶(UGT) 超家族, 主要以UDP-葡萄糖为糖基供体[31, 32]。糖基化反应属于末端修饰, 能改变化合物的理化性质, 直接或间接影响药用活性, 从而影响对活性成分的利用效率和方式[31]。UGT作为植物天然产物生物合成的关键修饰酶, 在植物基因组中的扩张和收缩存在种系特异性, 在植物次生代谢尤其以黄酮、花青素、三萜类和甾体皂苷类化合物等的结构多样性中起重要作用[32]。近年, 随着转录组和基因组数据的大量释放, UGT的研究得以深入开展, 从基因功能研究、晶体结构解析、催化机制研究以及蛋白质工程改造等方面均产生了许多突破成果[33-37]。植物的GT1家族可分为A-R共18个组(group), 不同物种UGT基因的总数, 在组间的分布均有差异[32, 38, 39], 这也暗示植物GT1家族基因在系统演化过程中的多样性和复杂性。以拟南芥的UGT (不包括两条UGT80家族的UGT) 和部分具有代表性且功能明确的植物来源UGT进行系统进化分析, 表明功能类似的UGT在系统进化树上通常处于较近的分支(图 3), 这为从植物中挖掘糖基转移酶进行结构修饰提供参考和指导。目前糖基化修饰研究最多的是黄酮类化合物, 其中O-糖基转移酶的研究较为深入[40]。近年不断从植物中克隆获得了一些具有较好活性的C-糖基转移酶[33, 34]和N-糖基转移酶[41]。北京大学医学部叶敏教授课题组[33]在光果甘草(Glycyrrhiza glabra) 中鉴定到一条具有双C-糖基转移酶GgCGT (UGT708B4) 的基因, 能够高效催化根皮素发生连续两步的C-糖基化反应, 生成相应的双碳苷化合物。据报道从多种植物克隆的C-糖基转移酶多为UGT708家族, 均能催化根皮素或2羟基黄酮的一步或两步的C-糖基化[42-44]。最近, 研究人员在银杏(Ginkgo biloba) 中发现了能够催化IAA和IAA-ASP的N-糖基转移酶GbNGT1, 完善了生长素的代谢通路和糖基转移酶的功能[41]。除了黄酮类化合物, 三萜皂苷类化合物的糖基化包括人参皂苷、三七皂苷、罗汉果甜苷等合成途径中糖基转移酶的研究也较深入。人参皂苷作为人参(Panax ginseng) 中重要的天然产物, 主要是原人参二醇和原人参三醇的糖基化产物。Yan等[45]从人参cDNA中找到第一个糖基转移酶UGTPg1, 发现其能够特异性催化原人参二醇的C20位羟基糖基化, 并使原人参三醇C20位羟基进行糖基化产生具有生物活性的人参皂苷F1。上海植物生理生态研究所周志华研究员团队[46]随后又从人参中分离得到4个新的糖基转移酶UGTPg100、UGTPg101、UGTPg102、UGTPg103, 其中UGTPg100能特异性催化原人参二醇的C6位羟基糖基化, 生成具有活性的人参皂苷Rh1, UGTPg101催化原人参三醇生成人参皂苷F1, 然后从F1生成人参皂苷Rg1。黄酮类化合物以及人参皂苷类生物合成途径糖基转移酶的系统研究能进一步指导其他苯丙素类化合物、三萜皂苷以及甾体皂苷生物合成研究。
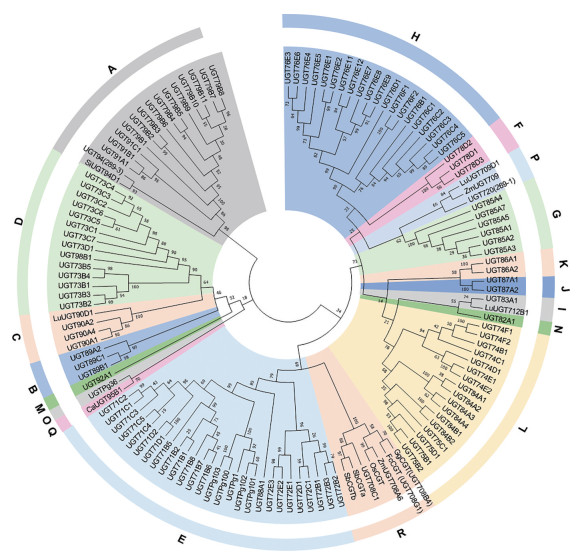
|
Figure 3 Phylogenetic analysis of UGTs in plant natural product biosynthesis pathway. UGTs from Arabidopsis thaliana were employed as references. Ca: Cicer arietinum; Lu: Linum usitatissimum; Si: Sesamum indicum; Zm: Zea mays; Fc: Fortunella crassifolia; Os: Oryza sativa japonica group; Pp: Prunus persica; Gg: Glycyrrhiza glabra; Pg: Panax ginseng; Sg: Siraitia grosvenorii; Sb: Scutellaria baicalensis; At: Arabidopsis thaliana. Arabidopsis UGTs was downloaded from the website www.p450.kvl.dk. Other UGTs were downloaded from NCBI or Unipro |
植物酰基转移酶包括两大家族: BAHD酰基转移酶和丝氨酸羧肽酶样(SCPL) 酰基转移酶, 分别以酰基辅酶A硫酯和1-O-β-葡萄糖苷为酰基供体[47, 48]。酰基转移酶可修饰多种植物天然产物, 包括黄酮类、奎宁酸/莽草酸、萜类、生物碱、醇类、脂类、蔗糖等[48]。药理学研究表明, 酰基化修饰能够提高化合物的稳定性和脂溶性, 并具有抵御植物病害以及发生应激反应的作用, 在植物的生长发育及次生代谢产物的结构修饰、生物活性中均发挥重要作用[49]。Vinorine synthase是一种乙酰转移酶, 在抗心律失常药单萜类吲哚生物碱ajmaline的生物合成中起到关键作用, 作为BAHD超家族中的一员, 具有可逆性催化16-epi-vellosimine生成vinoline的功能[50]。在紫杉醇生物合成过程中, 来自红豆杉属植物的N-苯甲酰基转移酶(NDTBT) 能够将苯甲酰基团从相应的辅酶A硫酯转移到N-debenzoyl-2'-deoxypaclitaxel中, 生成N-苯甲酰基衍生物。NDTBT具有底物杂泛性, 能够催化乙酰基、丁酰基、己酰基与紫杉烷类化合物反应生成对应的酰基取代化合物[51]。在最近的研究中, 四川大学张阳教授团队[52]发现菊苣酸的生物合成途径涉及了两大家族酰基转移酶BAHD和SCPL, 其中两个胞质中的BAHD家族成员EpHTT和EpHQT分别催化咖啡酰辅酶A和酒石酸及奎宁酸反应生成咖啡酰酒石酸和绿原酸, 并且当两种产物进入液泡后, 会被SCPL家族成员EpCAS催化, 生成菊苣酸和奎宁酸。值得注意的是, EpCAS酰基供体是绿原酸而不是SCPL酰基转移酶常见的酰基供体1-O-β-葡萄糖苷, 这一发现对SCPL家族酰基转移酶的认知具有十分重要的意义。目前酰基化取代在脂肪酰基(乙酰基、丙二酰基、丁酰基、己酰基等) 和芳香酰基(肉桂酰基、咖啡酰基、香豆酰基、阿魏酰基等)等天然产物结构修饰中被报道[53]。Wang等[48]较全面地综述了目前已进行功能研究的植物酰基转移酶, 并进行系统进化分析, 结果显示目前已报道功能(129个) 的酰基转移酶主要以底物结构聚类, 催化同样类型底物的酰基转移酶更倾向于聚在同一个分支, 该报道为植物天然产物生物合成中酰基转移酶的筛选和功能研究提供指导。
1.2.4 甲基转移酶甲基转移酶以S-腺苷甲硫氨酸(S-adenosy-L-methionine, SAM) 为甲基供体, 天然产物的甲基化修饰能够改善化合物的稳定性、溶解性和生物活性等, 在天然产物药物开发中起重要作用[54]。根据甲基化的靶原子不同, 可将甲基转移酶分为氧甲基转移酶(O-MTs)、碳甲基转移酶(C-MTs)、氮甲基转移酶(N-MTs)、硫甲基转移酶(S-MTs)、无机砷甲基转移酶(Cyt19) 等, 大多数甲基转移酶以底物结构聚类, 系统进化分析以及序列的相似性能为酶的功能研究提供初步参考(图 4)。在植物天然产物生物合成中研究较多的是O-MTs和N-MTs[55], 在苄基异喹啉类生物碱生物合成过程中, 通过O-MTs、N-MTs和P450的交替催化产生S-牛心果碱, 而S-牛心果碱是吗啡、可待因、诺司卡品、延胡索乙素等BIAs的共同前体[14]。暨南大学何蓉蓉教授团队[56]在苦茶(Cammelia assamica) 中找到苦茶碱合成过程中的关键酶9-N-甲基转移酶CkTcS, 并阐明其催化机制, 为培育富含苦茶碱的茶叶品种或异源合成苦茶碱奠定理论基础。目前研究人员在甲基转移酶的挖掘和应用方面取得较大进展, 实现了如苯丙烷类化合物、生物碱等多种高附加值天然产物的结构修饰, 但大多数甲基转移酶具有较强的催化杂泛性, 导致副产物产生, 使其在工程菌构建中应用受限[57], 因此甲基转移酶的改造提升是其开发利用的重要方向。随着蛋白质工程的逐渐发展以及定向突变和晶体结构的深入研究, 将为高效利用甲基转移酶作为生物催化剂提供更有效手段。
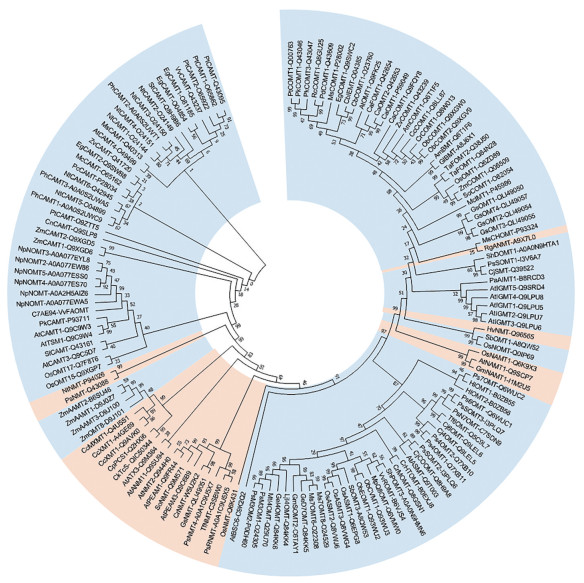
|
Figure 4 Phylogenetic analysis of methyltransferase genes in plant natural product biosynthesis pathway. O-MTs are in blue background. N-MTs are in pink background. Sequences were downloaded from NCBI or Unipro |
植物天然产物途径创建的关键是解析生物合成途径, 获得催化元件, 并根据元件催化特性对其进行适当改造, 从而为途径创建提供优质催化元件。植物天然产物的前体合成过程较保守, 且研究较透彻, 因此其下游合成途径的结构修饰酶的筛选和功能研究是关键。结构后修饰酶大多以基因家族的形式存在于植物基因组中, 其生物合成途径中基因的表达相对较弱, 因此从庞大组学数据中筛选出候选催化元件, 并进行功能研究具有一定的难度, 近年相关研究的积累为植物天然产物的生物合成途径解析提供了丰富的案例和参考。
2.1 生物合成途径元件的高效筛选植物天然产物生物合成途径研究过程中, 首要步骤是对基因元件进行挖掘和筛选。随着高通量测序技术的飞速发展, 转录组、代谢组、基因组等组学分析为生物元件的挖掘提供数据支持。元件挖掘常用的方法包括基于同源基因克隆和基于差异分析的比较组学筛选, 针对未知途径的解析主要依赖于后者, 依据不同发育阶段或不同组织部位成分含量差异的特点来筛选与代谢积累相关的候选基因。在研究秋水仙碱生物合成过程中, Nett等[58]通过代谢组学研究发现嘉兰(Gloriosa superba) 根茎部分秋水仙碱含量最多, 通过比较组学和共表达分析, 成功鉴定了8个与N-甲酰秋水仙胺生物合成相关的基因, 近乎完整地阐释了秋水仙碱的生物合成途径。Ono等[59]通过分析芝麻(Sesamum indicum) 6个生长发育阶段中芝麻素等木脂素类化合物的含量差异, 推测在种子发育后期, 芝麻素的生物合成才被激活, 通过基因表达谱与芝麻素的积累模式分析, 找到参与芝麻素生物合成的CYP81Q1基因。在研究吗啡生物合成途径的最后关键步骤中, Hagel等[60]将不含吗啡但高含蒂巴因、oripavine和可待因的罂粟突变品种与3个吗啡积累品种进行转录组比较, 发现只有一种编码为2-氧戊二酸/铁(Ⅱ) 依赖双加氧酶(DIOX1) 基因的转录水平在罂粟突变品种中显著低于其他3个吗啡积累品种, 通过功能研究找到thebaine 6-O-demethylase (T6ODM) 和codeine O-demethylase (CODM), 这是唯一报道的催化O-去甲基化的2-酮戊二酸/铁(Ⅱ) 依赖双加氧酶。这些基于组织表达差异分析、生长发育阶段差异分析、以及品种含量差异分析的基因筛选策略广泛应用于未知功能基因筛选, 能够有效缩小候选基因范围, 提高研究效率。
当植物受到生物或非生物因子刺激时, 会诱导次生代谢途径基因的表达和次生代谢产物的积累。作者课题组利用酵母和银离子诱导丹参(Salviae miltiorrhiae) 毛状根, 导致丹参酮大量积累, 同时丹参酮合成相关的上游途径基因表达上调, 基于此从比较转录组中筛选到多个参与丹参酮生物合成的P450基因, 有效推进了丹参酮生物合成途径的解析[25, 26, 61]。Han等[62]用茉莉酸甲酯诱导人参不定根使人参皂苷积累上调, 从上调基因中筛选到CYP716A47基因, 不仅能够在茉莉酸甲酯诱导下被转录激活, 还在过表达角鲨烯合酶的转基因人参中被激活, 功能鉴定证明CYP716A47基因具有催化达玛烯二醇氧化生成原人参二醇的功能。有研究发现利用酵母提取物和茉莉酸甲酯分别诱导蒺藜苜蓿(Medicago truncatula) 毛状根, 导致其三萜皂苷积累出现不同响应, 30多种三萜皂苷只有部分受茉莉酸甲酯诱导, 这些差异表型为三萜皂苷生物合成途径元件挖掘提供了非常好的筛选材料[63]。
2.2 生物合成途径催化元件功能研究获得候选基因后, 通常利用体外功能研究对其在生物合成途径的催化作用进行分析, 较常用的异源表达体系包括基于大肠杆菌和酵母细胞的微生物表达体系、以及近几年在植物天然产物生物合成途径解析中应用较多的烟草表达体系。通过克隆基因全长, 在异源体系中表达候选基因蛋白, 加入相应底物进行催化并检测, 从而确定生物合成途径元件的催化功能。
2.2.1 微生物表达系统应用于生物元件催化功能研究真核细胞酿酒酵母和原核细胞大肠杆菌是目前常用的微生物表达宿主。大肠杆菌拥有高效的异源蛋白表达效率、异源蛋白含量高、蛋白纯化体系成熟、有适合不同蛋白的商业化表达菌株和载体可选择, 并且培养周期短, 因此使用更广泛。大多数情况下, 包括萜类合酶、糖基转移酶和甲基转移酶等蛋白均能用大肠杆菌进行很好的表达, 并进行下一步的体外催化功能研究[40, 56, 64]。有研究者通过改造P450基因的膜定位序列, 也能实现P450在大肠杆菌中的表达和功能研究[65]。但由于P450主要定位于植物细胞内质网中, 因此用大肠杆菌进行P450基因的表达和功能研究仍具有一定的技术难度, 大多数情况下很难获得有功能的蛋白, 因此利用酿酒酵母来表达P450基因是最佳选择。P450是植物天然产物生物合成中参与度较高的一类酶, 参与植物次生代谢的P450需要P450还原酶或者细胞色素B5为其传递电子, 因此研究者基于P450的异源表达特点, 在酿酒酵母染色体上整合了一个来自拟南芥的P450还原酶基因(cytochrome P450 reductase, CPR), 构建了一个专用于植物P450表达和功能研究的酿酒酵母菌株WAT11[66]。目前这个菌株已经广泛用于P450基因的表达和功能研究, 并且由于酵母表达相对于早期使用的昆虫细胞表达体系操作更简单, 基本替代了昆虫细胞表达体系, 已被用于丹参酮、雷公藤、苄基异喹啉类生物碱等天然产物生物合成途径的功能基因研究[25, 61, 67, 68]。
2.2.2 烟草表达系统应用于生物元件催化功能研究除了微生物体系, 植物体系尤其是烟草表达体系也是近几年应用较多的植物功能基因异源表达体系[69, 70]。植物固有的光合作用和碳利用机制, 可以利用光合作用的还原能力驱动P450酶产生化学反应, 较微生物宿主表达P450s有很大改善[71]。与微生物相比, 植物具有蛋白表达和发挥功能的细胞器、辅酶、辅酶因子以及前体物质, 更利于植物来源蛋白表达和表达后修饰[4]。烟草作为应用较多的模式植物, 其转基因技术体系成熟, 自1989年第一次用烟草表达抗体, 烟草已经成为生产重组蛋白的主要工具, 其成熟且简单可行的瞬时表达体系, 为植物来源基因的表达和功能研究提供了新的体系。2015年发表在Science的合成依托泊苷苷元的研究中[72], 研究人员在本氏烟草(Nicotiana benthamiana) 中对桃儿七(Podophyllum hexandrum) 中的29个候选基因进行了组合表达, 成功鉴定出6个途径催化酶, 包括一个酮戊二酸依赖双加氧酶, 该双加氧酶能够闭合芳基四氢萘支架的核心环己烷环。Christ等[73]利用本氏烟草进行基因瞬时表达来筛选重楼(Paris polyphylla) 和葫芦巴(Trigonella foenum–graecum) 中的P450s, 成功鉴定出薯蓣皂苷元生物合成的关键P450基因。在秋水仙碱生物合成途径的研究中, Nett等[58]利用本氏烟草对合成途径的甲基转移酶和P450进行鉴定, 成功在本氏烟草中重构了一条含有16个基因的合成路径, 实现了从简单氨基酸到N-甲酰秋水仙胺的转化。随着生物合成途径解析的深入, 更多酶类型的发现, 烟草表达将成为植物来源基因的表达和功能研究更适合的体系。
2.2.3 催化元件的体内功能研究生物合成元件在植物体内发挥功能是研究植物天然产物形成及调控的基础, 但遗传转化体系不成熟限制了如RNA干扰、过表达或者CRISPR/Cas9等技术的应用。幸运的是随着越来越多的研究关注植物天然产物的合成, 不少植物渐渐构建了遗传转化体系, 比如丹参[61]、青蒿(Artemisia annua)[69]、颠茄(Atropa belladonna)[74]等。这些遗传转化体系的建立, 为药用活性成分生物合成途径的解析提供了技术平台。作者实验室从自交6代高度纯合的丹参植株基因组中找到4个CYP71D候选基因, 使用RNAi方法将其敲除, 获得转基因植株, 发现CYP71Ds-RNAi植株与野生型植株的根相比表现出明显的橙色表型, 进一步的生化研究表明CYP71Ds在丹参酮呋喃环形成中发挥重要作用[61]。海军军医大学张磊教授团队[75]利用CRISPR/Cas9技术对丹参酚酸类化合物合成途径中的迷迭香酸合成酶(SmRAS) 进行编辑, 获得突变毛状根, 检测发现迷迭香酸和丹酚酸B的含量降低, 证明SmRAS在丹参酚酸类化合物的生物合成途径中发挥重要作用。这些体内功能研究为系统了解植物天然产物代谢网络及调控机制提供支撑, 并为植物代谢工程改良奠定基础。
2.3 新技术新方法应用于功能基因筛选和功能研究计算生物学和合成生物学的发展、基因数据库的扩张以及生物合成途径功能元件的积累为植物天然产物生物合成途径解析提供了新方法和策略。基于合成生物学的植物天然产物途径边解析边重建的方法, 解决了代谢中间产物难以获取的问题。次丹参酮二烯作为丹参酮和雷公藤内酯的前体化合物, 在植物中含量非常低, 难以分离获取, 限制了途径的进一步解析, 通过构建次丹参酮二烯的酵母工程菌, 为下游P450功能基因研究提供了底盘和前体, 并为途径解析提供了基础平台[6, 76, 77]。除此之外, 研究者通过计算生物学的方法从数据库中挖掘天然产物生物合成途径可能的功能元件, 通过功能研究和途径重构, 实现了阿片类化合物[8]、苄基异喹啉类生物碱[78]等天然产物的生物合成, 计算生物学方法的应用能够更精确地获取候选基因并进行功能研究, 有效减少工作量、提高筛选效率。
基因组数据和转录组数据的积累、基因合成成本的下降为高通量的自动化筛选提供了数据支撑, 已应用于酶的改造和细胞工厂筛选。在采用随机或定向进化的方法提高酶和细胞工厂性能时, 突变文库的多样性以及可靠的高通量筛选方法是成败的关键因素[79, 80]。传统的筛选方法如琼脂平板和微孔板筛选法存在无法精准定量或者通量低、操作耗时等缺点, 而近年来开发的荧光激活细胞分选(FACS) 和液滴微流控分选(DMFS) 等超高通量筛选方法, 大大提高了筛选通量, 可用于酶和细胞工厂定向改造中大容量突变库的筛选[81]。Ma等[82]开发了一种超高通量双通道液滴微流控筛选系统, 每天可筛选多达107种酶突变体。基于该系统设计酯酶的对映选择性, 以优先生产具有抗炎作用的profens对映异构体, 在经历五轮定向进化后, 从两种荧光信号检测的筛选模式中确定了一种对(S)-profens选择性提高700倍的突变体, 新系统的应用显著提升了筛选效率。植物天然产物生物合成后修饰酶基因通常以基因家族的形式存在于基因组中, 候选基因数量较多, 具有普适性的高通量技术的开发应用能够更快推动功能基因的筛选和生物合成途径解析。
3 植物天然产物的合成生物学生产近年来, 随着生物合成途径的解析以及合成生物学使能技术的发展, 植物天然产物的合成生物学生产逐渐成为了生物合成研究的一个重要应用方向。从青蒿素的合成生物学生产到阿片类化合物在酵母中的途径创建, 合成生物学为天然产物的研究带来了一场新的革命, 为天然产物获取提供了新的方式。在微生物中实现植物天然产物高产需要从元件、途径、细胞三个层面进行系统改造, 最后利用发酵工程实现高效生产[83]。
3.1 生物元件的改造合成生物学元件(biological parts) 包括用于调控基因表达的调控元件(启动子、终止子、核糖体结合位点), 以及特定功能的结构元件(合成途径中特定的催化酶基因) 等[84]。虽然目前紫杉醇[85]、青蒿素[5]、吗啡[8]等植物天然产物生物合成途径的解析取得突破并正在逐步实现合成生物学生产, 但对于大多数植物天然产物, 其合成生物学产量仍然无法与植物提取相比。途径元件的改造和高效催化是亟需解决的问题, 作为研究热点, 酶的定向进化和理性改造在催化机制研究以及催化性能提升中发挥重要作用。
3.1.1 利用定向进化提升生物元件催化效率植物天然产物作为次生代谢产物, 在植物中的积累量较低, 而受限于次生代谢途径酶的天然特性, 相关基因如P450或萜类合酶等的表达量低, 催化活性弱, 其应用进一步受限。定向进化是一种应用广泛的改变酶催化特性的策略, 由于其在酶工程领域的重要作用, 于2018年获得诺贝尔化学奖。定向趋异进化是在定向进化和趋异进化理论上建立的一种重新设计酶催化功能的蛋白质工程学方法, 能够将天然蛋白改造成底物专一性各不相同的多种蛋白。中国科学院微生物研究所唐双焱研究员团队[86]利用蛋白质定向趋异进化策略对大肠杆菌单加氧酶HpaBC进行了改造, 获得了具有酪醇/酪胺羟化酶混杂催化活性的优良突变体H7, 它能够有效减少蛋白质过表达造成的细胞代谢负担, 显著提高羟基酪醇生物合成效率, 在没有优化发酵条件的情况下, 使羟基酪醇产量达到1 890 mg·L-1, 转化率可达到82%。定向进化与高通量筛选技术的结合共同提升酶的改造效率。Ellington团队[87]通过易错PCR构建了樟脑诱导型PCamR/CamR突变库, 筛选出能够特异性响应冰片、芬酚、桉油醇和莰烯等二环单萜的生物传感器, 实现了对二环单萜化合物的快速检测, 显著提升突变体库的筛选效率, 该策略为定向进化在酶改造研究中的应用提供案例。
3.1.2 生物元件的理性设计对于蛋白质晶体结构、催化机制较清晰的酶, 除了利用定向进化产生大量的突变体之外, 还可以采用理性设计的方法进行定向改造[83]。天津工业生物技术研究所孙媛霞研究员团队[35]在对罗汉果(Siraitia grosvenorii) 中糖基转移酶UGT74AC1的催化特性和晶体结构认识的基础上, 设计一系列突变体, 将该酶对三萜类化合物的催化效率提升100到10 000倍, 之后利用优势突变体的体外催化功能合成了一系列新的三萜皂苷化合物。随后孙媛霞研究员团队和孙周通研究员团队合作在罗汉果中发现了一个新的糖基转移酶UGT74AC2, 并采用聚焦理性迭代定点突变(focused rational iterative site-specific mutagenesis, FRISM) 的理性设计策略, 成功获得催化水飞蓟宾不同位置羟基的突变体, 实现了含氮和硫底物的催化功能[36]。定向的进化工程策略以及理性的设计方法可以有目标地改善酶的活性、热稳定性、对底物的亲和力和偏好性, 进一步提高从前体到产物的转化效率。
3.2 生物合成途径的设计与优化植物天然产物途径解析和关键元件的挖掘改造是其合成生物学生产的基础, 而在微生物中重构生物合成途径并进行设计和优化是其利用合成生物学高效生产的关键。合成途径长、催化酶类型多是影响产量提高的主要因素, 并且部分天然产物存在一定的细胞毒性, 导致产量提升困难, 因此途径创建和基于细胞整体的代谢流调控对于目标化合物产量提升非常重要。近几年对于天然产物合成生物学菌株改造的策略很多, 过表达关键酶基因和抑制竞争途径[5, 8, 76, 88]、蛋白融合[76]或蛋白支架[89]的方法是合成生物学基因工程菌构建的常用手段[90, 91], 最近针对降低代谢物细胞毒性的代谢产物区室化存储、传感器的应用、改善细胞内环境等产量提升策略也发挥越来越重要的作用[83]。
3.2.1 代谢流调控代谢流调控主要通过调控关键节点基因的表达从而提升目标化合物的产量, 包括过表达关键限速酶、降低或沉默竞争途径等。比如3-羟基-3-甲基戊二酰辅酶A还原酶(3-hydroxy-3-methylglutaryl coenzyme A reductase, HMGR) 是萜类生物合成的关键限速酶, 在工程菌中过表达HMGR基因的C-端结构域, 能显著提高萜类化合物的产量并提高细胞稳定性[92], 这一策略在青蒿素、丹参酮等天然产物基因工程菌构建中被广泛应用[5, 77]。在单萜生物合成中, Li等[93]通过将相对较弱的启动子替换为竞争途径元件的启动子, 使工程菌中单萜的产量提高了1.7~21.5倍。在苯丙素类化合物咖啡酸的工程菌构建中, 研究人员通过解除关键前体苯丙氨酸的反馈抑制并敲除竞争途径的关键基因使酵母中的咖啡酸产量提高2.6倍[94]。在脂肪酸的合成中, 中国科学院青岛能源研究所崔球研究员团队[95]通过过表达脂肪酸前体合成的关键酶6-磷酸葡萄糖脱氢酶(glucose-6-phosphate dehydrogenase, G6PDH) 和乙酰辅酶A羧化酶(acetyl-CoA carboxylase, ACC) 强化脂肪酸前体供给途径, 并且过表达脂肪酸贮存中的关键限速酶二酰基甘油酰基转移酶(diacylglycerol acyltransferase, DGAT), 结合弱化产物的竞争途径-脂肪酸合酶途径, 最终获得了合成高纯度二十二碳六烯酸(DHA) 的细胞工厂(331 mg·g-1)。虽然基因敲除或过表达等静态调控策略在提高天然产物生物合成的代谢通量方面具有非常有效的作用, 但容易引起代谢资源不足从而导致异源细胞生长迟缓。江南大学刘立明教授课题组[96]将稳定期启动子(SPP) 和蛋白质降解标签(PDT) 与CRISPR干扰系统结合, 构建了一个动态调控系统, 能够根据细胞的生理状态关闭多基因表达, 在5 L的发酵罐中得到了21 g·L-1的莽草酸。
3.2.2 减少有毒中间体的积累在异源表达天然产物时, 大量外源基因过表达或有毒中间体的积累可能会影响宿主细胞生长, 使用动态调控启动子是一种减少或消除有毒中间体积累的有效策略[97]。法呢基焦磷酸(farnesyl diphosphate, FPP) 和3-羟基-3-甲基戊二酰辅酶A (3-hydroxy-3-methylglutaryl coenzyme A reductase, HMG-CoA) 是萜类生物合成中的有毒中间体, Dahl等[98]通过使用动态调控启动子对有毒中间体进行实时调控, 在不使用任何诱导促进剂的情况下, 利用两个FPP反应促进剂, 有效减少有毒中间体的积累, 显著提高了基因工程菌中青蒿素前体紫穗槐二烯的产量。上海植物生理生态研究所陈晓亚研究员课题组[99]发现棉花中的糖代谢解毒酶(SPG) 在丢失N端的细胞器定位信号肽和谷胱甘肽(GSH) 结合位点后, 可以高效催化棉酚生物合成途径中的中间体芳香化且不需要辅因子参与, 并且SPG可以催化复杂反应, 具有潜在的解毒功能, 这些酶的发现为新型芳香剂合成提供了新的改造靶点。
3.2.3 代谢物的动态监测依赖于生物传感器(biosensor) 的代谢物动态监测是近年的研究热点, 在减少有毒中间体积累、提升目标产物产量中也发挥重要作用。生物传感器依靠对目标代谢物的特异敏感性, 可将代谢物的变化信息通过多种信号实时输出, 实现动态监测和反馈, 从而调节和平衡生物合成途径、提高天然产物产量[100]。通过与高通量筛选相结合能加快代谢途径以及生产菌株的选择过程, 是解决异源生产植物天然产物瓶颈的有效方法。Chou等[101]构建了一个动态控制全基因组突变率的传感器执行器电路, 当目标代谢物浓度较低时, 突变率增加, 在群体中产生多样性, 并导致新表型的进化; 当目标代谢物浓度增加时, 通过反馈调节导致突变率降低, 从而降低表型多样性, 利用该传感器成功使酪氨酸和番茄红素的产量增加5倍和3倍。上海交通大学赵心清教授课题组[102]以大肠杆菌木糖激活因子(XylR) 及相应结合位点(xylO) 为基础构建了突变型木糖传感器, 该突变型生物传感器不仅扩大了木糖的浓度检测范围, 也可用于β-半乳糖苷酶和番茄红素等生物合成途径的木糖诱导表达, 为其他生物传感器的设计和进化提供了新见解。目前生物传感器已广泛应用于生物质化学品的合成生物学工程菌构建和产量检测中, 相信未来针对天然产物积累或代谢流调控的生物传感器的开发和应用将有效助推天然产物基因工程菌产量提升研究。
3.2.4 亚细胞区室化策略许多结构复杂的天然产物在微生物的异源合成中含量较低, 易产生副反应, 这是由于在天然产物生物合成中, 部分酶需在特定的亚细胞区室(如细胞质、线粒体、内质网、过氧化物酶体、高尔基体、细胞壁或液泡等) 中定位发挥催化活性[103]。亚细胞区室化的优势在于不仅具有独特的生理化学环境、代谢物、酶和辅因子, 其细胞质间的物理间隔在消除代谢干扰以及增强区室化途径效率方面也具有良好效果[104]。斯坦福大学Smolke课题组[105]在研究托品烷类生物碱的生物合成时, 运用亚细胞区室化策略将来自酵母、细菌、植物和动物中的20多种酶定位到不同的亚细胞中(包括细胞质、线粒体、过氧化物酶体、液泡等), 解决了酶在不同环境中的适配性以及代谢物转运等问题, 实现了莨菪碱和东莨菪碱在酵母中的生物合成。单萜类化合物如柠檬酸、冰片等对细胞生长具有较大影响, 研究者通过将单萜途径定位至过氧化物酶体, 使酵母中单萜的产量提升到胞质表达的125倍[106]。该策略也成功应用于人参皂苷[107]以及BIAs合成生物学研究中。天津工业生物技术研究所张学礼研究员团队[107]在研究三萜化合物人参皂苷的合成途径时利用脂滴膜蛋白Pln1p将定位于内质网中的关键P450酶(PPDS) 定向到底物(达玛烯二醇-Ⅱ, DD) 存储的细胞器脂滴中, 使底物DD的转化率显著提高, 在导入人参皂苷Compound K生物合成高效模块后, 人参皂苷产量最终滴度达到5 g·L-1。去甲乌药碱合酶(NCS) 是BIAs合成途径的第一个关键酶, 但高活性的NCS突变体在酿酒酵母细胞质中表达时具有毒性。加利福尼亚大学Dueber课题组[108]将NCS定位至过氧化物酶体, 利用底物和产物在过氧化物酶体膜上的自由流动性, 降低了NCS细胞毒性, 之后进一步模拟脂肪酸诱导过氧化物酶体增殖, 将BIA产量提高了74%。除了过氧化物酶体, 线粒体工程在工程菌构建中亦有报道。线粒体中含有丰富的乙酰辅酶A和氧化还原等价物, 是进行萜类化合物生物合成的良好亚细胞区室, 但线粒体工程存在代谢负担, 会导致细胞生长不良。华东理工大学魏东芝教授课题组[109]在进行角鲨烯的生物合成时在酵母线粒体中引入甲羟戊酸途径并增强细胞质中甲羟戊酸的合成, 以此减轻线粒体区室化MVA途径引起的代谢负担, 促进了细胞生长, 最终发酵获得高产角鲨烯21.1 g·L-1。植物天然产物合成途径复杂、合成路径长, 酶的亚细胞定位或代谢途径的细胞区室化能在一定程度上聚集代谢途径、减少代谢物的交流、抑制竞争途径, 实现产量提升。
4 展望合成生物学技术经过20余年的发展, 在农业、医药、能源、环境等各领域发挥重要作用, 是十四五期间国家科技前沿重点攻关领域。近期国家发改委与工业和信息化部关于推动原料药产业高质量发展实施方案的通知将合成生物学和生物催化剂作为原料药的先进制造技术创新工程的重点发展任务。结合生物技术、计算机技术与合成生物学在不同层面(酶、途径和细胞) 对微生物合成过程进行设计、调控和优化, 不仅能够实现植物天然产物的途径创建和微生物生产, 而且能够对植物天然产物进行进一步的结构修饰和改造, 提升其开发利用价值。与植物天然产物的传统获取方式相比, 这种新的资源获取策略以青蒿素、β-榄香烯等为代表的工程菌构建和生产方面的应用, 体现其在资源可持续利用、稳定、高效等方面的显著优势, 实现了植物天然产物的绿色低碳制造。
植物天然产物生物合成研究一方面为合成生物学生产提供了元件和基础, 另一方面通过植物代谢工程也逐渐应用于农业、医药等领域。植物本身含有或编码酶基因, 具有类似结构和功能的细胞器、辅酶、辅酶因子以及前体物质, 有利于植物来源蛋白表达和翻译后修饰, 更适于复杂天然产物生物合成途径的解析以及基于基因编辑技术的药用植物改良, 减少可能存在的代谢途径不清晰等造成的困难, 并逐渐成为了植物天然产物合成生物学生产的方向[4]。比如番茄(Lycopersicon esculentum) 已被广泛用于如虾青素[110]、单萜[111]、花青素[112]、黄酮醇[113]等天然产物的生产。另外利用本氏烟草作为底盘重构多个天然产物的通路, 合成了如长春花碱[114]、鬼臼毒素[72]、红景天苷[115]等天然产物。未来随着植物基因组测序技术的成熟以及基因编辑工具的开发和编辑效率的提升, 在植物中以全局代谢网络为基础的代谢途径优化理念和操作的进一步突破, 将迎来植物源天然产物途径创建的新时代。
植物天然产物的生物合成及合成生物学研究已取得了突破进展, 成为了植物天然产物研究的重要方向之一, 但是目前仍有许多亟待解决的问题[116]: 生物合成途径长、结构修饰酶家族大等因素影响元件筛选和途径解析, 许多复杂小分子的生物合成途径仍然未完全解析; 即使途径已知, 但多个元件组装到一起仍需要克服组件间的协调问题; 因此随着途径工程越来越复杂, 途径工程的建设和优化过程也是异常艰巨; 有些植物天然产物对微生物细胞的增殖有影响, 难以实现高产。针对以上问题, 理论和技术的发展将不断促进这一领域的发展, 植物天然产物的产生是基因组复制和新功能化的结果, 由于植物基因组庞大、候选基因多, 迫切需要高通量检测技术的开发来配合基因功能研究, 从而推动元件的功能筛选; 人工智能已应用到药物的筛选和设计, 与药物与靶蛋白作用机制类似, 生物合成途径酶与底物的互作也符合分子动力学原理, 随着Alphafold2的应用、量子计算的发展, 我们已步入途径计算设计的时期; 定向进化、半理性设计的元件改造、底盘驯化、生物传感器、自动化高通量合成生物学工作站等技术的应用, 在植物天然产物基因工程菌产量提升上发挥越来越重要的作用; 定量合成生物学是合成生物学的前沿, 能够实现在基因工程菌中精准地调控、分配和亚细胞区室化生成多组分天然产物, 可以预见未来中药复方的合成生物学生产将成为中药创新制剂研发的一种方式。总之, 不断增加的科技投入将推动植物天然产物生物合成及合成生物学领域朝着高速、多元化的方向发展。
作者贡献: 郭娟、黄璐琦负责文章结构框架的设计以及文章的修改工作; 刘秀玉负责整体文章的撰写和CYP450系统进化树的构建; 罗凌龙和胡志敏负责CYP450数据的整理; 马莹负责文章思路的整理和内容修改; 卜俊玲负责甲基转移酶数据的整理; 孙术富、唐金富负责糖基转移酶数据的整理; 崔光红负责酰基转移酶数据的整理。
利益冲突: 本文的作者无任何利益冲突。
| [1] |
Liu X, Ding W, Jiang H. Engineering microbial cell factories for the production of plant natural products: from design principles to industrial-scale production[J]. Microb Cell Fact, 2017, 16: 125. DOI:10.1186/s12934-017-0732-7 |
| [2] |
Farnsworth NR, Akerele O, Bingel AS, et al. Medicinal plants in therapy[J]. Bull World Health Organ, 1985, 63: 965-981. |
| [3] |
De Luca V, Salim V, Atsumi SM, et al. Mining the biodiversity of plants: a revolution in the making[J]. Science, 2012, 33: 1658-1661. |
| [4] |
Fu R, Martin C, Zhang Y. Next-generation plant metabolic engineering, inspired by an ancient chinese irrigation system[J]. Mol Plant, 2018, 11: 47-57. DOI:10.1016/j.molp.2017.09.002 |
| [5] |
Paddon CJ, Westfall PJ, Pitera DJ, et al. High-level semi-synthetic production of the potent antimalarial artemisinins[J]. Nature, 2013, 496: 528-529. DOI:10.1038/nature12051 |
| [6] |
Dai Z, Liu Y, Huang L, et al. Production of miltiradiene by metabolically engineered Saccharomyces cerevisiae[J]. Biotechnol Bioeng, 2012, 109: 2845-2853. DOI:10.1002/bit.24547 |
| [7] |
Ro DK, Paradise EM, Ouellet M, et al. Production of the antimalarial drug precursor artemisinic acid in engineered yeast[J]. Nature, 2006, 440: 940-943. DOI:10.1038/nature04640 |
| [8] |
Stephanie G, Kate T, Isis JT, et al. Complete biosynthesis of opioids in yeast[J]. Science, 2015, 349: 1095-1100. DOI:10.1126/science.aac9373 |
| [9] |
Cai T, Sun H, Qiao J, et al. Cell-free chemoenzymatic starch synthesis from carbon dioxide[J]. Science, 2021, 373: 1523-1527. DOI:10.1126/science.abh4049 |
| [10] |
Huang LQ, Gao W, Zhou YJ. Application of synthetic biology to sustainable utilization of Chinese materia medica resources[J]. Acta Pharm Sin (药学学报), 2014, 49: 37-43. |
| [11] |
Jiang FL, Gong T, Chen JJ, et al. Synthetic biology of plants-derived medicinal natural products[J]. Chin J Biotech (生物工程学报), 2021, 37: 1931-1951. |
| [12] |
Neelam, Khatkar A, Sharma KK. Phenylpropanoids and its derivatives: biological activities and its role in food, pharmaceutical and cosmetic industries[J]. Crit Rev Food Sci Nutr, 2020, 60: 2655-2675. DOI:10.1080/10408398.2019.1653822 |
| [13] |
Hu Z, Liu X, Tian M, et al. Recent progress and new perspectives for diterpenoid biosynthesis in medicinal plants[J]. Med Res Rev, 2021, 41: 2971-2997. DOI:10.1002/med.21816 |
| [14] |
Dastmalchi M, Park MR, Morris JS, et al. Family portraits: the enzymes behind benzylisoquinoline alkaloid diversity[J]. Phytochem Rev, 2018, 17: 249-277. DOI:10.1007/s11101-017-9519-z |
| [15] |
Chang WC, Song H, Liu HW, et al. Current development in isoprenoid precursor biosynthesis and regulation[J]. Curr Opin Chem Biol, 2013, 17: 571-579. DOI:10.1016/j.cbpa.2013.06.020 |
| [16] |
Tohge T, Watanabe M, Hoefgen R, et al. The evolution of phenylpropanoid metabolism in the green lineage[J]. Crit Rev Biochem Mol, 2013, 48: 123-152. DOI:10.3109/10409238.2012.758083 |
| [17] |
Ziegler J, Facchini PJ. Alkaloid biosynthesis: metabolism and trafficking[J]. Annu Rev Plant Biol, 2008, 59: 735-769. DOI:10.1146/annurev.arplant.59.032607.092730 |
| [18] |
Nelson D, Werck-Reichhart D. A P450-centric view of plant evolution[J]. Plant J, 2011, 66: 194-211. DOI:10.1111/j.1365-313X.2011.04529.x |
| [19] |
Mizutani M, Ohta D. Diversification of P450 genes during land plant evolution[J]. Annu Rev Plant Biol, 2010, 61: 291-315. DOI:10.1146/annurev-arplant-042809-112305 |
| [20] |
Hamberger B, Bak S. Plant P450s as versatile drivers for evolution of species-specific chemical diversity[J]. PhilosTrans R Soc Lond B Biol Sci, 2013, 368: 20120426. DOI:10.1098/rstb.2012.0426 |
| [21] |
Ma Y, Cai Y, Ma XJ, et al. Research progress of P450 in the biosynthesis of bioactive compound of medicinal plants[J]. Acta Pharm Sin (药学学报), 2020, 55: 1573-1589. |
| [22] |
Coon MJ. Cytochrome P450: nature's most versatile biological catalyst[J]. Annu Rev Pharmacol, 2005, 45: 1-25. DOI:10.1146/annurev.pharmtox.45.120403.100030 |
| [23] |
Croteau R, Ketchum R, Long RM, et al. Taxol biosynthesis and molecular genetics[J]. Phytochem Rev, 2006, 5: 75-97. DOI:10.1007/s11101-005-3748-2 |
| [24] |
Pateraki I, Andersen-Ranberg J, Jensen NB, et al. Total biosynthesis of the cyclic AMP booster forskolin from Coleus forskohlii[J]. Elife, 2017, 6: e23001. DOI:10.7554/eLife.23001 |
| [25] |
Guo J, Ma X, Cai Y, et al. Cytochrome P450 promiscuity leads to a bifurcating biosynthetic pathway for tanshinones[J]. New Phytol, 2016, 210: 525-534. DOI:10.1111/nph.13790 |
| [26] |
Guo J, Zhou YJ, Hillwig ML, et al. CYP76AH1 catalyzes turnover of miltiradiene in tanshinones biosynthesis and enables heterologous production of ferruginol in yeasts[J]. Proc Natl Acad Sci U S A, 2013, 110: 12108-12113. DOI:10.1073/pnas.1218061110 |
| [27] |
Ikezawa N, Iwasa K, Sato F. Molecular cloning and characterization of CYP80G2, a cytochrome P450 that catalyzes an intramolecular C-C phenol coupling of (S)-reticuline in magnoflorine biosynthesis, from cultured Coptis japonica cells[J]. J Biol Chem, 2008, 283: 8810-8821. DOI:10.1074/jbc.M705082200 |
| [28] |
Ikezawa N, Iwasa K, Sato F. CYP719A subfamily of cytochrome P450 oxygenases and isoquinoline alkaloid biosynthesis in Eschscholzia californica[J]. Plant Cell Rep, 2009, 28: 123-133. DOI:10.1007/s00299-008-0624-8 |
| [29] |
Seki H, Sawai S, Ohyama K, et al. Triterpene functional genomics in licorice for identification of CYP72A154 involved in the biosynthesis of glycyrrhizin[J]. Plant Cell, 2011, 23: 4112-4123. DOI:10.1105/tpc.110.082685 |
| [30] |
Wüst M, Little DB, Schalk M, et al. Hydroxylation of limonene enantiomers and analogs by recombinant (-)-limonene 3- and 6-hydroxylases from mint (Mentha) species: evidence for catalysis within sterically constrained active sites[J]. Arch Biochem Biophys, 2001, 387: 125-136. DOI:10.1006/abbi.2000.2248 |
| [31] |
Chen X, Fu RR, Zhang RR, et al. Research progress of UGT family in medicinal plants[J]. Biot Resour (生物资源), 2018, 40: 443-449. |
| [32] |
Wilson AE, Tian L. Phylogenomic analysis of UDP-dependent glycosyltransferases provides insights into the evolutionary landscape of glycosylation in plant metabolism[J]. Plant J, 2019, 100: 1273-1288. DOI:10.1111/tpj.14514 |
| [33] |
Zhang M, Li FD, Li K, et al. Functional characterization and structural basis of an efficient di-C-glycosyltransferase from Glycyrrhiza glabra[J]. J Am Chem Soc, 2020, 142: 3506-3512. DOI:10.1021/jacs.9b12211 |
| [34] |
Chen D, Fan S, Chen R, et al. Probing and engineering key residues for bis-C-glycosylation and promiscuity of a C-glycosyltransferase[J]. ACS Catal, 2018, 8: 4917-4927. DOI:10.1021/acscatal.8b00376 |
| [35] |
Li J, Yang J, Mu S, et al. Efficient O-glycosylation of triterpenes enabled by protein engineering of plant glycosyltransferase UGT74AC1[J]. ACS Catal, 2020, 10: 3629-3639. DOI:10.1021/acscatal.9b05232 |
| [36] |
Li J, Qu G, Shang N, et al. Near-perfect control of the regioselective glucosylation enabled by rational design of glycosyltransferases[J]. Green Synth Catal, 2021, 2: 45-53. DOI:10.1016/j.gresc.2021.01.005 |
| [37] |
Xie K, Zhang X, Sui S, et al. Exploring and applying the substrate promiscuity of a C-glycosyltransferase in the chemo-enzymatic synthesis of bioactive C-glycosides[J]. Nat Commun, 2020, 11: 5162. DOI:10.1038/s41467-020-18990-9 |
| [38] |
Caputi L, Malnoy M, Goremykin V, et al. A genome-wide phylogenetic reconstruction of family 1 UDP-glycosyltransferases revealed the expansion of the family during the adaptation of plants to life on land[J]. Plant J, 2012, 69: 1030-1042. DOI:10.1111/j.1365-313X.2011.04853.x |
| [39] |
Ross J, Li Y, Lim E, et al. Higher plant glycosyltransferases[J]. Genome Biol, 2001, 2: reviews3004. |
| [40] |
Liu S, Zhang M, Bao Y, et al. Characterization of a highly selective 2''-O-galactosyltransferase from trollius chinensis and structure-guided engineering for improving UDP-glucose selectivity[J]. Org Lett, 2021, 23: 9020-9024. DOI:10.1021/acs.orglett.1c02581 |
| [41] |
Yin Q, Zhang J, Wang S, et al. N-Glucosyltransferase GbNGT1 from ginkgo complements the auxin metabolic pathway[J]. Hort Res, 2021, 8: 229. DOI:10.1038/s41438-021-00658-0 |
| [42] |
Ito T, Fujimoto S, Suito F, et al. C-Glycosyltransferases catalyzing the formation of di-C-glucosyl flavonoids in citrus plants[J]. Plant J, 2017, 91: 187-198. DOI:10.1111/tpj.13555 |
| [43] |
Wang ZL, Gao HM, Wang S, et al. Dissection of the general two-step di-C-glycosylation pathway for the biosynthesis of (iso)schaftosides in higher plants[J]. Proc Natl Acad Sci U S A, 2020, 117: 30816-30823. DOI:10.1073/pnas.2012745117 |
| [44] |
Nagatomo Y, Usui S, Ito T, et al. Purification, molecular cloning and functional characterization of flavonoid C-glucosyltransferases from Fagopyrum esculentum M. (buckwheat) cotyledon[J]. Plant J, 2014, 80: 437-448. DOI:10.1111/tpj.12645 |
| [45] |
Yan X, Fan Y, Wei W, et al. Production of bioactive ginsenoside compound K in metabolically engineered yeast[J]. Cell Res, 2014, 24: 770-773. DOI:10.1038/cr.2014.28 |
| [46] |
Wei W, Wang P, Wei Y, et al. Characterization of Panax ginseng UDP-glycosyltransferases catalyzing protopanaxatriol and biosyntheses of bioactive ginsenosides F1 and Rh1 in metabolically engineered yeasts[J]. Mol Plant, 2015, 8: 1412-1424. DOI:10.1016/j.molp.2015.05.010 |
| [47] |
Bontpart T, Cheynier V, Ageorges A, et al. BAHD or SCPL acyltransferase? What a dilemma for acylation in the world of plant phenolic compounds[J]. New Phytol, 2015, 208: 695-707. DOI:10.1111/nph.13498 |
| [48] |
Wang L, Chen K, Zhang M, et al. Catalytic function, mechanism, and application of plant acyltransferases[J]. Crit Rev Biotechnol, 2021. DOI:10.1080/07388551.2021.1931015 |
| [49] |
D'auria JC. Acyltransferases in plants: a good time to be BAHD[J]. Curr Opin Plant Biol, 2006, 9: 331-340. DOI:10.1016/j.pbi.2006.03.016 |
| [50] |
Ma X, Koepke J, Panjikar S, et al. Crystal structure of vinorine synthase, the first representative of the BAHD superfamily[J]. J Biol Chem, 2005, 280: 13576-13583. DOI:10.1074/jbc.M414508200 |
| [51] |
Nevarez DM, Mengistu YA, Nawarathne IN, et al. An N-aroyltransferase of the BAHD superfamily has broad aroyl CoA specificity in vitro with analogues of N-dearoylpaclitaxel[J]. J Am Chem Soc, 2009, 131: 5994-6002. DOI:10.1021/ja900545m |
| [52] |
Fu R, Zhang P, Jin G, et al. Versatility in acyltransferase activity completes chicoric acid biosynthesis in purple coneflower[J]. Nat Commun, 2021, 12: 1563. DOI:10.1038/s41467-021-21853-6 |
| [53] |
Liu YY, Mo T, Wang XH, et al. Research progress of plant BAHD acyltransferase family[J]. China J Chin Mater Med (中国中药杂志), 2016, 41: 2175-2182. |
| [54] |
Yang Y, Yuan JS, Ross J, et al. An Arabidopsis thaliana methyltransferase capable of methylating farnesoic acid[J]. Arch Biochem Biophys, 2006, 448: 123-132. DOI:10.1016/j.abb.2005.08.006 |
| [55] |
Wang ZG, Wu JX. DNA methyltransferases: classification, functions and reseach progress[J]. Hereditas (遗传), 2009, 31: 903-912. |
| [56] |
Zhang YH, Li YF, Wang Y, et al. Identification and characterization of N9-methyltransferase involved in converting caffeine into non-stimulatory theacrine in tea[J]. Nat Commun, 2020, 11: 1473. DOI:10.1038/s41467-020-15324-7 |
| [57] |
Zhang XY, Shen XL, Sun XX, et al. Application of methyltransferases in microbial synthesis of natural products[J]. Chin J Biotech (生物工程学报), 2021, 37: 18. |
| [58] |
Nett RS, Lau W, Sattely ES. Discovery and engineering of colchicine alkaloid biosynthesis[J]. Nature, 2020, 584: 148-153. DOI:10.1038/s41586-020-2546-8 |
| [59] |
Ono E, Nakai M, Fukui Y, et al. Formation of two methylenedioxy bridges by a Sesamum CYP81Q protein yielding a furofuran lignan, (+)-sesamin[J]. Proc Natl Acad Sci U S A, 2006, 103: 10116-10121. DOI:10.1073/pnas.0603865103 |
| [60] |
Hagel JM, Facchini PJ. Dioxygenases catalyze the O-demethylation steps of morphine biosynthesis in opium poppy[J]. Nat Chem Biol, 2010, 6: 273-275. DOI:10.1038/nchembio.317 |
| [61] |
Ma Y, Cui G, Chen T, et al. Expansion within the CYP71D subfamily drives the heterocyclization of tanshinones synthesis in Salvia miltiorrhiza[J]. Nat Commun, 2021, 12: 685. DOI:10.1038/s41467-021-20959-1 |
| [62] |
Han JY, Kim HJ, Kwon YS, et al. The Cyt P450 enzyme CYP716A47 catalyzes the formation of protopanaxadiol from dammarenediol-Ⅱ during ginsenoside biosynthesis in Panax ginseng[J]. Plant Cell Physiol, 2011, 52: 2062-2073. DOI:10.1093/pcp/pcr150 |
| [63] |
Naoumkina MA, Modolo LV, Huhman DV, et al. Genomic and coexpression analyses predict multiple genes involved in triterpene saponin biosynthesis in Medicago truncatula[J]. Plant Cell, 2010, 22: 850-866. DOI:10.1105/tpc.109.073270 |
| [64] |
Zhao Y, Hu T, Liu R, et al. Biochemical characterization and function of eight microbial type terpene synthases from lycophyte Selaginella moellendorffii[J]. Int J Mol Sci, 2021, 22: 605. DOI:10.3390/ijms22020605 |
| [65] |
Zi J, Peters RJ. Characterization of CYP76AH4 clarifies phenolic diterpenoid biosynthesis in the Lamiaceae[J]. Org Biomol Chem, 2013, 11: 7650-7652. DOI:10.1039/c3ob41885e |
| [66] |
Urban P, Mignotte C, Kazmaier M, et al. Cloning, yeast expression, and characterization of the coupling of two distantly related Arabidopsis thaliana NADPH-cytochrome P450 reductases with P450 CYP73A5[J]. J Biol Chem, 1997, 272: 19176-19186. DOI:10.1074/jbc.272.31.19176 |
| [67] |
Liu X, Bu J, Ma Y, et al. Functional characterization of (S)-N-methylcoclaurine 3'-hydroxylase (NMCH) involved in the biosynthesis of benzylisoquinoline alkaloids in Corydalis yanhusuo[J]. Plant Physiol Biochem, 2021, 168: 507-515. DOI:10.1016/j.plaphy.2021.09.042 |
| [68] |
Tu L, Su P, Zhang Z, et al. Genome of Tripterygium wilfordii and identification of cytochrome P450 involved in triptolide biosynthesis[J]. Nat Commun, 2020, 11: 971. DOI:10.1038/s41467-020-14776-1 |
| [69] |
Farhi M, Marhevka E, Ben-Ari J, et al. Generation of the potent anti-malarial drug artemisinin in tobacco[J]. Nat Biotechnol, 2011, 29: 1072-1074. DOI:10.1038/nbt.2054 |
| [70] |
Reed J, Osbourn A. Engineering terpenoid production through transient expression in Nicotiana benthamiana[J]. Plant Cell Rep, 2018, 37: 1431-1441. DOI:10.1007/s00299-018-2296-3 |
| [71] |
Ikram NK, Zhan X, Pan XW, et al. Stable heterologous expression of biologically active terpenoids in green plant cells[J]. Front Plant Sci, 2015, 6: 129. |
| [72] |
Lau W, Sattely ES. Six enzymes from mayapple that complete the biosynthetic pathway to the etoposide aglycone[J]. Science, 2015, 349: 1224-1228. DOI:10.1126/science.aac7202 |
| [73] |
Christ B, Xu C, Xu M, et al. Repeated evolution of cytochrome P450-mediated spiroketal steroid biosynthesis in plants[J]. Nat Commun, 2019, 10: 3206. DOI:10.1038/s41467-019-11286-7 |
| [74] |
Zhang Q, Liang M, Liu Y, et al. Development of homozygous transgenic Atropa belladonna plants with glyphosate resistance and high-yield scopolamine using metabolic engineering[J]. Ind Crop Prod, 2021, 171: 113953. DOI:10.1016/j.indcrop.2021.113953 |
| [75] |
Zhou Z, Tan H, Li Q, et al. CRISPR/Cas9-mediated efficient targeted mutagenesis of RAS in Salvia miltiorrhiza[J]. Phytochemistry, 2018, 148: 63-70. DOI:10.1016/j.phytochem.2018.01.015 |
| [76] |
Zhou YJ, Gao W, Rong Q, et al. Modular pathway engineering of diterpenoid synthases and the mevalonic acid pathway for miltiradiene production[J]. J Am Chem Soc, 2012, 134: 3234-3241. DOI:10.1021/ja2114486 |
| [77] |
Hu T, Zhou J, Tong Y, et al. Engineering chimeric diterpene synthases and isoprenoid biosynthetic pathways enables high-level production of miltiradiene in yeast[J]. Metab Eng, 2020, 60: 87-96. DOI:10.1016/j.ymben.2020.03.011 |
| [78] |
Hafner J, Payne J, Mohammadipeyhani H, et al. A computational workflow for the expansion of heterologous biosynthetic pathways to natural product derivatives[J]. Nat Commun, 2021, 12: 1760. DOI:10.1038/s41467-021-22022-5 |
| [79] |
Zeng W, Guo L, Xu S, et al. High-throughput screening technology in industrial biotechnology[J]. Trends Biotechnol, 2020, 38: 888-906. DOI:10.1016/j.tibtech.2020.01.001 |
| [80] |
Tan Y, Zhang Y, Han Y, et al. Directed evolution of an α1, 3-fucosyltransferase using a single-cell ultrahigh-throughput screening method[J]. Sci Adv, 2019, 5: eaaw8451. DOI:10.1126/sciadv.aaw8451 |
| [81] |
Yang JH, Su XL, Zhu LL. Advances of high-throughput screening system in reengineering of biological entities[J]. Chin J Biotech (生物工程学报), 2021, 37: 2197-2210. |
| [82] |
Ma F, Chung MT, Yao Y, et al. Efficient molecular evolution to generate enantioselective enzymes using a dual-channel microfluidic droplet screening platform[J]. Nat Commun, 2018, 9: 1030. DOI:10.1038/s41467-018-03492-6 |
| [83] |
Dai ZB, Wang Y, Zhou ZH, et al. Synthetic biology for production of plant-derived natural products[J]. Bull Chin Acad Sci (中国科学院院刊), 2018, 33: 1228-1238. |
| [84] |
Wang JF, Meng HL, Xiong ZQ, et al. Design and construction of artificial biological systems for complex natural products biosynthesis[J]. Chin J Biotech (生物工程学报), 2013, 29: 1146-1160. |
| [85] |
Guerra-Bubb J, Croteau R, Williams RM. The early stages of taxol biosynthesis: an interim report on the synthesis and identification of early pathway metabolites[J]. Nat Prod Rep, 2012, 29: 683. DOI:10.1039/c2np20021j |
| [86] |
Chen W, Yao J, Meng J, et al. Promiscuous enzymatic activity-aided multiple-pathway network design for metabolic flux rearrangement in hydroxytyrosol biosynthesis[J]. Nat Commun, 2019, 10: 960. DOI:10.1038/s41467-019-08781-2 |
| [87] |
D'oelsnitz S, Nguyen V, Alper HS, et al. Evolving a generalist biosensor for bicyclic monoterpenes[J]. bioRxiv, 2021, DOI. DOI:10.1101/2021.08.20.457167 |
| [88] |
Liu Q, Yu T, Li X, et al. Rewiring carbon metabolism in yeast for high level production of aromatic chemicals[J]. Nat Commun, 2019, 10: 4976. DOI:10.1038/s41467-019-12961-5 |
| [89] |
Ji D, Li J, Xu F, et al. Improve the biosynthesis of baicalein and scutellarein via manufacturing self-assembly enzyme reactor in vivo[J]. ACS Synth Bio, 2021, 10: 1087-1094. DOI:10.1021/acssynbio.0c00606 |
| [90] |
Cravens A, Payne J, Smolke CD. Synthetic biology strategies for microbial biosynthesis of plant natural products[J]. Nat commun, 2019, 10: 2142. DOI:10.1038/s41467-019-09848-w |
| [91] |
Yang D, Park SY, Park YS, et al. Metabolic engineering of Escherichia coli for natural product biosynthesis[J]. Trends Biotechnol, 2020, 38: 745-765. DOI:10.1016/j.tibtech.2019.11.007 |
| [92] |
Polakowski T, Stahl U, Lang C. Overexpression of a cytosolic hydroxymethylglutaryl-CoA reductase leads to squalene accumulation in yeast[J]. Appl Microbiol Biotechnol, 1998, 49: 66-71. DOI:10.1007/s002530051138 |
| [93] |
Li R, Wang K, Wang D, et al. Production of plant volatile terpenoids (rose oil) by yeast cell factories[J]. Green Chem, 2021, 23: 5088-5096. DOI:10.1039/D1GC00917F |
| [94] |
Zhou P, Yue C, Shen B, et al. Metabolic engineering of Saccharomyces cerevisiae for enhanced production of caffeic acid[J]. Appl Microbiol Biotechnol, 2021, 105: 5809-5819. DOI:10.1007/s00253-021-11445-1 |
| [95] |
Wang Z, Wang S, Feng Y, et al. Obtaining high-purity docosahexaenoic acid oil in thraustochytrid Aurantiochytrium through a combined metabolic engineering strategy[J]. J Agric Food Chem, 2021, 69: 10215-10222. DOI:10.1021/acs.jafc.1c03781 |
| [96] |
Gao C, Guo L, Hu G, et al. Engineering a CRISPRi circuit for autonomous control of metabolic flux in Escherichia coli[J]. ACS Synth Bio, 2021, 10: 2661-2671. DOI:10.1021/acssynbio.1c00294 |
| [97] |
Luo Y, Enghiad B, Zhao H. New tools for reconstruction and heterologous expression of natural product biosynthetic gene clusters[J]. Nat Prod Rep, 2016, 33: 174-182. DOI:10.1039/C5NP00085H |
| [98] |
Dahl RH, Zhang F, Alonso-Gutierrez J, et al. Engineering dynamic pathway regulation using stress-response promoters[J]. Nat Biotechnol, 2013, 31: 1039-1046. DOI:10.1038/nbt.2689 |
| [99] |
Huang JQ, Fang X, Tian X, et al. Aromatization of natural products by a specialized detoxification enzyme[J]. Nat Chem Biol, 2020, 16: 250-256. DOI:10.1038/s41589-019-0446-8 |
| [100] |
Wang D, Dai ZB, Zhang XL. Production of plant-derived natural products in yeast cells - a review[J]. Acta Microbiol Sin (微生物学报), 2016, 56: 516-529. |
| [101] |
Chou HH, Keasling JD. Programming adaptive control to evolve increased metabolite production[J]. Nat Commun, 2013, 4: 2595. DOI:10.1038/ncomms3595 |
| [102] |
Tang RQ, Wagner JM, Alper HS, et al. Design, evolution, and characterization of a xylose biosensor in Escherichia coli using the XylR/xylO system with an expanded operating range[J]. ACS Synth Biol, 2020, 9: 2714-2722. DOI:10.1021/acssynbio.0c00225 |
| [103] |
Cao X, Yang S, Cao C, et al. Harnessing sub-organelle metabolism for biosynthesis of isoprenoids in yeast[J]. Syn Syst Biotechnol, 2020, 5: 179-186. DOI:10.1016/j.synbio.2020.06.005 |
| [104] |
Hammer SK, Avalos JL. Harnessing yeast organelles for metabolic engineering[J]. Nat Chem Biol, 2017, 13: 823-832. DOI:10.1038/nchembio.2429 |
| [105] |
Srinivasan P, Smolke CD. Biosynthesis of medicinal tropane alkaloids in yeast[J]. Nature, 2020, 585: 614-619. DOI:10.1038/s41586-020-2650-9 |
| [106] |
Dusséaux S, Wajn WT, Liu Y, et al. Transforming yeast peroxisomes into microfactories for the efficient production of high-value isoprenoids[J]. Proc Natl Acad Sci U S A, 2020, 117: 31789-31799. DOI:10.1073/pnas.2013968117 |
| [107] |
Shi Y, Wang D, Li R, et al. Engineering yeast subcellular compartments for increased production of the lipophilic natural products ginsenosides[J]. Metab Eng, 2021, 67: 104-111. DOI:10.1016/j.ymben.2021.06.002 |
| [108] |
Grewal PS, Samson JA, Baker JJ, et al. Peroxisome compartmentalization of a toxic enzyme improves alkaloid production[J]. Nat Chem Biol, 2021, 17: 96-103. DOI:10.1038/s41589-020-00668-4 |
| [109] |
Zhu ZT, Du MM, Gao B, et al. Metabolic compartmentalization in yeast mitochondria: burden and solution for squalene overproduction[J]. Metab Eng, 2021, 68: 232-245. DOI:10.1016/j.ymben.2021.10.011 |
| [110] |
Huang JC, Zhong YJ, Liu J, et al. Metabolic engineering of tomato for high-yield production of astaxanthin[J]. Metab Eng, 2013, 17: 59-67. DOI:10.1016/j.ymben.2013.02.005 |
| [111] |
Gutensohn M, Nguyen TT, Mcmahon RD, et al. Metabolic engineering of monoterpene biosynthesis in tomato fruits via introduction of the non-canonical substrate neryl diphosphate[J]. Metab Eng, 2014, 24: 107-116. DOI:10.1016/j.ymben.2014.05.008 |
| [112] |
Butelli E, Titta L, Giorgio M, et al. Enrichment of tomato fruit with health-promoting anthocyanins by expression of select transcription factors[J]. Nat Biotechnol, 2008, 26: 1301-1308. DOI:10.1038/nbt.1506 |
| [113] |
Luo J, Butelli E, Hill L, et al. AtMYB12 regulates caffeoyl quinic acid and flavonol synthesis in tomato: expression in fruit results in very high levels of both types of polyphenol[J]. Plant J, 2008, 56: 316-326. DOI:10.1111/j.1365-313X.2008.03597.x |
| [114] |
Caputi L, Franke J, Farrow SC, et al. Missing enzymes in the biosynthesis of the anticancer drug vinblastine in Madagascar periwinkle[J]. Science, 2018, 360: 1235-1239. DOI:10.1126/science.aat4100 |
| [115] |
Liu X, Tang Y, Zeng J, et al. Biochemical characterization of tyrosine aminotransferase and enhancement of salidroside production by suppressing tyrosine aminotransferase in Rhodiola crenulata[J]. Ind Crop Prod, 2021, 173: 114075. DOI:10.1016/j.indcrop.2021.114075 |
| [116] |
Kwok R. Five hard truths for synthetic biology[J]. Nature, 2010, 463: 288-290. DOI:10.1038/463288a |
 2021, Vol. 56
2021, Vol. 56


