2. 中国中医科学院中药研究所, 北京 100700
2. Institute of Chinese Meteria Medica, China Academy of Chinese Medical Sciences, Beijing 100700, China
MADS-box基因普遍存在于动物、植物和真菌中, 是一类在植物成花诱导及花器官发育等方面具有重要调控作用的转录因子, 能够调控生长发育和信号转导[1, 2]。MADS-box转录因子家族主要分为Ⅰ和Ⅱ两类, Ⅰ类具有MADS (M)、Intervening (I)、Keratin-like (K) 和Carboxyl-terminal (C) 4个结构域, 故亦称MIKC基因[3]。该基因家族具有高度保守的结构域, 其中MADS结构域对其功能至关重要, K域是蛋白二聚化的关键结构域, 它们分别是DNA结合和蛋白复合物形成所不可缺少的[4, 5]。MIKC型MADS-box基因可根据I区序列的不同分为MIKCc和MIKC*两个亚家族, MIKC*通常在雄配子发育中发挥重要作用, MIKCc基因则在花器官生长发育等方面相对于研究更多具有重要意义[6, 7]。在目前与植物花发育分子机制相关的ABCDE模型中, MADS-box基因所涉及的同源二聚体和异源二聚体(花的四聚体), 它们能调节不同花序中的特定表达过程[8]。MIKC基因在花发育的各个阶段都发挥作用, 大多数MIKC型MADS-box成员均是花器官决定基因, 在花形态建成、调节开花时间等方面具有重要作用[9]。
大麻(Cannabis sativa L.) 是大麻科(Cannabinaceae) 大麻属(Cannabis) 一年生草本植物, 栽培历史悠久, 在世界各地均有种植[10]。大麻的用途十分广泛, 不但在各种工业产品、生物建筑、隔热材料等方面有应用[11, 12], 在药理特性方面如在治疗癫痫、破伤风、风湿病和失眠等方面同样具有良好的功效, 常常被称为天然的治疗草药[13-15]。
在药用植物大麻中大麻素是一种具有精神活性的酚萜类物质, 它们主要在腺毛中合成和积累[16]。随着大麻全基因组序列的公布, 考虑到MADS-box基因家族在植物开花调控中的重要作用, 以及该基因家族在药用开花植物中的基础信息缺乏, 对大麻MIKC型MADS-box基因家族全序列进行较为全面的研究, 有助于更好地挖掘相关基因的生物信息学功能, 为进一步探究大麻MIKC型基因在花发育调控、形态建成中的作用提供理论基础。
材料与方法材料 从NCBI (https://www.ncbi.nlm.nih.gov) 网站上获取大麻(GCA_900626175.1) 和拟南芥(GCA_000005425.2) 的全基因组及注释文件, 品种为CRBRx的雌株。转录组数据分为两部分, 9个不同品种腺毛的RNA-seq数据(PRJNA498707) 获取于NCBI数据库[17], 品种名称分别为Black Berry Kush (BB)、Black Lime (BL)、Canna Tsu (CT)、Mama Thai (MT)、Valley Fire (VF)、Cherry Chem (CC)、Terple (T)、Sour Diesel (SD) 和White Cookies (WC); 同一品种Diku植株的茎、叶、苞片和雌花的RNA-seq数据是由课题组通过测序获得, Diku品种的全称为Dinamed Kush CBD Autoflowering, 是Purple Kush和Dinamed Autoflowering CBD的杂交品种。
大麻MIKC型MADS-box家族成员的鉴定、蛋白特征分析及染色体定位 大麻CsMADS基因家族鉴定使用TBtools[18]软件提取大麻蛋白序列, 利用PlantTFDB网站进行转录因子预测, 将预测得到的目的家族基因成员进行筛选, 去除可变剪切导致的重复转录本, 并利用TBtools软件获取大麻CsMADS的蛋白序列并解析大麻的注释文件, 同时使用在线生物信息学工具ExPASy-ProSite (http://web.expasy.org/protparam/) 对39个CsMADS蛋白的理化性质进行预测。利用Cell-PLoc (http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/) 网站进行亚细胞定位预测。同时从基因组注释文件中筛选出基因家族成员ID, 并利用TBtools软件确定每个CsMADS基因的位置信息并绘制其所对应的染色体物理位置图。
大麻MIKC型MADS-box基因保守结构域、基因结构分析与GO功能分析 利用MEME (http://meme-suite.org/tools/meme) 对39个CsMADS蛋白质保守基序进行挖掘与分析, 目标保守基序数量设置为10。利用基因组注释文件对大麻CsMADS基因进行解析, 最终通过TBtools软件对大麻MIKC家族的基因进行基因结构、保守结构域和保守基序三者可视化分析。利用OmicsBox分析工具对所有鉴定出的CsMADS转录因子序列进行氨基酸相似性比对、鉴定和筛选, 并对筛选出的转录因子进行GO功能分类。
大麻MIKC型MADS-box系统发育树构建及共线性分析 利用39个CsMADS的蛋白序列, 与TAIR数据库(https://www.arabidopsis.org/) 搜索到的拟南芥(Arabidopsis thaliana) 45个MIKC家族基因蛋白序列, 应用MEGA-X比对工具采取NJ (Neighbour-join) 方法构建分子进化树, 并进行了重复1 000次的Bootstrap检验, 所有的设定均采用默认值。使用TBtools软件对大麻和拟南芥共线性关系进行可视化, 获得两者的同源基因对。
大麻MIKC型MADS-box基因顺式作用元件预测及表达模式分析 使用TBtools软件提取39个CsMADS基因上游2 000 bp启动子序列, 利用PlantCare (http://www.plantcare.co.uk/) 在线网站对其进行顺式作用元件预测, 通过TBtools软件对其进行可视化构图。基于实验室测序获得的一个杂交品种Diku的雌花、苞片、茎和叶的转录组数据以及9个不同栽培品种雌性花序的转录组数据分析基因的差异表达情况, 利用TBtools软件进行表达模式热图的绘制, 对CsMADS成员基因表达量(FPKM) 使用log2(FPKM+1) 对数据进行标准化处理。
大麻MIKC型MADS-box基因实时荧光定量PCR分析 利用Liu等[19]筛选的EF1a作为内参基因, 对转录组数据中存在表达量差异的CsMADS4、CsMADS7、CsMADS12、CsMADS29进行实时荧光定量PCR (quantitative real-time PCR, qRT-PCR) 验证, 利用NCBI设计引物(表 1)。实验样品为大麻(Diku) 的雌花、苞片、茎和叶, 每组试验3个重复, 采用WARYONG试剂盒提取样品RNA, 用TransGen试剂盒进行cDNA合成, 以及TransStart试剂盒进行实时荧光定量PCR分析。反应程序为: 95 ℃酶激活30 s, 95 ℃变性5 s, 56 ℃退火15 s, 72 ℃延伸10 s, 循环次数40。CsMADS的相对表达量采用2-ΔΔCt法计算。
结果与分析 1 大麻MIKC型MADS-box基因的鉴定与染色体分布从大麻基因组数据库筛选并鉴定出63个MIKC型MADS-box基因, 去除可变剪切导致的重复转录本和结构域确认后, 最终获得39个MIKC型MADS-box基因家族成员, 依次命名为CsMADS1~CsMADS39。利用大麻MIKC家族的基因ID, 使用TBtools软件获得CsMADS的蛋白序列。结果如表 2所示, 其中氨基酸数量最少的是CsMADS34, 包含146个氨基酸残基, 数量最多的为CsMADS38, 由503个氨基酸组成。蛋白质等电点最小的为CsMADS26, 其PI为5.09, 最大的是CsMADS20, 其PI为10.12。其中所有蛋白的分子质量在16 739.35~57 070.56 Da之间。亚细胞定位预测结果显示39个CSMADS蛋白均集中在细胞核定位。
CsMADS基因家族39个基因不均匀地分布在9条染色体上(NC_044370.1、NC_044371.1、NC_044372.1、NC_044373.1、NC_044374.1、NC_044375.1、NC_044377.1、NC_044378.1、NC_044379.1分别分布了6、3、5、5、3、4、1、1、11个CsMADS基因) (图 1)。每条染色体排列的基因数目差别很大, 其中NC_044379.1染色体上CsMADS基因家族数目最多, 含有11个基因, 最少的是NC_044377.1和NC_044378.1均只分布1个基因。并发现NC_044370.1染色体上的CsMADS15和CsMADS16, NC_044375.1染色体上的CsMADS33和CsMADS35、CsMADS32和CsMADS34; NC_044379.1染色体上的CsMADS12、CsMADS7和CsMADS3出现了基因成簇排列现象。另外在同一条染色体中出现的多基因存在串联重复特征, 如NC_044372.1染色体上的CsMADS20和CsMADS22、NC_044379.1染色体上的CsMADS5和CsMADS8。
| Table 1 Primer sequences of CsMADS |
| Table 2 The information of 39 CsMADS genes in Cannabis sativa |

|
Figure 1 Distribution of 39 CsMADS genes on nine chromosomes in Cannabis sativa |
基因家族成员是通过不断拷贝进化而来, 在序列上具有保守性和相关性[20]。通过MEME在线网站分析基因序列, 获得10个相对保守性较高的基序(图 2)。其中motif之间的位置具有一定规律, 在所鉴定的39个基因中均存在高度保守的MADS区(motif 1), 而且除了MIKC*亚类、CsMADS15和CsMADS16之外, 其余基因均存在较为保守的K-box区, 在AGL15、AGL17、API和AGL6亚类中具有motif1-motif3-motif5-motif2-motif4的相对稳定结构, 并未出现保守基序增加、替换或缺失现象, 但在不同亚类中保守基序数量存在不同, 说明在基因家族进化过程中结构特征存在差异。
通过分析39个CsMADS基因的非翻译区(UTR, untranslated region)、内含子和外显子结构, 发现大多数基因UTR数量位于0~2之间, 其中87.2%的基因含有2个, 约7.7%的基因只含有1个; 外显子数量较为保守, 除了CsMADS26由11个外显子组成外, 其他大多数基因的外显子数量处于6~8之间; 大部分基因包含3~9个不等的内含子, 其中CsMADS2、CsMADS12以及CsMADS30的单个内含子长度较大, 但有3个CsMADS基因(CsMADS15、CsMADS16和CsMADS38) 没有内含子, 只有一个外显子; 同一进化支上基因的结构较为保守, 例如SEP/AGL2 (CsMADS9、CsMADS10、CsMADS21和CsMADS24) 和AGL6 (CsMADS7、CsMADS12、CsMADS29) 进化支上的基因均由8个外显子和7个内含子组成(图 3)。
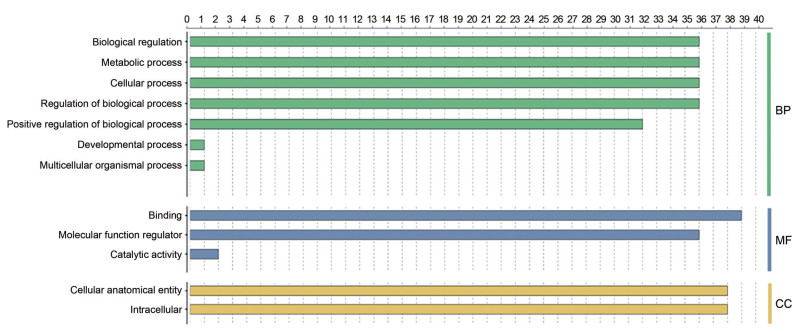
3 大麻MIKC型MADS-box基因GO富集分析利用OmicsBox软件进行GO功能注释分析, 结果显示39个CsMADS基因共注释到331个GO terms (图 4), 生物过程(biological process, BP)、分子功能(molecular function, MF) 和细胞组分(cellular component, CC) 涉及的GO terms分别为: 178、77和76个。其中DNA binding富集个数最多, 在所有基因中均有注释; 细胞组分中细胞结构组分和胞内组分均富集到38个转录因子, 生物过程中细胞过程也相对较多, 富集数目为36个。
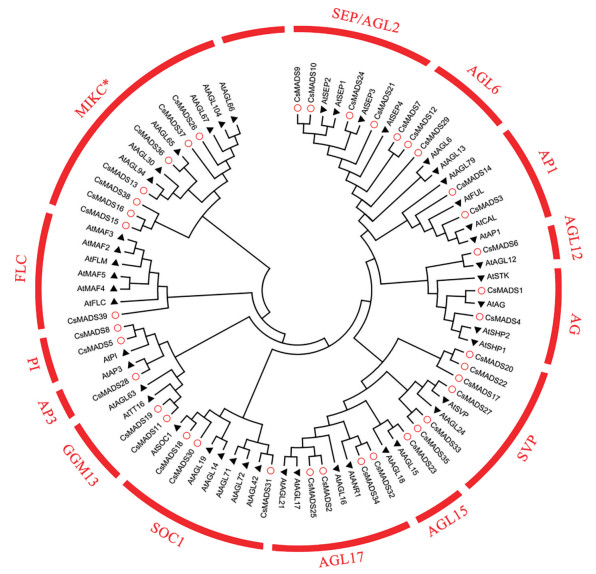
4 大麻MIKC型MADS-box蛋白系统发育树的构建及家族进化特性为探究大麻MIKC型MADS-box基因家族的进化关系, 使用MEGA-X软件对大麻39个和拟南芥45个蛋白序列构建Neighbor-join (NJ) 树。大麻39个CsMADS基因明显分为14个进化支, 即AG、AGL12、MIKC*、AGL15、AGL17、API、AGL6、SEP/AGL2、PI、SVP、AP3、FLC、SOC1和GGM13, FLC进化支在拟南芥中共有6个基因, 是春化开花调控途径的关键基因, 但是在大麻中该进化支上只有1个同源基因, 即CsMADS39 (图 5)。不同进化支上大麻分布的基因数量略有差异, 如在调控开花时间的SVP进化支中, 大麻的基因数目明显多于拟南芥, 在拟南芥中有2个基因, 在大麻中却有6个同类基因; AGL17和SEP/AGL2进化支中, 拟南芥和大麻基因数量相同, 均有4个基因; 而在PI进化支中, 大麻的基因数量较少, 只有2个, 即CsMADS5和CsMADS8, 这类基因均为控制花形态建成的ABCDE模型中的同源异型基因, 推测大麻在花器官形态建成中可能存在更加复杂和精细的调控机制。

|
Figure 2 The conservation domain of 39 CsMADS genes in Cannabis sativa |

|
Figure 3 Conserved motifs and gene structure analyses of 39 CsMADS genes of Cannabis sativa |
|
Figure 4 GO categorization and annotation of 39 CsMADS genes |
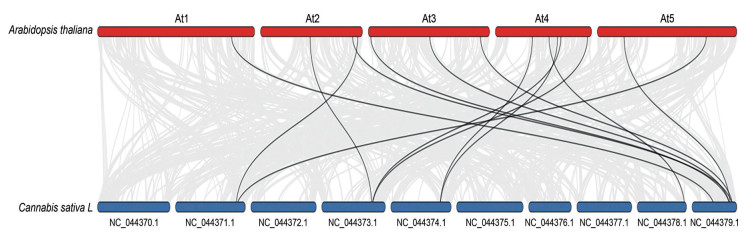
利用共线性原理, 探究大麻CsMADS基因和拟南芥基因家族成员的共线性关系并可视化, 结果显示, 大麻和拟南芥间共有14对同源基因, 分别为CsMADS18/AtSOC1、CsMADS18/AtAGL72、CsMADS27/AtSVP、CsMADS27/AtAGL24、CsMADS25/AtAGL21、CsMADS30/AtAGL19、CsMADS30/AtAGL14、CsMADS1/AtAG、CsMADS3/AtAP1、CsMADS4/AtSHP2、CsMADS4/AtSHP1、CsMADS9/AtSEP2、CsMADS9/AtSEP1、CsMADS14/AtAGL79 (图 6), 与大麻和拟南芥构建的系统发育树结果一致。
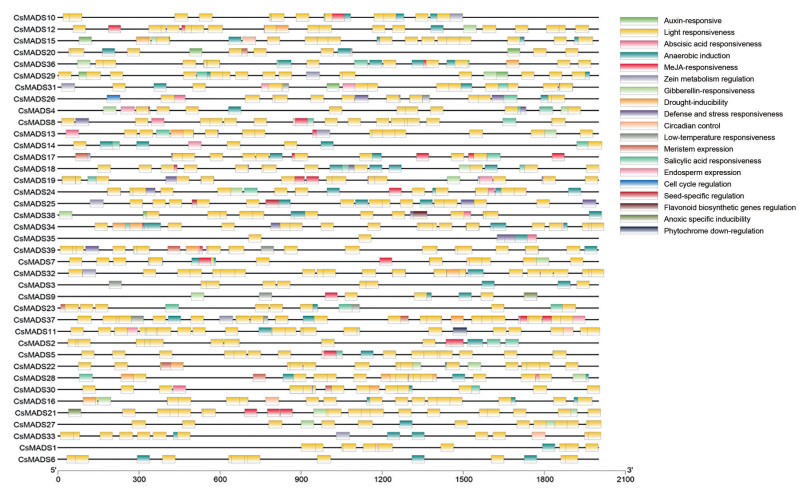
6 大麻MIKC型MADS-box家族基因顺式元件预测顺式作用元件可以调控基因表达, 分析启动子及其上游顺式作用元件能预测相应的基因功能。CsMADS基因成员启动子序列(基因上游2 000 bp) 预测到的顺式元件共19类, 其中参与光响应的顺式元件最多, 共574个, 在所有CsMADS基因中均有涉及(图 7)。其次, 预测到的响应脱落酸元件有112个、厌氧诱导顺式元件有89个; 数目最少的有光敏色素下调表达的顺式作用元件、类黄酮生物合成基因调控元件以及参与细胞周期调控作用元件, 都只含有1个, 分别存在于CsMADS11、CsMADS38和CsMADS26启动子区。另外在其他基因启动区还存在大量参与生长素反应元件、响应茉莉酸甲酯调控元件和参与水杨酸反应性顺式作用等多种顺式调控元件。
|
Figure 5 Phylogenetic trees of MIKC-type genes in Cannabis sativa and Arabidopsis thaliana |
|
Figure 6 The collinear relationship of CsMADS genes between A. thaliana and C. sativa |
|
Figure 7 cis-Acting elements within the promoter of 39 CsMADS genes in Cannabis sativa |
为揭示CsMADS基因的表达情况, 将9个大麻品种的腺毛以及大麻(Diku) 的花、苞片、茎和叶的转录组数据, 绘制基因表达热图, 并将表达模式进行聚类。在不同品种中, CsMADS29和CsMADS14在SD、CT、BL、VF、CC、WC、BB、MT和T品种中表达最高, 而CsMADS17和CsMADS20则在Diku品种中表达最高(图 8A)。在同一品种的不同部位中, CsMADS表达模式存在差异, CsMADS29和CsMADS7在花和苞片中表达上调(图 8B), CsMADS20和CsMADS22在茎中表达较高, 而CsMADS34、CsMADS38、CsMADS11、CsMADS26、CsMADS28、CsMADS33在花、苞片、茎和叶中均没有表达。表达模式分析结果表明大麻CsMADS基因成员在不同组织器官和品种中表达均存在差异性, 暗示其功能存在分化。

|
Figure 8 Expression pattern of MIKC-type MADS-box genes in Cannabis sativa. A: Indicates the expression pattern of CsMADS in female flowers of ten breeds; B: Indicates the expression pattern of CsMADS in different organs of same breed |
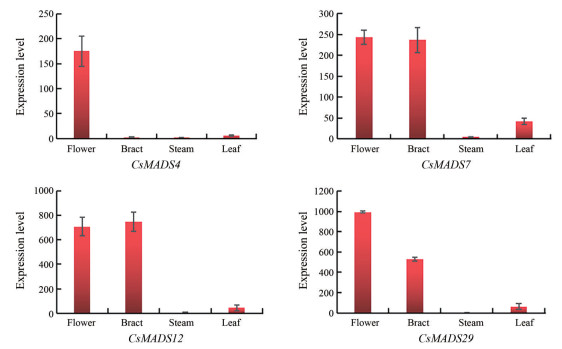
大麻腺毛位于雌花苞片的表面, 是富含大麻素和萜类物质的树脂生物合成和储存的主要场所, 而CsMADS4、CsMADS7、CsMADS12、CsMADS29正是在雌花或苞片中明显上调的基因, 利用qRT-PCR检测这4个基因在大麻(Diku) 不同组织部位的表达情况, 结果显示, CsMADS7、CsMADS12、CsMADS29在大麻Diku品种各组织部位的表达情况与转录组趋势略有不同, 但整体来说这3个基因在雌花和苞片中的表达量高于在茎和叶中的表达, 且CsMADS12和CsMADS29在花和苞片中表达量极高; CsMADS4具有明显的组织差异性, 只在花中极显著表达, 在其他部位几乎无表达(图 9)。
讨论在植物中, MIKC型MADS-box转录因子包含MADS-域和K-box域, 在植物发育和生长等多种生物过程中发挥着关键作用[21, 22]。近年来, MIKC型MADS-box基因家族的鉴定和进化分析在许多植物中均有报道, 例如在大豆(Glycine max)[23]、葡萄(Vitis vinifera)[24]、石榴(Punica granatum)[25]中, 分别鉴定到57、38、36个MIKC基因。本研究共鉴定了大麻39个CsMADS候选基因, 分布于9条染色体上, 基因之间存在串联重复现象, 编码的氨基酸大小位于146~503氨基酸之间, 理化性质存在差异, GO功能分析中, 主要富集于生物过程调控, 亚细胞定位均预测在细胞核中, 说明该家族可能参与大麻遗传代谢调控。
MIKC型MADS-box基因家族在进化过程中具有较高的保守性, 在大麻和拟南芥共线性分析中, 39个CsMADS基因共有14对旁系同源基因, 进一步与拟南芥MIKC型MADS-box家族成员共同构建系统进化树, 可分成14个亚族, 其中CsMADS3和CsMADS14属于AP1亚家族, 根据该亚族在小麦(Triticum aestivum) 中具有调控营养和生殖结构功能[26], 进一步推测该同源基因可能在调控大麻开花时间方面发挥重要作用; CsMADS5和CsMADS8基因属于PI亚家族, 该亚族在兰科(Orchidaceae) 植物花器官、萼片、花瓣、唇瓣、合蕊柱和未发育子房中均发挥作用[27]; AG亚家族基因对于子房授粉后胚珠发育具有调控作用[28], 在大麻中CsMADS1和CsMADS4属于该亚家族, 进一步推测可能对雄蕊、雌蕊和柱头的形态发育具有一定的影响。
39个CsMADS基因的保守序列分析表明, 同一亚家族成员的基序数目和序列相似, 保守基序1编码的MADS结构域, 是最保守的结构域, 其次, K-box结构域是第二保守的结构域。基因结构分析表明, 同一亚家族具有一定的相似性, CsMADS基因普遍包含6~7个内含子, 0~2个外显子。顺式作用元件存在于基因启动子中, 与转录因子形成特异性结合, 在调控靶基因表达中发挥重要作用[29]。CsMADS基因启动子序列存在种类丰富和功能多样的顺式作用元件, 其中富含光响应元件, 进一步证明最佳光质量、光量和光周期对于大麻生物量的旺盛生长是非常重要的, 光的质量和数量对光合作用有深刻的影响, 最终影响植物的生长发育[30], 另外CsMADS基因能够响应脱落酸、水杨酸、茉莉酸甲酯等激素的调控, 表明这些激素可能在大麻生长发育和应对非生物胁迫中发挥重要作用。据文献[16]记载大麻腺毛位于雌花苞片的表面, 是富含7种大麻素和萜类物质的树脂生物合成和储存的主要场所, 转录组表达谱聚类分析显示CsMADS基因具有明显的组织特异性, CsMADS7、CsMADS12和CsMADS29在花和苞片中表达上调, CsMADS4只在雌花中显著表达, 在其他部位几乎无表达, 说明其可能在植物开花或苞片的生长发育过程中发挥关键作用, 推测其可能调控大麻素生物合成; CsMADS34、CsMADS38、CsMADS11、CsMADS26、CsMADS28、CsMADS33在花、苞片、茎、叶和种子中均没有表达, 推测以上未表达的基因在该品种某一特定的生长发育过程中未发挥调控作用。通过RT-qPCR进行验证, CsMADS7、CsMADS12和CsMADS29在花和苞片中相对表达较高, 且进化树分析都聚类在一起, 均属于AGL6亚家族, 根据拟南芥相关功能推测CsMADS7、CsMADS12和CsMADS29可能在大麻开花期间促进了侧枝器官发育和苞片的形成[31]。
|
Figure 9 The expression levels of the CsMADS genes in different tissues |
综上所述, 本研究基于大麻全基因组数据, 筛选和鉴定出39个大麻MIKC型MADS-box基因, 对其进行全面的生物信息学分析, 包括序列特征、染色体定位、GO富集分类、保守结构域、基因结构、系统发育、共线性分析、顺势元件预测以及表达模式的分析和验证, 能够初步了解大麻MIKC型MADS-box基因数量、功能分类以及表达趋势, 对后续进一步开展MIKC型MADS-box基因对药用植物大麻生长发育调控研究具有一定理论价值。
作者贡献: 万志庭负责文章撰写及修改; 鲁梦负责qRT-PCR实验部分; 吴沙沙指导文章撰写并提出修改意见; 米要磊负责实验设计及指导撰写思路; 翟俊文负责文章的选题、指导文章撰写及修改。
利益冲突: 所有作者均声明不存在利益冲突。
| [1] |
Elena R, Alvarez B, Soraya P, et al. An ancestral MADS-box gene duplication occurred before the divergence of plants and animals[J]. Proc Natl Acad Sci U S A, 2000, 97: 5328-5333. DOI:10.1073/pnas.97.10.5328 |
| [2] |
Annette B, Günter T. The major clades of MADS-box genes and their role in the development and evolution of flowering plants[J]. Mol Phylogenet Evol, 2003, 29: 464-489. DOI:10.1016/S1055-7903(03)00207-0 |
| [3] |
Ma H, Yanofsky MF, Meyerowitz EM. AGL1-AGL6, an Arabidopsis gene family with similarity to floral homeotic and transcription factor genes[J]. Genes Dev, 1991, 5: 484-495. DOI:10.1101/gad.5.3.484 |
| [4] |
Kaufmann K, Melzer R, Theissen G. MIKC-type MADS-domain proteins: structural modularity, protein interactions and network evolution in land plants[J]. Gene, 2005, 347: 183-198. DOI:10.1016/j.gene.2004.12.014 |
| [5] |
Tang QL, Li NZ, Song M, et al. Identification of acting domains mediating the protein interactions between SVP and FLC in Brassica juncea Coss[J]. Acta Hort Sin (园艺研究), 2012, 39: 2395-2403. |
| [6] |
Gramzow L, Theissen G. A hitchhiker's guide to the MADS world of plants[J]. Gen Biol, 2010, 11: 214-225. DOI:10.1186/gb-2010-11-6-214 |
| [7] |
Kwantes M, Liebsch D, Verelst W. How MIKC* MADS-box genes originated and evidence for their conserved function throughout the evolution of vascular plant gametophyte[J]. Mol Biol Evol, 2012, 29: 293-302. DOI:10.1093/molbev/msr200 |
| [8] |
Soltis DE, Ma H, Frohlich MW, et al. The floral genome: an evolutionary history of gene duplication and shifting patterns of gene expression[J]. Trends Plant Sci, 2007, 12: 358-367. DOI:10.1016/j.tplants.2007.06.012 |
| [9] |
Guo P. The Phylogenetic Analysis of MADS-box Gene Family in Brassiceae Plants (十字花科MADS-box基因家族系统进化分析)[D]. Fuzhou: Fujian Agriculture & Forestry University, 2018.
|
| [10] |
McPartland JM, Guy GW, Hegman W. Cannabis is indigenous to Europe and cultivation began during the Copper or Bronze age: a probabilistic synthesis of fossil pollen studies[J]. Veg Hist Archaeobot, 2018, 27: 635-648. DOI:10.1007/s00334-018-0678-7 |
| [11] |
William L, Zhang PZ, Ying DY, et al. Extrusion improves the phenolic profile and biological activities of hempseed (Cannabis sativa L.) hull[J]. Food Chem, 2021, 346: 128606. DOI:10.1016/j.foodchem.2020.128606 |
| [12] |
Irakli M, Tsaliki E, Kalivas A, et al. Effect of genotype and growing year on the nutritional, phytochemical, and antioxidant properties of industrial hemp (Cannabis sativa L.) seeds[J]. Antioxidants, 2019, 8: 491. DOI:10.3390/antiox8100491 |
| [13] |
ElSohly MA, Sladea D. Chemical constituents of marijuana: the complex mixture of natural cannabinoids[J]. Life Sci, 2005, 78: 539-548. DOI:10.1016/j.lfs.2005.09.011 |
| [14] |
Flores-Sanchez IJ, Verpoorte R. Secondary metabolism in cannabis[J]. Phytochem Rev, 2008, 7: 615-639. DOI:10.1007/s11101-008-9094-4 |
| [15] |
Giacoppo S, Mandolino G, Galuppo M, et al. Cannabinoids: new promising agents in the treatment of neurological diseases[J]. Molecules, 2014, 19: 18781-18816. DOI:10.3390/molecules191118781 |
| [16] |
Bai YJ, Zhou XY, Yuan Y, et al. Origin of medicinal Cannabis sativa and its early spread[J]. Chin Tradit Herb Drugs (中草药), 2019, 50: 5071-5079. |
| [17] |
Zager JJ, Lange I, Srividya N, et al. Gene networks underlying cannabinoid and terpenoid accumulation in Cannabis[J]. Plant Physiol, 2019, 180: 1877-1897. DOI:10.1104/pp.18.01506 |
| [18] |
Chen CJ, Chen H, Zhang Y, et al. TBtools: an integrative toolkit developed for interactive analyses of big biological data[J]. Mol Plant, 2020, 13: 1194-1202. DOI:10.1016/j.molp.2020.06.009 |
| [19] |
Liu MQ, Sun W, Meng XX, et al. Identification and expression analysis of the C2H2 gene family in Cannabis sativa L[J]. Acta Pharm Sin (药学学报), 2021, 56: 1486-1496. |
| [20] |
Ren ZY, Yu DQ, Yang ZE, et al. Genome-wide identification of the MIKC-type MADS-box gene family in Gossypium hirsutum L. unravels their roles in flowering[J]. Front Plant Sci, 2017, 8: 384-400. |
| [21] |
de Folter S, Shchennikova AV, Franken J, et al. A Bsister MADS-box gene involved in ovule and seed development in petunia and Arabidopsis[J]. Plant J, 2006, 47: 934-946. DOI:10.1111/j.1365-313X.2006.02846.x |
| [22] |
Mondragón-Palomino M, Theissen G. Conserved differential expression of paralogous DEFICIENS- and GLOBOSA-like MADS-box genes in the lowers of Orchidaceae: refining the 'orchid code'[J]. Plant J, 2011, 66: 1008-1019. DOI:10.1111/j.1365-313X.2011.04560.x |
| [23] |
Hu RB, Fan CM, Li HY, et al. Analysis of MIKC-type MADS-box genes in Soybean (Glycine max)[J]. Mol Plant Breeding (分子植物育种), 2009, 7: 429-436. |
| [24] |
Díaz-Riquelme J, Lijavetzky D, Martínez-Zapater JM, et al. Genome-wide analysis of MIKCC-type MADS-box genes in grapevine[J]. Plant Physiol, 2009, 149: 354-369. DOI:10.1104/pp.108.131052 |
| [25] |
Zhao YJ, Zhao HL, Wang YY, et al. Genome-wide identification and expression analysis of MIKC-Type MADS-Box gene family in Punica granatum L.[J]. Agronomy, 2020, 10: 1-10. |
| [26] |
Susanne S, Alice K, Sirui P, et al. Genome-wide analysis of MIKC-type MADS-box genes in wheat: pervasive duplications, functional conservation and putative neofunctionalization[J]. New Phytol, 2020, 225: 511-529. DOI:10.1111/nph.16122 |
| [27] |
He CM, Si C, Jaime A, et al. Genome-wide identification and classification of MIKC-type MADS-box genes in Streptophyte lineages and expression analyses to reveal their role in seed germination of orchid[J]. BMC Plant Biol, 2019, 19: 223. DOI:10.1186/s12870-019-1836-5 |
| [28] |
Li CR, Dong N, Li XP, et al. A review of MADS-box genes, the molecular regulatory genes for floral organ development in Orchidaceae[J]. Acta Hort Sin (园艺学报), 2020, 47: 2047-2062. |
| [29] |
Riechmann JL, Heard J, Martin G, et al. Arabidopsis transcription factors: genome-wide comparative analysis among eukaryotes[J]. Science, 2000, 290: 2105-2110. DOI:10.1126/science.290.5499.2105 |
| [30] |
Chandra S, Lata H, Khan IA, et al. Photosynthetic response of Cannabis sativa L. to variations in photosynthetic photon flux densities, temperature and CO2 conditions[J]. Physiol Mol Biol Plants, 2008, 14: 299-306. DOI:10.1007/s12298-008-0027-x |
| [31] |
Shen GX, Yang CH, Shen CY, et al. Origination and selection of ABCDE and AGL6 subfamily MADS-box genes in gymnosperms and angiosperms[J]. Biol Res, 2019, 52: 25. DOI:10.1186/s40659-019-0233-8 |
 2021, Vol. 56
2021, Vol. 56


