红花(Carthamus tinctorius L.), 为1年生菊科红花属草本植物, 是我国传统的中草药, 具有活血通经、祛瘀止痛之功效, 近年来, 红花也是一种新型油用和工业资源植物[1, 2]。黄酮类化合物是红花中最主要的化学成分, 广泛存在于自然界各种植物中, 具有抗菌、杀菌和抗氧化的作用[3, 4]。文献报道称目前有大约10 000种不同的黄酮类化合物已经被分离鉴定, 这些不同的黄酮类化合物主要分为黄酮醇类、二氢黄酮醇类、异黄酮类、查耳酮类、黄烷醇类和花青素类[5, 6]。其中黄酮醇类和花青素类是两类研究较多的黄酮类化合物, 对植物自身的生长发育起着重要的作用, 例如, 黄酮醇类化合物在植物生长素运输调控方面, 抗紫外线方面都起着关键作用[7], 花青素是高等植物花发育及花色形成的一类重要的黄酮类化合物。
对于植物中黄酮类化合物的生物合成途径目前已被阐述的较为清楚, 黄酮醇和花青素的形成处于类黄酮生物合成途径的两个分支上, 其中二氢黄酮醇是一个重要的中间代谢产物, 由黄烷酮3-羟化酶(flavanone-3-hydroxylase, F3H) 通过催化黄烷酮所生成。黄酮醇合成酶(flavonol synthase, FLS) 和二氢黄酮醇4-还原酶(dihydroflavonol 4-reductase, DFR) 共同竞争二氢黄酮醇这一底物, 分别生成黄酮醇和无色花青素, 无色花青素再由花青素合成酶(anthocyanidin synthase, ANS) 催化生成有色的花色苷。其中, F3H、FLS和ANS基因均属于植物2-酮戊二酸依赖性双加氧酶(2-oxoglutarate-dependent oxygenase, 2-ODD) 超级家族中的成员, 在植物类黄酮生物合成通路中属于该家族成员的还有黄酮合成酶I (FNSI) 基因, 该基因是伞形科物种中一类特有的酶类, 通过F3H基因复制而来, 与F3H基因具有高度的结构相似性[8, 9]。2-酮戊二酸依赖性双加氧酶超级家族是植物基因组中第二大酶家族, 基于氨基酸序列相似性将该基因家族中的成员分为三大类: DOXA、DOXB和DOXC, 其中类黄酮生物合成通路上的2-ODD基因属于DOXC类, 按照系统进化树将其进一步划分, F3H和FNSI基因属于DOXC28, FLS和ANS基因属于DOXC47, FLS和ANS蛋白之间具有50%~60%的序列相似性, 而与F3H或FNSI蛋白之间的序列相似性低于35%[9, 10]。
目前对植物类黄酮生物合成通路中的2-ODD家族基因的功能研究较多, Hammerbacher等[11]在云杉中鉴定出1个PaF3H基因, 发现该基因所编码的酶可以催化圣草酚转化成紫杉叶素, 以及云杉在受到真菌感染后PaF3H基因会被高度的表达, PaF3H基因在云杉防御化合物的生物合成中起着关键作用。Park等[7]从水稻中分离得到OsFLS基因, 并发现OsFLS基因同时具有黄酮醇合成酶和黄烷酮3-羟化酶的活性, 是一个双功能酶, 将OsFLS基因转入烟草中, 发现转基因烟草的花瓣颜色要比野生型烟草的花瓣颜色浅, 说明OsFLS基因调控着花色。Li等[12]从丹参中克隆了一个SmANS基因, 并通过亚细胞定位发现该蛋白在内质网、质体、高尔基体、质膜和细胞核中均有分布, 并且该基因在紫花丹参的花瓣中表达最高, 在白花丹参的花瓣中几乎不表达。虽然对植物类黄酮生物合成途径上2-ODD家族的基因有着较多的功能研究, 但对该通路上的2-ODD基因家族所有成员序列的挖掘分析鲜有相关的文献报道, 而基于红花基因组数据库对其类黄酮生物合成途径中的2-ODD基因家族成员的鉴定和分析未见有其文献报道。
本研究利用生物信息学的方法, 从红花基因组数据库中鉴定了红花类黄酮生物合成通路上的2-ODD家族基因, 并分析了其理化性质、基因结构、系统进化、顺式作用元件等, 利用荧光定量PCR技术, 对鉴定出来的基因在红花的不同花期及叶片进行表达模式分析, 以期为深入挖掘研究红花类黄酮生物合成通路上的2-ODD家族基因的功能奠定基础。
材料与方法材料 实验材料为红花, 种植于吉林农业大学红花试验田, 品种为“吉红一号”。于2020年7月份采取红花的幼嫩叶片及其花蕾期、初花期、盛花期和衰落期四个花期的花瓣, 用锡箔纸包好迅速放于液氮中冷冻保存, 随后放于实验室-80 ℃冰箱中保存备用, 用于后续的定量分析。
红花类黄酮生物合成通路2-ODD基因家族成员鉴定与理化性质分析 从本课题组保存的红花基因组数据库中调取红花类黄酮生物合成通路上的2-ODD基因家族序列, 将其编码区序列利用DNAMAN软件中的翻译功能翻译成相应的氨基酸序列, 利用NCBI数据库中的CD-search和Pfam数据库(http://pfam.xfam.org/) 对所得到的候选蛋白序列进行保守结构域的分析, 去除不具有完整保守结构域的蛋白序列, 最终得到16条具有2-ODD基因家族保守结构域的蛋白序列。利用在线工具Expasy (http://web.expasy.org/protparam/) 对红花类黄酮生物合成通路2-ODD基因家族所编码的蛋白序列进行基本理化性质分析, 包括理论等电点、分子质量和不稳定系数等。利用在线工具WOLF PSORT (https://www.genscript.com/psort.html) 和Cell-PLoc 2.0 (http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/) 对红花类黄酮生物合成通路2-ODD基因家族所编码的蛋白进行亚细胞定位预测。
红花类黄酮生物合成通路2-ODD基因家族系统进化分析 为了获得红花类黄酮生物合成通路2-ODD基因家族中各成员的系统进化关系, 从NCBI数据库中下载了亚麻FLS (AFN53718.1)、银杏FLS (ACY00393.1)、水稻FLS (BAD17324.1)、小麦FLS (AHW49495.1)、玉米FLS (NP_001140915.1)、高粱FLS (EES07584.1)、拟南芥FLS (NP_196481.1, NP_201163.1, NP_201164.1, NP_680463.1, NP_001032131.1, NP_680388.1)、可可树FLS (EOY09743.1)、柑橘FLS (XP_006485472.1, XP_006466183.1)、洋葱FLS (AQR58516.1)、葡萄FLS (BAE75807.1)、苜蓿FLS (AES97739.1, AES71332.1)、大豆FLS (NP_001237419.1)、海棠FLS (AAX89401.1)、梨FLS (ABB70118.1)、桃树FLS (AJO70134.1)、佛手柑FLS (ABH07784.1)、金鱼草FLS (ABB53382.1)、洋桔梗FLS (BAD34463.1)、马铃薯FLS (CAA63092.1)、矮牵牛FLS (CAA80264.1)、烟草FLS (ABE28017.1)、向日葵FLS (OTG15245.1)、高粱ANS (XP_002451336.1)、玉米ANS (NP_001106074.1)、水稻ANS (CAA69252.1)、小麦ANS (BAE98276.1)、洋葱ANS (ABM66367.1, ABR24157.1)、紫苏ANS (BAA20143.1)、彩叶草ANS (ABP57079.1)、拟南芥ANS (NP_194019.1)、大豆ANS (AAR26525.1)、苜蓿ANS (ABU40983.1)、梨ANS (ABB70119.1)、海棠ANS (AAD26205.1)、桃树ANS (ABX89943.1)、佛手柑ANS (AAU12369.1)、葡萄ANS (NP_001268147.1)、可可树ANS (ADD51355.1)、柑橘ANS (NP_001275784.1)、洋桔梗ANS (BAJ08929.2)、拟南芥F3H (NP_190692.1)、苜蓿F3H (ACR15123.1)、马铃薯F3H (NP_001274930.1)、矮牵牛F3H (AAC49929.1)、柑橘F3H (BAA36553.1)、桃树F3H (AQX36284.1)、烟草F3H (AAC15414.1)、小麦F3H (ABR13013.1)、玉米F3H (AAA91227.1) 25个物种中的58条类黄酮生物合成通路上的2-ODD蛋白序列, 利用MEGA-X内置的ClustalW软件对以上蛋白质进行氨基酸多序列比对分析, 然后利用MEGA-X软件中的邻接法(neighbor-joining, NJ) 构建系统进化树, 重复次数设定为1 000, 其他参数为系统默认值。使用EvolView v.2 (https://www.evolgenius.info/evolview-v2/#login) 在线工具对进化树进行分类及注释。
红花类黄酮生物合成通路2-ODD基因家族保守基序和基因结构分析 利用MEME (http://meme-suite.org/tools/meme) 在线软件, 对红花类黄酮生物合成通路2-ODD基因家族所编码的蛋白质序列进行保守基序的分析, 搜索基序的数量为10, 其余为默认值, 然后利用TBtools软件对其保守基序结合其系统进化树进行可视化作图。将红花类黄酮生物合成通路上的2-ODD基因家族的CDS序列和其对应的转录本序列分别导入GSDS2.0 (http://gsds.gao-lab.org/) 在线网站中, 对其进行基因结构的分析。
红花类黄酮生物合成通路2-ODD基因家族启动子顺式作用元件分析 提取红花类黄酮生物合成通路2-ODD家族基因上游2 000 bp的核苷酸序列, 利用在线网站Plant CARE (http://bioinformatics.psb.ugent.be/webtools/plantcare/html/) 对获得的核苷酸序列进行顺式作用元件预测, 再利用TBtools软件对其进行可视化作图。
红花类黄酮生物合成通路2-ODD家族基因表达模式分析 利用TaKaRa公司的RNAiso Plus试剂提取本研究所述植物材料的总RNA, 用NanoDrop 2000分光光度计检测所提取总RNA的OD260/OD280的比值和浓度以及用1%琼脂糖凝胶电泳检测总RNA的完整性。将符合要求的总RNA利用反转录试剂盒PrimeScript RT reagent Kit (TaKaRa) 说明书合成cDNA, 其反转录产物cDNA总量定为1 000 ng。以稀释20倍的cDNA为模板, 18srRNA为内参基因, 对红花类黄酮生物合成通路上的2-ODD家族的基因进行实时荧光定量PCR分析, 定量引物见表 1。qRT-PCR的反应体系及反应条件参照莫纳公司的荧光定量PCR试剂盒说明书进行, 利用Agilent Mx3000P荧光定量PCR仪进行反应。相对定量的计算采用2-△△Ct方法。
| Table 1 The qRT-PCR primers of 2-ODD gene family involved in flavonoid biosynthesis pathway in safflower |
根据红花基因组数据库的注释结果, 初步注释到29个与红花类黄酮生物合成通路有关的2-ODD基因, 分别为黄烷酮3-羟化酶基因(F3H), 黄酮醇合成酶基因(FLS) 和花青素合成酶基因(ANS)。根据其相应的基因编号, 从基因组数据库中搜索得到对应的编码区序列, 再将其翻译成相应的氨基酸序列, 利用pfam和NCBI CDD进一步对这29个基因对应的蛋白质序列进行保守结构域的分析, 去除不具有2-ODD保守结构域序列和不具有完整2-ODD保守结构域的序列, 最终得到红花类黄酮生物合成通路上2-ODD家族相关基因共有16个, 分别命名为CtFLS1~CtFLS8、CtANS1~CtANS7和CtF3H (表 2), 其中, CtFLS1和CtF3H基因在NCBI数据库中已有相关报道, 其登录号分别为AUH25685.1和AHY99670.1。
| Table 2 Information and characteristics of 2-ODD gene family involved in flavonoid biosynthesis in safflower |
通过Expasy在线分析软件得到红花类黄酮生物合成通路2-ODD家族基因所编码蛋白的理化性质见表 2。由表 2可以看出红花类黄酮生物合成通路2-ODD家族基因的编码区长度为852 (CtFLS2)~1 329 bp (CtANS6), 编码氨基酸的数量为283 (CtFLS2)~442 aa (CtANS6), 蛋白的分子质量在32 062.05 (CtFLS2)~48 245.49 Da (CtANS6) 之间, 16个之中有15个2-ODD蛋白的理论等电点在4.99 (CtANS4)~6.21 (CtANS6) 之间, 为酸性蛋白, CtANS7的理论等电点为7.05, 属于中性蛋白。对16个Ct2ODD-IFB (Ct2ODD-involved in flavonoid biosynthesis) 蛋白稳定系数的预测结果表明, 有7个不稳定系数大于40, 为不稳定蛋白, 剩余9个为稳定蛋白。预测结果显示红花类黄酮生物合成通路上2-ODD蛋白的亲水性平均值均为负值, 表明Ct2ODD-IFB蛋白均为亲水性蛋白。利用WOLF PSORT和Cell-PLoc 2.0在线软件对红花类黄酮生物合成通路上2-ODD基因家族成员进行亚细胞定位预测, 预测结果显示, 有12个Ct2ODD-IFB蛋白定位于细胞质, 占比75%。有2个预测定位于细胞核, 分别是CtFLS7和CtFLS8蛋白。CtANS1和CtANS3蛋白预测定位于叶绿体。
3 红花类黄酮生物合成通路2-ODD基因家族系统进化分析为了研究红花类黄酮生物合成通路上2-ODD基因家族的系统进化关系, 在NCBI数据库中选取了拟南芥、向日葵、大豆等25个物种类黄酮生物合成通路上的58条2-ODD蛋白序列构建系统发育树。结果如图 1所示, 红花类黄酮生物合成通路上的2-ODD家族被分成3个亚家族, 其中, CtF3H基因与其余物种中的F3H基因聚在一起, 并且与烟草中的F3H基因有着较高的同源性, CtANS2基因与其余物种中的ANS基因聚为一类, 而其余的CtFLS基因和CtANS基因独自与亚麻中的FLS基因聚为一类。以上结果表明红花类黄酮生物合成通路上的大多数2-ODD基因与其余物种中该家族的基因有着比较远的亲缘关系。

|
Figure 1 Phylogenetic analysis of 2-ODD gene family involved in flavonoid biosynthesis pathway in safflower |
利用MEME在线软件对红花类黄酮生物合成通路上的16个2-ODD蛋白进行保守基序的分析, 保守基序鉴定的个数上限为10, 分别命名为motif1~motif10。保守基序在每个Ct2ODD-IFB蛋白序列上的分布结果如图 2A所示。图 2A中结果显示, 红花类黄酮生物合成通路上所有2-ODD蛋白都具有motif4、motif5、motif1和motif2, 其中motif1和motif2中分别包含与Fe2+离子结合的H-x-D-xn-H保守残基和与2-酮戊二酸结合的R-x-S保守残基, 如图 2B所示。除CtANS6和CtFLS5蛋白外, 其余蛋白序列都包含motif3, 除CtFLS2、CtANS6蛋白序列不包含motif7, CtFLS5蛋白序列不包含motif6, 其余蛋白序列均包含上述两个保守基序。并且上述保守基序(motif1~motif7) 在每条蛋白序列上的分布位置、分布顺序和分布间隔都具有高度的一致性, 由此推断这一部分红花类黄酮生物合成通路上的2-ODD基因家族成员在某些生物学功能上具有相似性。此外, motif8、motif9和motif10这3个保守基序不均匀的分布在某些Ct2ODD-IFB蛋白序列中, 表明红花类黄酮生物合成通路上的2-ODD蛋白在具有功能上的相似性外同样也具有功能差异性。

|
Figure 2 Conserved protein motifs of 2-ODD gene family involved in flavonoid biosynthesis pathway in safflower. A: Conserved motifs distribution of Ct2ODDs-IFB protein; B: 10 conserved motif sequences of Ct2ODDs-IFB protein |
利用GSDS在线工具对红花类黄酮生物合成通路上的16条2-ODD基因家族成员进行基因结构分析, 如图 3结果显示, 该家族成员基因长度介于1 386 (CtFLS3)~7 731 bp (CtANS7)。基因的外显子与内含子研究发现, 大多数CtANS基因的基因结构组成为5+4 (外显子+内含子), CtFLS基因的基因结构组成比较分散, 有3+2型(CtFLS2和CtFLS5)、4+3型(CtFLS6, CtFLS8和CtFLS1)、2+1型(CtFLS3和CtFLS4), CtF3H基因的基因结构组成为3+2型, 此外, CtFLS6、CtANS2、CtFLS1和CtFLS3基因的上下游都具有不同长度的非编码区片段。以上研究结果表明, 红花类黄酮生物合成通路上2-ODD家族成员的基因结构较为多样, 这预示该家族基因可能具有多种功能。

|
Figure 3 Gene structure of 2-ODD gene family involved in flavonoid biosynthesis pathway in safflower |
利用PlantCARE在线网站对红花类黄酮生物合成通路2-ODD家族基因上游2 000 bp的启动子序列进行分析, 预测该家族基因的顺式作用元件。结果如图 4所示, 红花类黄酮生物合成通路2-ODD基因家族存在着不同种类的顺式作用元件, 如与光响应有关的元件G-box, 与逆境胁迫相关的元件LTR (响应低温)、TC-rich repeats (抗病和胁迫响应元件), 与植物激素调节相关的元件TCA-element (水杨酸响应元件)、ABRE (脱落酸响应元件)、CGTCA-motif (茉莉酸甲酯诱导响应元件)、TGACG-motif (茉莉酸甲酯诱导响应元件)、TGA-element (生长素诱导元件)、P-box (赤霉素响应元件), 此外, 还具有与MYB转录因子相识别和结合的位点CCGTTG、CAACAG。以上结果表明, 红花类黄酮生物合成通路上的2-ODD基因可能会受到光、温度及不同内源植物激素所调控, 进而去应对植物在生长发育过程中所受到的一些相关胁迫。而且MYB转录因子也可以与启动子区上的相关位点进行识别和结合, 进而驱动下游基因的表达。
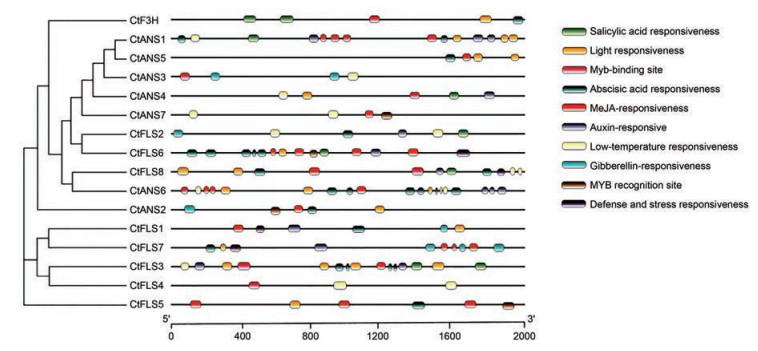
|
Figure 4 The cis-acting elements within the promoter of 2-ODD family genes involved in flavonoid biosynthesis pathway in safflower |
为了研究红花类黄酮生物合成通路2-ODD家族基因在红花不同花期及叶片中的表达模式, 利用荧光定量PCR技术对其家族中的基因进行表达模式的分析, 结果如图 5所示, 红花类黄酮生物合成通路2-ODD家族中的基因在不同花期及叶片均有不同程度的表达, CtFLS2、CtFLS7、CtANS1、CtANS4、CtANS5、CtANS7和CtF3H基因在花蕾期表达较高, 在初花期大部分基因的表达量都较低, CtFLS1、CtANS2和CtANS6这3个基因在盛花期表达量最高, 而在其余花期的表达量都很低, CtFLS2、CtFLS3、CtFLS4、CtFLS5、CtFLS6、CtFLS7、CtFLS8、CtANS3、CtANS5和CtANS7这10个基因在红花衰落期时表达较高, 除了CtFLS7、CtANS1、CtANS4和CtANS5这四个基因外, 其余该家族中的基因在叶片中的表达量都很低。以上研究结果表明, 红花类黄酮生物合成通路2-ODD基因家族中的成员可能参与不同花期的调控并且其功能存在分化。
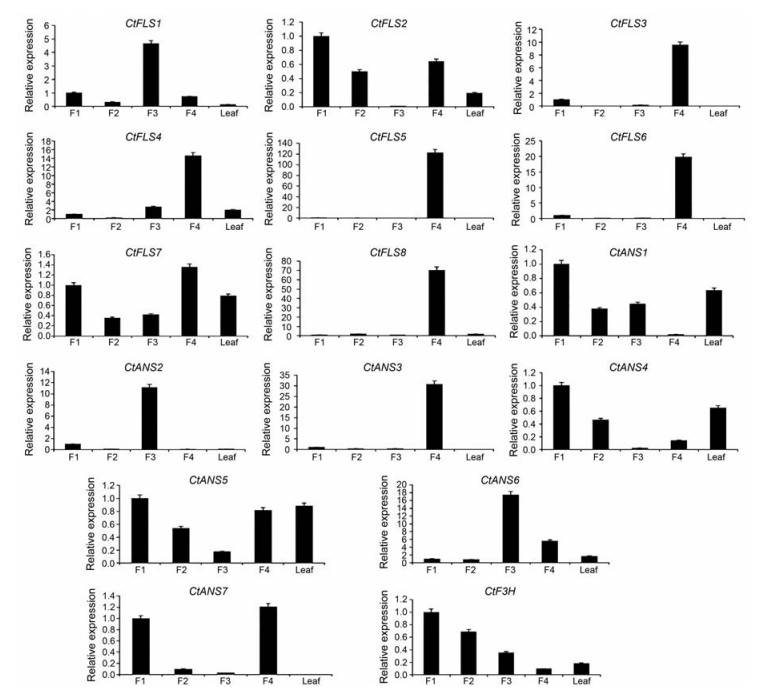
|
Figure 5 qRT-PCR analysis of 2-ODD gene family involved in flavonoid biosynthesis pathway in safflower. F1: Bud; F2: Initial; F3: Full; F4: Fading |
2-ODD超级家族是植物中第二大酶家族, 该家族成员在DNA去甲基化、脯氨酸羟基化、植物激素合成和特殊代谢产物合成过程中起着重要的作用[9]。本研究从红花基因组数据库中共筛选鉴定出16条与类黄酮生物合成通路有关的2-ODD家族基因, 其中8条CtFLS基因、7条CtANS基因和1条CtF3H基因。而该通路上的2-ODD基因还包括黄酮合成酶I (FNSI) 基因, 但在红花基因组数据库中并未注释到这一基因, 分析其原因可能是由于红花属于菊科植物, 而目前有关文献报道称FNSI基因仅存在于伞形科物种以及水稻中[13], 因此并未在红花基因组数据库中鉴定到这一基因。
基于生物信息学方法, 对红花中鉴定到的类黄酮生物合成通路上的2-ODD基因家族成员进行理化性质、系统进化、保守基序等的分析。其理化性质分析结果表明, 该家族基因编码区的平均长度为1 096.5 bp, 氨基酸数量平均为364.5, 均为亲水性蛋白, 并且大多数基因所编码的蛋白质定位在细胞质中, 这与大多数文献报道结果相一致[14-16]。系统进化分析结果表明, 红花类黄酮生物合成通路上的2-ODD家族基因大多自己聚为一类, 这与文献报道中的聚类结果有所差异[5]。分析其原因可能是由于同一物种中的ANS与FLS基因有着50%~60%的序列相似性[7], 而与其他物种中的FLS或ANS基因的序列相似性较低, 因此红花类黄酮生物合成通路上的大多数FLS和ANS蛋白在系统进化中自己聚为一类。蛋白保守基序结果显示, 红花类黄酮生物合成通路上的2-ODD家族基因都具有两个保守基序motif1和motif2, 这两个保守基序上含有与Fe2+离子结合的H-x-D-xn-H保守残基和与2-酮戊二酸结合的R-x-S保守残基, 这与Deng等[17]的研究结果相一致。基因结构分析结果表明, 红花类黄酮生物合成通路上的2-ODD家族基因的外显子数量较为多样, 有2~6个外显子不等, 而有关其他物种该通路上2-ODD家族基因的文献报道, 外显子数量大多是比较恒定的, 为2~3个外显子, 这可能与系统进化有关, 在系统进化树中, CtANS2基因与其他物种中的ANS基因聚在一起, 其基因结构表现出与其他物种中的ANS基因结构相一致的现象, 其基因结构组成为2+1型[5], 同样, CtF3H基因与其他物种中的F3H基因聚在一起, 其结构组成也相同, 为3+2型[5, 17], 而其余基因的结构差异可能是由于外显子/内含子获得/丢失, 外显子化/假外显子化以及插入/缺失这三种主要机制引起的, 有关文献报道称重复基因在进化中所产生的结构差异非常普遍, 而其基因结构的改变则会导致其基因功能的多样性[18]。通过qRT-PCR分析发现, 大部分CtFLS基因在红花的衰落期表达量较高, 这与其他物种中的FLS基因表达量有所差异, 在黄芩中分离得到的一条SbFLS基因在其根部和花中的表达量最高[19], 而在葡萄风信子中鉴定到的一个MaFLS基因主要在花发育早期表达[20]。CtANS基因在红花的不同花期及叶片均有不同程度的表达, 本课题组前期通过转录本数据库成功克隆了一条CtANS基因, 并发现该基因在红花的初花期和盛花期表达量较高[21]。而通过基因组数据库鉴定出来的这些CtANS基因, 只有CtANS2和CtANS6这两个基因在红花的盛花期表达量最高。以上结果表明, 红花类黄酮生物合成通路2-ODD基因家族中的成员可能参与不同花期的调控并且其功能存在分化。
综上所述, 本研究在红花基因组数据库中共筛选鉴定了16个与类黄酮生物合成通路有关的2-ODD基因, 所编码的蛋白均为亲水性蛋白, 且该家族成员均含有H-x-D-xn-H和R-x-S两个保守残基, 并且该家族基因受到光、温度等环境因素及水杨酸、茉莉酸甲酯等植物激素的调控。基于qRT-PCR结果, 红花类黄酮生物合成通路上的大部分2-ODD基因在花蕾期和衰落期的表达量较高。该研究可为深入挖掘红花类黄酮生物合成途径上的2-ODD基因的功能奠定基础。
作者贡献: 第一作者马鑫彤负责论文设计、实验、数据分析及论文撰写; 通讯作者刘秀明、姚娜负责论文设计、数据分析和论文指导; 朱枚桦、王南、张馨月参与实验及数据分析; 张庆宇、王宇菲参与样品采集; 侯钰莹、李志凌参与数据分析。所有作者参与论文修改。
利益冲突: 所有作者均声明不存在利益冲突。
| [1] |
Zhao G, Wang AH. Safflower resource and its medicinal value[J]. Chin Wild Plant Resour (中国野生植物资源), 2004, 23: 24-25. |
| [2] |
Yuan QQ, Liu WY. Research progress on the active components and functional properties of safflower (Carthamus tinctorius L.)[J]. Sci Technol Food Ind (食品工业科技), 2020, 41: 332-338. |
| [3] |
Li YZ, Hu WZ, SaRen GW, et al. Research progress on antioxidant components and antioxidant mechanism of flavonoids[C]. Qingdao: Abstracts of the 15th Annual Meeting of CIFST (中国食品科学技术学会第十五届年会), 2018: 2.
|
| [4] |
Yi SY, Guan LL, Yang J, et al. Research advances in pharmacological function and development and application of Carthamus tinctorius L.[J]. North Hort (北方园艺), 2015(05): 191-195. |
| [5] |
Wang Z, Wang S, Wu M, et al. Evolutionary and functional analyses of the 2-oxoglutarate-dependent dioxygenase genes involved in the flavonoid biosynthesis pathway in tobacco[J]. Planta, 2019, 249: 543-561. DOI:10.1007/s00425-018-3019-2 |
| [6] |
Zhao XW, Liu PY, Liu D, et al. Research progress in structure-activity relationship of flavoniods[J]. Chin Tradit Herb Drugs (中草药), 2015, 46: 3264-3271. |
| [7] |
Park S, Kim D, Park B, et al. Molecular and functional characterization of Oryza sativa flavonol synthase (OsFLS), a bifunctional dioxygenase[J]. J Agric Food Chem, 2019, 67: 7399-7409. DOI:10.1021/acs.jafc.9b02142 |
| [8] |
Gebhardt YH, Witte S, Steuber H, et al. Evolution of flavone synthase I from parsley flavanone 3β-hydroxylase by site-directed mutagenesis[J]. Plant Physiol, 2007, 144: 1442-1454. DOI:10.1104/pp.107.098392 |
| [9] |
Kawai Y, Ono E, Mizutani M. Evolution and diversity of the 2-oxoglutarate-dependent dioxygenase superfamily in plants[J]. Plant J, 2014, 78: 328-343. DOI:10.1111/tpj.12479 |
| [10] |
Turnbull JJ, Nakajima J, Welford RW, et al. Mechanistic studies on three 2-oxoglutarate-dependent oxygenases of flavonoid biosynthesis: anthocyanidin synthase, flavonol synthase, and flavanone 3β-hydroxylase[J]. J Biol Chem, 2004, 279: 1206-1216. DOI:10.1074/jbc.M309228200 |
| [11] |
Hammerbacher A, Kandasamy D, Ullah C, et al. Flavanone-3-hydroxylase plays an important role in the biosynthesis of spruce phenolic defenses against bark beetles and their fungal associates[J]. Front Plant Sci, 2019, 10: 208. DOI:10.3389/fpls.2019.00208 |
| [12] |
Li H, Liu J, Pei T, et al. Overexpression of SmANS enhances anthocyanin accumulation and alters phenolic acids content in Salvia miltiorrhiza and Salvia miltiorrhiza Bge f.alba plantlets[J]. Int J Mol Sci, 2019, 20: 1-21. |
| [13] |
Martens S, Preuss A, Matern U. Multifunctional flavonoid dioxygenases: flavonol and anthocyanin biosynthesis in Arabidopsis thaliana L[J]. Phytochemistry, 2010, 71: 1040-1049. DOI:10.1016/j.phytochem.2010.04.016 |
| [14] |
Huang QL, Cai C. Bioinformatics analysis of flavonol synthase from various plants[J]. Guangdong Agric Sci (广东农业科学), 2014, 41: 140-143. |
| [15] |
Gai JT, Huang JF, Dang ZG, et al. Identification of ANS gene in Mangifera indica Linnaeus and comparative analysis with other plants[J]. Jiangsu Agric Sci (江苏农业科学), 2017, 45: 43-49. |
| [16] |
Li P, Wang RX, Wang H. Bioinformatics analysis of flavanone-3-hydroxylase (F3H)[J]. J Biol (生物学杂志), 2015, 32: 25-29. |
| [17] |
Deng Y, Li C, Li H, et al. Identification and characterization of flavonoid biosynthetic enzyme genes in Salvia miltiorrhiza (Lamiaceae)[J]. Molecules, 2018, 23: 1467. DOI:10.3390/molecules23061467 |
| [18] |
Xu G, Guo C, Shan H, et al. Divergence of duplicate genes in exon-intron structure[J]. Proc Natl Acad Sci U S A, 2012, 109: 1187-1192. DOI:10.1073/pnas.1109047109 |
| [19] |
Kim YB, Kim K, Kim Y, et al. Cloning and characterization of a flavonol synthase gene from Scutellaria baicalensis[J]. Sci World J, 2014, 2014: 980740. |
| [20] |
Liu H, Su B, Zhang H, et al. Identification and functional analysis of a flavonol synthase gene from grape hyacinth[J]. Molecules, 2019, 24: 1579. DOI:10.3390/molecules24081579 |
| [21] |
Liu XM, Yang WT, Zhao LD, et al. Cloning and expression analysis of anthocyanidin synthase in safflower[J]. Chin J Cell Biol (中国细胞生物学学报), 2014, 36: 766-772. |
 2021, Vol. 56
2021, Vol. 56


