2. 青海大学三江源生态与高原农牧业国家重点实验室, 青海 西宁 810016
2. State Key Laboratory of Plateau Ecology and Agriculture, Qinghai University, Xining 810016, China
高原环境具有低压、低氧、强辐射、寒冷等特征, 其中低氧是影响人类生命活动的主要因素[1]。高原低氧条件下机体发生一系列生理和病理性变化, 导致药物体内代谢动力学特征、药物代谢酶和转运体的活性及表达均发生显著改变, 进一步影响药物的治疗作用[2]。
肠道菌群是数以万亿计微生物栖息于机体肠道形成的复杂生态群落, 影响机体正常生理和疾病的易感性, 不仅可以促进营养物质吸收, 还具有免疫调节、保护黏膜屏障等功能[3]。肠道菌群的结构和多样性取决于与宿主的共生关系, 受遗传、年龄、药物和环境等因素的影响, 存在显著的个体差异[4]。肠道菌群的研究主要集中在肥胖、糖尿病等慢性代谢性疾病及溃疡性结肠炎、克罗恩病等炎症性肠病方面, 近年来其在药物代谢中的作用也受到广泛关注[5]。
高原低氧条件下, 机体胃肠道蠕动减慢, 胃肠黏膜病理性损伤, 屏障功能障碍, 从而导致肠道菌群发生紊乱, 其结构和多样性发生显著变化[6-8]。高原低氧环境, 机体肠道菌群的改变能否进一步调节药物的体内代谢值得深入探讨。本文综述了高原低氧对肠道菌群的影响, 探讨肠道菌群介导的高原低氧对药物代谢的调节作用, 并提出了以肠道菌群为核心的高原低氧影响药物代谢的分子机制, 为合理解释高原低氧环境中的药物代谢和临床合理用药提供参考依据。
1 高原低氧对肠道菌群的影响肠道菌群是由寄居在胃肠道内的微生物组成的复杂生态系统, 主要有厚壁、拟杆、变形、放线和梭杆等优势菌门, 其中厚壁菌门和拟杆菌门占90%以上, 各菌群间相互制约, 相互依存, 形成质和量的动态平衡[9, 10]。肠道菌群是一个高度动态的系统, 也是一个相对脆弱的“隐形器官”, 已知多种因素影响肠道微生物群的组成, 包括抗生素、益生菌以及环境等[11]。Zhang等[12]研究肠道菌群介导的阿莫西林与阿司匹林相互作用发现, 大鼠给予阿莫西林, 显著减少幽门螺杆菌和普氏菌丰度, 阿司匹林代谢产物水杨酸的生成量增加, 药代动力学参数血药浓度曲线下面积(AUC) 提高91.38%, 峰浓度(Cmax) 增加60.43%, 而清除率(CL) 下降43.55%。提示阿莫西林与阿司匹林合用, 发生由肠道菌群介导的相互作用, 这可能是影响药物疗效的重要因素之一。另外, Kim等[13]证实口服益生菌对小鼠粪便微生物群组成存在影响, 口服路氏乳杆菌增加梭菌、双歧杆菌和肠球菌数量, 降低对乙酰氨基酚AUC, 表明益生菌可导致同服药物的药代动力学参数发生改变。肠道菌群不仅参与内源性物质如胆固醇、胆汁酸、色氨酸等的代谢以维持肠道内环境稳态及机体的代谢平衡, 还通过肠道菌-宿主共代谢或肠道菌代谢参与药物的处置, 影响药效或毒性[7]。除抗生素、益生菌等影响因素外, 近年来越来越多研究表明, 高原低氧环境中机体肠道菌群发生显著变化。
1.1 高原低氧环境下实验鼠肠道菌群的变化高原低氧环境, 可一定程度改变机体肠道菌群的动态平衡、肠黏膜完整性和功能[6, 8]。氧气是调节氧化应激和肠道炎症的重要因素, 在维持肠道上皮细胞有氧和厌氧之间的平衡中发挥着关键作用[14]。肠上皮细胞的高厌氧状态降低了需氧菌的数量, 有利于肠腔微环境中厌氧菌的生长[15, 16]。Adak等[17]研究发现, 大鼠连续暴露于4.8 km高原模拟环境30天后, 低氧应激促进肠道厌氧状态, 导致可有效利用碳水化合物和耐受氧化应激的兼性厌氧菌和厌氧菌的种群规模明显扩大, 肠内气体形成增多, 引起酸积累和消化不良。同时, 低氧应激使需氧菌种群密度显著降低, 提示氧气在调节肠道菌群稳态中发挥重要作用。
Zhang等[18]通过模拟5 km高原环境分析小鼠肠道微生物群落的变化, 其中放线菌门、ε-变形菌纲、丹毒丝菌纲、螺杆菌属和另枝菌属变化显著, Alpha多样性结果表明, 高原低氧环境可显著减少肠道菌群的种类和数量, 进一步揭示高原低氧环境对小鼠肠道微生物群落的影响。Ma等[19]发现, 高原低氧环境, 大鼠肠道微生物群落的结构和多样性呈动态变化, 与碳水化合物代谢相关菌群如普氏菌、拟杆菌等的丰富度明显增加, 表明高原低氧环境影响肠道菌群平衡。课题组利用实地高原环境, 通过16S rDNA基因测序技术探讨不同海拔高度及低氧处理时间对大鼠肠道菌群结构及多样性的影响, 研究高原低氧环境对大鼠肠道菌群的影响。结果发现, 进入高原低氧环境后, 大鼠肠道菌群多样性显著增加, 疣微菌门、阿克曼菌属相对丰度显著降低, 而厚壁菌门、乳杆菌属相对丰度显著升高。此外, 随低氧暴露时间延长, 大鼠肠道中格氏乳杆菌和罗伊乳杆菌呈先降后升趋势, 证实低氧条件下乳杆菌在肠道更易存活, 提示高原低氧环境中大鼠肠道菌群可能通过调节益生菌维持肠道内的相对平衡。另外, 肠道差异菌如梭状芽孢杆菌_42_27、毛螺菌_28_4和另枝菌属的富集可能与低氧诱导的相关疾病形成与发展有关[8]。
1.2 高原低氧环境下人体肠道菌群的变化人体肠道菌群的结构和多样性也受到高原低氧环境的影响。Li等[20]发现, 平原和高原地区汉族人肠道菌群的组成存在显著差异, 高原汉族人中厚壁菌门、丁酸弧菌属、螺杆菌属和放线菌属的相对丰度较高, 拟杆菌门、萨特氏菌属和丁酸单胞菌属的相对丰度较低。另外, 高原地区汉族和藏族人肠道菌群中普雷沃菌属的相对丰度显著高于平原地区汉族[21], 与Moeller等[22]研究结果一致, 提示高原低氧环境对肠道菌群的塑造起重要作用。2.8~4.5 km不同海拔梯度研究表明, 生活在青藏高原不同地区藏族人肠道菌群间存在显著差异, 且随海拔升高, 肠道菌群多样性增加。拟杆菌门、厚壁菌门、变形菌门和放线菌门等门分类水平菌的相对丰度分别为60.00%、29.04%、5.40%和3.85%, 以普雷沃菌属、Faecalibacterium和Blautia为高原低氧地区优势菌属, 并推测梭菌目可能是高原低氧环境的敏感微生物[23]。Jia等[24]研究还发现, 高原低氧环境对长期或短期生活在高原个体的肠道菌群都有显著影响, 即使离开高原, 这种影响也可能持续相当长的一段时间。
综上, 高原低氧是一个重要的环境因素, 可以调节肠道菌群的结构, 改变肠道微生物的多样性, 进一步影响宿主肠道微生态的平衡。
2 高原低氧对药物代谢的影响 2.1 高原低氧对药物代谢动力学的影响药物的体内生物转化是一个耗氧的过程, 在高原低氧条件下, 机体肝脏的代谢能力受到限制, 大多数药物的生物转化减慢, 清除率降低。本课题组研究表明, 人体急进高原后血浆磺胺甲噁唑浓度明显降低, 急、慢性低氧使其蛋白结合率显著升高, 平均驻留时间(MRT)、AUC增大, CL降低[25]。比较平原汉族、世居高原汉族和藏族志愿者体内磺胺甲噁唑的药代动力学特征发现, 磺胺甲噁唑在高原世居汉族和藏族健康男性志愿者体内的药物代谢动力学均发生显著变化, 主要表现为半衰期(t1/2) 延长, CL降低[26]。比较高原低氧环境盐酸二甲双胍和对乙酰氨基酚在大鼠体内药代动力学参数变化发现, 急、慢性低氧组大鼠血浆盐酸二甲双胍t1/2分别延长27.8%和56.6%, 消除速率(Ke) 分别下降22.9%和37.1%; 急、慢性低氧组对乙酰氨基酚t1/2分别延长21.7%和40.9%, AUC分别增加91.9%和133.9%, Cmax分别增加76.8%和93.7%, CL分别降低48.1%和56.9%, 表观分布容积(Vd) 分别降低37.6%和38.7%[26]。地西泮和地塞米松的药代动力学研究表明, 低氧条件下地西泮在大鼠体内的AUC和MRT显著升高, 地塞米松的t1/2和MRT增加, Ke和Cmax明显降低[27]。高原低氧环境中布洛芬的体内代谢也发生一定改变, 表现为Vd、t1/2和MRT增加, AUC、Cmax和CL降低[28]。奥美拉唑在低氧环境中的代谢明显减慢, CL下降70%[29]。截至目前, 已知尚有呋塞米[2]、泼尼松龙[2]、罗红霉素[26]、哌替啶[30]、普萘洛尔[31]、美托洛尔[31]、阿莫西林[32]、乙酰水杨酸[33]、庆大霉素[33]、苯巴比妥[33]、乙酰唑胺[33]、诺氟沙星[34]、茶碱[35]和地尔硫卓[36]等药物被证实高原低氧条件下的体内代谢减慢。
在高原低氧环境中, 大多数药物的体内代谢减慢, 药物代谢动力学参数发生显著变化, 主要表现为t1/2和MRT显著延长, AUC显著增加, CL显著降低。研究高原低氧环境中药物的药代动力学, 能够为高原地区临床合理用药和个体化用药做出合理指导。高原低氧环境中的药物代谢减慢提示高原地区临床用药时, 应重新评估药物的代谢动力学特征, 适当调整给药剂量。
2.2 高原低氧对药物代谢酶的影响药物代谢酶根据其参与体内药物代谢的过程分为Ⅰ相代谢酶和Ⅱ相代谢酶。Ⅰ相代谢酶主要参与氧化、还原、水解等反应, 其中细胞色素P450 (cytochrome P450, CYP450) 是最主要的Ⅰ相代谢酶, CYP1A2、CYP2C9、CYP2C19、CYP2D6、CYP2E1和CYP3A4代谢90%以上的药物[2]。Ⅱ相代谢酶主要催化葡萄糖醛酸化、硫酸化、乙酰化等反应, 主要有N-乙酰基转移酶(N-acetyltransferase, NATs)、谷胱甘肽巯基转移酶(glutathione S-transferase, GSTs) 以及葡萄糖醛酸转移酶(UDP-glucuronyl transferases, UGTs) 等[37]。
CYP450是参与药物代谢反应的主要酶系, 易受到氧气浓度的影响。低氧环境中CYP450酶的活性和表达发生显著变化, Fradette等[38]采用低氧氧舱模拟低氧环境, 研究发现急性低氧条件下家兔CYP3A6表达升高, CYP1A1、CYP1A2、CYP2B4、CYP2C5和CYP2C16表达降低。Kurdi等[39]进一步证实, 家兔急性缺氧48 h后, CYP1A1和CYP1A2的活性和蛋白表达均显著下调。课题组利用高原实际环境, 综合急性缺氧、慢性缺氧、中海拔和高海拔等因素, 系统研究了高原环境对药物代谢酶的影响, 证实在2.8 km中海拔和4.3 km高海拔高原地区, 急、慢性低氧使大鼠CYP1A2的活性、蛋白和mRNA表达均显著降低。2.8 km中海拔和4.3 km高海拔环境中, 慢性低氧使CYP2D1的活性和表达显著升高, 而CYP3A1和CYP2E1的活性和表达显著降低, 急性低氧对CYP2D1、CYP3A1和CYP2E1的活性和表达无明显影响[40]。体外研究同样发现, 在低氧条件下, 人胚肺成纤维细胞中CYP3A4和CYP3A7的mRNA表达均处于被抑制状态[41]。Legendre等[42]也证实, 在低氧条件下HepaRG细胞中CYP1A2、CYP2C9、CYP3A4和CYP2E1的mRNA表达均显著降低。
高原低氧对Ⅱ相代谢酶的影响也有一定报道, 研究已证实大鼠NAT2的活性和mRNA表达在2.8 km高原环境中分别降低23.1%和55.8%, 在4.3 km高原环境中分别降低28.6%和19.5%[40]。大鼠UGT1A1的研究也发现, 高原急性、慢性低氧使其蛋白表达显著降低[31]。
综上, 高原低氧环境中CYP1A1、CYP1A2、CYP2B4、CYP2C5、CYP2C16、CYP2E1和CYP3A4等大部分CYP450酶的活性和表达降低, 唯一例外的是CYP2D6和CYP3A6的活性和表达升高。药物代谢酶活性及表达的改变是引起药物体内代谢发生变化的重要因素, 对研究高原低氧环境中的药物代谢变化情况有重要意义。高原低氧条件下CYP1A2、CYP2B6、CYP3A1、NAT2和UGT1A1的活性和表达降低, 其对应底物咖啡因、苯妥英钠、咪达唑仑、磺胺甲噁唑和对乙酰氨基酚的体内代谢相应减慢[43], 提示高原低氧条件下药物代谢酶活性和表达的变化与其底物的药代动力学特征变化一致。
3 肠道菌群在药物代谢中的作用近年来越来越多的研究表明肠道菌群在药物代谢中发挥重要作用, 对药物及内源性化合物有较强的代谢能力[44, 45]。目前已发现多种药物的代谢受肠道菌群的影响, 如止痛药对乙酰氨基酚、强心药地高辛、抗病毒药物替诺福韦、催眠药氯硝西泮和抗震颤麻痹药左旋多巴的代谢分别与艰难梭菌[46]、Eggerthella lenta[47]、拟杆菌[48]、梭菌[49]和梭状芽孢杆菌[50]有关, 见表 1[7, 46-57]。
| Table 1 Summary of drugs metabolized by gut microbiota. ↑: Increase; ↓: Decrease |
肠道菌分泌的酶可以与药物相互作用, 影响药物在体内的吸收、代谢等[58]。β-葡萄糖醛酸酶(β-glucuronidase, GUS) 是肠道菌最高产的酶之一, 可催化葡萄糖醛酸结合物发生水解反应[53, 59]。Yip等[51]研究发现, GUS处理组大鼠血浆他克林AUC和Cmax分别显著升高69%和77%, 万古霉素和亚胺培南预处理大鼠则得到相反的结果, 提示肠道菌产生的GUS会显著影响他克林的肝毒性。伊立替康(irinotecan, CPT-11) 的胃肠道毒性也与肠道菌GUS活性密切相关, 肠道菌GUS可将CPT-11的无毒代谢产物7-乙基-10-羟基喜树碱-O-葡萄糖醛酸转化为有毒的7-乙基-10-羟基喜树碱, 最终导致肠上皮细胞损伤, 引起胃肠道反应[52]。GUS对非甾体抗炎药物如吲哚美辛和双氯芬酸的胃肠道毒性反应同样有重要影响, 其可催化非甾体抗炎药物的葡萄糖醛酸水解, 释放出的苷元可被肠道再次吸收, 从而形成药代动力学上的“双峰现象”[53]。
除GUS外, 另一种代表性的微生物酶是羧酸酯酶(carboxylesterases, CES)。依那普利在肠道菌CES的作用下转化为依那普利拉, 肠道中转化率达32%~51%, 提示肠道菌CES的去酯化作用可能在一定程度上促进依那普利的代谢, 这种作用也适用于其他血管紧张素转换酶抑制剂前药, 如雷米普利、培哚普利、莫西普利和福辛普利[7]。阿司匹林的体内代谢也与肠道菌产生的CES息息相关, Kim等[54]发现阿司匹林可被肠道CES水解生成水杨酸, 当阿司匹林与抗菌药物氨苄西林联用时会抑制肠道CES的活性, 使大鼠血浆中阿司匹林的tmax、Cmax和AUC分别增加了150%、109%和151%, 吸收和生物利用度提高, 从而增强抗血栓作用。另外, 偶氮还原酶也广泛存在于肠道菌中, 参与多种药物的代谢过程, 如作为治疗炎症性肠病的药物柳氮磺胺吡啶, 只有在偶氮还原酶的作用下转化为5-氨基水杨酸后才能发挥药理作用[55]。此外, 磺胺类药物也需经偶氮还原酶裂解, 才能生成具有抗菌活性的磺胺[56]。
3.2 肠道菌群代谢物对药物代谢的影响肠道菌群与宿主相互作用会产生大量的代谢产物, 如短链脂肪酸、对甲酚、石胆酸和色氨酸代谢物等, 其在维持肠道稳态和调节药物代谢等方面具有重要作用[60]。He等[57]利用小鼠肿瘤模型发现, 抗菌药物的使用会破坏肠道菌群的稳态, 可显著减少短链脂肪酸等肠道菌群代谢产物的丰度, 并影响化疗药奥沙利铂的疗效。相关机制研究表明, 短链脂肪酸(丁酸) 能够显著增强肿瘤微环境中CD8+ T细胞的增殖和功能, 进而促进奥沙利铂的疗效, 提示肠道菌群可通过其代谢产物影响抗肿瘤药物疗效。止痛药对乙酰氨基酚的代谢也会受到肠道菌代谢物对甲酚的影响, 对甲酚会与对乙酰氨基酚竞争与磺基转移酶的结合, 这种竞争性结合会阻碍肝脏对对乙酰氨基酚的解毒能力, 从而引起肝毒性[61, 62]。
肠道菌代谢物不仅能与药物竞争药物代谢酶, 还可作为信号分子通过激活孕烷X受体(pregnane X receptor, PXR)、组成型雄烷受体(constitutive androstane receptor, CAR) 和芳香烃受体(aryl hydrocarbon receptor, AHR) 等核受体间接调控药物代谢[63, 64]。Toda等[65]证实了石胆酸作为PXR和CAR的主要胆汁酸配体, 可诱导PXR和CAR的激活, 调控相关药物代谢酶的表达, 加速体内药物的代谢。进一步研究发现, 使用环丙沙星等抗菌药物会降低石胆酸的丰度, 导致PXR和CAR的激活受到抑制, 使CYP3A的mRNA表达降低[66]。研究同样表明肠道菌色氨酸代谢物吲哚及吲哚丙酸也可作为内源性配体激活PXR和AHR, 从而调控靶基因CYP1A2和CYP3A11的表达[67, 68]。
3.3 肠道菌群对药物代谢酶的影响肠道菌群除了直接影响药物代谢外, 还可以改变宿主Ⅰ、Ⅱ相药物代谢酶的表达间接介导药物代谢。Toda等[65]证实了无菌(GF) 小鼠中CYP1A2、CYP2A4、CYP2B9、CYP2B13、CYP2C37、CYP3A11、CYP3A16和CYP3A44的mRNA表达均显著低于无特定病原体(SPF) 小鼠。Selwyn等[69]利用转录组测序技术深入研究表明, GF小鼠中CYP1A2、CYP2A5和CYP2A22的mRNA表达分别增加了51%、143%和33%, 而CYP2B10、CYP3A11、CYP3A16、CYP3A44和CYP3A59的mRNA表达分别降低了57%、87%、86%、87%和11%, CYP2B10和CYP3A11的蛋白表达也显著降低, 除CYP1A2外, 与Toda等[65]研究结果一致。Kuno等[70]发现GF小鼠和抗生素处理小鼠中CYP2B10的蛋白表达分别降低了0.04和0.45倍, CYP3A11的蛋白表达分别降低了0.17和0.12倍, 其中CYP2B10对底物Benzyloxyresorufin的代谢减慢, 表现为Vmax、Km和CL均显著降低, 进一步表明肠道菌群的任何改变都可能会影响相关药物代谢酶的表达和活性。值得注意的是, CYP3A作为机体最重要的药物代谢酶, 其亚家族成员CYP3A11、CYP3A16、CYP3A25、CYP3A44和CYP3A59等在GF小鼠中的表达均降低, 提示肠道菌群在调节CYP450酶介导的药物代谢中的作用不容小觑。
一些Ⅱ相药物代谢酶的表达也受到肠道菌群的影响。研究发现, GSTs和UGTs是GF小鼠肝脏中变化最大的Ⅱ相药物代谢酶, GSTA1、GSTP1、GSTP2、UGT2B35和UGT2B38的mRNA表达分别降低了48%、66%、64%、32%和11%[69]。UGT1A1作为一种重要的Ⅱ相药物代谢酶可通过增加药物的水溶性促进药物排出体外, 现已证实GF小鼠肠道和肝脏中UGT1A1的表达明显更高, 提示当缺少肠道菌群时, UGT1A1的葡萄糖醛酸作用可能会加强, 从而减少药物的肝肠循环, 减轻药物的不良反应[47]。
综上, 肠道菌群可与药物发生直接或间接的相互作用, 从而调节药物的体内代谢(图 1)。一是肠道菌会产生降解或改变药物化学结构的酶, 影响药物在胃肠道的代谢。二是肠道菌的代谢产物如短链脂肪酸、对甲酚和胆汁酸等参与、干扰药物代谢。三是肠道菌群可通过调节药物代谢酶的表达和活性影响药物代谢。因此, 对肠道微生物微环境的任何干扰因素都会直接或间接地影响药物体内代谢, 了解肠道微生物在药物代谢中的作用有助于预测和控制药物的不良反应, 能更好地指导临床合理用药。

|
Figure 1 The sites and enzymatic processes involved in the drug metabolism by gut microbiota. There are both direct and indirect mechanisms through which the gut microbiome can influencedrugs metabolism to modulate efficacy, absorption, and bioavailability |
高原低氧影响药物代谢相关机制的研究集中在药物代谢酶和转运体, 二者的调控机制相似, 主要通过炎症因子、核因子κB (nuclear factor-kappa B, NF-κB)、核受体和miRNA等进行调控。高原低氧环境中一些炎症因子如白细胞介素(IL-1、IL-6、IL-4和IL-1β) 和肿瘤坏死因子-α等显著升高, 从而激活NF-κB通路, 通过磷酸化、泛素化等途径降解通路中的抑制元件IκB, 进而调控药物代谢酶的表达[71]。Fradette等[72]证实炎症因子IL-1β和干扰素-γ等是CYP1A1、CYP1A2活性和表达下调的调节因子。Rahman等[73]研究也证实低氧条件下CYP1A表达的下调与IL-1β等炎症因子有关。低氧条件下PXR和CAR的表达均降低, 并进一步调节药物代谢酶的活性和表达[74]。课题组通过构建过表达/抑制表达和沉默PXR、CAR的HepG2细胞模型, 进一步证实PXR和CAR介导低氧对CYP1A2、CYP2C9、CYP2E1、CYP3A4和UGT1A1的转录调节作用[75]。另外, 课题组综述了低氧环境下miRNA的表达发生显著变化, miRNA可负调控药物代谢酶、转运体和与药物代谢相关的基因, 推测提出了以miRNA为核心的低氧影响药物代谢的分子机制[37]。
综上所述, 高原低氧环境中炎症因子、NF-κB、核受体和miRNA等对药物代谢酶和转运体具有重要调控作用, 肠道菌群的结构和多样性也会受到高原低氧环境的影响, 且越来越多的研究表明肠道菌群在药物代谢中发挥重要作用。由此推测, 在高原低氧条件下, 肠道菌群可直接调控药物体内代谢, 也可介导药物代谢酶的表达间接调控药物体内代谢。
本文以此提出在高原低氧环境下肠道菌群可能会直接或间接调节药物代谢的相关机制。①肠道菌群中的一些低氧敏感微生物的种类和相对丰度发生改变, 影响前药的激活和药物的生物转化, 从而直接对药物体内代谢产生影响。②高原低氧环境会导致胆汁酸、短链脂肪酸和对甲酚等肠道菌代谢物的吸收和代谢受损, 而PXR、CAR和AHR等核受体均表现出肠道菌代谢物的特异敏感性, 肠道菌代谢物的细微变化都有可能会影响核受体的表达和活性, 从而影响药物代谢酶的转录, 调节药物体内代谢。③肠道菌群是炎症反应的重要调节者, 且低氧的信号传导途径与炎症过程之间存在多种联系, 随着信号的传递, 释放的各种炎症因子(TNF-α、IL-1β、IL-6、IL-4和IL-10) 加重肠道黏膜的炎症反应, 使NF-κB信号通路被激活, NF-κB直接与类视黄醇X受体α (retinoic acid X receptor α, RXRα) 的DNA结合域结合, 干扰PXR/RXRα、CAR/RXRα和AHR/RXRα异源二聚体的形成, 调节靶基因药物代谢酶的转录活化, 间接调节药物体内代谢。因此, 高原低氧条件下肠道菌群对药物代谢会产生影响, 伴随其上游调控因子炎症因子、NF-κB和核受体的改变以及整体代谢网络发生迁移。肠道菌群可能是高原低氧影响药物代谢的又一关键机制, 将为研究高原合理用药提供新思路。
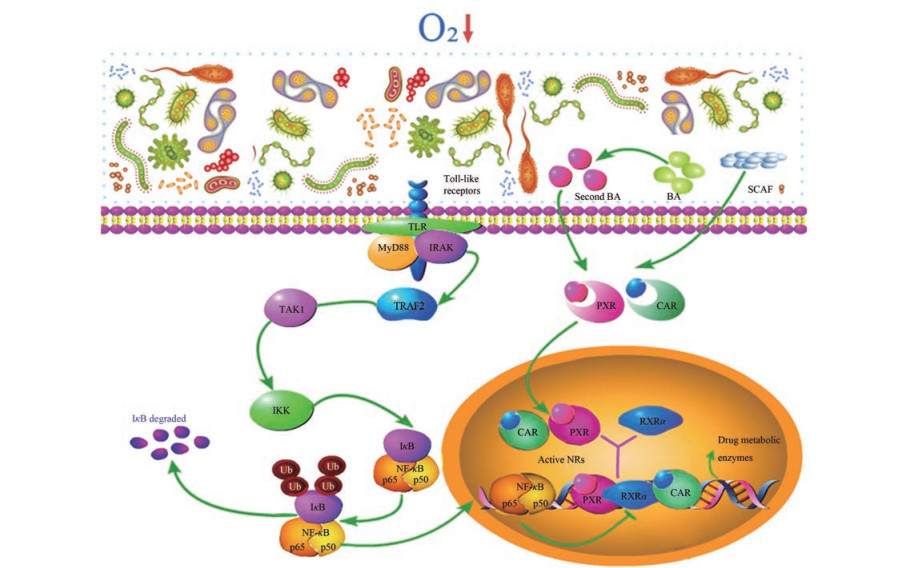
5 小结与展望研究高原低氧环境中肠道菌群对药物代谢的调节作用, 可以预测药物相互作用, 提高用药安全性, 避免不良反应的发生, 无论是在新药研发还是临床安全用药中都有重要意义。探讨高原低氧-肠道菌群-转录因子(炎症因子、核受体)-药物代谢之间的关系, 将是高原低氧影响药物代谢研究的新方向。高原低氧引起药代动力学特征的变化以及肠道菌群失衡都会影响药物的体内代谢, 根据现有文献报道和研究成果, 本文推测并提出了肠道菌群介导高原低氧调节药物代谢的分子机制, 如图 2所示。
|
Figure 2 A hypothetical network view on the interactions between gut microbiota and transcription factors under hypoxia. The network contains gut microbiota and its target genes. Hypoxia condition affects the function and composition of the gut microbiota. Inflammation activates the family of transcription factor called nuclear factor-kappa B (NF-κB). The IκB kinase (IKK) complex is the signal integration hub for NF-κB activation. IKB: NF-κB inhibitory protein. After ligand binding, NRs and some components of the chaperone complex translocate to the nucleus, where NRs binds DNA-responsive elements to control gene expression |
随着对肠道菌群的代谢功能以及宿主、菌群和药物之间复杂多维互作关系认识的不断深入, 肠道菌群在药物代谢中的作用越来越受到关注。值得注意的是, 由于肠道菌群组成的复杂性和分析技术的限制, 使得肠道菌代谢药物的研究仍面临巨大挑战。肠道菌群的组成存在显著的个体差异并受多种因素的影响, 将肠道菌代谢纳入生理药动学模型, 定量评估个体药物代谢, 实现药物在临床上的个体化治疗显得尤为重要[76]。此外, 传统的粪便菌体外代谢研究难以准确评估肠道菌对药物代谢的贡献, 探索参与药物代谢的特征菌和代谢酶, 推动肠道菌代谢药物的研究向着系统性、精细化快速发展, 不仅需要16S rDNA基因测序技术、宏观基因组学、代谢组学、培养组学、微生态模拟和生物信息学等技术的支持, 还需要综合药物微生物组学、药理学和毒理学等学科知识[77, 78]。
目前, 肠道菌群影响药物代谢的研究多集中在低海拔地区, 高原低氧环境中肠道菌群介导的药物代谢仍处于探索阶段, 存在很大的研究空间。高原环境中除低氧外, 强辐射、寒冷等因素是否会影响肠道菌群的组成和功能, 目前尚未明确。影响肠道微生物因素的混杂性, 使得研究高原条件下肠道菌群介导的药物代谢难度加剧。因此, 哪些影响因素最终能在肠道菌群组成和功能上留下印记, 引起行为表型和生理特性的变化, 从而影响高原低氧环境中的药物代谢, 未来还需进一步探究。
明确高原低氧条件下肠道微生物群落对药物代谢的具体影响仍面临一些挑战。研究大多采用无菌动物或通过抗生素构建的伪无菌动物等模型, 均存在各自的局限性, 无菌动物成本昂贵且对动物房设施要求严格, 高原地区恶劣的环境和艰苦的条件, 大大限制了对无菌动物的应用。另外, 动物与人体存在一定差异, 且实验动物难以全面客观反映人体真实特征, 动物研究结果是否与人体研究结果一致, 也值得进一步探讨。
肠道菌群对药物代谢的影响机制复杂而难以捉摸。近年来, 研究已证实肠道菌群在宿主药物代谢和转运中具有重要的作用, 其可通过释放可溶性因子、细菌外膜囊泡或代谢产物影响宿主代谢酶和转运体的表达和活性[79]。高原低氧不仅会调节机体各组织药物代谢酶和转运蛋白的转录和表达, 而且肠道菌群的结构和多样性也会受到高原低氧环境的影响。在高原低氧条件下, 肠道菌失调是否介导药物代谢酶和转运蛋白的表达, 目前尚缺少直接证据, 相关效应分子及具体作用机制也是未来的研究重点。
探讨高原低氧环境中肠道菌群对药物代谢的影响及相关机制, 以及指导高原人群合理有效用药将是未来研究的热点。应综合多技术、多方法和多学科系统性深入的研究高原低氧环境中肠道菌群影响药物代谢的机制。
作者贡献: 白雪负责全文撰写并总结图表; 刘贵琴、杨建鑫参与论文内容核对; 段雅彬、朱俊博参与论文相关内容的补充; 李向阳负责对论文进行整体的指导和修改。
利益冲突: 无任何利益冲突。
| [1] |
Li XY, Liu YN, Li YP, et al. Pharmacokinetic study of sulfamethoxazole in healthy Han volunteers living at plain and native Han and Tibetan healthy volunteers living at high altitude[J]. Acta Pharm Sin (药学学报), 2011, 46: 1117-1122. |
| [2] |
Zhang JL, Li XY. A review of drug metabolism under hypoxia environment at high altitude[J]. Acta Pharm Sin (药学学报), 2015, 50: 1073-1079. |
| [3] |
Lozupone CA, Stombaugh JI, Gordon JI, et al. Diversity, stability and resilience of the human gut microbiota[J]. Nature, 2012, 489: 220-230. DOI:10.1038/nature11550 |
| [4] |
Benson AK, Kelly SA, Legge R, et al. Individuality in gut microbiota composition is a complex polygenic trait shaped by multiple environmental and host genetic factors[J]. Proc Natl Acad Sci U S A, 2010, 107: 18933-18938. DOI:10.1073/pnas.1007028107 |
| [5] |
Shreiner AB, Kao JY, Young VB. The gut microbiome in health and in disease[J]. Curr Opin Gastroenterol, 2015, 31: 69-75. DOI:10.1097/MOG.0000000000000139 |
| [6] |
Xing JY, Ying YQ, Mao CX, et al. Hypoxia induces senescence of bone marrow mesenchymal stem cells via altered gut microbiota[J]. Nat Commun, 2018, 9: 2020-2033. DOI:10.1038/s41467-018-04453-9 |
| [7] |
Sun CN, Chen L, Zu S. Mechanisms of gastrointestinal microflora on drug metabolism in clinical practice[J]. Saudi Pharm J, 2019, 27: 1146-1156. DOI:10.1016/j.jsps.2019.09.011 |
| [8] |
Liu GQ, Bai X, Duan YB, et al. Changes in the intestinal flora of rats under high altitude hypoxia[J]. Acta Pharm Sin (药学学报), 2021, 56: 1100-1108. |
| [9] |
Choi MS, Yu JS, Yoo HH, et al. The role of gut microbiota in the pharmacokinetics of antihypertensive drugs[J]. Pharmacol Res, 2018, 130: 164-171. DOI:10.1016/j.phrs.2018.01.019 |
| [10] |
Magne F, Gotteland M, Gauthier L, et al. The firmicutes/bacteroidetes ratio: a relevant marker of gut dysbiosis in obese patients[J]. Nutrients, 2020, 12: 1474. DOI:10.3390/nu12051474 |
| [11] |
Willyard C. When drugs unintentionally affect gut bugs[J]. Nat Rev Drug Discov, 2018, 17: 383-384. DOI:10.1038/nrd.2018.88 |
| [12] |
Zhang JH, Sun YM, Wang R, et al. Gut microbiota-mediated drug-drug interaction between amoxicillin and aspirin[J]. Sci Rep, 2019, 9: 16194. DOI:10.1038/s41598-019-52632-5 |
| [13] |
Kim JK, Choi MS, Jeong JJ, et al. Effect of probiotics on pharmacokinetics of orally administered acetaminophen in mice[J]. Drug Metab Dispos, 2018, 46: 122-130. DOI:10.1124/dmd.117.077222 |
| [14] |
Mu CL, Zhu WY. Antibiotic effects on gut microbiota, metabolism, and beyond[J]. Appl Microbiol Biotechnol, 2019, 103: 9277-9285. DOI:10.1007/s00253-019-10165-x |
| [15] |
Loiacono LA, David SS. Detection of hypoxia at the cellular level[J]. Crit Care Clin, 2010, 26: 409-412. DOI:10.1016/j.ccc.2009.12.001 |
| [16] |
Anand AC, Sashindran VK, Mohan L. Gastrointestinal problems at high altitude[J]. Trop Gastroenterol, 2006, 27: 147-153. |
| [17] |
Adak A, Maity C, Ghosh K, et al. Alteration of predominant gastrointestinal flora and oxidative damage of large intestine under simulated hypobaric hypoxia[J]. Z Gastroenterol, 2014, 52: 180-186. DOI:10.1055/s-0033-1336007 |
| [18] |
Zhang W, Jiao LF, Liu RX, et al. The effect of exposure to high altitude and low oxygen on intestinal microbial communities in mice[J]. PLoS One, 2018, 13: e0203701. DOI:10.1371/journal.pone.0203701 |
| [19] |
Ma Y, Ma S, Shang CX, et al. Effects of hypoxic exposure on rats' gut microbiota[J]. Microbiol China (微生物学通报), 2019, 46: 120-129. |
| [20] |
Li L, Zhao X. Comparative analyses of fecal microbiota in Tibetan and Chinese Han living at low or high altitude by barcoded 454 pyrosequencing[J]. Sci Rep, 2015, 5: 14682. DOI:10.1038/srep14682 |
| [21] |
Li K, Dan Z, Gesang L, et al. Comparative analysis of gut microbiota of native Tibetan and Han populations living at different altitudes[J]. PLoS One, 2016, 11: e0155863. DOI:10.1371/journal.pone.0155863 |
| [22] |
Moeller AH, Li Y, Mpoudi NE, et al. Rapid changes in the gut microbiome during human evolution[J]. Proc Natl Acad Sci U S A, 2014, 111: 16431-16435. DOI:10.1073/pnas.1419136111 |
| [23] |
Lan DL, Ji WH, Lin BS, et al. Correlations between gut microbiota community structures of Tibetans and geography[J]. Sci Rep, 2017, 7: 16982. DOI:10.1038/s41598-017-17194-4 |
| [24] |
Jia Z, Zhao X, Liu X, et al. Impacts of the plateau environment on the gut microbiota and blood clinical indexes in Han and Tibetan individuals[J]. mSystems, 2020, 5: e00660-19. |
| [25] |
Li XY, Gao F, Li ZQ, et al. Comparison of the pharmacokinetics of sulfamethoxazole in male chinese volunteers at low altitude and acute exposure to high altitude versus subjects living chronically at high altitude: an open-label, controlled, prospective study[J]. Clin Ther, 2009, 31: 2744-2754. DOI:10.1016/j.clinthera.2009.11.019 |
| [26] |
Nian YQ. Effect of Simulated High Altitude Hypoxia on Pharmacokinetics of Metformin Hydrochloride and Acetaminophen (模拟高原低氧对盐酸二甲双胍和对乙酰氨基酚药代动力学特征的影响)[D]. Qinghai: Qinghai University, 2019.
|
| [27] |
Gong WW, Liu SH, Xu PX, et al. Simultaneous quantification of diazepam and dexamethasone in plasma by high-performance liquid chromatography with tandem mass spectrometry and its application to a pharmacokinetic comparison between normoxic and hypoxic rats[J]. Molecules, 2015, 20: 6901-6912. DOI:10.3390/molecules20046901 |
| [28] |
Gola S, Gupta A, Keshri GK, et al. Evaluation of hepatic metabolism and pharmacokinetics of ibuprofen in rats under chronic hypobaric hypoxia for targeted therapy at high altitude[J]. J Pharm Biomed Anal, 2016, 121: 114-122. DOI:10.1016/j.jpba.2016.01.018 |
| [29] |
Webster LK, Jones DB, Mihaly GW, et al. Effect of hypoxia on oxidative and reductive pathways of omeprazole metabolism by the isolated perfused rat liver[J]. Biochem Pharmacol, 1985, 34: 1239-1245. DOI:10.1016/0006-2952(85)90501-5 |
| [30] |
Ritschel WA, Paulos C, Arancibia A, et al. Pharmacokinetics of meperidine in healthy volunteers after short-and long-term exposure to high altitude[J]. J Clin Pharmacol, 1996, 36: 610-616. DOI:10.1002/j.1552-4604.1996.tb04225.x |
| [31] |
Zhang JH, Wang R, Xue H, et al. Effect of acute exposure to high altitude on pharmacokinetics of propranolol and metoprolol in rats[J]. J South Med Univ (南方医科大学学报), 2014, 34: 1616-1620. |
| [32] |
Zhang JH, Wang R, Xie H, et al. Effects of aminophylline on the pharmacokinetic parameters at high altitude[J]. Pharm J Chin PLA (解放军药学学报), 2014, 30: 125-131. |
| [33] |
Anjana GV, Krishna K, Joginder D. Effect of intermittent hypobaric hypoxia on efficacy and clearance of drug[J]. Indian J Med Res, 2012, 135: 211-216. |
| [34] |
Luo BF, Wang R, Li WB, et al. Pharmacokinetic changes of norfloxacin based on expression of MRP2 after acute exposure to high altitude at 4 300 m[J]. Biomed Pharmacother, 2017, 89: 1078-1085. DOI:10.1016/j.biopha.2017.02.092 |
| [35] |
Richer M, Lam YW. Hypoxia, arterial pH and theophylline disposition[J]. Clin Pharmacokinet, 1993, 25: 283-299. DOI:10.2165/00003088-199325040-00004 |
| [36] |
du Souich P, Hartemann D, Saunier C. Effect of acute and chronic moderate hypoxia on diltiazem kinetics and metabolism in the dog[J]. Pharmacology, 1993, 47: 378-385. DOI:10.1159/000139121 |
| [37] |
Duan YB, Zhu JB, Yang JX, et al. Effect of hypoxia on drug metabolizing enzymes and transporters and the role of microRNA[J]. Acta Pharm Sin (药学学报), 2021, 56: 50-60. |
| [38] |
Fradette C, Batonga J, Teng S, et al. Animal models of acute moderate hypoxia are associated with a down-regulation of CYP1A1, 1A2, 2B4, 2C5, and 2C16 and up-regulation of CYP3A6 and P-glycoprotein in liver[J]. Drug Metab Dispos, 2007, 35: 765-771. DOI:10.1124/dmd.106.013508 |
| [39] |
Kurdi J, Maurice H, Elkadi AO, et al. Effect of hypoxia alone or combined with inflammation and 3-methylcholanthrene on hepatic cytochrome P450 in conscious rabbits[J]. Br J Pharmacol, 1999, 128: 365-373. DOI:10.1038/sj.bjp.0702795 |
| [40] |
Li XY, Wang XJ, Li YP, et al. Effect of exposure to acute and chronic high-altitude hypoxia on the activity and expression of CYP1A2, CYP2D6, CYP2C9, CYP2C19 and NAT2 in rats[J]. Pharmacology, 2014, 93: 76-83. DOI:10.1159/000358128 |
| [41] |
Suzuki E, Matsunaga T, Aonuma A, et al. Effects of hypoxia-inducible factor-1α chemical stabilizer, CoCl2 and hypoxia on gene expression of CYP3As in human fetal liver cells[J]. Drug Metab Pharmacokinet, 2012, 27: 398-404. DOI:10.2133/dmpk.DMPK-11-RG-074 |
| [42] |
Legendre C, Hori T, Loyer P, et al. Drug-metabolising enzymes are down-regulated by hypoxia in differentiated human hepatoma HepaRG cells: HIF-1α involvement in CYP3A4 repression[J]. Eur J Cancer, 2009, 45: 2882-2892. DOI:10.1016/j.ejca.2009.07.010 |
| [43] |
Du SP, Fradette C. The effect and clinical consequences of hypoxia on cytochrome P450, membrane carrier proteins activity and expression[J]. Expert Opin Drug Metab Toxicol, 2011, 7: 1083-1100. DOI:10.1517/17425255.2011.586630 |
| [44] |
Carmody RN, Turnbaugh PJ. Host-microbial interactions in the metabolism of therapeutic and diet-derived xenobiotics[J]. J Clin Invest, 2014, 124: 4173-4181. DOI:10.1172/JCI72335 |
| [45] |
Li H, He J, Jia W. The influence of gut microbiota on drug metabolism and toxicity[J]. Expert Opin Drug Metab Toxicol, 2016, 12: 31-40. DOI:10.1517/17425255.2016.1121234 |
| [46] |
Malfatti MA, Kuhn EA, Murugesh DK, et al. Manipulation of the gut microbiome alters acetaminophen biodisposition in mice[J]. Sci Rep, 2020, 10: 4571. DOI:10.1038/s41598-020-60982-8 |
| [47] |
Haiser HJ, Gootenberg DB, Chatman K, et al. Predicting and manipulating cardiac drug inactivation by the human gut bacterium Eggerthella lenta[J]. Science, 2013, 341: 295-298. DOI:10.1126/science.1235872 |
| [48] |
Klatt NR, Cheu R, Birse K, et al. Vaginal bacteria modify HIV tenofovir microbicide efficacy in African women[J]. Science, 2017, 356: 938-945. DOI:10.1126/science.aai9383 |
| [49] |
Elmer GW, Remmel RP. Role of the intestinal microflora in clonazepam metabolism in the rat[J]. Xenobiotica, 1984, 14: 829-840. DOI:10.3109/00498258409151481 |
| [50] |
Maini RV, Bess EN, Bisanz JE, et al. Discovery and inhibition of an interspecies gut bacterial pathway for levodopa metabolism[J]. Science, 2019, 364: 6445. |
| [51] |
Yip LY, Aw CC, Lee SH, et al. The liver-gut microbiota axis modulates hepatotoxicity of tacrine in the rat[J]. Hepatology, 2018, 67: 282-295. DOI:10.1002/hep.29327 |
| [52] |
Tobin P, Clarke S, Seale JP, et al. The in vitro metabolism of irinotecan (CPT-11) by carboxylesterase and beta-glucuronidase in human colorectal tumours[J]. Br J Clin Pharmacol, 2006, 62: 122-129. DOI:10.1111/j.1365-2125.2005.02477.x |
| [53] |
Pellock SJ, Redinbo MR. Glucuronides in the gut: sugar-driven symbioses between microbe and host[J]. J Biol Chem, 2017, 292: 8569-8576. DOI:10.1074/jbc.R116.767434 |
| [54] |
Kim IS, Yoo DH, Jung IH, et al. Reduced metabolic activity of gut microbiota by antibiotics can potentiate the antithrombotic effect of aspirin[J]. Biochem Pharmacol, 2016, 122: 72-79. DOI:10.1016/j.bcp.2016.09.023 |
| [55] |
Chung KT, Stevens SE, Cerniglia CE. The reduction of azo dyes by the intestinal microflora[J]. Crit Rev Microbiol, 1992, 18: 175-190. DOI:10.3109/10408419209114557 |
| [56] |
Zhang JH, Zhang JM, Wang R. Gut microbiota modulates drug pharmacokinetics[J]. Drug Metab Rev, 2018, 50: 357-368. DOI:10.1080/03602532.2018.1497647 |
| [57] |
He Y, Fu LH, Li YP, et al. Gut microbial metabolites facilitate anticancer therapy efficacy by modulating cytotoxic CD8+ T cell immunity[J]. Cell Metab, 2021, 33: 988-1000. DOI:10.1016/j.cmet.2021.03.002 |
| [58] |
Fu ZD, Selwyn FP, Cui JY, et al. RNA-Seq profiling of intestinal expression of xenobiotic processing genes in germ-free mice[J]. Drug Metab Dispos, 2017, 45: 1225-1238. DOI:10.1124/dmd.117.077313 |
| [59] |
Dabek M, McCrae SI, Stevens VJ, et al. Distribution of beta-glucosidase and beta-glucuronidase activity and of beta-glucuronidase gene gus in human colonic bacteria[J]. FEMS Microbiol Ecol, 2008, 66: 487-495. DOI:10.1111/j.1574-6941.2008.00520.x |
| [60] |
Alexander C, Swanson KS, Fahey GC, et al. Perspective: physiologic importance of short-chain fatty acids from nondigestible carbohydrate fermentation[J]. Adv Nutr, 2019, 10: 576-589. DOI:10.1093/advances/nmz004 |
| [61] |
Poesen R, Evenepoel P, De LH, et al. Metabolism, protein binding, and renal clearance of microbiota-derived p-cresol in patients with CKD[J]. Clin J Am Soc Nephrol, 2016, 11: 1136-1144. DOI:10.2215/CJN.00160116 |
| [62] |
Clayton TA, Baker D, Lindon JC, et al. Pharmaco-metabonomic identification of a significant host-microbiome metabolic interaction affecting human drug metabolism[J]. Proc Natl Acad Sci U S A, 2009, 106: 14728-14733. DOI:10.1073/pnas.0904489106 |
| [63] |
Pavlovic N, Golocorbin KS, danic M, et al. Bile acids and their derivatives as potential modifiers of drug release and pharmacokinetic profiles[J]. Front Pharmacol, 2018, 9: 1283-1305. DOI:10.3389/fphar.2018.01283 |
| [64] |
Zarrinpar A, Chaix A, Xu ZZ, et al. Antibiotic-induced microbiome depletion alters metabolic homeostasis by affecting gut signaling and colonic metabolism[J]. Nat Commun, 2018, 9: 2872. DOI:10.1038/s41467-018-05336-9 |
| [65] |
Toda T, Saito N, Ikarashi N, et al. Intestinal flora induces the expression of CYP3A in the mouse liver[J]. Xenobiotica, 2009, 39: 323-334. DOI:10.1080/00498250802651984 |
| [66] |
Toda T, Ohi K, Kudo T, et al. Ciprofloxacin suppresses CYP3A in mouse liver by reducing lithocholic acid-producing intestinal flora[J]. Drug Metab Pharmacokinet, 2009, 24: 201-208. DOI:10.2133/dmpk.24.201 |
| [67] |
Morgan ET, Dempsey JL, Mimche SM, et al. Physiological regulation of drug metabolism and transport: pregnancy, microbiome, inflammation, infection, and fasting[J]. Drug Metab Dispos, 2018, 46: 503-513. DOI:10.1124/dmd.117.079905 |
| [68] |
Illes P, Krasulova K, Vyhlidalova B, et al. Indole microbial intestinal metabolites expand the repertoire of ligands and agonists of the human pregnane X receptor[J]. Toxicol Lett, 2020, 334: 87-93. DOI:10.1016/j.toxlet.2020.09.015 |
| [69] |
Selwyn FP, Cheng SL, Klaassen CD, et al. Regulation of hepatic drug-metabolizing enzymes in germ-free mice by conventionalization and probiotics[J]. Drug Metab Dispos, 2016, 44: 262-274. DOI:10.1124/dmd.115.067504 |
| [70] |
Kuno T, Hirayama KM, Ito S, et al. Effect of intestinal flora on protein expression of drug-metabolizing enzymes and transporters in the liver and kidney of germ-free and antibiotics-treated mice[J]. Mol Pharm, 2016, 13: 2691-2701. DOI:10.1021/acs.molpharmaceut.6b00259 |
| [71] |
Zhou XJ, Nian YQ, Qiao YJ, et al. Hypoxia plays a key role in the pharmacokinetic changes of drugs at high altitude[J]. Curr Drug Metab, 2018, 19: 960-969. DOI:10.2174/1389200219666180529112913 |
| [72] |
Fradette C, Bleau AM, Pichette V, et al. Hypoxia-induced down-regulation of CYP1A1/1A2 and up-regulation of CYP3A6 involves serum mediators[J]. Br J Pharmacol, 2002, 137: 881-891. DOI:10.1038/sj.bjp.0704933 |
| [73] |
Rahman MS, Thomas P. Effects of hypoxia exposure on hepatic cytochrome P450 1A (CYP1A) expression in atlantic croaker: molecular mechanisms of CYP1A down-regulation[J]. PLoS One, 2012, 7: e40825. DOI:10.1371/journal.pone.0040825 |
| [74] |
Fradette C, Du SP. Effect of hypoxia on cytochrome P450 activity and expression[J]. Curr Drug Metab, 2004, 5: 257-271. DOI:10.2174/1389200043335577 |
| [75] |
Duan YB, Zhu JB, Yang JX, et al. Regulation of high-altitude hypoxia on the transcription of CYP450 and UGT1A1 mediated by PXR and CAR[J]. Front Pharmacol, 2020, 11: 574176. DOI:10.3389/fphar.2020.574176 |
| [76] |
Yan R, Yang Y, Chen YJ. Pharmacokinetics of Chinese medicines: strategies and perspectives[J]. Chin Med, 2018, 13: 24. DOI:10.1186/s13020-018-0183-z |
| [77] |
Zimmermann M, Zimmermann-Kogadeeva M, Wegmann R, et al. Separating host and microbiome contributions to drug pharmacokinetics and toxicity[J]. Science, 2019, 363: aat9931. DOI:10.1126/science.aat9931 |
| [78] |
Lagier JC, Khelaifia S, Alou MT, et al. Culture of previously uncultured members of the human gut microbiota by culturomics[J]. Nat Microbiol, 2016, 1: 16203. DOI:10.1038/nmicrobiol.2016.203 |
| [79] |
Jia YF, Wang PP, Chen YJ, et al. Advances in gut microbial drug metabolism[J]. Prog Pharm Sci (药学进展), 2020, 44: 83-99. |
 2021, Vol. 56
2021, Vol. 56


