2. 邯郸市第一医院药学部, 河北 邯郸 056002
2. Department of Pharmacy, Handan First Hospital, Handan 056002, China
蛋白质二硫键异构酶(protein disulfide isomerase, PDI) 家族属于硫氧还蛋白(thioredoxin, TRX) 超家族。PDI家族有20多个成员, 包括PDIA1、PDIA3等, 其中PDIA1是该家族的原型蛋白, 一般称为PDI。PDI主要存在于内质网(endoplasmic reticulum, ER), 具有分子伴侣功能, 帮助蛋白正确折叠, 形成天然构象。当在某些病理情况下出现蛋白折叠错误或未折叠时, 会引起ER应激(ER stress)、氧化应激、未折叠蛋白应答(unfolded protein response, UPR) 和内质网相关降解(ER-associated degradation, ERAD) 等, PDI在这些过程中具有重要的调节作用。脊髓侧索硬化症(amyotrophic lateral sclerosis, ALS) 是一种神经退行性疾病, 主要特征是蛋白折叠错误、异常聚集, 并引起ER应激等反应, 最终导致神经元病变甚至死亡。近年来, 人们在ALS患者中发现了PDI突变体, 并证实了PDI在ALS疾病中的重要作用, 其不仅可抑制ALS相关蛋白的异常聚集, 而且可影响神经生长和突触功能等。本文重点总结PDI蛋白在ALS疾病中的研究进展, 以期为ALS疾病的治疗提供新思路。
1 PDI简介PDI由P4HB (prolyl 4-hydroxylase subunit beta) 基因编码, 是脯氨酸-4-羟化酶P4H蛋白的β亚基, 故又称P4HB。PDI主要由活性结构域和底物结合结构域构成, 整体呈U型排布, 其亚细胞定位不局限于ER, 也存在于其他部位, 并具有生物活性包括二硫键异构酶活性和分子伴侣功能等。基于对PDI蛋白结构和功能的认识, 人们也找到了许多PDI的调节剂如LOC14、16F16等。
1.1 PDI的结构PDI蛋白分子量约为57 kDa, 主要包含4个TRX样结构域(a、b、b'和a') 以及羧基末端c。a和a'是活性结构域, 包含活性位点CGHC; b和b'是底物结合结构域, 通过疏水作用力与底物结合, 其中b'在底物结合过程中具有更重要的作用。c末端有KDEL序列, 使PDI滞留于ER中。b'和a'之间的连接区称为x-linker, 由19个氨基酸组成; x-linker可与b'结构域的底物结合位点结合, 有capped和uncapped两种状态而影响底物结合。PDI蛋白整体呈U型或马蹄形, 活性结构域a和a'相对, 底物结合结构域b和b'位于内侧。PDI蛋白结构如图 1所示。
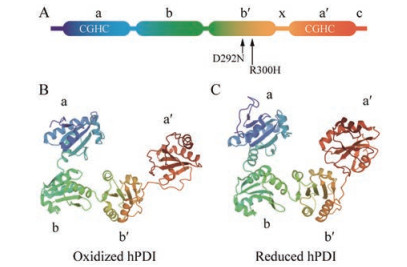
|
Figure 1 Overall structure of human protein disulfide isomerase (hPDI). A: Major structural domains of hPDI including a-b-b'-x-a'-c. Active sites CGHC locate in a and a' domains. Amyotrophic lateral sclerosis (ALS) related PDI mutants D292N and R300H locate in the b' domain; B: Crystal structure of oxidized hPDI (PDB code: 4EL1); C: Crystal structure of reduced hPDI (PDB code: 4EKZ) |
X射线衍射和高速原子力显微镜分别从晶体和液态的角度观察到氧化型和还原型两种不同状态的人源hPDI蛋白: 氧化型PDI蛋白结构较开阔, 而还原型较紧凑[1, 2]。氧化型是指PDI蛋白活性位点CGHC中的半胱氨酸残基的巯基形成二硫键, 还原型是指半胱氨酸残基的巯基处于游离状态。氧化型PDI蛋白可形成二聚体, 二聚体中间是很大的疏水空间, 同时又有多个活性位点, 因此可容纳更大的底物分子, 或多个二聚体同时工作完成底物蛋白的氧化折叠[2]。还原型PDI与底物可能的作用模式为活性位点C1GHC2中的C1与底物巯基结合形成二硫键, C2再打开此二硫键[3]。微粒体甘油三酯转运蛋白(microsomal triglyceride transfer protein, MTP) 是异源二聚体, 由MTP的α亚基和β亚基组成, 其中β亚基就是PDI蛋白。PDI与MTP的α亚基相互作用时整体结构开阔, CGHC处于还原态[4]。因此, 有研究者认为PDI主要有底物结合和不结合两种状态, 而不是氧化型和还原型[4]。
虽然PDI蛋白有多个结构域, 但作为一个整体, 这些结构域是协调一致的。b和b'会增强a和a'的活性, a和a'对于b和b'结合大分子底物也至关重要。另外, PDI与底物的相互作用方式是多样的, 这可能受到底物驱动和氧化还原环境的影响。
1.2 PDI的功能“PDI既是酶又是分子伴侣”的概念是王志珍院士在1993年首次提出的[5]。PDI的酶学分类编号是EC: 5.3.4.1, 通过活性位点CGHC形成分子间或分子内二硫键, 发挥二硫键异构酶活性, 如PDI通过酶的催化作用使变性的RNase形成正确二硫键并恢复活性[6]。PDI的分子伴侣功能是指蛋白辅助功能, 辅助分子内不存在二硫键和巯基的蛋白正确折叠更能体现PDI的分子伴侣特性。例如P4H的α亚基主要和底物结合发挥酶解作用, 而β亚基即PDI主要通过分子伴侣功能辅助α亚基更好地发挥作用。Tsai等[7]通过研究PDI对霍乱毒素的调节作用, 提出PDI是“氧化还原依赖的分子伴侣”。霍乱毒素A1亚基从毒素上分离后, 由ER进入胞浆的过程需要PDI辅助, 这依赖于还原型PDI, 因此认为PDI是氧化还原依赖的分子伴侣。
对PDI蛋白来说, 酶或分子伴侣是功能上的定义, 氧化、还原或异构二硫键是结构上的定义, 两者并不矛盾。PDI的a和a'结构域活性位点CGHC主要体现其酶活性和氧化还原状态, b和b'结构域的底物结合功能主要体现其分子伴侣作用。对于某一底物来说, PDI蛋白各结构域之间通过协作的方式来发挥其生物功能。作为一个集酶活性和分子伴侣功能于一身的蛋白, PDI注定是个多功能的重要大分子。
1.3 PDI的亚细胞定位PDI蛋白主要存在于ER, 但也出现在ER以外的区域如细胞外、细胞膜、细胞质、细胞核等。Detwiler课题组[8]在1992年首次发现PDI可从血小板分泌且具有生物活性, 后来发现血小板分泌的PDI对血栓的形成有调节作用。除了血小板以外, 还有很多细胞也都可以分泌PDI蛋白, 如神经元、胶质细胞等[9]。ALS患者脑脊液和血液中的PDIA1 (PDI) 和PDIA3 (ERp57) 蛋白表达量升高, 提示PDI确实可以分泌出来, 甚至可以作为诊断ALS疾病的生物标记物。另外, PDI和ALS的相关蛋白如超氧化物歧化酶1 (superoxide dismutase 1, SOD1)、核酸结合蛋白TDP-43/TARDBP (transactive response DNA-binding protein 43 kDa) 的共定位发生在细胞质中, 而不是ER, 这也说明PDI可以离开ER。
PDI蛋白离开ER的机制目前还不清楚。有研究发现[10], 过表达reticulon-4A (NOGO-A) 可以使PDI离开ER并进入囊泡, 形成PDI点状复合物, 这对于SOD1G93A小鼠是有保护意义的。如果将ALS模型小鼠SOD1G93A中的NOGO-A和NOGO-B敲除, 将导致ALS病情恶化, 这意味着reticulon-4A可通过调节PDI的分布对SOD1突变体相关的ALS病情产生保护作用[10]。另有文献报道[11], 在人神经母细胞瘤细胞SH-SY5H中过表达RTN-1C (reticulon protein 1-C) 也可使PDI从ER进入囊泡, 与此同时PDI活性增加, 亚硝基化水平降低。
1.4 PDI的调节剂迄今为止, 人们已开发了许多PDI蛋白调节剂如LOC14、16F16、BMC、异槲皮素等, 它们或是人工合成或是来自天然化合物, 部分化合物结构如图 2所示。这些调节剂对神经退行性疾病如亨廷顿病(Huntington disease, HD)、阿尔茨海默病(Alzheimer's disease, AD) 和ALS是有一定神经保护作用的。
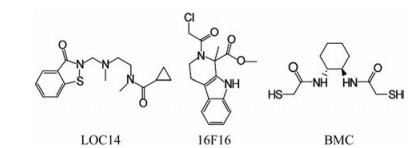
|
Figure 2 Structures of PDI modifiers including LOC14, 16F16, and BMC |
LOC14是作用较强的PDI抑制剂, 半数有效抑制浓度(IC50) 值是0.5 μmol·L-1, 解离常数Kd值为62 nmol·L-1[12]。构效关系研究发现[12], 硫原子对于LOC14和PDI的结合至关重要, 如果把硫原子替换成氧原子, 将降低两者亲和力。LOC14与a结构域的可逆性结合使PDI保持在氧化状态。氧化状态的PDI具有更好的分子伴侣活性, 有利于蛋白折叠, 缓解ER应激。LOC14可缓解过表达多聚谷氨酰胺亨廷顿突变蛋白(mHTT-Q73) 对大鼠中型棘神经元的毒性作用。小鼠体内的药代动力学研究结果显示, LOC14与血浆蛋白结合率低, 可通过血脑屏障, 在肝脏和血浆中比较稳定。LOC14对PDI的特异调节作用以及良好的药代动力学特性预示着其潜在的成药可能性。
16F16通过不可逆结合a结构域来抑制PDI活性, 缓解亨廷顿多聚谷氨酰胺突变蛋白Q103引起的PC12神经细胞凋亡[13]。Hoffstrom等[13]认为Q103蛋白可使PDI在线粒体相关的ER膜上聚集, 导致线粒体外膜通透性(mitochondrial outer-membrane permeabilization, MOMP) 增加, 引起细胞凋亡。PDI的MOMP作用可能与Bax/Bak蛋白复合物或者Bcl-2蛋白有关, 16F16通过抑制该作用发挥细胞保护作用。构效关系研究发现[13], 16F16结构中的氯乙酰基是主要官能团, 与PDI发生共价结合。若将16F16结构中形成酯键的甲基换成乙基并不影响其活性。16F16对HTT突变蛋白和Aβ突变蛋白(主要引起AD) 诱导的大鼠脑神经元死亡都有保护作用。
BMC是根据PDI的生物活性结构域CGHC设计的, 可以催化蛋白质分子中二硫键的氧化、还原和异构[14]。在神经退行疾病如ALS、HD、AD等晚期, 患者体内可检测到亚硝基化PDI蛋白分子, 这种PDI已失去了生物活性, 如果此时给予PDI蛋白mimics应可缓解患者症状[15]。研究发现, 给予BMC可抑制ALS相关SOD1突变蛋白的异常聚集[16]。
2 PDI在ALS疾病中的作用ALS是一种神经退行性疾病, 成年时发病并伴随运动神经元快速退化, 主要临床表现是疲惫、肌无力、肌萎缩直至瘫痪、死亡。目前针对ALS的药物只有利鲁唑(riluzole) 和依达拉奉(edaravone)[17]。利鲁唑是1995年美国食品药品监督管理局FDA (Food and Drug Administration) 批准的第1个用来治疗ALS的药物。它是苯并噻唑类化合物, 通过抑制脑内兴奋性氨基酸的释放来发挥神经元保护作用, 其相对安全、不良反应较轻, 但只能延长ALS患者2~3个月的生存期, 不能改善肌肉症状[18]。依达拉奉是一种自由基清除剂, 长期用于改善脑梗死所致的神经症状, 后来发现它可显著改善ALS早期症状[19]。2015年, 依达拉奉在日本被批准用于治疗ALS, 这是第2个针对ALS疾病的药物。
到目前为止, ALS的发病机制并不完全清楚。约有5%~10%的ALS患者是由多基因突变引起的, 是家族性的, 称为fALS (familial ALS); 而大部分患者是散发性的, 称为sALS (sporadic ALS)。其中, C9ORF72 (chromosome 9 open reading frame 72) 六核苷酸重复序列和铜锌SOD1基因突变是fALS最常见的病因, 二者约共占fALS的50%。TDP-43和肉瘤融合/脂肪肉瘤转运蛋白FUS/TLS (fused in sarcoma/translocated in liposarcoma protein) 基因突变约各占fALS的5%。
ALS的主要特征是上述相关蛋白如SOD1、TDP-43、FUS等的错误折叠、异常聚集, 从而引起ER应激、氧化应激、UPR以及ERAD障碍等反应, 导致神经元病变甚至死亡[20]。在ALS疾病中PDI蛋白表达升高可以抑制蛋白异常聚集而起到保护作用, 但在疾病晚期时PDI也会出现变性如亚硝基化。不仅如此, 最近在ALS中还发现了PDI突变基因, 这充分说明PDI在ALS中的重要作用。
2.1 ALS中的PDI突变基因应用DNA测序的方法在英国及其他欧盟国家的ALS患者中筛选候选基因时, 首次发现P4HB基因非编码区的单核苷酸多态性(single nucleotide polymorphism, SNP) 与fALS和sALS相关[21]。P4HB基因的2个SNP位点rs876016和rs2070872与fALS存在基因型显著相关性, 其中rs876016与ALS (包括fALS和sALS) 存在等位基因显著相关性, 携带rs2070872次要等位基因型(AG或GG) 的fALS患者生存时间明显降低。P4HB的4个SNP位点rs876017、rs876016、rs2070872和rs8324分别位于内含子4、6以及3' UTR (untranslated region) 非翻译区, 存在遗传连锁不平衡。其中AAAC单体型频率在健康人中较高而在ALS患者中显著降低。7个单体型(AGAC、GGGC、AAAA、GAAA、GGAA、AGGC和GAAC) 与fALS显著相关, 1个单体型(AGGA) 与sALS显著相关, 还有1个单体型(AAGC) 在SOD1阳性fALS患者高表达。SOD1是第1个发现的与ALS相关的基因, 迄今为止已发现170多个ALS相关的SOD1突变体。这些SNP位点说明P4HB可能是ALS的危险因素。
应用聚合酶链反应限制性片段长度多态性(polymerase chain reaction-restriction fragment length polymorphism, PCR-RFLP) 分型法在中国汉族sALS患者中也发现了PDI的2个SNP位点: rs876016和rs2070872[22]。rs876016 (C) 和rs2070872 (G) 等位基因频率在sALS患者中显著升高, rs876016/rs2070872 (C/G) 基因型频率也与sALS显著相关, 说明PDI突变基因可能影响sALS的发生发展过程。
Gonzalez-Perez等[23]通过基因测序的方法发现P4HB基因编码区(即外显子) SNP位点所导致的PDI突变蛋白与ALS相关: 通过DNA测序方法在美国ALS患者中鉴定出2个PDI突变蛋白p.D292N和p.R300H, 接着通过外显子测序方法在美国和加拿大ALS患者中发现了另外7个PDI突变蛋白。这些突变蛋白的存在说明PDI与ALS密切相关, 可能成为ALS潜在的治疗靶点。
2.2 ALS相关PDI突变蛋白的活性改变PDID292N和PDIR300H是ALS相关的2个PDI突变体, 其整体结构没有太大改变, 但是与底物的结合受到了破坏, 因为突变位点都位于底物结合结构域b'[24] (图 1)。PDID292N突变体的x-linker阻碍底物接近结合位点, 而突变体PDIR300H反而更有利于底物结合。ERO1Lα (ER oxidoreductin 1Lα) 与PDID292N的结合增强, 但与PDIR300H的结合减弱, 两者都导致H2O2的生成减少; BPTI (bovine pancreatic trypsin inhibitor) 与PDID292N和PDIR300H的相互作用改变, BPTI氧化复活受到抑制[24]。这些数据说明PDID292N和PDIR300H突变体蛋白与底物结合力改变将导致其活性降低。
PDI的氧化还原活性对于ALS疾病至关重要[25]。在小鼠脑神经瘤细胞Neuro-2a中表达氧化还原敏感的生物感受器蛋白(redox biosensor) 的方法显示PDI-WT具有氧化还原活性, 而活性位点突变体PDI-QUAD (4个活性位点的半胱氨酸C全部突变为丝氨酸S) 及2个与ALS相关的突变体PDI-D292N、PDI-R300H都没有氧化还原活性, 但保留了分子伴侣功能[25]。PDI的氧化还原活性不仅对ALS TDP-43突变体或SOD1突变体引起的蛋白错误折叠、ER应激、ER-高尔基转运、细胞凋亡反应有保护作用, 而且对TDP-43突变体胞质错误定位以及表达SOD1突变体的斑马鱼的运动功能损伤都有保护意义[25]。与ALS相关的突变体PDI-D292N和PDI-R300H由于缺乏氧化还原活性对TDP-43或SOD1突变体所引起的细胞毒性抵抗力降低[25]。
ALS模型SOD1G93A小鼠和ALS患者脊髓中出现PDI蛋白亚硝基化(s-nitrosylation) 即SNO-PDI。SNO-PDI是指在PDI蛋白活性位点半胱氨酸残基加上NO基团, 从而使PDI的蛋白酶活性和分子伴侣功能降低。其他神经退行性疾病如AD、HD等也出现SNO-PDI[26], 说明SNO-PDI是神经退行性疾病中一种常见的现象。SNO-PDI的产生可能与病理状态下NOS活性增加或线粒体过度激活产生过多NO有关。另外, 细胞表面PDI可以通过SNO-PDI的方式帮助NO进入细胞, 从而导致更多的蛋白亚硝基化[15]。所以SNO-PDI不仅使PDI蛋白失去活性, 还可能导致其他蛋白亚硝基化而使ALS病情进一步恶化。
ALS模型SOD1G93A小鼠的小胶质细胞中PDI的升高与NOX (NADPH oxidase) 活性增加、ROS (reactive oxygen species) 水平升高相关, 应用PDI抑制剂抑制其活性或通过siRNA敲低PDI都可降低NOX和ROS活性[27]。这提示PDI也与氧化应激密切相关, 且胶质细胞PDI蛋白活性升高并不一定有细胞保护作用, 也可能产生细胞毒性。
2.3 ALS相关PDI突变蛋白对神经生长的影响斑马鱼中表达ALS相关的突变蛋白PDID292N和PDIR300H不仅会改变运动神经元的形态, 还会影响其功能[24]。表达PDIR300H的斑马鱼神经元轴突变短, 分支增加, 说明PDI蛋白影响神经突起的生长。PDIR300H斑马鱼在TEER (touch-evoked escape response) 逃跑实验中反应迟钝、逃跑速度降低, 可能是由于神经肌肉联系受到损伤所致。在体外实验中还发现[24], PDID292N和PDIR300H也会影响小鼠运动神经元NSC-34、大鼠脊髓运动神经元以及人胚胎干细胞HuESC 3 Hb9:: GFP诱导分化的运动神经元神经突起的生长。
无独有偶, PDI还可调节背根神经节(dorsal root ganglion, DRG) 神经元神经突起的生长[9]。P-half-somite鸡胚细胞表面存在功能性PDI (cell surface PDI, csPDI)。当DRG生长椎的伪足与P-half-somite细胞表面接触时会产生排斥反应, 伪足回缩, 生长锥重新定位远离P-half-somite细胞。无论应用siRNA技术敲低PDI, 还是化学小分子PACMA31抑制PDI蛋白活性, 都可使神经元轴突横穿P-half-somite。进一步研究发现, csPDI可将P-half-somite细胞外的NO转运到DRG生长椎的伪足胞内, NO可能通过亚硝基化MAP1B亚基LC1, 抑制dynein活性, 从而产生排斥反应, 改变神经生长方向。这些数据说明csPDI可通过NO转运作用调节神经突起的接触抑制行为。
Renthal等[28]通过RNA-seq技术也发现外周神经损伤后, DRG神经元中PDI的转录水平发生变化。本课题组通过Western blot实验发现, 在坐骨神经损伤后, DRG组织的PDI蛋白表达量升高。这说明PDI蛋白在神经发育、神经生长、突触联系等方面发挥着重要作用。
2.4 PDI可抑制ALS相关蛋白的异常聚集ALS疾病模型SOD1G93A小鼠和ALS患者脊髓中PDI表达升高, 在小鼠运动神经元细胞株NSC-34中过表达PDI可降低SOD1异常聚集, 敲低PDI可增加SOD1聚集[16], 说明PDI蛋白通过抑制SOD1蛋白异常聚集来阻止ALS疾病的进展。野生型同源二聚体SOD1蛋白有4个半胱氨酸(C6、C57、C111、C146), C57和C146形成分子内二硫键, C6和C111是游离巯基。C111暴露在二聚体分界面, 可能与突变型SOD1分子聚合物的形成有关, PDI对突变型SOD1聚集的抑制作用可能与C111位点有关, 但这需要进一步求证。
SNO-PDI出现在ALS模型SOD1G93A小鼠和ALS患者的脊髓中[16]。亚硝基化使PDI蛋白酶活性降低, PDI不仅失去正常的保护作用如抑制SOD1异常聚集, 而且使ALS病情加重, 这也从侧面印证了PDI在ALS中的保护作用。在PDI蛋白自身异常的情况下, 如果给予能够模拟PDI正常活性的小分子化合物, 应该能够缓解ALS病情。事实上, 给予BMC确实能抑制SOD1突变蛋白的异常聚集[16]。BMC是一个小分子PDI活性模拟化合物, 可模拟PDI蛋白二硫键异构酶活性和分子伴侣功能, 有效帮助蛋白折叠, 维护其活性天然构象[14]。SNO-PDI以及BMC的数据说明PDI蛋白完整的生物活性是其抑制SOD1突变蛋白异常聚集、发挥ALS保护作用所必需的。
TDP-43主要存在于细胞核中, 参与RNA代谢过程[29]。ALS患者运动神经元细胞质中出现TDP-43错误定位和异常聚集, 引起一系列应激反应, 导致细胞功能异常。TDP-43胞质聚合物已成为ALS的标志。PDI可通过氧化还原活性抑制TDP-43突变体在细胞质的错误定位和异常聚集, 从而缓解ALS疾病症状[25]。肉瘤融合/脂肪肉瘤转运蛋白FUS/TLS也是一种核酸结合蛋白, 与TDP-43的正常工作模式和ALS致病模式相似。PDI与FUS在sALS和fALS患者脊髓神经元胞质中也有共定位, 但两蛋白之间的功能研究并不多[30]。
3 PDI家族其他蛋白在ALS中的作用PDI家族除了PDI, 还有PDIA3 (即ERp57或GRP58)、PDIA12 [即TMX2 (thioredoxin-related transmembrane protein 2)] 等20多个成员, 它们至少含有一个TRX样结构域, 主要位于ER上, 辅助蛋白折叠, 调节ER稳态。目前, 除了PDI与ALS高度相关外, 还发现ERp57和TMX2与ALS疾病相关。ERp57与PDI的同源性高, 结构功能相似, 在ALS中的作用也相似, 可抑制蛋白异常聚集、影响神经肌肉突触联系等。TMX2是跨膜型蛋白二硫键异构酶, 对ALS相关基因C9ORF72引起的细胞毒性有保护作用。
3.1 ERp57在ALS中的作用ERp57与PDI有高度同源性, 与PDI有相似的结构域顺序a-b-b'-a'-c, a和a'的活性位点也是CGHC。ERp57和PDI不仅结构相似, 功能也相近。在ALS疾病中, ERp57对SOD1突变蛋白的异常聚集有保护作用[31]。过表达ERp57可抑制SOD1异常聚集和由此引起的ER应激、泛素蛋白酶系统功能异常和细胞凋亡等, 敲低ERp57却能增强这些细胞毒性[31]。在sALS患者脊髓中, ERp57和TDP-43也有部分共定位[31]。
Gonzalez-Perez等[23]在ALS患者中除了发现9个PDI突变体外, 还发现了7个ERp57突变体包括ERp57D217N和ERp57Q481K等。ERp57D217N和ERp57Q481K的突变位点分别位于b和a'结构域, 它们与钙联蛋白(calnexin, CNX) 和钙网蛋白(calreticulin, CRT) 的作用增强[24]。D217N是一个突变的糖基化位点, 但其意义尚不清楚[24]。
与PDIR300H相似, 表达ERp57D217N和ERp57Q481K的斑马鱼神经元轴突变短, 分支增加。ERP57Q481K斑马鱼的运动功能和神经肌肉联系受到损伤[24]。与ALS相关的PDI突变蛋白相似, 这2个ERp57突变蛋白也影响神经突起的生长。条件性敲除神经系统ERp57的小鼠神经肌肉接头形态和肌电图都变得异常, 运动功能受损, 体重下降, 死亡率增加[24]。神经系统ERp57缺陷小鼠神经肌肉接头处乙酰胆碱受体表达降低, 说明肌肉的神经支配减少; SV2蛋白表达改变, 出现异常聚集, 说明突触功能受损[24]。
在ALS疾病的晚期阶段过表达ERp57可减少SOD1突变蛋白的异常聚集; 而在早期阶段, 过表达ERp57的SOD1G93A小鼠神经肌肉突触联系的损伤延迟, 运动功能可得到改善[32]。这说明ERp57不是通过抑制SOD1突变蛋白聚集来保护神经肌肉突触的。蛋白质组学研究结果显示, 过表达ERp57可能是通过提高突触蛋白和细胞骨架蛋白水平来发挥神经保护作用的[32]。
3.2 TMX2对ALS的影响TMX2是PDI家族中一类特殊的、滞留在ER中的跨膜蛋白, 与ER应激密切相关。TMX2活性位点是SNDC, 这有别于PDI的活性位点CGHC。目前对TMX2的研究还比较少。fALS相关基因C9ORF72六核苷酸重复序列GGGGCC可通过RAN (repeat-associated non-AUG) 翻译的方式从正反两个方向翻译出多聚二肽重复蛋白(dipeptide repeat proteins, DPRs)。DPRs有5种: GA (glycine-alanine)、GR (glycine-arginine)、PA (proline-alanine)、PR (proline-arginine) 和GP (glycine-proline)。其中富集精氨酸的GR和PR细胞毒性尤为严重, 会引起ER应激和核仁应激等反应, 是ALS主要的致病因素。多聚PR和GR可聚集于核仁使其结构和功能改变, 如核仁大小改变、核仁相关蛋白移位以及核糖体合成障碍, 引起核仁应激和细胞死亡[33]。
敲低TMX2能显著抑制DPRs (主要是PRs和GRs) 对人类髓系白血病细胞K562和小鼠初级皮层神经元的细胞毒性[34]。RNA测序技术发现, 在敲除TMX2基因的细胞中表达PR50可以使一系列促进生存的UPR通路基因上调, 钙结合基因和凋亡基因下调[34]。这一初步结果说明, 下调TMX2对细胞的保护作用可能与DPRs引起的ER应激反应有关。
Kramer等[34]通过转基因技术从C9orf72 ALS患者的iPSCs (induced pluripotent stem cells) 细胞诱导出iMNs (induced motor neurons) 细胞, 再通过shRNA方法敲低iMNs细胞的TMX2, 结果发现, 降低TMX2蛋白表达不仅可以缓解DPRs引起的ER应激, 而且可以提高C9-ALS iMNs细胞的存活率[34]。这一结果再次证明TMX2对C9orf72-ALS疾病进程的调节作用。
4 小结与展望随着研究的不断深入, 人们对PDI结构、功能以及在ALS疾病中的作用有了一定认识。PDI作为ER上重要的蛋白二硫键异构酶和分子伴侣, 不仅对ALS相关蛋白的正确折叠具有重要作用, 而且对运动神经元突起生长以及神经肌肉突触联系都有重要影响。ALS相关PDI突变蛋白和PDI蛋白的亚硝基化也会影响PDI蛋白活性从而成为ALS疾病的危险因素。另外, PDI蛋白家族的其他成员如ERp57和TMX2也对ALS疾病有重要影响。这些数据说明PDI可能成为ALS疾病治疗的潜在靶点, PDI蛋白调节剂可能为ALS疾病治疗的药物研发提供新方向。
作者贡献: 张永雪负责文章撰写和修改; 蒲泽瑶、安亚婷和张薇负责作图和修改; 王艺颖和郝瀚负责格式编辑和修改; 杜肖娜、张海林和张会然提供思路和指导。
利益冲突: 所有作者均声明不存在利益冲突。
| [1] |
Wang C, Li W, Ren J, et al. Structural insights into the redox-regulated dynamic conformations of human protein disulfide isomerase[J]. Antioxid Redox Signal, 2013, 19: 36-45. DOI:10.1089/ars.2012.4630 |
| [2] |
Okumura M, Noi K, Kanemura S, et al. Dynamic assembly of protein disulfide isomerase in catalysis of oxidative folding[J]. Nat Chem Biol, 2019, 15: 499-509. DOI:10.1038/s41589-019-0268-8 |
| [3] |
Neves RPP, Fernandes PA, Ramos MJ. Mechanistic insights on the reduction of glutathione disulfide by protein disulfide isomerase[J]. Proc Natl Acad Sci U S A, 2017, 114: E4724-E4733. DOI:10.1073/pnas.1618985114 |
| [4] |
Biterova EI, Isupov MN, Keegan RM, et al. The crystal structure of human microsomal triglyceride transfer protein[J]. Proc Natl Acad Sci U S A, 2019, 116: 17251-17260. DOI:10.1073/pnas.1903029116 |
| [5] |
Wang CC, Tsou CL. Protein disulfide isomerase is both an enzyme and a chaperone[J]. FASEB J, 1993, 7: 1515-1517. DOI:10.1096/fasebj.7.15.7903263 |
| [6] |
Hillson DA, Lambert N, Freedman RB. Formation and isomerization of disulfide bonds in proteins: protein disulfide-isomerase[J]. Methods Enzymol, 1984, 107: 281-294. |
| [7] |
Tsai B, Rodighiero C, Lencer WI, et al. Protein disulfide isomerase acts as a redox-dependent chaperone to unfold cholera toxin[J]. Cell, 2001, 104: 937-948. DOI:10.1016/S0092-8674(01)00289-6 |
| [8] |
Chen K, Lin Y, Detwiler TC. Protein disulfide isomerase activity is released by activated platelets[J]. Blood, 1992, 79: 2226-2228. DOI:10.1182/blood.V79.9.2226.2226 |
| [9] |
Cook GM, Sousa C, Schaeffer J, et al. Regulation of nerve growth and patterning by cell surface protein disulphide isomerase[J]. Elife, 2020, 9: e54612. DOI:10.7554/eLife.54612 |
| [10] |
Yang YS, Harel NY, Strittmatter SM. Reticulon-4A (Nogo-A) redistributes protein disulfide isomerase to protect mice from SOD1-dependent amyotrophic lateral sclerosis[J]. J Neurosci, 2009, 29: 13850-13859. DOI:10.1523/JNEUROSCI.2312-09.2009 |
| [11] |
Bernardoni P, Fazi B, Costanzi A, et al. Reticulon1-C modulates protein disulphide isomerase function[J]. Cell Death Dis, 2013, 4: e581. DOI:10.1038/cddis.2013.113 |
| [12] |
Kaplan A, Gaschler MM, Dunn DE, et al. Small molecule-induced oxidation of protein disulfide isomerase is neuroprotective[J]. Proc Natl Acad Sci U S A, 2015, 112: E2245-E2252. DOI:10.1073/pnas.1500439112 |
| [13] |
Hoffstrom BG, Kaplan A, Letso R, et al. Inhibitors of protein disulfide isomerase suppress apoptosis induced by misfolded proteins[J]. Nat Chem Biol, 2010, 6: 900-906. DOI:10.1038/nchembio.467 |
| [14] |
Lukesh JC, Andersen KA, Wallin KK, et al. Organocatalysts of oxidative protein folding inspired by protein disulfide isomerase[J]. Org Biomol Chem, 2014, 12: 8598-8602. DOI:10.1039/C4OB01738B |
| [15] |
Halloran M, Parakh S, Atkin JD. The role of s-nitrosylation and s-glutathionylation of protein disulphide isomerase in protein misfolding and neurodegeneration[J]. Int J Cell Biol, 2013, 2013: 797914. |
| [16] |
Walker AK, Farg MA, Bye CR, et al. Protein disulphide isomerase protects against protein aggregation and is S-nitrosylated in amyotrophic lateral sclerosis[J]. Brain, 2010, 133: 105-116. DOI:10.1093/brain/awp267 |
| [17] |
Jaiswal MK. Riluzole and edaravone: a tale of two amyotrophic lateral sclerosis drugs[J]. Med Res Rev, 2019, 39: 733-748. DOI:10.1002/med.21528 |
| [18] |
Saitoh Y, Takahashi Y. Riluzole for the treatment of amyotrophic lateral sclerosis[J]. Neurodegener Dis Manag, 2020, 10: 343-355. DOI:10.2217/nmt-2020-0033 |
| [19] |
Yoshino H. Edaravone for the treatment of amyotrophic lateral sclerosis[J]. Expert Rev Neurother, 2019, 19: 185-193. DOI:10.1080/14737175.2019.1581610 |
| [20] |
Kim G, Gautier O, Tassoni-Tsuchida E, et al. ALS genetics: gains, losses, and implications for future therapies[J]. Neuron, 2020, 108: 822-842. DOI:10.1016/j.neuron.2020.08.022 |
| [21] |
Kwok CT, Morris AG, Frampton J, et al. Association studies indicate that protein disulfide isomerase is a risk factor in amyotrophic lateral sclerosis[J]. Free Radic Biol Med, 2013, 58: 81-86. DOI:10.1016/j.freeradbiomed.2013.01.001 |
| [22] |
Yang Q, Guo ZB. Polymorphisms in protein disulfide isomerase are associated with sporadic amyotrophic lateral sclerosis in the Chinese Han population[J]. Int J Neurosci, 2016, 126: 607-611. |
| [23] |
Gonzalez-Perez P, Woehlbier U, Chian RJ, et al. Identification of rare protein disulfide isomerase gene variants in amyotrophic lateral sclerosis patients[J]. Gene, 2015, 566: 158-165. DOI:10.1016/j.gene.2015.04.035 |
| [24] |
Woehlbier U, Colombo A, Saaranen MJ, et al. ALS-linked protein disulfide isomerase variants cause motor dysfunction[J]. EMBO J, 2016, 35: 845-865. DOI:10.15252/embj.201592224 |
| [25] |
Parakh S, Shadfar S, Perri ER, et al. The redox activity of protein disulfide isomerase inhibits ALS phenotypes in cellular and zebrafish models[J]. iScience, 2020, 23: 101097. DOI:10.1016/j.isci.2020.101097 |
| [26] |
Perri ER, Thomas CJ, Parakh S, et al. The unfolded protein response and the role of protein disulfide isomerase in neurodegeneration[J]. Front Cell Dev Biol, 2015, 3: 80. |
| [27] |
Jaronen M, Vehvilainen P, Malm T, et al. Protein disulfide isomerase in ALS mouse glia links protein misfolding with NADPH oxidase-catalyzed superoxide production[J]. Hum Mol Genet, 2013, 22: 646-655. DOI:10.1093/hmg/dds472 |
| [28] |
Renthal W, Tochitsky I, Yang L, et al. Transcriptional reprogramming of distinct peripheral sensory neuron subtypes after axonal injury[J]. Neuron, 2020, 108: 128-144. DOI:10.1016/j.neuron.2020.07.026 |
| [29] |
Birsa N, Bentham MP, Fratta P. Cytoplasmic functions of TDP-43 and FUS and their role in ALS[J]. Semin Cell Dev Biol, 2020, 99: 193-201. DOI:10.1016/j.semcdb.2019.05.023 |
| [30] |
Farg MA, Soo KY, Walker AK, et al. Mutant FUS induces endoplasmic reticulum stress in amyotrophic lateral sclerosis and interacts with protein disulfide-isomerase[J]. Neurobiol Aging, 2012, 33: 2855-2868. DOI:10.1016/j.neurobiolaging.2012.02.009 |
| [31] |
Parakh S, Jagaraj CJ, Vidal M, et al. ERp57 is protective against mutant SOD1-induced cellular pathology in amyotrophic lateral sclerosis[J]. Hum Mol Genet, 2018, 27: 1311-1331. DOI:10.1093/hmg/ddy041 |
| [32] |
Rozas P, Pinto C, Martinez Traub F, et al. Protein disulfide isomerase ERp57 protects early muscle denervation in experimental ALS[J]. Acta Neuropathol Commun, 2021, 9: 21. DOI:10.1186/s40478-020-01116-z |
| [33] |
Aladesuyi Arogundade O, Nguyen S, Leung R, et al. Nucleolar stress in C9orf72 and sporadic ALS spinal motor neurons precedes TDP-43 mislocalization[J]. Acta Neuropathol Commun, 2021, 9: 26. DOI:10.1186/s40478-021-01125-6 |
| [34] |
Kramer NJ, Haney MS, Morgens DW, et al. CRISPR-Cas9 screens in human cells and primary neurons identify modifiers of C9ORF72 dipeptide-repeat-protein toxicity[J]. Nat Genet, 2018, 50: 603-612. DOI:10.1038/s41588-018-0070-7 |
 2021, Vol. 56
2021, Vol. 56


