2. 西藏藏医药大学, 西藏 拉萨 850000;
3. 北京中医药大学, 北京 100029
2. Tibetan Traditional Medical College, Lhasa 850000, China;
3. Beijing University of Chinese Medicine, Beijing 100029, China
在长期的生活与医疗活动中, 藏族人民不断认识身边高山植物、动物及矿物的药用价值, 积累了丰富的实践经验, 逐步形成了独具特色的藏医药学。藏医药学是祖国民族医药学的重要组成部分, 具有丰富的科学与藏族文化内涵。深入开展藏药品种整理, 可为藏药材客观、准确的鉴定, 活性物质研究及质量标准的制定与完善等工作打下坚实基础, 具有重要的科学意义。但由于种种原因, 藏药品种整理等工作尚不能适应藏医药学发展的需要, 当务之急是开展相关深入的基础性研究工作。
藏药解吉(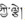


长梗秦艽G. waltonii Burk., 我国高山特有种, 仅在西藏自治区有分布, 生于海拔3 000~4 800 m的高山草坡、山坡砾石及林下[5]。全萼秦艽G. lhassica Burk., 我国高山特有种, 在西藏自治区及青海省有分布, 生于海拔4 200~5 050 m的高山草甸[5]。秦艽组植物的花色常分为两类, 一类常为蓝至蓝紫色, 一类则为灰白色或浅黄至黄绿色, 这也是将解吉分为解吉那保与解吉嘎保两个品种的主要依据。长梗秦艽与全萼秦艽的花同为蓝紫色, 花均具花梗, 排列成疏松的花序。尽管植物地上部分形态特征有一定区别, 但对于根部药材鉴定而言, 传统方法有其局限性。同时, 作为高山药用植物, 生存环境严酷, 加之人为采挖, 野生资源状况不容乐观。课题组前期开展了长梗秦艽及全萼秦艽的初步生药学研究[6]; 但有关上述两种高山特有物种的遗传背景研究及深入的种质资源评价等工作, 目前尚未见系统报道。
绿色植物的叶绿体拥有自身完整的一套基因组, 随着烟草Nicotiana tabacum[7]和地钱Marchantia polymorpha[8]等叶绿体全基因组序列的发表, 植物叶绿体基因结构逐渐被人们所熟知。大多数被子植物的叶绿体基因结构为双链环状[9, 10], 包括大单拷贝区(LSC)、小单拷贝区(SSC) 以及两个反向重复区(IR)。其大小、结构和基因种类一般较为保守[11, 12], 编码区和非编码区进化速率与模式具有物种鉴别意义[13], 在研究植物系统进化、物种遗传背景分析、物种DNA分子标记筛选等方面具有其独特优势[14, 15]。
为此, 在民族植物学考察、标本采集与分类学鉴定等工作基础[4, 16, 17]上, 进行长梗秦艽及全萼秦艽叶绿体全基因组序列测定及遗传背景分析, 筛选物种DNA分子标记、构建特异性PCR鉴定方法, 以期为长梗秦艽及全萼秦艽物种DNA分子鉴定、藏药“解吉”品质评价、高山濒危物种种质资源保护及龙胆科龙胆属系统发育分析等工作提供基础科学资料。
材料与方法样品采集及分类学鉴定 叶绿体基因组测序样品, 长梗秦艽G. waltonii采集日期2018-08-08, 西藏拉萨, 高山草坡, 海拔4 364 m, 样点地理坐标N 29°46.132', E 91°10.148', 凭证标本: 董博然等XZ2018CG001 (4)。全萼秦艽G. lhassica采集日期2018-08-10, 西藏米拉山, 高寒草甸, 海拔5 018 m, 样点地理坐标N 29°49.596', E 92°20.829', 凭证标本: 赵志礼, 倪梁红Z2018XZ003 (5)。同时, 采集龙胆属秦艽组其他8个近缘种标本, 每个居群至少取3株样品(2016XZ001除外), 总计10种22个居群91份。
去英国皇家植物园(Kew) 植物标本馆查阅长梗秦艽G. waltonii、全萼秦艽G. lhassica及其近缘物种的模式标本, 以确保物种分类学鉴定的准确性。凭证标本存放于上海中医药大学中药学院药用植物标本室(表 1, 图 1)
| Table 1 Samples and voucher specimens |

|
Figure 1 Gentiana lhassica and Gentiana waltonii. A: Habit of G. lhassica; B: Type of G. lhassica in Kew! C: Habit of G. waltonii; D: Lectotype of G. waltonii in Kew! |
总DNA提取 硅胶快速干燥叶片, 液氮中研磨, 改良CTAB法[18]提取总DNA。
叶绿体全基因组序列的获得及物理图谱绘制 参考被子植物叶绿体全基因组测序通用引物[19], Sanger法测序[20]。其中通用引物无法扩出的片段以及gaps, 参考相关序列[16, 21, 22]基于Primer Select (Lasergene 7.0) 软件, 重新设计引物, 其中长梗秦艽21对, 全萼秦艽7对。扩增出叶绿体基因组各片段序列, 应用SeqMan软件进行正反向拼接。针对叶绿体基因组结构的特征, 设计IR区与LSC区、SSC区的4个接口位置的特异性引物, 经PCR扩增、测序后再次进行序列准确性验证。所用引物序列及验证区域见表 2。
| Table 2 Primers for validation of the boundary regions |
叶绿体基因组在线注释软件DOGMA[23]对样品叶绿体全基因组进行初步注释; 参考近缘种序列[16, 21, 22], 结合编码蛋白基因起始密码子、终止密码子, 进行BLAST[24]手工校正; tRNA基因利用在线软件tRNAscan-SE 2.0[25]进行验证。将注释完成的基因组序列上传至NCBI, 应用在线绘图软件OGDRAW[26], 分别绘制出长梗秦艽及全萼秦艽叶绿体基因组物理图谱。
基于龙胆属秦艽组叶绿体全基因组结构分析的物种DNA分子标记筛选 基于秦艽组已报道的叶绿体全基因组序列, 应用MEGA[27]软件分析长梗秦艽、全萼秦艽的基因结构差异, 进而序列对比筛选分子标记位点; MrBayes软件[28], 设置GTR+I+G模型, 以夹竹桃科Apocynaceae植物长春花Catharanthus roseus为外类群, 构建龙胆科21个分类群的贝叶斯系统进化树。DnaSP软件计算秦艽组10个物种叶绿体全基因组的核苷酸多样性(Pi), 以寻找潜在的分子标记位点, Pi值设置: > 0.02。
基于特异性引物的长梗秦艽与全萼秦艽DNA分子鉴定 对初筛的分子标记进行进一步分析, 筛选原则: ①差异片段尽可能长; ②尽可能避免重复序列。基于分析选定的2个分子标记, 设计物种特异性鉴定引物, 优化PCR扩增程序, 通过琼脂糖凝胶电泳对目的条带长度进行比较。先选取秦艽组10个物种14个居群, 每个居群单株样品共14株, 对DNA分子标记进行初步验证; 在此基础上, 扩大样本量至龙胆属秦艽组10种22个居群91份样品, 再次验证分子标记的专属性, 最终建立鉴别方法。
结果与分析 1 长梗秦艽、全萼秦艽叶绿体基因组序列结构IR区与LSC区、SSC区的4个接口位置验证实验结果表明, 拼接所得叶绿体基因组序列相应接口位置均准确无误, 最终获得长梗秦艽、全萼秦艽叶绿体全基因组序列。长梗秦艽序列总长148 705 bp (MK780032), 包括两个反向重复区域(IRa、IRb: 25 304 bp)、一个大单拷贝区(LSC: 81 068 bp) 和一个小单拷贝区(SSC: 17 029 bp); 全萼秦艽序列总长148 652 bp (MK790135), 包括两个反向重复区域(IRa、IRb: 25 302 bp)、一个大单拷贝区(LSC: 80 997 bp) 和一个小单拷贝区(SSC: 17 051 bp) (表 3)。
| Table 3 Chloroplast genome structure in G. waltonii and G. lhassica |
两个物种的基因种类及数量相同, 包括78个编码蛋白的基因(CDS: coding sequence)、30个tRNA基因、4个rRNA基因, 以及2个假基因(pseudogene: ψrps16, ψinfA) (表 4)。基因组物理图谱见图 2。
| Table 4 Chloroplast genes in G. waltonii and G. lhassica. *: Genes containing introns; (×2): Two gene copies in the IRs; ψ: Pseudogenes |
|
Figure 2 Chloroplast genome maps of G. waltonii and G. lhassica. Genes inside the circle are transcribed clockwise, whereas those outside the circle counterclockwise. Dashed area in the inner circle indicates the G+C content of the genome |
以长梗秦艽为参照, 利用MEGA软件将其余秦艽组9种植物的叶绿体基因组序列进行差异位点的人工比对, 差异类型分为4种: 插入、缺失、碱基转换、碱基颠换。结果显示, 全萼秦艽与长梗秦艽间序列差异性最小, 二者的差异主要是碱基替换; 而与黄管秦艽G. officinalis差异性最大(表 5)。
| Table 5 SNPs and InDels in plastome sequences within section Cruciata spp. SNP Tr: SNP transition; SNP Trv: SNP transversion |
DnaSP软件计算秦艽组10个物种叶绿体全基因组的Pi (Pi值设置: > 0.02)。多态性分布的热点区域常位于大单拷贝区和小单拷贝区, 而IR区相对保守。叶绿体基因中的IGS (trnH-psbA)、IGS (trnS-trnG)、IGS (ndhC-trnV)、ycf1、rpl2基因, 可以作为秦艽组组内潜在的分子标记。
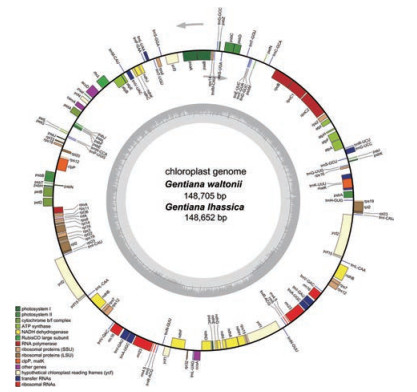
2.3 分子系统学分析基于龙胆科龙胆属Gentiana秦艽组(Sect. Cruciata) 10个物种、多枝组(Sect. Kudoa) 8个分类群及獐牙菜属Swertia 3个物种的叶绿体基因组序列, 以夹竹桃科植物长春花Catharanthus roseus为外类群, 构建贝叶斯系统进化树(图 3)。
|
Figure 3 Bayesian phylogenetic analysis based on 21 plastomes within Gentianaceae, with Catharanthus roseus as the outgroup. Numbers at nodes are posterior probabilities |
进化树中各类群间的拓扑结构与经典分类学观点基本相一致: 首先外类群夹竹桃科长春花被分出, 而另一大支为龙胆科各类群。进而龙胆科獐牙菜属Swertia和龙胆属Gentiana分成两支; 其中龙胆属一支除短柄龙胆G. stipitata的归属外, 其他各类群又被分为秦艽组(Sect. Cruciata) 与多枝组(Sect. Kudoa) 两小支。同时, 长梗秦艽和全萼秦艽显示了更为接近的亲缘关系。
3 基于叶绿体基因组的长梗秦艽、全萼秦艽物种分子标记及其鉴定秦艽组组内10个物种叶绿体基因组序列对比分析显示, 长梗秦艽与全萼秦艽两个物种序列的同源性、相似度极高, 秦艽组内难于直接将这两个物种进行区分。两步法鉴定策略: ①设计DNA特异性引物, 将长梗秦艽、全萼秦艽归为一类, 与其他8个近缘种有效区分。②再设计特异性引物实现两个近缘种之间的鉴定。
“第一步”预筛出5个片段(表 6); “第二步”预筛出3个片段(表 7)。从中再复筛并各确定1个片段: IGS (trnT-UGU, trnL-UAA) 间隔区一个特异性“75 bp deletion/insertion”以及ycf1基因中的一个特异性“21 bp deletion/insertion”。进而扩大样本量至秦艽组10种91个个体进行验证实验, 其中, G. waltonii 7居群31个个体, G. lhassica 7居群36个个体。由此构建如下鉴定方法:
| Table 6 Candidate loci for identifying G. waltonii and G. lhassica within section Cruciata spp |
| Table 7 Candidate loci for differentiating between G. waltonii and G. lhassica |
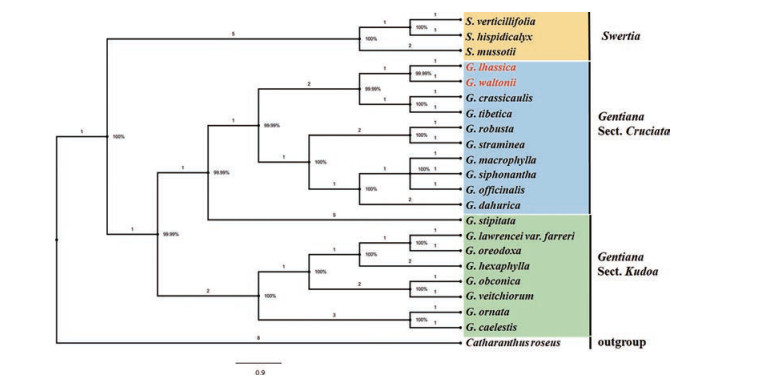
第一步: 引物(Primer 2-8) 5'-CGACCGTTCAAGT ATGCAAAAGGTT及5'-TAGTTTATTGTGAGGTCG CTCCG; PCR程序为94 ℃, 5 min; 42× (94 ℃, 30 s; 60 ℃, 40 s; 72 ℃, 1~3 min); 72 ℃, 7 min。凝胶电泳条件: 2.0%琼脂糖凝胶, 120 V, 45 min, 65 mA。长梗秦艽与全萼秦艽目的条带长度为173 bp, 而秦艽组其他8种植物为248 bp, 差距明显(75 bp), 图 4。
|
Figure 4 A group-specific PCR profile of Gentiana section Cruciata spp. Bands in the box only for G. waltonii and G. lhassica. M: DNA Ladder Mix; 0: Negative control; G. waltonii: 1-5 (A), 17-21 (B), 33-39 (C), 49-51 (E), 64 (F), 65-69 (K), 78-82 (I); G. lhassica: 6-10 (L), 22-26 (O), 27-29 (P), 40-45 (R), 52-60 (N), 70-74 (S), 83-85 (Q); 11-13 (J): G. crassicaulis; 14-16 (Z): G. robusta; 30-32 (M): G. straminea; 46-48 (G): G. siphonantha; 61-63 (H): G. officinalis; 75-77 (T): G. tibetica; 86-88 (X): G. dahurica; 89-91 (D): G. macrophylla; Bracket: Pop. ID |
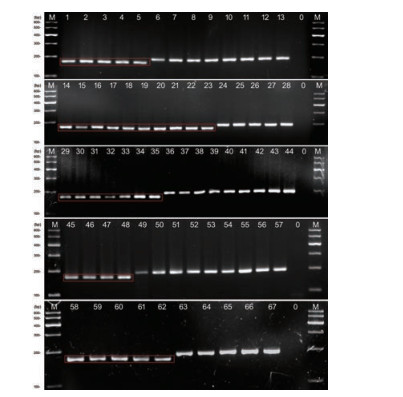
第二步: 引物(Primer 1-1) 5'-CGGATTGTTGGTA ATACCGTACC及5'-CCAAATAGGCGGTGGCAAAT TCGC; PCR程序为94 ℃, 5 min; 42× (94 ℃, 30 s; 59.9 ℃, 40 s; 72 ℃, 1~3 min); 72 ℃, 7 min。凝胶电泳条件: 2.7%琼脂糖凝胶, 145 V, 45 min, 106 mA。长梗秦艽目的条带长度为172 bp, 全萼秦艽为193 bp, 差距较为明显(21 bp), 图 5。
|
Figure 5 A species-specific PCR profile of G. waltonii and G. lhassica. Bands in the box only for G. waltonii. M: DNA Marker-B; 0: Negative control; G. waltonii: 1-5 (A), 14-18 (B), 19-23 (I), 29-35 (C), 45-47 (E), 48 (F), 58-62 (K); G. lhassica: 6-10 (L), 11-13 (P), 24-28 (O), 36-44 (N), 49-51 (Q), 52-57 (R), 63-67 (S); Bracket: Pop. ID |
最终视琼脂糖凝胶电泳目的条带的位置, 完成物种鉴别。
讨论植物叶绿体基因组序列含有丰富的植物系统学信息。本文基于龙胆科21个分类群的质体基因组相关资料构建的贝叶斯系统发育树, 拓扑关系与经典分类学观点基本一致。秦艽组内, 长梗秦艽G. waltonii则与全萼秦艽G. lhassica互为姐妹种, 后验概率99.99%, 亲缘关系更为接近; 两者的序列分析进一步显示出高度的同源性。因此, 在构建物种条形码时, 工作较为复杂、困难。本文工作, 筛选出位于IGS (trnT-trnL) 和ycf1基因中的两个分子标记, 大样本验证, 由此构建长梗秦艽、全萼秦艽的特异性引物PCR两步鉴定法, 可在秦艽组10个物种间有效鉴定姐妹种: 长梗秦艽与全萼秦艽, 方法稳定, 易于操作, 准确性高。
多态性分布的热点区域分析结果提示: 叶绿体基因中的IGS (trnH-psbA)、IGS (trnS-trnG)、IGS (ndhC-trnV) 及ycf1、rpl2基因, 可以作为秦艽组组内潜在的分子标记。
全萼秦艽基因组内在IGS (rps16-trnQ) 中出现了两个基因型。I型: 该片段序列完整, II型: 出现一47 bp的碱基缺失, 两者比例为15∶21。该基因分型的遗传学意义及是否与植株生长环境和产地等影响因子有关, 值得深入研究。
多枝组(Sect. Kudoa) 短柄龙胆G. stipitata在系统树中被聚入秦艽组(Sect. Cruciata) 一支中, 与经典分类学处理有所不同。龙胆属物种间自然杂交现象多有报道[29, 30], 短柄龙胆是否为一杂交起源物种或两近缘物种同域分布个体间的杂交类型?需要更多的证据支持。
综上所述, 叶绿体基因组序列与结构特性在龙胆科及龙胆属系统发育研究及构建物种DNA条形码方面同样具有重要的意义。龙胆属是龙胆科的第一大属, 国产近250种[5], 药用价值高。但目前已报道的质体基因组相关资料有限, 有必要广泛深入的开展相关序列测定及分析工作。
每个物种均有其独特的形成历史及复杂的生物学特性, 因此, 具体的鉴定对象, 应具体分析其遗传背景, 构建个性化的DNA条形码。面对丰富多样的植物类群及中药基原植物, 探究并构建准确、高效的DNA条形码鉴定体系还有很长的路要走。
作者贡献: 董博然是本文的第一作者, 负责分子生物学实验及稿件撰写; 倪梁红负责样品采集工作; 嘎务负责民族植物学考察工作; 刘铜华参与研究工作总体设计; 赵志礼为本文的通讯作者, 负责样品分类学鉴定, 研究工作总体设计及稿件修改等工作。
利益冲突: 本文的研究内容无任何利益冲突。
| [1] |
Yutok Yonten Gonpo. The Four Medical Tantras (四部医典)[M]. Shanghai: Shanghai Science & Technology Press, 1987: 42.
|
| [2] |
Dimaer Danzeng Pengcuo. Jing Zhu Ben Cao (晶珠本草)[M]. Shanghai: Shanghai Science & Technology Press, 1986: 119.
|
| [3] |
Northwest Institute of Plateau Biology, Chinese Academy of Sciences. Tibetan Medicine (藏药志)[M]. Xining: Qinghai People Press, 1991: 9-12.
|
| [4] |
Zhao ZL, Dorje Gaawe, Wang ZT. Identification of medicinal plants used as Tibetan Traditional Medicine Jie-Ji[J]. J Ethnopharmacol, 2010, 132: 122-126. DOI:10.1016/j.jep.2010.07.051 |
| [5] |
Ho TN. Gentianaceae in Flora Reipublicae Popularis Sinicae: Vol 62(中国植物志: 62卷)[M]. Beijing: Science Press, 1988: 14-64.
|
| [6] |
Gu CY, Zhao ZL, Wu JR, et al. Pharmacognostical study on Gentiana lhassica Burk. and Gentiana waltonii Burk.[J]. Chin J Inf Tradit Chin Med (中国中医药信息杂志), 2010, 17: 41-43. |
| [7] |
Shinozaki K, Ohme M, Tanaka M, et al. The complete nucleotide sequence of the tobacco chloroplast genome: its gene organization and expression[J]. EMBO J, 1986, 5: 2043-2049. DOI:10.1002/j.1460-2075.1986.tb04464.x |
| [8] |
Ohyama K, Fukuzawa H, Kohchi T, et al. Chloroplast gene organization deduced from complete sequence of Liverwort Marchantia polymorpha chloroplast DNA[J]. Nature, 1986, 322: 572-574. DOI:10.1038/322572a0 |
| [9] |
Palmer JD, Stein DB. Conservation of chloroplast genome structure among vascular plants[J]. Curr Genet, 1986, 10: 823-833. DOI:10.1007/BF00418529 |
| [10] |
Palmer JD. Plastid chromosomes: structure and evolution. In: Vasil IK, Bogorad L (eds) Cell Culture and Somatic Cell Genetics in Plants, the Molecular Biology of Plastids, 7A[M]. San Diego, Academic Press, 1991: 5-53.
|
| [11] |
Clegg MT, Gaut BS, Learn GH, et al. Rates and patterns of chloroplast DNA evolution[J]. Proc Natl Acad Sci U S A, 1994, 91: 6795-6801. DOI:10.1073/pnas.91.15.6795 |
| [12] |
Daniell H, Lin CS, Yu M, et al. Chloroplast genomes: diversity, evolution, and applications in genetic engineering[J]. Genome Biol, 2016, 17: 134. DOI:10.1186/s13059-016-1004-2 |
| [13] |
Sabater B. Evolution and function of the chloroplast. current investigations and perspectives[J]. Int J Mol Sci, 2018, 19: 3095. DOI:10.3390/ijms19103095 |
| [14] |
Zhang YJ, Li DZ. Advances in phylogenomics based on complete chloroplast genomes[J]. Plant Divers Resour (植物分类与资源学报), 2011, 33: 365-375. |
| [15] |
Ni LH, Zhao ZL, Mi M. Advances in chloroplast genome of medicinal plants[J]. J Chin Med Mater (中药材), 2015, 38: 1990-1994. |
| [16] |
Ni LH, Zhao ZL. A morphometric comparison of three closely related species of Gentiana (Gentianaceae), endemic to the region of the Qinghai-Tibet Plateau[J]. Botany, 2018, 96: 209-215. DOI:10.1139/cjb-2017-0166 |
| [17] |
Zong FF, Zhao ZL, Ni LH, et al. DNA fingerprinting and genetic diversity in a threatened alpine medicinal plant, Gentiana crassicaulis (Gentianaceae) using AFLP[J]. Acta Pharm Sin (药学学报), 2020, 55: 1941-1950. |
| [18] |
Doyle JJ, Doyle JL. A rapid DNA isolation procedure for small quantities of fresh leaf material[J]. Phyochem Bull, 1987, 19: 11-15. |
| [19] |
Qian J, Song J, Gao H, et al. The complete chloroplast genome sequence of the medicinal plant Salvia miltiorrhiza[J]. PLoS One, 2013, 8: e57607. DOI:10.1371/journal.pone.0057607 |
| [20] |
Sanger F, Nicklen S, Coulson AR. DNA sequencing with chain-terminating inhibitors[J]. Proc Natl Acad Sci U S A, 1977, 74: 5463-5467. DOI:10.1073/pnas.74.12.5463 |
| [21] |
Ni LH, Zhao ZL, Xu HX, et al. The complete chloroplast genome of Gentiana straminea (Gentianaceae), an endemic species to the Sino-Himalayan subregion[J]. Gene, 2015, 577: 281-288. |
| [22] |
Zhou T, Wang J, Jia Y, et al. Comparative chloroplast genome analyses of species in Gentiana section Cruciata (Gentianaceae) and the development of authentication markers[J]. Int J Mol Sci, 2018, 19: 1962. DOI:10.3390/ijms19071962 |
| [23] |
Wyman SK, Jansen RK, Boore JL. Automatic annotation of organellar genomes with DOGMA[J]. Bioinformatics, 2004, 20: 3252-3255. DOI:10.1093/bioinformatics/bth352 |
| [24] |
Altschul SF, Gish W, Miller W, et al. Basic local alignment search tool (BLAST)[J]. J Mol Biol, 1990, 215: 403-410. DOI:10.1016/S0022-2836(05)80360-2 |
| [25] |
Goremykin VV, Barbara H, Hirsch-Ernst KI, et al. Analysis of Acorus calamus chloroplast genome and its phylogenetic implications[J]. Mol Biol Evol, 2005, 22: 1813-1822. DOI:10.1093/molbev/msi173 |
| [26] |
Lohse M, Drechsel O, Bock R. Organellar Genome DRAW (OGDRAW): a tool for the easy generation of high-quality custom graphical maps of plastid and mitochondrial genomes[J]. Curr Genet, 2007, 52: 267-274. DOI:10.1007/s00294-007-0161-y |
| [27] |
Tamura K, Peterson D, Peterson N, et al. MEGA5:molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Mol Biol Evol, 2011, 28: 2731-2739. DOI:10.1093/molbev/msr121 |
| [28] |
Ronquist F, Huelsenbeck JP. MrBayes 3:Bayesian phylogenetic inference under mixed models[J]. Bioinformatics, 2003, 19: 1572-1574. DOI:10.1093/bioinformatics/btg180 |
| [29] |
Xiong, B, Zhao ZL, Ni LH, et al. DNA-based identification of Gentiana robusta and related species[J]. China J Chin Mater Med (中国中药杂志), 2015, 40: 4680-4685. |
| [30] |
Li XJ, Wang LY, Yang HL, et al. Confirmation of natural hybrids between Gentiana straminea and G. siphonantha (Gentianaceae) based on molecular evidence[J]. Acta Bot Yunnan (云南植物研究), 2007, 29: 91-97. |
 2021, Vol. 56
2021, Vol. 56


