2. 国家药品监督管理局化学药品质量研究与评价重点实验室, 北京 102629
2. NMPA Key Laboratory for Quality Research and Evaluation of Chemical Drugs, Beijing 102629, China
脱酰胺是大多数多肽或蛋白类药物在生产、储存及运输过程中发生的常见的降解形式, 这类药物氨基酸序列中通常含有天冬酰胺(asparagine, Asn, N) 残基或谷氨酰胺(glutamine, Gln, Q) 残基, 发生脱酰胺反应时其侧链酰胺基水解转变为羧基[1]。由于脱酰胺化改变了多肽或蛋白质原本的结构, 所以其活性、功能、稳定性、溶解性等性质可能发生相应的改变[2-4], 甚至在免疫原性、神经生物学等方面也会产生重要影响[5, 6], 对该类药品的安全性与有效性带来巨大的挑战。因此, 需要关注蛋白及多肽类药物中的脱酰胺杂质及其水平, 尤其是Asn的脱酰胺问题, Gln发生脱酰胺化的情况较少且转化速率远低于Asn[7-9]。
科博肽(cobratide) 是从中华眼镜蛇蛇毒中分离纯化的神经毒蛋白, 是一种用于晚期癌症疼痛、慢性关节痛、坐骨神经痛等慢性疼痛的多肽药物, 因其镇痛时间长、不良反应小, 尤其是成瘾性低等优点而被广泛应用于临床, 它的一级结构由62个氨基酸四组二硫键组成[10]。科博肽的氨基酸序列含有多个Asn残基, 并且第48位点的Asn残基右侧为甘氨酸(glycine, Gly, 图 1), 极易发生脱酰胺化, 所以科博肽脱酰胺化的检测尤为重要, 但目前尚无科博肽的脱酰胺检测方法的研究。

|
Figure 1 Amino acid sequence of cobratide |
随着生物质谱技术的发展, 应用质谱分析技术对药物杂质进行定性和定量的分析越来越普遍[11]。目前蛋白质脱酰胺的分析研究主要采用自上而下(top-down) 和自下而上(bottom-up) 两种策略。top-down是利用色谱联用质谱或质谱等分析技术, 直接对完整蛋白水平分析脱酰胺杂质, 包括翻译后修饰蛋白质和其他大片段蛋白质; bottom-up则是将大蛋白片段水解/酶解为肽段, 然后进行多肽水平的分析。top-down策略不需要复杂的前处理过程, 即可获得真实的脱酰胺水平, 但与bottom-up策略相比存在多个方面的缺陷。第一, 它对质谱分辨率的要求更高(因为蛋白与其脱酰胺产物最低仅相差0.98 Da, 在质谱中与同位素难以区分); 第二, 酶解完整蛋白与脱酰胺产物不易分离, 采用色谱联用质谱方式分析, 需要较为繁重的分析与分离方法的优化过程; 第三, 完整蛋白的质谱响应较弱, 导致top-down策略检测灵敏度较低; 第四, 完整蛋白质的断裂效率随着蛋白质分子质量的增加而降低, 导致大蛋白质脱酰胺位点的定位困难。但bottom-up策略也有其局限性, 其中最主要的问题就是前处理过程造成脱酰胺定量结果偏高的问题[12]。
目前, 传统的Asn脱酰胺化的检测步骤一般为: 先用内切蛋白酶将完整蛋白或多肽片段酶解成多个短肽, 再将肽段进一步处理后, 用不同分析仪器进行脱酰胺位点的定位和定量测定[13]。但是, 酶解反应中较为常用的蛋白酶(如胰蛋白酶、Lys-C、糜蛋白酶等) 均采用缓冲液碱性条件, 而碱性条件就会促进脱酰胺化的发生[14, 15], 尤其是当Asn的羧基侧有一个简单氨基酸如甘氨酸(NG) 时(如科博肽), 该位点在碱性条件下发生脱酰胺的量会高于其他位点[9, 16], 造成测定结果的准确度较低。
与上述内切酶相比, 蛋白内切酶Glu-C具有明显优势。该酶可适用的pH范围较宽, 酸性与碱性条件下均可催化水解反应。科博肽氨基酸序列含有多个谷氨酸残基, 可以利用Glu-C酶在较低pH的醋酸铵缓冲液中将肽链切割成多个短肽, 然后基于高分辨质谱技术定位并定量检测样品中真实水平的脱酰胺化比例。本文通过系统优化基于Glu-C酶的bottom-up分析方法, 建立了定性与定量分析科博肽的脱酰胺杂质的方法, 为保证该类药物的安全有效和质量一致性奠定了基础。
材料与方法试剂与仪器 超纯水(Milli-Q超纯水机制备, 购自美国Millipore公司); 乙腈(LC/MS级, 203500)、甲酸(202674)、胰蛋白酶(TG267381), 赛默飞世尔科技(中国) 有限公司); Tris (1022C435, 北京欣经科生物技术有限公司); 盐酸(20180427)、醋酸(20201021), 国药集团化学试剂有限公司; 碳酸氢铵(K51759005016)、醋酸铵(BCCB9847)、二硫苏糖醇(DL-dithiothreitol, DTT, BCCC6733)、碘乙酰胺(iodoacetamide, IAM, SLCD4031)、盐酸胍(SLCC3338)、Glu-C酶(40725220), 美国Sigma-Aldrich公司; 流动相A、B两相分别为0.1%甲酸-水溶液和0.1%甲酸-乙腈溶液。
色谱柱(美国Waters公司, XBridge® BEH C18, 2.5 μm, 2.1 mm×150 mm, 序列号01833011218521); Thermo Ultimate 3000-Q Extractive液相色谱联用高分辨质谱仪(数据采集软件: TraceFinder 4.1 General Quan, 数据处理软件: Thermo BioPharma Finder 4.0)、pH计、离心机, 赛默飞世尔科技(中国) 有限公司; 超滤管(截留相对分子质量3 000, 美国PALL公司); 恒温水浴槽, 优莱博技术(北京) 有限公司; 十万分之一电子天平, 瑞士梅特勒-托利多公司; 涡旋混合器, 美国Scientific Industries公司等。
还原烷基化处理 科博肽原料药来自国内某生产厂家不同批次的样品, 在-20 ℃下保存。取科博肽原料药固体粉末适量, 精密称定, 加超纯水溶解成终浓度为10 mg·mL-1的供试品溶液。取供试品溶液10 μL, 加1 mol·L-1 Tris-HCl缓冲液(pH 7.6) 10 μL, 再加8 mol·L-1盐酸胍溶液80 μL使蛋白变性, 最后加1 mol·L-1 DTT溶液1 μL还原二硫键, 充分混匀上述溶液后, 于56 ℃水浴中反应30 min。水浴结束后, 将溶液冷却至室温, 再加1 mol·L-1 IAM溶液2 μL封闭自由的巯基, 充分混匀后室温避光反应30 min。将还原烷基化后的样品转移至超滤管内, 12 000 r·min-1离心10 min。
脱酰胺位点确认 取还原烷基化样品加入0.1 mol·L-1 Tris-HCl缓冲液(pH 7.6) 0.1 mL, 12 000 r·min-1离心10 min, 重复该步骤1次, 离心结束后需确认超滤管内无液体存留, 否则需继续离心至全部液体滤除。加入缓冲液0.2 mL, 振摇使蛋白复溶于新的缓冲体系中, 再加入0.5 μg·μL-1胰蛋白酶10 μL (酶∶底物=1∶40), 混匀后, 37 ℃孵育, 样品重复制备两份, 于4和18 h时分别取样100 μL, 加甲酸2 μL终止酶切后进质谱检测。
Glu-C酶酶解缓冲液pH条件优化 分别配制pH值为4.0、4.5、5.0、5.5和6.0的醋酸铵缓冲液(浓度均为0.1 mol·L-1), 及2 mol·L-1碳酸氢铵缓冲液(待碳酸氢铵溶解后直接测其pH值为7.929)。
制备还原烷基化样品6支(均4倍体积制备), 分别加入上述缓冲液0.1 mL, 12 000 r·min-1离心10 min, 重复该步骤1次。再分别加入缓冲液0.4 mL, 振摇后加入1 μg·μL-1 Glu-C酶20 μL (酶∶底物=1∶20), 混匀后, 37 ℃孵育, 于2、4、8和18 h时分别取样100 μL, 加甲酸2 μL终止酶切后进质谱检测。
Glu-C酶酶切时间及酶量优化 根据Glu-C酶在不同缓冲液中的酶切效率及科博肽的脱酰胺率综合得出Glu-C酶酶切科博肽的最佳pH 5.5, 在此基础上继续对酶切时间及酶量进行优化。
制备还原烷基化的样品4支(均7倍体积制备), 分别加入pH 5.5的醋酸铵缓冲液(0.1 mol·L-1) 0.1 mL, 12 000 r·min-1离心10 min, 重复该步骤1次, 再加入缓冲液0.7 mL, 振摇后分别加入1、0.5、0.25和0.125 μg·μL-1的Glu-C酶35 μL (酶∶底物分别为1∶20、1∶40、1∶80和1∶160), 混匀后, 37 ℃孵育, 于1、2、4、6、8和18 h时分别取样100 μL, 加甲酸2 μL终止酶切后进质谱检测。
方法学验证 根据酶切效率及酶的成本两方面综合考虑, 得到最佳酶∶底物的比例为1∶40, 酶切时间6 h。确定好酶切条件后, 即进行方法学验证。
专属性 因样品为超纯水溶解, 故取超纯水10 μL, 加1 mol·L-1 Tris-HCl缓冲液(pH 7.6) 10 μL, 后续按还原烷基化步骤进行, 再在优化后的酶切条件下进行孵育, 加甲酸终止酶切后进质谱检测。
重复性 称取科博肽原料药固体粉末6份, 加超纯水溶解成终浓度10 mg·mL-1的供试品溶液, 分别取10 μL, 加1 mol·L-1 Tris-HCl缓冲液(pH 7.6) 10 μL, 后续按还原烷基化步骤进行, 再在优化后的酶切条件下进行孵育, 加甲酸终止酶切后进质谱检测。
精密度 制备还原烷基化样品1支(按2倍体积制备), 再在优化后的酶切条件下进行孵育, 加甲酸终止酶切后进质谱检测, 平行进样6次。
稳定性 将酶切结束并猝灭反应后的样品于室温放置14天后再进样检测。
色谱条件 流动相A、B两相分别为0.1%甲酸-水溶液和0.1%甲酸-乙腈溶液。流速为0.2 mL·min-1, 柱温45 ℃, 进样量20 μL, 进样器温度10 ℃, 洗脱梯度为: 0~3 min, 2% B; 3~45 min, 2%~32% B; 45~50 min, 32%~90% B; 50~51 min, 90%~100% B; 51~64 min, 100% B; 64~65 min, 100%~2% B; 65~70 min, 2% B。
质谱条件 采用四极杆-静电场轨道阱高分辨质谱仪, 配备电喷雾离子源(ESI) 进行测定。使用正离子扫描模式, ESI离子源参数如下: 鞘气流速30 L·min-1, 辅助气流速10 L·min-1, 喷雾电压3.8 kV, 毛细管温度320 ℃, 辅助气温度200 ℃。扫描类型为Full MS/dd-MS2, 扫描时间3~65 min, Full MS参数: 分辨率70 000, 最大离子注入时间20 ms, 扫描范围200~1 500 m/z; dd-MS2/dd-SIM参数: 分辨率17 500, 最大离子注入时间100 ms, 循环次数10, 隔离窗口: 1.8 m/z, NCE碰撞能量28; dd参数: 启用同位素峰排除, 动态排除8.0 s。
样品测定及结果对比 取同一厂家不同批次的科博肽原料药样品3批(A、B、C), 用优化好的酶切条件酶解样品并检测, 每批样品进样2次。在前期的工作中建立了毛细管电泳-质谱(CE-MS) 的方法分离并鉴定了科博肽及其脱酰胺产物, 该方法经过方法学验证, 可以用于科博肽脱酰胺水平的定量分析[17]。取上述三批样品, 采用毛细管电泳的方法进行分析, 再将二者结果进行对比。
数据处理 采用Thermo BioPharma Finder软件(版本号: 4.0) 对酶解后的肽段进行分析。在编辑氨基酸序列时选择可变修饰: Deamidation (N), 利用软件自动计算并汇总所有Asn残基上发生的脱酰胺情况。该软件计算翻译后修饰肽段相对丰度的方法为: 用鉴定到的翻译后修饰肽段的峰面积除以所有相关肽段总的峰面积。
结果 1 脱酰胺位点确认及胰蛋白酶酶切结果用胰蛋白酶在碱性条件下酶解科博肽以确定科博肽脱酰胺位点, 数据处理软件显示仅第48位的Asn发生了脱酰胺化, 其他位点中未发现脱酰胺化产物, 胰蛋白酶酶切18 h后科博肽的序列覆盖情况如图 2。

|
Figure 2 Cobratide sequence coverage map after 18 hours of digestion with trypsin |
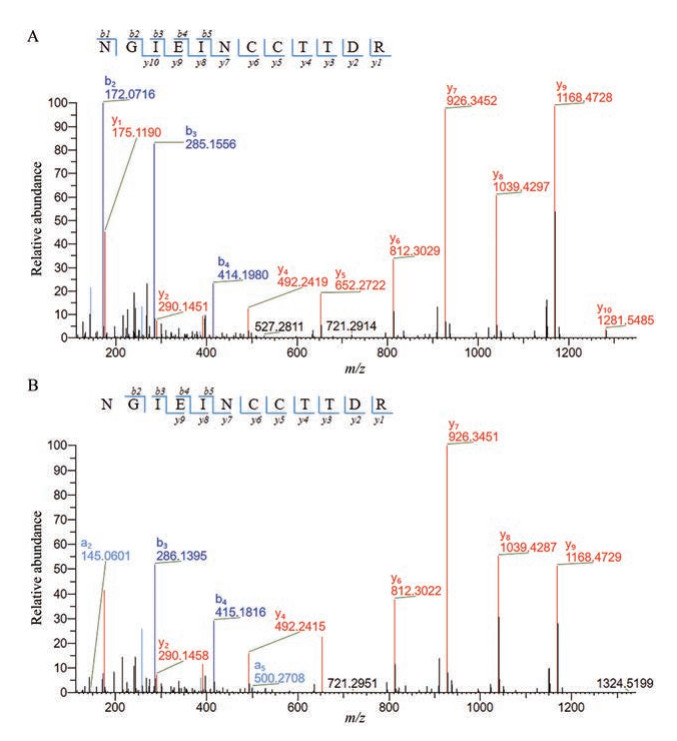
为进一步准确确定脱酰胺位点, 除依靠BioPharma Finder软件进行计算外, 还需对包含了Asn脱酰胺位点的肽段的二级质谱进行手动分析, 验证软件分析的结果。在N48-R59肽段(氨基酸序列: NGIEINCCTTDR) 中含有2个Asn残基, 肽段未发生脱酰胺化时m/z为726.814 1 (z = 2) (图 3A), 发生脱酰胺化后m/z为727.306 4 (z = 2) (图 3B), 质量数增加了0.984 6 Da, 说明N48-R59肽段有1个Asn残基发生了脱酰胺化。未发生脱酰胺化的肽段中, 检测到了包含N48位点的b3离子m/z为285.155 6 (z = 1), 包含N53位点的y7离子m/z为926.345 2 (z = 1) (图 4A); 发生脱酰胺化的肽段中, 检测到了包含N48位点的b3离子m/z为286.139 5 (z = 1), 包含N53位点的y7离子m/z为926.345 1 (z = 1) (图 4B)。包含了N48位点的b3离子质量数增加了0.983 9Da, 而包含N53位点的y7离子质荷比未发生变化, 由此可推测发生脱酰胺化的Asn残基在48位, 而53位的Asn残基未发生脱酰胺化, 与软件分析结果相互印证(表 1)。

|
Figure 3 Molecular mass analysis of N48-R59 peptide by LC-MS. A: m/z of the peptide (without deamidation). B: m/z of the peptide (deamidation) |
|
Figure 4 Identification of the deamidation sites by tandem mass spectra. A: MS/MS spectra of the N48-R59 peptide ion (without deamidation); B: MS/MS spectra of the N48-R59 peptide ion (deamidation) |
| Table 1 Observed molecular mass and expected molecular mass of molecular ion peak and fragment ion of the N48-R59 peptide |
用胰蛋白酶酶切样品4 h脱酰胺检测结果为: 30.35%、27.96%, 18 h脱酰胺检测结果为54.45%、59.46%, 检测结果不稳定, 重复性低。由图 2也可看出, 用胰蛋白酶酶切科博肽序列覆盖率不能达到100%, 中间还有肽段丢失, 也可能影响脱酰胺率的计算。
2 Glu-C酶酶切缓冲液pH条件优化为提升酶切效率, 减少人为脱酰胺对测定结果的影响, 首先将科博肽与Glu-C酶置于不同pH缓冲液体系下进行酶切, 然后比较不同时间各缓冲体系中科博肽的脱酰胺率和Glu-C酶的酶切效率, 脱酰胺率采用BioPharma Finder软件自动计算, 酶切效率以PeptideMapQuality值评价(表 2)。
| Table 2 Comparison of the deamidation rate and PeptideMapQuality values of cobratide digested by Glu-C enzyme in different pH buffer systems |
由表 2信息可知, 缓冲液pH为4.0时, Glu-C酶的活性较低, 肽段质量较差(酶切效率低于40%), 所以数据处理软件无法识别并检测科博肽的脱酰胺情况, 故脱酰胺率为0。在18 h内, 缓冲液pH为4.5和5.0的酶切效率低于其他缓冲液条件, pH为7.9的碳酸氢铵缓冲液酶切效率随时间增长很快, 但其脱酰胺率明显高于实际水平, 到18 h甚至接近100% (证明pH 7.9时, 科博肽酶切过程会产生较大量的脱酰胺)。缓冲液pH为5.5和6.0的酶切效率相近, 但当酶切时间超过4 h后, 缓冲液pH为6.0时测得的脱酰胺率逐渐增加, 而缓冲液pH为5.5时测得的脱酰胺率则基本稳定。综合考虑酶切效率和脱酰胺率的检测情况, 最终选择酶解缓冲液pH为5.5。
3 Glu-C酶酶切时间及酶量优化出于检验成本的考虑, 在准确度与灵敏度能够满足检测要求的前提下, 应尽可能减少酶的用量和酶促反应时间, 因此需要对酶用量与酶切时间进行优化。当底物浓度一定时, 酶量过多会使酶达到饱和, 不能再增加酶促反应速率; 酶切反应时间过长, 会引起脱酰胺化增加及其他副反应的发生(如异构化、氧化等)。根据上述研究结果, 在缓冲液pH 5.5时, 科博肽的脱酰胺率基本恒定, 所以可仅依据酶切效率(即PeptideMapQuality值) 确定最佳的酶切时间和酶量。
实验结果证明酶浓度越大, 酶切效率越高, 但酶切时间超过6 h后, 酶的活性逐渐降低, 会导致酶切效率增长速度显著降低。综合考虑酶的成本、副反应及酶切效率, 确定酶: 底物比例为1:40, 酶切时间为6 h。
以上述最优条件酶解得到的肽段覆盖率统计图如图 5所示, 可与科博肽有效匹配的肽段为64个, 序列覆盖率达100%, 错切、漏切肽段的比例较少, 整个肽段在相应的位点被切割成4条主要短肽, 每个短肽的响应(由一级质谱响应强度、CID碎裂产生的b离子和y离子与理论值的匹配程度等决定) 都很高, N48的脱酰胺率主要由软件自动依据R39-E51肽段和R39-N62肽段中脱酰胺比例计算。

|
Figure 5 Sequence coverage map of cobratide digested in optimal conditions |
配制空白溶液进行相同操作, 以验证方法的专属性, 结果证明, 空白溶液中并无影响脱酰胺率的干扰因素, 该方法专属性良好(图 6)。
|
Figure 6 Sequence coverage map of blank sample |
重复制备6份样品检测, 6份样品脱酰胺率的平均值为15.81%, RSD = 5.19%, 说明前处理方法重复性良好。
4-3 精密度制备1份样品重复进样6次, 6次样品脱酰胺率RSD = 2.55%, 说明仪器方法精密度良好。
4-4 稳定性样品酶解反应采用甲酸进行淬灭。将淬灭后溶液于室温放置14天后再次检测脱酰胺率由14.72%变为14.79%, 基本一致, 说明待测溶液淬灭后稳定性良好。
5 样品测定及结果对比采用本文实验方法, 对3批样品进行了测定。此外, 为验证方法的准确度, 本文与前期经过系统优化建立的基于毛细管电泳的科博肽中脱酰胺杂质的分析方法结果进行了对比(表 3)。两种方法中相同批次的样品脱酰胺率测定结果较为接近, 误差均在1%以内, 说明本文所建立的方法准确度良好。
| Table 3 Comparison of cobratide deamidation rate detected by LC-HRMS and CE |
通过本方法的研究, 建立了科博肽原料药中脱酰胺化的定位与定量测定方法。科博肽由62个氨基酸残基组成, 无法在完整蛋白水平下, 直接通过质谱对完整序列的检测以获得脱酰胺率。首先, 利用液相色谱要将科博肽及其脱酰胺产物完全分开较为困难; 其次, 由于科博肽48位Asn残基侧链的氨基(-NH2) 转变为羟基(-OH) 后与科博肽自身分子质量仅相差约0.98 Da, 与科博肽自身的同位素峰无法实现分离。在方法研究过程中, 本课题组尝试了在完整蛋白水平下, 直接提取科博肽和脱酰胺产物一级质谱离子流, 通过离子流图的峰面积来计算脱酰胺比例。但因质谱无法区分科博肽的同位素峰与脱酰胺产物的分子离子峰而失败。科博肽同位素峰质荷比为1 390.803 1 (z = 5), 脱酰胺产物响应最高的分子离子峰为1 390.803 5 (z = 5), QE高分辨质谱分辨率仅为140 000, 即使调整质量精度为10-6, 仍无法实现两者的分离。
在样品前处理过程中, 还原烷基化所用的缓冲液为Tris-HCl溶液(pH 7.6), 该pH条件下, 会导致科博肽在还原烷基化过程中的脱酰胺化。三(2-羧乙基) 膦(TCEP) 也是一种常用的还原剂[18, 19], 最适pH范围较宽, 可在酸性条件下还原二硫键, 因此本研究尝试利用TCEP还原科博肽中二硫键, 但因无法完全还原科博肽4组二硫键, 未能得到质量较好的图谱。本方法最终选择DTT作为还原剂, 优化并严格控制反应温度与时间, 在尽量不造成脱酰胺增加的前提下, 实现科博肽4组二硫键的完全还原。
科博肽的脱酰胺杂质对酶解缓冲液的pH非常敏感, 在pH 5.5的醋酸铵缓冲液中, 脱酰胺率在18 h内并未增加。但pH为6.0时, 脱酰胺水平随着酶切时间的延长逐渐增加, 在酶切18 h时由15%增加到40%, 所以实验过程中需严格控制缓冲液的pH值。鉴于醋酸铵缓冲液放置一段时间pH值会发生改变, 所以缓冲液应定期更换。
尽管在前期的工作中, 毛细管电泳的检测方法可以实现科博肽及其脱酰胺产物的分离, 但是需要经过复杂的分析方法优化过程, 无法适用于其他蛋白及多肽类产品中脱酰胺杂质的分析, 分析方法的开发效率相对较低; 但是, 由于液相-高分辨质谱法也存在检测仪器成本较高的问题, 需要更多的实验室投入。
本文以科博肽为研究对象, 通过对酶解缓冲液pH值、酶浓度、酶切时间进行优化, 建立了基于Glu-C酶的脱酰胺杂质测定方法, 方法学验证与不同方法的对比结果表明, 所建方法重复性好, 准确度高, 操作简单, 本研究为科博肽及其相似结构中氨基酸的脱酰胺杂质的检测与控制提供了重要参考。
作者贡献: 黄露负责实验设计、实验操作、数据整理及文章撰写; 刘博负责实验设计、数据处理及文章修改; 范慧红负责提出研究思路、指导论文写作并对论文进行了修改和检查。
利益冲突: 所有作者均声明不存在利益冲突。
| [1] |
di Salvo ML, Delle Fratte S, Maras B, et al. Deamidation of asparagine residues in a recombinant serine hydroxymethyltransferase[J]. Arch Biochem Biophys, 1999, 372: 271-279. DOI:10.1006/abbi.1999.1512 |
| [2] |
Aswad DW, Paranandi MV, Schurter BT. Isoaspartate in peptides and proteins: formation, significance, and analysis[J]. J Pharm Biomed Anal, 2000, 21: 1129-1136. DOI:10.1016/S0731-7085(99)00230-7 |
| [3] |
Lindner H, Helliger W. Age-dependent deamidation of asparagine residues in proteins[J]. Exp Gerontol, 2001, 36: 1551-1563. DOI:10.1016/S0531-5565(01)00140-1 |
| [4] |
Lampi KJ, Wilmarth PA, Murray MR, et al. Lens β-crystallins: the role of deamidation and related modifications in aging and cataract[J]. Prog Biophys Mol Biol, 2014, 115: 21-31. DOI:10.1016/j.pbiomolbio.2014.02.004 |
| [5] |
Mamula MJ, Gee RJ, Elliott JI, et al. Isoaspartyl post-translational modification triggers autoimmune responses to self-proteins[J]. J Biol Chem, 1999, 274: 22321-22327. DOI:10.1074/jbc.274.32.22321 |
| [6] |
Kim E, Lowenson JD, MacLaren DC, et al. Deficiency of a protein-repair enzyme results in the accumulation of altered proteins, retardation of growth, and fatal seizures in mice[J]. Proc Natl Acad Sci U S A, 1997, 94: 6132-6137. DOI:10.1073/pnas.94.12.6132 |
| [7] |
Joshi AB, Kirsch LE. The relative rates of glutamine and asparagine deamidation in glucagon fragment 22-29 under acidic conditions[J]. J Pharm Sci, 2002, 91: 2331-2345. |
| [8] |
Robinson NE, Robinson AB. Prediction of primary structure deamidation rates of asparaginyl and glutaminyl peptides through steric and catalytic effects[J]. J Pept Res, 2004, 63: 437-448. DOI:10.1111/j.1399-3011.2004.00148.x |
| [9] |
Bischoff R, Kolbe HV. Deamidation of asparagine and glutamine residues in proteins and peptides: structural determinants and analytical methodology[J]. J Chromatogr B Biomed Appl, 1994, 662: 261-278. DOI:10.1016/0378-4347(94)00203-7 |
| [10] |
Cheng SY, Le JJ, Li J, 等. Study on methods and establishment for reference standard of cobratide[J]. Chin J Pharm Anal (药物分析杂志), 2007, 27: 1212-1214. |
| [11] |
Olsen JV, Mann M. Status of large-scale analysis of post-translational modifications by mass spectrometry[J]. Mol Cell Proteomics, 2013, 12: 3444-3452. DOI:10.1074/mcp.O113.034181 |
| [12] |
Jin Y, Yi Y, Yeung B. Mass spectrometric analysis of protein deamidation-A focus on top-down and middle-down mass spectrometry[J]. Methods, 2020. DOI:10.1016/j.ymeth.2020.08.002 |
| [13] |
Li H, He YN, Ma JS, 等. Overview of determination methods for deamination reaction of biotech drugs[J]. Her Med (医药导报), 2019, 38: 147-152. |
| [14] |
Ren D, Pipes GD, Liu D, et al. An improved trypsin digestion method minimizes digestion-induced modifications on proteins[J]. Anal Biochem, 2009, 392: 12-21. DOI:10.1016/j.ab.2009.05.018 |
| [15] |
Krokhin OV, Antonovici M, Ens W, et al. Deamidation of -Asn-Gly- sequences during sample preparation for proteomics: consequences for MALDI and HPLC-MALDI analysis[J]. Anal Chem, 2006, 78: 6645-6650. DOI:10.1021/ac061017o |
| [16] |
Cournoyer JJ, Lin C, Bowman MJ, et al. Quantitating the relative abundance of isoaspartyl residues in deamidated proteins by electron capture dissociation[J]. J Am Soc Mass Spectrom, 2007, 18: 48-56. DOI:10.1016/j.jasms.2006.08.008 |
| [17] |
Liu B, Wang WT, Huang L, et al. Separation, identification and quantification of associated impurities in cobratide using sheathless CE-MS and CE-UV[J]. Anal Methods, 2021. DOI:10.1039/d1ay00717c |
| [18] |
Wang XY, Yan GQ, Zhou XW, 等. Location of peptide disulfide bonds by chemical cleavage combined with biological mass spectrometry[J]. Chem J Chin Univ (高等学校化学学报), 2020, 41: 1505-1512. |
| [19] |
Liu ZH, Liang SP. Location of the huwentoxin disulfide bonds by partial reduction and step-by-step sequencing[J]. Chem J Chin Univ (高等学校化学学报), 2003, 24: 1815-1819. |
 2021, Vol. 56
2021, Vol. 56


