2. 华中科技大学同济医学院药学院, 湖北 武汉 430030
2. School of Pharmacy, Tongji Medical College, Huazhong University of Science and Technology, Wuhan 430030, China
随着分子生物学技术的不断发展, 基因突变和缺陷与疾病之间的关联性获得了越来越深入的认识。核酸因为在疾病诊断和治疗过程中显示出巨大的应用潜力而备受关注。核酸药物是指人工合成的具有疾病治疗功能的DNA或RNA片段。此类药物能够直接作用于致病靶基因或致病靶mRNA, 在基因水平上发挥治疗疾病作用。与传统小分子药物和抗体药物相比, 核酸药物可从根源上调控致病基因的表达, 具有“治标治本”的特点。核酸药物还具有高效性、低毒性、高特异性等明显优势[1]。自1998年以来, 已有多个核酸药物相继被批准用于临床治疗。目前全球上市的核酸药物主要有反义核酸(anti-sense oligonucleotides, ASO)、小干扰RNA (small interfering RNA, siRNA)、核酸适配体(nucleic acid adapters)[2]。ASO具有与靶RNA互补的碱基序列, 可以与其特异性结合。ASO一般包括反义DNA (anti-sense DNA) 和反义RNA (anti-sense RNA)。已被批准上市的ASO药物, 如福米韦生(fomivirsen, 商品名为Vitravene)、米泊美生(mipomersen, 商品名为Kynamro) 和依特立生(eteplirsen, 商品名为Exondys 51) 等[1]。siRNA是对靶RNA进行切割所产生具有特定长度和序列的小片段RNA。这些siRNA可以特异性诱导靶mRNA降解。已获批上市的siRNA药物, 如帕替斯坦(patisiran, 商品名为Onpattro) 等[1]。核酸适配体是可与有机小分子、DNA、RNA、多肽或者蛋白质等多种靶分子进行高亲和力、高特异性结合的寡聚核苷酸[3]。派加他尼(pegaptanib, 商品名为Macugen) 是首例获批上市的核酸适配体药物[1]。除核酸适配体外(可能超过30个核苷酸), 核酸药物通常为12~30个核苷酸组成的寡聚核苷酸, 也称为寡核苷酸药物[4, 5]。此外, 微小RNA (microRNA, miRNA)、核酶(ribozyme) 与脱氧核酶(deoxyribozyme) 等在多种疾病的治疗中也显示出巨大的开发价值[2]。核酸药物已成为当今生物医药研发中最具发展前景的领域之一。Wang等[6]分析了目前全球431个核酸药物在研项目, 其中63%处于非临床研究阶段, 32%处于早期临床试验(I期或II期), 3%处于III期临床, 5种药物处于新药申请阶段。
生物分析利用色谱法、色谱-质谱联用法、配体结合法等技术对生物样品中的药物及其代谢产物或内源性物质进行定量分析。已被广泛用于药代动力学(pharmacokinetics, PK)、毒代动力学(toxicokinetics, TK)、生物等效性试验、临床治疗药物监测等领域。随着科学仪器和分子生物学技术的发展与进步, 许多分析技术被用于生物基质中核酸药物的检测。本文综述近年来核酸药物生物分析领域的研究进展, 介绍当前各项分析技术的应用和发展趋势以及在法规指导下的规范性核酸药物生物分析研究所面临的挑战及解决方案。
1 基于核酸分子杂交技术分析方法核酸分子杂交(hybridization) 是利用核酸分子的变性和复性的性质, 使不同来源的DNA (或RNA) 片段, 按碱基互补关系形成杂交双链分子(如DNA与DNA、RNA与DNA等) 后, 利用相应的技术进行检测。多种基于分子杂交技术的方法已应用于核酸药物的生物分析, 例如: 核酸分子杂交-酶联免疫吸附法(hybridization-enzyme linked immunosorbent assay, hybridization-ELISA) 和实时荧光定量PCR (real-time quantitative PCR, qPCR)。
1.1 核酸分子杂交-酶联免疫吸附测定法在hybridization-ELISA中, 杂交双链由待测核酸和探针组成。探针是带有标记酶(如碱性磷酸酯酶, alkaline phosphatase, AP) 的特异性核酸序列片段。利用该探针可以进行特异性的靶序列检测。待测核酸的检测信号来自酶联免疫吸附法中的标记酶与底物发生的反应。因此, 基于ELISA的核酸分子杂交分析技术也被称为hybridization-ELISA。与其他核酸生物分析方法相比, 该法不但具有较高的灵敏度, 而且几乎不需要样品处理。对于组织样品中的核酸测定, 可先采用液-液萃取法(如苯酚和氯仿) 除去组织样品中的蛋白和其他干扰物质。如果组织样品量较小(如200 mg·mL-1的组织匀浆液50 μL), 可加入蛋白酶k (proteinase k) 和非离子表面活性剂破坏细胞膜脂质双分子层后直接进行hybridization-ELISA测定。此外, 该方法无需纯化血浆样品中的待测核酸, 可直接测定其含量[4]。
核酸分子杂交法具有高灵敏度、高特异性和高通量等特点, 已广泛用于支持药代动力学和毒代动力学研究的生物样品分析[7, 8]。目前, 用于生物样品中核酸药物分析的分子杂交法主要包括三明治杂交法(sandwich hybridization assay)、杂交-连接法(hybridization-ligation assay)、杂交荧光分析法(hybridization-based fluorescence assay)、竞争性杂交分析法(competitive hybridization assay)。
1.1.1 三明治杂交法三明治杂交分析法是一种双杂交探针法, 采用捕获探针和检测探针分别结合目标物的部分核酸片段后进行检测(图 1A)。其中, 生物素(biotin) 修饰的捕获探针固定于固定载体, 按照碱基互补关系与待测核酸的部分片段结合。核酸的另一部分与检测探针结合。三明治杂交法操作简便, 适用于复杂的或高度化学修饰的核酸药物检测。

|
Figure 1 Schematic description of sandwich hybridization assay (A), hybridization-ligation assay (B), hybridization-based fluorescence assay (C) and competitive enzyme hybridization assay (D) |
Efler等[9]应用3'-端生物素标记的检测探针和5'-端含有氨基连接体(amino linker) 的捕获探针定量测定人血浆中脱氧寡核苷酸(CGP 7909)。该方法测得定量下限(lower limit of quantification, LLOQ) 为7.8 pg·mL-1, 优于毛细管凝胶电泳-紫外检测法(70 ng·mL-1)。对于三明治杂交法, 当待测核酸链较短时, 使用的检测探针和捕获探针也相对较短。此时, 可利用锁核酸(locked nucleic acid, LNA) 探针增强该核酸药物与探针形成杂合双链分子的稳定性[10]。
1.1.2 杂交-连接法杂交-连接分析法又称为基于核酸连接的分子杂交。主要依靠5'-端额外连接含有9个核苷酸的模板探针和与之互补配对的含有9个核苷酸的连接探针。杂交-连接法的简要过程如图 1B所示。首先, 模板探针的3'-端的生物素与链霉亲和素包被板(streptavidin-coated plate) 结合, 使其固定于固相载体, 待测核酸与模板探针结合。然后, 在T4 DNA连接酶(ligase) 作用下, 连接探针的5'-端磷酸基团与待测核酸3'-端羟基结合。同时, 连接探针互补结合模板探针。随后加入酶标记抗体和底物, 检测反应信号[8]。杂交-连接法不受3'-端分解的核酸代谢产物干扰。虽然该方法可以检测到5'-端分解的核酸代谢产物, 但是在体内3'→5'核酸外切酶(3'→5' exonuclease) 作用下, 核酸代谢主要发生在3'-端[11, 12]。Yu等[8]测定大鼠静脉给予Alicaforsen (ISIS 2302) 后血浆中药物浓度, 计算得到其t1/2分别为39.7 h (剂量3 mg·kg-1) 和54.4 h (剂量10 mg·kg-1)。作者观察到随着给药剂量的增加, t1/2也明显增加。该方法的LLOQ为50 pmol·L-1, 因此可以检测到给药后170 h的血药浓度。
1.1.3 杂交荧光分析法杂交荧光分析法根据荧光染料(Hoechst或者溴化乙啶) 与单链DNA (ssDNA) 作用时不产生荧光或荧光很弱, 而与双链DNA (dsDNA) 作用时荧光增强的原理, 将待测ssDNA与互补ssDNA杂交形成dsDNA, 实现对ssDNA的检测(图 1C)[13, 14]。与其他核酸分子杂交法不同, 该方法并不依赖酶循环扩增。Wei等[15]建立并验证一种杂交荧光分析法定量人血浆中GTI2040 (LLOQ为50 pmol·L-1), 并成功用于急性骨髓性白血病患者的药动学研究。患者静脉滴注GTI2040 (5 mg·kg-1·d-1) 14 h后, 测得其血药浓度。结果显示, GTI2040在患者体内符合二室药代动力学模型特征。t1/2α和t1/2β分别为0.71 h和32 h。该分析方法可以检测静脉滴注停药后48 h血浆中GTI2040浓度, 实现准确描述其末端消除相。
1.1.4 竞争性杂交分析法竞争性杂交分析法利用待测核酸和与其序列相同的示踪探针竞争固定微孔板上的有义链(图 1D) [7]。示踪探针为5'-端生物素标记的反义链。乙酰胆碱酯酶-链霉亲和素(AchE-streptavidin) 复合物与示踪探针为5'-端生物素结合。采用酶免疫分析技术(如Ellman's乙酰胆碱酯酶活性测定法) 测定乙酰胆碱酯酶活性[16]。在竞争性杂交分析法中, 待测核酸的浓度与响应信号成反比。若生物样品中待测核酸的浓度越低, 则结合在有义链上的酶标记物越多, 酶活性就越强。Deverre等[7]应用此方法测定小鼠血浆中的Oligo A。Oligo A为Friend型鼠白血病病毒的反义寡核苷酸。药动学结果显示, Oligo A快速从血中消除, t1/2仅为4.8 min。
在三明治杂交法和杂交-连接法中, 当待测核酸链较短时, 使用捕获探针和检测探针也相对较短, 导致分子杂交反应特异性降低。此时, 竞争性杂交分析法可作为三明治杂交法和杂交-连接法的替代方法。
1.2 实时荧光定量PCRqPCR是指在DNA扩增反应中引入荧光标记分子, 并使荧光信号强度与PCR产物量成正比, 对每一反应时刻的荧光信号进行实时分析, 计算出PCR产物量。目前, qPCR被公认为标准的已知序列核酸定量检测方法[17]。对于信使RNA (messenger RNA, mRNA) 和miRNA的定量分析, 需采用逆转录PCR技术(reverse transcription RCR, RT-PCR)。RT-PCR首先以RNA为模板, 在逆转录酶的作用下合成互补DNA (complementary DNA, cDNA), 再以cDNA为模板通过PCR反应扩增目的基因。虽然RT-PCR是对已知序列的RNA定量分析的有效方法, 但是对于miRNA和siRNA的分析则面临一个挑战性问题。由于miRNA和siRNA的长度仅为18~25个核苷酸, 常规的PCR技术无法实现核苷酸序列的扩增。因此, 必须要延长待测miRNA和siRNA的长度, 构建出一个足够长的PCR模板, 才能进一步应用PCR技术定量分析[18]。为此, 一些针对qPCR的改进方法应运而生。
1.2.1 茎环RT-PCR法茎环RT-PCR法(stem-loop RT-PCR) 是利用茎环结构的引物对miRNA进行反转录, 用3'末端所含的与miRNA反向互补的茎环引物进行miRNA片段的逆转录, 新合成miRNA-cDNA杂合体, 进一步用于定量PCR扩增(图 2A)。因为茎环状结构的引物对成熟的miRNA 3'端具有特异性, 能够将短的miRNA扩展, 赋予该技术更高的特异性, 有助于降低信号的背景噪音[19, 20]。正因如此, 需针对每个miRNA设计相应的引物。
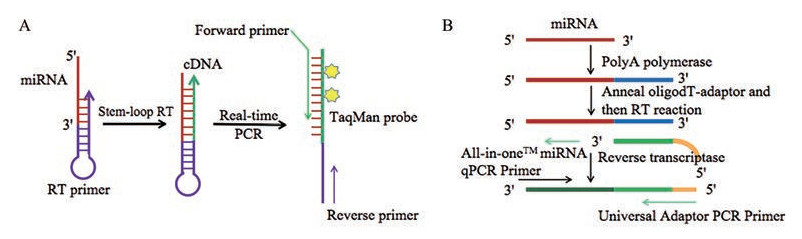
|
Figure 2 Schematic description of stem-loop RT-qPCR (A) and polyA tailing based RT-PCR (B) |
化学修饰使核酸药物在效价、结合亲和性、抗酶解稳定性和毒性等方面不断得到改善[2]。然而在qPCR分析时, 核酸药物的化学修饰可能影响cDNA合成反应的效率以及干扰PCR反应中酶对核苷与磷酸二酯键的识别。Landesman等[21]用改良茎环RT-PCR法测定大鼠血浆和肝脏中化学修饰的siRNA。方法的灵敏度为70 pg·g-1肝脏或70 pg·mL-1血浆, 定量范围可达5个数量级。
1.2.2 PolyA聚合酶加尾法PolyA聚合酶加尾法(PolyA tailing based RT-PCR) 是依靠PolyA聚合酶和逆转录酶的共同参与来完成[20, 22]。在polyA聚合酶的作用下, miRNA的3'端加上一段寡聚腺苷酸的尾巴, 形成一个与mRNA相似的结构, 再用5'端带有oligo (dT) 的通用引物反转录生成miRNA-cDNA杂合体, 然后进行实时PCR反应(图 2B)。PolyA加尾法可对样品中的miRNA进行无差别加尾, 一次逆转录即可完成所有qPCR模板的制备, 实现对样品中多个miRNA的快速检测。
此外, 其他qPCR的改进方法亦有报道, 如引物延伸qPCR (primer-extension qPCR)[23]、竞争性定量qPCR (competitive quantification qPCR)[24]、基于DNA探针连接的qPCR法(ligation-based qPCR)[25]等。
虽然qPCR具有操作简单、检测速度快、灵敏度高和特异性好等特点, 但qPCR实验涉及多个过程, 每一步都可能会影响qPCR数据的质量, 如: 实验设计存在缺陷、对照和重复不足、实验条件和操作技术规范不统一、引物选择不合适、数据处理和统计分析不当等。为获得准确的、可重复的实验结果, Bustin等[26]发表的qPCR实验操作指南, 即MIQE (minimum information for publication of quantitative real-time PCR/digital PCR experiments), 已被广泛接受和应用。
2 基于液相色谱分离技术分析方法液相色谱法可将生物样品中的各组分(如核酸药物及其代谢产物) 分离, 而后采用适当的检测方法逐个进行分析。核酸药物的色谱分离和检测是此类方法需要考虑的重要因素, 它们与分析方法的选择性和灵敏度密切相关。核酸药物常用的分析技术主要包括: 高效液相色谱-紫外/荧光检测法(high performance liquid chromatography-ultraviolet/fluorescence, HPLC-UV/FL)、高效液相色谱-质谱联用法(high performance liquid chromatography tandem mass spectrometry, LC-MS) 等。基于液相色谱分离技术的分析方法具有高选择性、高灵敏度、重复性好、线性范围宽等特点。
2.1 高效液相色谱-紫外/荧光法HPLC法是最早被用于核酸药物分析的色谱分析方法。核酸的液相色谱分离常选择反相离子对色谱法(ion-pair reversed-phased HPLC)[27]和阴离子交换色谱法(anion-exchange HPLC)[28]。与紫外检测相比, 荧光检测灵敏度更高。Arora等[29]将血浆样品提取后冻干, 并用含有5'荧光素DNA探针的溶液复溶, 该探针能与分析物(AVI-4126) 按碱基互补关系形成杂交双链分子, 分析方法LLOQ可达40 ng·mL-1。
2.2 高效液相色谱-质谱联用法LC-MS集HPLC的高分离效率与MS的高灵敏度、高选择性于一体, 已成为生物基质中核酸药物及其代谢物的定性和定量研究强有力的分析工具之一。核酸药物的多聚阴离子磷酸骨架结构, 使核酸药物的LC-MS分析多采用电喷雾离子源(electrospray ionization, ESI) 负离子模式。同时, 样品处理方法、液相色谱和质谱条件的选择也与液相色谱的保留机制、离子化方法、离子化效率等密切相关。
选择与LC-ESI-MS兼容的液相色谱分离方法是首先需要考虑的问题。其中, 反相离子对色谱法采用ESI兼容的有机碱作为流动相, 使有机碱与待测核酸形成离子对, 增强其在反相色谱柱的保留能力, 实现核酸药物及其代谢物的色谱分离。三乙基铵盐常作为离子对试剂, 如三乙基碳酸氢铵(triethylammonium bicarbonate, TEAB) 和三乙基醋酸铵(triethylammonium acetate, TEAA)[30]。LC-ESI-MS的灵敏度受到离子强度的影响, 随着离子对试剂的浓度升高, 离子抑制越明显[31]。迄今, 已有研究证明了1, 1, 1, 3, 3, 3-六氟-2-丙醇-三乙基铵盐(1, 1, 1, 3, 3, 3-hexa-fluoro-2-propanol, HFIP/TEA) 离子对是最行之有效的反相离子对系统之一[30, 31]。HFIP/TEA既实现核酸药物在反相色谱柱的保留, 又能降低离子抑制。根据待测核酸药物的长度及浓度范围的不同, 优化获得HFIP和TEA的最佳浓度[32-34]。
近年来, 随着亲水相互作用色谱(hydrophilic interaction liquid chromatography, HILIC) 的发展, HILIC模式开始用于核酸的LC-MS分析[35]。HILIC采用强极性固定相, 以高比例有机相/低比例水相为流动相, 实现强极性化合物(如寡核苷酸) 在色谱柱上保留。另一方面, 流动相中高比例有机相, 有利于提高ESI-MS的灵敏度[36, 37]。
在dsDNA和siRNA分析中, 色谱柱温是另一个值得关注的问题。当柱温超过dsDNA或siRNA的解链温度时(45~60 ℃), 双分子链结构会被破坏[38-40]。因此, 色谱分离和样品处理过程中须保持温度低于解链温度[38]。
适当的生物样品处理方法可以除去生物基质中含有的大量内源性及外源性干扰物质, 达到从生物样品中分离与浓集待测核酸及其代谢产物的目的。此外, 应用于LC-MS的样品处理方法还应尽量避免基质效应的干扰。蛋白沉淀法(protein precipitation) 因其较低的回收率和明显的基质效应, 较少使用[30]。Chen等[28]采用液-液萃取法(liquid-liquid extraction, LLE), 先以蛋白酶k消化组织样品后用苯酚-二氯甲烷(1∶1, v/v) 提取组织样品中的核酸。此方法从脑、心脏、肾脏各组织中提取待测核酸的回收率范围在50%~85%之间。Turnpenny等[41]在以苯酚和二氯甲烷为提取溶剂的基础上, 加入阳离子型表面活性剂1-(4-硝基苄基)-4-(4-二乙胺苯偶氮) 溴吡啶, 有助于破坏核酸药物与蛋白间的结合, 提高了方法的回收率。结果显示, 8种反义寡核苷酸在血浆中的平均回收率为89% ± 2%。已有多种固相萃取吸附剂(solid phase extraction, SPE) 用于生物样品中寡核苷酸的提取, 如Clarity OTX SPE[42, 43]、Waters Oasis WAX[44]、Waters Oasis HLB[45]等。Clarity OTX SPE是弱的阴离子交换吸附剂。Ramanathan等[42]用Clarity OTX SPE提取石蟹猴血浆中的siRNA, 平均回收率为94.8%, 内标归一化基质因子接近于1, RSD小于15%。Waters Oasis WAX是适用于强酸性化合物的混合模式弱阴离子交换吸附剂, Hemsley等[44]用该吸附剂提取人血浆中15-mer未修饰的DNA寡核苷酸, 平均回收率为62.7%, 内标归一化基质因子1.7, RSD为4.7%。SPE技术方便、省时, 同时可以避免LLE产生的乳化现象, 而且便于自动化操作[44]。SPE吸附剂的筛选及其程序的优化直接影响提取回收率及基质效应, 致使在分析方法开发阶段耗时较长。此外, SPE尽量采用与ESI-MS兼容的试剂, 避免某些试剂对信号产生离子抑制。有时单一的样品处理技术无法获得满意的实验结果, 不同技术的合用则可以实现扬长避短[46, 47]。最近, 一些前瞻性的核酸提取技术也见诸报道, 如反胶束液-液萃取[48]、磁离子液体萃取[49]、生物素化的捕获探针偶联磁珠杂交法[50]等。
与小分子有机药物的生物分析相似, 液相色谱串联三重四极杆质谱(liquid chromatography-triple quadrupole tandem mass spectrometry, LC-MS/MS) 是核酸药物生物分析的强有力工具[34, 47, 51]。其多反应监测模式(multiple reaction monitoring, MRM) 可同时监测多个离子对(前体离子-产物离子), 极大提高分析的选择性和灵敏度, 可对核酸药物及其代谢物进行准确定量。Deng等[34]首先采用苯酚/二氯甲烷液-液萃取结合固相萃取方法提取血浆中PF-OND (开发用于治疗肺癌的硫代反义寡聚核苷酸药物) 及其5'N-1/3'N-1, 5'N-2和5'N-3代谢物, 以含2.85 mmol·L-1 TEA和100 mmol·L-1 HFIP的水(pH 7.8) 和甲醇为流动相进行梯度洗脱, 使用LC-MS/MS的MRM模式同时检测原形核酸及其代谢物。方法定量线性范围4.0~2 000 ng·mL-1, 批内和批间精密度均小于12.2%。MacNeill等[52]使用Waters Oasis WAX固相萃取柱样品处理, HILIC色谱柱进行分离, MRM模式定量分析人血浆中的寡核苷酸(RM1)。方法色谱柱为Waters Acquity UPLC BEH Amide (2.1 mm×50 mm, 1.7 μm), 水(0.02%氨水和10 mmol·L-1甲酸铵) 和乙腈为流动相, 柱温30 ℃, LLOQ为10 nmol·L-1。Gallo Cantafio等[53]用LC-MS/MS检测静脉给药后, LNA-i-miR-221 (875 mg·kg-1) 在石蟹猴血浆中的浓度。该方法的LLOQ为50 ng·mL-1。由血药浓度计算得LNA-i-miR-221在石蟹猴体内t1/2为12.83 h。
高分辨质谱的快速发展, 使得液相色谱串联高分辨质谱(liquid chromatography-high resolution-mass spectrometry, LC-HRMS) 越来越多地用于核酸药物的生物分析领域。分辨率是质谱分开相邻质荷比离子的能力。高分辨率质谱可以进行同位素质量及有机分子质量的准确测定(质量偏差小于5 ppm)。因此, LC-HRMS为核酸分析提供更好的选择性。用于核酸分析的高分辨质谱主要有: 飞行时间质谱(time-of-flight, TOF)[54]、四级杆串联飞行时间质谱(quadrupole time-of-flight, Q-TOF)[42]、线性离子阱串联静电场轨道阱质谱(linear ion trap quadrupole-Orbitrap, LTQ-Orbitrap) 和四级杆串联静电场轨道阱质谱(Q-Exactive Orbitrap)[55, 56]。Ramanathan等[42]用LC-TOF-MS的全扫描模式(full scan mode) 同时测定食蟹猴血浆和组织中siRNA的有义链和反义链。方法以高分辨(分辨率 > 10 000) 提取离子色谱图(extracted ion chromatogram, XIC) 定量, 累加待测核酸不同电荷态离子[M-mH]m-信号强度获得最佳灵敏度(定量下限为10 ng·mL-1)。另外, Q-TOF的MRM模式也可用于生物大分子定量分析[57]。此时与三重四级杆质谱相似, 四级杆过滤前体离子(precursor ion), TOF扫描产物离子(product ion)。该模式下, 由于Q1选择前体离子, 无法对分析物不同电荷状态离子[M-mH]m-信号强度累加。与Q-TOF相比, Q-Exactive具有更高的分辨率(~140 000)。对于定量复杂生物基质中的核酸, Q-Exactive的高分辨率(~70 000或更高) 足以消除生物基质或代谢物对分析产生的干扰[58]。因此, Q-Exactive的全扫描模式和选择离子检测模式(selected ion monitoring, SIM) 同样适用于核酸分析[55, 59]。Sun等[60]采用LC-Q-Exactive MS定量大鼠血浆中米泊美生。Q-Exactive MS的全扫描模式(分辨率70 000) 用以检测其电荷分布情况, 平行反应监测模式(parallel reaction monitoring, PRM) 用于定量分析(分辨率17 500)。
高分辨质谱能够采集药物及其代谢物精确质量数据, 这些数据有助于计算代谢物的精确质量转移, 确定元素组成, 为确定代谢物的分子式、预测代谢物的结构特征提供重要的信息。Zou等[61]用LTQ-Orbitrap表征名为HBV263的siRNA在体外人血清和肝微粒体中的代谢产物。Liu等[62]利用Q-Exactive研究REVERSIR在大鼠和石蟹猴体内代谢物。数据分析中当分辨率设置为35 000时, 即可消除每个电荷态离子的同位素重叠效应。代谢物Rev-N1-N9的质谱峰经过去卷积后获得单同位素质量数为3 002.386 Da, 与其理论值3 002.385 Da相比, 质量偏差仅为0.3 ppm。
3 其他分析技术方法 3.1 基于杂交的高效液相色谱荧光法基于杂交的高效液相色谱法(hybridization-based HPLC-fluorescence assays, hybridization LC-FL) 能将两种技术(核酸分子杂交和高效液相色谱) 整合并用于定量生物样品中的核酸药物。待测核酸首先与带有特异性核酸序列的荧光探针杂交, 接着杂交双链经过高效液相色谱分离, 最后被荧光检测器检测[58]。
Tian等[63]利用肽核酸探针(peptide nucleic acid probe, PNA probe) 与siRNA的反义链形成PNA/RNA杂交双链。样品分析采用DNAPac® PA 200 (4 mm×250 mm) 色谱柱; 乙腈: 25 mmol·L-1 Tris-HCl缓冲溶液(含1 mmol·L-1 EDTA) (50∶50, v/v) 为流动相A, 流动相A中加入1.6 mol·L-1 NaClO4为流动相B, 梯度洗脱; 荧光检测波长为426 nm/484 nm (激发波长/发射波长)。结果显示, 血浆样品中待测核酸在1.0~1 000 ng·mL-1线性良好。在一项多剂量爬坡Ⅱ期临床试验中, Zhang等[64]采用hybridization LC-FL法, 测定静脉给药后人血浆中帕替斯坦浓度。药动学研究结果显示, 帕替斯坦的t1/2α和t1/2β分别为0.947~1.26 h和1.65~2.48 d。帕替斯坦血药浓度以二相的方式消除, 并在较短时间内从血浆快速分布到组织, 随后是一个较慢的处置阶段, 消除半衰期为1.65~2.48 d。血浆中较长的末端消除半衰期代表了组织向血浆室的再分布, 反映了组织细胞向细胞外液和血浆室的缓慢转移[5]。
尽管核酸分子杂交和qPCR具有较高的灵敏度, 可是难以区分核酸原形药物及其代谢产物。LC-MS可以提高分析方法的选择性, 却在灵敏度方面稍显不足。杂交-液相色谱荧光法将分子杂交技术和液相色谱法的优点合二为一。从分析方法的灵敏度方面来看, 杂交-液相色谱荧光法与核酸分子杂交法不分伯仲(1 ng·mL-1)[63]。而且, 此法能弥补核酸分子杂交和qPCR选择性差的不足。通过液相色谱可以将核酸药物及其代谢产物分离。与HPLC和LC-MS相比, 该法不但可以提供较高的灵敏度, 而且无需从生物样品提取待测核酸药物, 简化样品处理过程, 有利于提高分析通量[63]。杂交-液相色谱荧光法不足之处在于分子杂交需要设计探针, 导致分析方法开发时间延长。探针的长度是探针设计值得注意的一个问题。Ji等[65]发现, 肽核酸探针会影响色谱峰形和分离度。在高效液相色谱中由短链肽核酸探针形成的杂交双分子链具有更出色的峰形和分离度。这是由于探针链越长, 核酸及其代谢物形成的杂交双分子链的物理化学性质越接近, 色谱分离难度越大[65]。
3.2 毛细管凝胶电泳毛细管凝胶电泳(capillary gel electrophoresis, CGE) 是将板上的凝胶填充到毛细管中作支持物, 在电压的驱动下将核酸根据分子量进行分离。因此, CGE的高分辨率适用于生物基质中的核酸及其代谢产物的分离。与液相色谱法相似, CGE也需要对生物样品进行处理后进样分析。Reyderman等[66]用CGE分离激光诱导荧光(laser-induced fluorescence) 检测大鼠血浆中的寡核苷酸(GS522), 方法的LLOQ为20 ng·mL-1。同样, CGE-MS也能够为核酸分析提供较高分辨率和灵敏度[67]。但是, CGE所需的高浓度缓冲盐也对质谱的灵敏度造成影响。
3.3 基质辅助激光解吸电离-飞行时间质谱基质辅助激光解吸电离(matrix assisted laser desorption ionization, MALDI) 的原理是将分析物分散在基质分子中使二者形成共结晶, 当激光照射晶体时, 基质分子吸收能量, 导致能量蓄积并迅速升温, 从而使基质晶体升华, 致使基质和分析物进入气相, 此时被分析物和基质分子一起被释放出来, 在此过程中生物大分子与基质分子同时发生了质子转移, 使生物大分子被离子化。MALDI的关键是选择合适的基质, 基质的具体作用包括: ①生物大分子样品被基质分散从而减小生物大分子间的相互作用; ②基质从激光束中吸收能量并转化为固态熔体体系的激发能, 使微量样品产生瞬间相变并吸收过剩的能量; ③使生物大分子离子化[68]。最近, Herkt等[69]用MALDI-TOF-MS测定anti-microRNA (antimiR132) 在小鼠血浆中的含量, 并对其进行药动学研究。血浆样品经过蛋白酶k消化和乙醇沉淀蛋白后, 离心所得上清液与基质溶液(2, 4, 6-三羟基苯乙酮) 混合, 滴加到MALDI样品靶上, 由TOF-MS分析检测。虽然该方法的灵敏度(LLOQ 1 pmol·μL-1) 明显逊色于LC-MS方法(LLOQ 10 fmol·μL-1), 但MALDI-TOF-MS的分析速度快, 15 min可以检测100个样品[69]。
4 生物样品分析方法验证可靠的分析方法是进行生物样品定量分析的基础。生物样品分析方法用于实际样品的分析前, 须对方法进行方法学验证。《中国药典》(2020版) 四部通则9012收载了《生物样品定量分析方法指导原则》, 针对色谱分析方法和配体结合分析方法, 指导原则提供了方法验证的要求[70]。此外, 欧洲药品管理局(EMA) 于2012年发布了《生物分析方法验证指导原则》[71]。美国食品药品监督管理局(FDA) 曾于2001年发布《生物分析方法验证指导原则》, 并于2013年发布了修订版(草案), 于2018年生效[72]。人用药品注册技术要求国际协调会(ICH) 于2019年2月最新发布ICH协调指导原则M10《生物分析方法验证(草案)》, 正处于征求意见阶段[73]。
目前, 在核酸药物的生物样品定量分析方法验证中, 生物分析研究人员主要依照上述指导原则对色谱分析方法和配体结合分析方法验证的要求进行。例如: 基于液相色谱分离技术分析方法(HPLC和LC-MS) 和基于杂交的高效液相色谱荧光法参照色谱分析方法验证的要求进行。然而, 上述指导原则并未对qPCR法作出明确规定。虽然Bustin等[26]发表的qPCR实验操作指南(MIQE), 已被学术界广泛接受和应用, 但是MIQE不足以支持开展规范性生物分析研究。近年, 生物分析研究人员设计并采用基于“目的适用性评估的生物分析方法验证”确保qPCR分析方法的可靠性和可行性。从1990年至今, 国际上每年召开生物分析当前问题研讨会(workshop on recent issues in bioanalysis, WRIB)。WRIB由制药工业界、学术界和监管部门共同参与并针对生物分析面临问题展开讨论, 提出可能的解决方法并达成一致。WRIB分别于2018年[74]、2019年[75]和2020年[76]发表白皮书, 对基于qPCR技术的生物分析方法验证进行讨论。例如: 方法验证应明确测定方法的灵敏度、特异性和重现性; 方法验证的接受标准, 可以具体问题具体分析, 但应当在方法验证前明确; 推荐PCR扩增效率应在90%~110%内; 含有内参基因的加样回收率样品应能够评价样品基质是否对分析物的回收有干扰等[76]。
5 总结与展望开发可靠的且具有高特异性、选择性和灵敏度的分析方法用以支持规范性的生物分析研究是生物分析研究人员不断追求的目标。本文主要围绕基于核酸分子杂交的酶联免疫法、实时荧光定量PCR法和基于液相色谱的分离技术法介绍核酸药物的生物分析方法, 它们各有优势, 也各有不足(表 1)。在这些分析技术中, hybridization-ELISA和qPCR拥有出色的灵敏度, LC-MS技术则在选择性方面优势明显。核酸药物分析技术的类型以及待测核酸药物化学结构是决定核酸药物生物分析方法的灵敏度和选择性的重要因素。通常, 分子杂交法的灵敏度随着待测核酸的长度增加显著提升, 而LC-MS法则恰恰与之相反。因为长链核酸在ESI-MS中易产生多电荷离子, 导致其信号强度被分散, 灵敏度降低[58]。然而, 分子杂交法对较大的核酸代谢产物(如n-1或n-2) 却缺乏足够的选择性。此外, 核酸药物的化学修饰可能影响基于酶反应的分子杂交法。核酸分子杂交-液相色谱荧光法将分子杂交技术和液相色谱法合二为一, 取长补短。与LC-MS法相比, 提升了检测的灵敏度; 与分子杂交法相比, 又改善了方法的选择性, 但是分析方法开发时间较长。因此, LC-MS适用于链长较短的核酸药物(8~18-mer)定量分析, 核酸分子杂交-液相色谱荧光法适于中等链长的核酸药物分析(14~30-mer), 而对于长链(> 24-mer) 且未经修饰的核酸药物(如内源性的miRNA或sirRNA), 分子杂交法则更具优势[58]。生物分析研究人员应综合考虑待测核酸药物的结构特征及试验需求(如灵敏度) 合理地选择分析技术。
| Table 1 Summary and comparison of different methods for the bioanalysis of therapeutic oligonucleotides |
数字PCR技术(digital PCR, dPCR) 是在PCR基础上发展, 可用于核酸绝对定量的新方法[77]。与RT-PCR相比, dPCR无需标准曲线即可绝对定量miRNA。dPCR既表现出与RT-PCR相当的灵敏度, 又具有更高的精密度[78]。同时, dPCR不易受到PCR抑制剂的影响, 如肝素、十二烷基硫酸钠(SDS) 等[79]。采用dPCR方法定量生物基质中的miRNA和长链非编码RNA (long non-coding RNA, lncRNA) 日益受到生物分析研究人员的青睐[80, 81]。随着纳米技术和微流体技术的发展, 微滴式数字PCR (droplet digatil PCR, ddPCR) 应运而生[77]。ddPCR的优势在于高精密度的定量分析。与qPCR相似, ddPCR的可靠性仍依赖良好的实验设计和内参基因的质量控制。分支链DNA信号放大技术(branched DNA, bDNA) 是一种不依赖PCR扩增的核酸杂交信号放大检测技术。该技术可用于多种生物样品(例如血、细胞、组织等) 中mRNA的定量检测。当前bDNA技术已经发展至第三代, 其灵敏度得到进一步提升[82-84]。bDNA技术克服了传统的实时PCR技术中的缺陷和不确定因素(如抽提核酸的损失、操作次数过多导致的误差), 无需抽提纯化RNA, 无需反转录, 无需PCR扩增, 只需要将样品用特定的裂解液裂解后, 经探针杂交与信号放大即可迅速得到基因定量结果[85]。因此, dPCR、ddPCR及bDNA技术在高精密度的核酸药物生物分析领域表现出一定的优势和巨大的应用潜力。
LC-MS/MS和LC-HRMS因其高选择性、良好的重复性和重现性、宽线性范围和易于自动化等特点, 备受生物分析研究人员的青睐。纳升液相色谱-高分辨质谱联用(nano-LC-HRMS) 提高了miRNA分析的灵敏度[86]。然而, 较长的样品分析时间限制其在生物分析研究的应用。微流液相色谱-质谱联用(microflow LC-MS/MS) 有望在提高分析方法灵敏度的同时, 实现高通量分析[87]。另外, 随着自动化在线SPE-LC-MS技术的发展, 势必会进一步提高核酸生物分析的工作效率。
作者贡献: 程忠哲负责论文的文献查阅、撰写与修改; 姜宏梁负责论文构思、修改及审阅。
利益冲突: 作者声明没有竞争性经济利益冲突。
| [1] |
Wang J, Wang L, Lv J, 等. Progress in efficacy analysis and development of listed nucleic acid drugs[J]. Chin J New Drugs (中国新药杂志), 2019, 28: 2217-2224. |
| [2] |
He J. Current progress and prospects of nucleic acid-based therapeutics[J]. J Int Pharm Res (国际药学研究杂志), 2017, 44: 1028-1051. |
| [3] |
Zhou JH, Rossi J. Aptamers as targeted therapeutics: current potential and challenges[J]. Nat Rev Drug Discov, 2017, 16: 181-202. DOI:10.1038/nrd.2016.199 |
| [4] |
Tremblay GA, Oldfield PR. Bioanalysis of siRNA and oligonucleotide therapeutics in biological fluids and tissues[J]. Bioanalysis, 2009, 1: 595-609. DOI:10.4155/bio.09.66 |
| [5] |
Tang XG, Guan XD, Chen R, 等. Progress in clinical pharmacology of oligonucleotides[J]. Acta Pharm Sin (药学学报), 2020, 55: 218-225. |
| [6] |
Wang F, Zuroske T, Watts JK. RNA therapeutics on the rise[J]. Nat Rev Drug Discov, 2020, 19: 441-442. DOI:10.1038/d41573-020-00078-0 |
| [7] |
Deverre JR, Boutet V, Boquet D, et al. A competitive enzyme hybridization assay for plasma determination of phosphodiester and phosphorothioate antisense oligonucleotides[J]. Nucleic Acids Res, 1997, 25: 3584-3589. DOI:10.1093/nar/25.18.3584 |
| [8] |
Yu RZ, Baker B, Chappell A, et al. Development of an ultrasensitive noncompetitive hybridization-ligation enzyme-linked immunosorbent assay for the determination of phosphorothioate oligodeoxynucleotide in plasma[J]. Anal Biochem, 2002, 304: 19-25. DOI:10.1006/abio.2002.5576 |
| [9] |
Efler SM, Zhang L, Noll BO, et al. Quantification of oligodeoxynucleotides in human plasma with a novel hybridization assay offers greatly enhanced sensitivity over capillary gel electrophoresis[J]. Oligonucleotides, 2005, 15: 119-131. DOI:10.1089/oli.2005.15.119 |
| [10] |
You Y, Moreira BG, Behlke MA, et al. Design of LNA probes that improve mismatch discrimination[J]. Nucleic Acids Res, 2006, 34: e60. DOI:10.1093/nar/gkl175 |
| [11] |
Eder PS, Devine RJ, Dagle JM, et al. Substrate specificity and kinetics of degradation of antisense oligonucleotides by a 3' exonuclease in plasma[J]. Antisense Res Dev, 1991, 1: 141-151. DOI:10.1089/ard.1991.1.141 |
| [12] |
Wójcik M, Cieślak M, Stec WJ, et al. Nucleotide pyrophosphatase/phosphodiesterase 1 is responsible for degradation of antisense phosphorothioate oligonucleotides[J]. Oligonucleotides, 2007, 17: 134-145. DOI:10.1089/oli.2007.0021 |
| [13] |
Kandimalla ER, Pandey RK, Agrawal S. Hybridization-based fluorescence assay allows quantitation of single-stranded oligodeoxynucleotides in low nanomolar range[J]. Anal Biochem, 2004, 328: 93-95. DOI:10.1016/j.ab.2004.01.005 |
| [14] |
Zeng GP, Xiang DS, He ZK. Fluorimetric method for the determination of sequence-specific DNA with the fluorescent dye Hoechst 33258[J]. Acta Chim Sin (化学学报), 2011, 69: 1450-1456. |
| [15] |
Wei XH, Dai GW, Marcucci G, et al. A specific picomolar hybridization-based ELISA assay for the determination of phosphorothioate oligonucleotides in plasma and cellular matrices[J]. Pharm Res, 2006, 23: 1251-1264. DOI:10.1007/s11095-006-0082-3 |
| [16] |
Ellman GL, Courtney KD, Andres V, et al. A new and rapid colorimetric determination of acetylcholinesterase activity[J]. Biochem Pharmacol, 1961, 7: 88-95. DOI:10.1016/0006-2952(61)90145-9 |
| [17] |
Ma M, Balasubramanian N, Dodge R, et al. Challenges and opportunities in bioanalytical support for gene therapy medicinal product development[J]. Bioanalysis, 2017, 9: 1423-1430. DOI:10.4155/bio-2017-0116 |
| [18] |
Luo X, Zhang J, Wang H, et al. PolyA RT-PCR-based quantification of microRNA by using universal TaqMan probe[J]. Biotechnol Lett, 2012, 34: 627-633. DOI:10.1007/s10529-011-0813-3 |
| [19] |
Chen C, Ridzon DA, Broomer AJ, et al. Real-time quantification of microRNAs by stem-loop RT-PCR[J]. Nucleic Acids Res, 2005, 33: e179. DOI:10.1093/nar/gni178 |
| [20] |
Chen Y, Wang Y. Current research in methods for the quantitative detection of microRNA[J]. J Guangdong Pharm Univ (广东药学院学报), 2016, 32: 666-670. |
| [21] |
Landesman Y, Svrzikapa N, Cognetta A, et al. In vivo quantification of formulated and chemically modified small interfering RNA by heating-in-triton quantitative reverse transcription polymerase chain reaction (HIT qRT-PCR)[J]. Silence, 2010, 1: 16. DOI:10.1186/1758-907X-1-16 |
| [22] |
Shi R, Sun YH, Zhang XH, et al. Poly(T) adaptor RT-PCR[J]. Methods Mol Biol, 2012, 822: 53-66. |
| [23] |
Raymond CK, Roberts BS, Garrett-Engele P, et al. Simple, quantitative primer-extension PCR assay for direct monitoring of microRNAs and short-interfering RNAs[J]. RNA, 2005, 11: 1737-1744. DOI:10.1261/rna.2148705 |
| [24] |
Liu WL, Stevenson M, Seymour LW, et al. Quantification of siRNA using competitive qPCR[J]. Nucleic Acids Res, 2009, 37: e4. DOI:10.1093/nar/gkn903 |
| [25] |
Zhang JY, Li ZP, Wang H, et al. Ultrasensitive quantification of mature microRNAs by real-time PCR based on ligation of a ribonucleotide-modified DNA probe[J]. Chem Commun (Camb), 2011, 47: 9465-9467. DOI:10.1039/c1cc13466c |
| [26] |
Bustin SA, Benes V, Garson JA, et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments[J]. Clin Chem, 2009, 55: 611-622. DOI:10.1373/clinchem.2008.112797 |
| [27] |
Bigelow JC, Chrin LR, Mathews LA, et al. High-performance liquid chromatographic analysis of phosphorothioate analogues of oligodeoxynucleotides in biological fluids[J]. J Chromatogr A, 1990, 533: 133-140. DOI:10.1016/S0378-4347(00)82193-3 |
| [28] |
Chen SH, Qian M, Brennan JM, et al. Determination of antisense phosphorothioate oligonucleotides and catabolites in biological fluids and tissue extracts using anion-exchange high-performance liquid chromatography and capillary gel electrophoresis[J]. J Chromatogr B Biomed Sci Appl, 1997, 692: 43-51. DOI:10.1016/S0378-4347(96)00499-9 |
| [29] |
Arora V, Knapp DC, Reddy MT, et al. Bioavailability and efficacy of antisense morpholino oligomers targeted to c-myc and cytochrome P-4503A2 following oral administration in rats[J]. J Pharm Sci, 2002, 91: 1009-1018. DOI:10.1002/jps.10088 |
| [30] |
van Dongen WD, Niessen WM. Bioanalytical LC-MS of therapeutic oligonucleotides[J]. Bioanalysis, 2011, 3: 541-564. DOI:10.4155/bio.11.8 |
| [31] |
Apffel A, Chakel JA, Fischer S, et al. Analysis of oligonucleotides by HPLC-electrospray ionization mass spectrometry[J]. Anal Chem, 1997, 69: 1320-1325. DOI:10.1021/ac960916h |
| [32] |
Dai G, Wei X, Liu Z, et al. Characterization and quantification of Bcl-2 antisense G3139 and metabolites in plasma and urine by ion-pair reversed phase HPLC coupled with electrospray ion-trap mass spectrometry[J]. J Chromatogr B Analyt Technol Biomed Life Sci, 2005, 825: 201-213. DOI:10.1016/j.jchromb.2005.05.049 |
| [33] |
Zhang G, Lin J, Srinivasan K, et al. Strategies for bioanalysis of an oligonucleotide class macromolecule from rat plasma using liquid chromatography-tandem mass spectrometry[J]. Anal Chem, 2007, 79: 3416-3424. DOI:10.1021/ac0618674 |
| [34] |
Deng P, Chen XY, Zhang GD, et al. Bioanalysis of an oligonucleotide and its metabolites by liquid chromatography-tandem mass spectrometry[J]. J Pharm Biomed Anal, 2010, 52: 571-579. DOI:10.1016/j.jpba.2010.01.040 |
| [35] |
Goyon A, Yehl P, Zhang K. Characterization of therapeutic oligonucleotides by liquid chromatography[J]. J Pharm Biomed Anal, 2020. DOI:10.1016/j.jpba.2020.113105 |
| [36] |
Gong LZ, Mccullagh JSO. Analysis of oligonucleotides by hydrophilic interaction liquid chromatography coupled to negative ion electrospray ionization mass spectrometry[J]. J Chromatogr A, 2011, 1218: 5480-5486. DOI:10.1016/j.chroma.2011.06.044 |
| [37] |
Easter RN, Kroning KK, Caruso JA, et al. Separation and identification of oligonucleotides by hydrophilic interaction liquid chromatography (HILIC)-inductively coupled plasma mass spectrometry (ICPMS)[J]. Analyst, 2010, 135: 2560-2565. DOI:10.1039/c0an00399a |
| [38] |
Mccarthy SM, Gilar M, Gebler J. Reversed-phase ion-pair liquid chromatography analysis and purification of small interfering RNA[J]. Anal Biochem, 2009, 390: 181-188. DOI:10.1016/j.ab.2009.03.042 |
| [39] |
Beverly M, Hartsough K, Machemer L, et al. Liquid chromatography electrospray ionization mass spectrometry analysis of the ocular metabolites from a short interfering RNA duplex[J]. J Chromatogr B Analyt Technol Biomed Life Sci, 2006, 835: 62-70. DOI:10.1016/j.jchromb.2006.03.008 |
| [40] |
Beverly M, Hartsough K, Machemer L. Liquid chromatography/electrospray mass spectrometric analysis of metabolites from an inhibitory RNA duplex[J]. Rapid Commun Mass Spectrom, 2005, 19: 1675-1682. DOI:10.1002/rcm.1972 |
| [41] |
Turnpenny P, Rawal J, Schardt T, et al. Quantitation of locked nucleic acid antisense oligonucleotides in mouse tissue using a liquid-liquid extraction LC-MS/MS analytical approach[J]. Bioanalysis, 2011, 3: 1911-1921. DOI:10.4155/bio.11.100 |
| [42] |
Ramanathan L, Shen H. LC-TOF-MS methods to quantify siRNAs and major metabolite in plasma, urine and tissues[J]. Bioanalysis, 2019, 11: 1983-1992. DOI:10.4155/bio-2019-0134 |
| [43] |
Jiao KS, Rashid A, Basu SK, et al. Quantitative analysis of dicer substrate oligonucleotides in mouse liver by ultra-high-performance liquid chromatography-electrospray ionization tandem mass spectrometry[J]. Assay Drug Dev Technol, 2012, 10: 278-288. DOI:10.1089/adt.2011.0435 |
| [44] |
Hemsley M, Ewles M, Goodwin L. Development of a bioanalytical method for quantification of a 15-mer oligonucleotide at sub-ng/ml concentrations using LC-MS/MS[J]. Bioanalysis, 2012, 4: 1457-1469. DOI:10.4155/bio.12.117 |
| [45] |
Johnson JL, Guo W, Zang J, et al. Quantification of raf antisense oligonucleotide (rafAON) in biological matrices by LC-MS/MS to support pharmacokinetics of a liposome-entrapped rafAON formulation[J]. Biomed Chromatogr, 2005, 19: 272-278. DOI:10.1002/bmc.450 |
| [46] |
Cen YY, Li XL, Liu D, et al. Development and validation of LC-MS/MS method for the detection and quantification of CpG oligonucleotides 107(CpG ODN107) and its metabolites in mice plasma[J]. J Pharm Biomed Anal, 2012, 70: 447-455. DOI:10.1016/j.jpba.2012.06.022 |
| [47] |
Ewles M, Goodwin L, Schneider A, et al. Quantification of oligonucleotides by LC-MS/MS: the challenges of quantifying a phosphorothioate oligonucleotide and multiple metabolites[J]. Bioanalysis, 2014, 6: 447-464. DOI:10.4155/bio.13.319 |
| [48] |
Maruyama T, Ishizu N, Eguchi Y, et al. Liquid-liquid extraction of enzymatically synthesized functional RNA oligonucleotides using reverse micelles with a DNA-surfactant[J]. Chem Commun (Camb), 2016, 52: 12376-12379. DOI:10.1039/C6CC06985A |
| [49] |
Clark KD, Nacham O, Yu HL, et al. Extraction of DNA by magnetic ionic liquids: tunable solvents for rapid and selective DNA analysis[J]. Anal Chem, 2015, 87: 1552-1559. DOI:10.1021/ac504260t |
| [50] |
Li P, Gong YQ, Kim J, et al. Hybridization liquid chromatography-tandem mass spectrometry: an alternative bioanalytical method for antisense oligonucleotide quantitation in plasma and tissue samples[J]. Anal Chem, 2020, 92: 10548-10559. DOI:10.1021/acs.analchem.0c01382 |
| [51] |
Basiri B, Bartlett MG. LC-MS of oligonucleotides: applications in biomedical research[J]. Bioanalysis, 2014, 6: 1525-1542. DOI:10.4155/bio.14.94 |
| [52] |
Macneill R, Hutchinson T, Acharya V, et al. An oligonucleotide bioanalytical LC-SRM methodology entirely liberated from ion-pairing[J]. Bioanalysis, 2019, 11: 1157-1170. |
| [53] |
Gallo Cantafio ME, Nielsen BS, Mignogna C, et al. Pharmacokinetics and pharmacodynamics of a 13-mer LNA-inhibitor-miR-221 in mice and non-human primates[J]. Mol Ther Nucleic Acids, 2016, 5: 1-13. |
| [54] |
Rentel C, Dacosta J, Roussis S, et al. Determination of oligonucleotide deamination by high resolution mass spectrometry[J]. J Pharm Biomed Anal, 2019, 173: 56-61. DOI:10.1016/j.jpba.2019.05.012 |
| [55] |
Taoka M, Yamauchi Y, Nobe Y, et al. An analytical platform for mass spectrometry-based identification and chemical analysis of RNA in ribonucleoprotein complexes[J]. Nucleic Acids Res, 2009, 37: e140. DOI:10.1093/nar/gkp732 |
| [56] |
Goyon A, Zhang K. Characterization of antisense oligonucleotide impurities by ion-pairing reversed-phase and anion exchange chromatography coupled to hydrophilic interaction liquid chromatography/mass spectrometry using a versatile two-dimensional liquid chromatography setup[J]. Anal Chem, 2020, 92: 5944-5951. DOI:10.1021/acs.analchem.0c00114 |
| [57] |
Morin LP, Mess JN, Garofolo F. Large-molecule quantification: sensitivity and selectivity head-to-head comparison of triple quadrupole with Q-TOF[J]. Bioanalysis, 2013, 5: 1181-1193. DOI:10.4155/bio.13.87 |
| [58] |
Wang L, Ji C. Advances in quantitative bioanalysis of oligonucleotide biomarkers and therapeutics[J]. Bioanalysis, 2016, 8: 143-155. DOI:10.4155/bio.15.234 |
| [59] |
Miyaguchi H, Yamamuro T, Ohta H, et al. Genotyping of toxic pufferfish based on specific PCR-RFLP products as determined by liquid chromatography/quadrupole-orbitrap hybrid mass spectrometry[J]. J Agric Food Chem, 2015, 63: 9363-9371. DOI:10.1021/acs.jafc.5b03703 |
| [60] |
Sun YC, Nitta S, Saito K, et al. Development of a bioanalytical method for an antisense therapeutic using high-resolution mass spectrometry[J]. Bioanalysis, 2020, 12: 1739-1756. DOI:10.4155/bio-2020-0225 |
| [61] |
Zou Y, Tiller P, Chen IW, et al. Metabolite identification of small interfering RNA duplex by high-resolution accurate mass spectrometry[J]. Rapid Commun Mass Spectrom, 2008, 22: 1871-1881. DOI:10.1002/rcm.3561 |
| [62] |
Liu J, Li J, Tran C, et al. Oligonucleotide quantification and metabolite profiling by high-resolution and accurate mass spectrometry[J]. Bioanalysis, 2019, 11: 1967-1981. DOI:10.4155/bio-2019-0137 |
| [63] |
Tian QG, Rogness J, Meng M, et al. Quantitative determination of a siRNA (AD00370) in rat plasma using peptide nucleic acid probe and HPLC with fluorescence detection[J]. Bioanalysis, 2017, 9: 861-872. DOI:10.4155/bio-2017-0017 |
| [64] |
Zhang XP, Goel V, Robbie GJ. Pharmacokinetics of patisiran, the first approved RNA interference therapy in patients with hereditary transthyretin-mediated amyloidosis[J]. J Clin Pharmacol, 2020, 60: 573-585. |
| [65] |
Ji Y, Liu Y, Xia W, et al. Importance of probe design for bioanalysis of oligonucleotides using hybridization-based LC-fluorescence assays[J]. Bioanalysis, 2019, 11: 1917-1925. DOI:10.4155/bio-2019-0154 |
| [66] |
Reyderman L, Stavchansky S. Quantitative determination of short single-stranded oligonucleotides from blood plasma using capillary electrophoresis with laser-induced fluorescence[J]. Anal Chem, 1997, 69: 3218-3222. DOI:10.1021/ac970280+ |
| [67] |
Willems AV, Deforce DL, van Peteghem CH, et al. Analysis of nucleic acid constituents by on-line capillary electrophoresis-mass spectrometry[J]. Electrophoresis, 2005, 26: 1221-1253. DOI:10.1002/elps.200410278 |
| [68] |
Yang Y, Zheng P, Ye L. Biological mass spectrometry and its application in the field of nucleic acid[J]. Chin J Pharm Anal (药物分析杂志), 2008, 28: 642-646. |
| [69] |
Herkt M, Foinquinos A, Batkai S, et al. Pharmacokinetic studies of antisense oligonucleotides using MALDI-TOF mass spectrometry[J]. Front Pharmacol, 2020, 11: 220. DOI:10.3389/fphar.2020.00220 |
| [70] |
Chinese Pharmacopoeia Commission. Pharmacopoeia of the People's Republic of China (中华人民共和国药典)[S]. Vol 4. 2020 Ed. Beijing: China Medical Science Press, 2020: 466-472.
|
| [71] |
EMA. Guideline on bioanalytical method validation[S/OL]. London: European Medicines Agencey, 2012[2021-07-07]. https://www.ema.europa.eu/en/documents/scientific-guideline/guideline-bioanalytical-method-validation_en.pdf.
|
| [72] |
FDA. Bioanalytical Method Validation Guidance for Industry[S/OL]. USA: FDA, 2018[2021-07-07]. https://www.fda.gov/media/70858/download.
|
| [73] |
ICH. ICH harmonised guideline. M10: Bioanalytical Method Validation[S/OL]. Switzerland: ICH, 2019[2021-07-07]. https://database.ich.org/sites/default/files/M10_EWG_Draft_Guideline.pdf.
|
| [74] |
Stevenson L, Richards S, Pillutla R, et al. 2018 White paper on recent issues in bioanalysis: focus on flow cytometry, gene therapy, cut points and key clarifications on BAV (Part 3-LBA/cell-based assays: immunogenicity, biomarkers and PK assays)[J]. Bioanalysis, 2018, 10: 1973-2001. DOI:10.4155/bio-2018-0287 |
| [75] |
Piccoli S, Mehta D, Vitaliti A, et al. 2019 White paper on recent issues in bioanalysis: FDA immunogenicity guidance, gene therapy, critical reagents, biomarkers and flow cytometry validation (Part 3-recommendations on 2019 FDA immunogenicity guidance, gene therapy bioanalytical challenges, strategies for critical reagent management, biomarker assay validation, flow cytometry validation & CLSI H62)[J]. Bioanalysis, 2019, 11: 2207-2244. DOI:10.4155/bio-2019-0271 |
| [76] |
Corsaro B, Yang TY, Murphy R, et al. 2020 White paper on recent issues in bioanalysis: vaccine assay validation, qPCR assay validation, QC for CAR-T flow cytometry, NAb assay harmonization and ELISpot validation (Part 3-recommendations on immunogenicity assay strategies, NAb assays, biosimilars and FDA/EMA immunogenicity guidance/guideline, gene & cell therapy and vaccine assays)[J]. Bioanalysis, 2021, 13: 415-463. DOI:10.4155/bio-2021-0007 |
| [77] |
Postel M, Roosen A, Laurent-Puig P, et al. Droplet-based digital PCR and next generation sequencing for monitoring circulating tumor DNA: a cancer diagnostic perspective[J]. Expert Rev Mol Diagn, 2018, 18: 7-17. DOI:10.1080/14737159.2018.1400384 |
| [78] |
Hindson CM, Chevillet JR, Briggs HA, et al. Absolute quantification by droplet digital PCR versus analog real-time PCR[J]. Nat Methods, 2013, 10: 1003-1005. DOI:10.1038/nmeth.2633 |
| [79] |
Dingle TC, Sedlak RH, Cook L, et al. Tolerance of droplet-digital PCR vs real-time quantitative PCR to inhibitory substances[J]. Clin Chem, 2013, 59: 1670-1672. DOI:10.1373/clinchem.2013.211045 |
| [80] |
Dodd DW, Gagnon KT, Corey DR. Digital quantitation of potential therapeutic target RNAs[J]. Nucleic Acid Ther, 2013, 23: 188-194. DOI:10.1089/nat.2013.0427 |
| [81] |
Ferracin M, Lupini L, Salamon I, et al. Absolute quantification of cell-free microRNAs in cancer patients[J]. Oncotarget, 2015, 6: 14545-14555. DOI:10.18632/oncotarget.3859 |
| [82] |
Collins ML, Irvine B, Tyner D, et al. A branched DNA signal amplification assay for quantification of nucleic acid targets below 100 molecules/ml[J]. Nucleic Acids Res, 1997, 25: 2979-2984. DOI:10.1093/nar/25.15.2979 |
| [83] |
Zhou L, Cryan EV, Minor LK, et al. A branched DNA signal amplification assay to quantitate messenger RNA of human uncoupling proteins 1, 2, and 3[J]. Anal Biochem, 2000, 282: 46-53. DOI:10.1006/abio.2000.4587 |
| [84] |
Tsongalis GJ. Branched DNA technology in molecular diagnostics[J]. Am J Clin Pathol, 2006, 126: 448-453. DOI:10.1309/90BU6KDXANFLN4RJ |
| [85] |
Hou Y, Liang D, Peng L, 等. bDNA-based Alu quantitative assay of plasma cell-free DNA level in sepsis patients[J]. Lab Med (检验医学), 2012, 27: 1050-1053. |
| [86] |
Kullolli M, Knouf E, Arampatzidou M, et al. Intact microRNA analysis using high resolution mass spectrometry[J]. J Am Soc Mass Spectrom, 2014, 25: 80-87. DOI:10.1007/s13361-013-0759-x |
| [87] |
Needham SR, Valaskovic GA. Microspray and microflow LC-MS/MS: the perfect fit for bioanalysis[J]. Bioanalysis, 2015, 7: 1061-1064. DOI:10.4155/bio.15.42 |
 2021, Vol. 56
2021, Vol. 56


