2. 陕西中医药大学药学院, 陕西 咸阳 712046;
3. 咸阳师范大学化学与化工学院, 陕西 咸阳 712000
2. School of Pharmacy, Shaanxi University of Chinese Medicine, Xianyang 712046, China;
3. School of Chemistry and Chemical Engineering, Xianyang Normal University, Xianyang 712000, China
牛尾独活(Rhizoma et Radix Heraclei) 为地方性常用药材, 药材牛尾独活的来源记载并不统一, 有伞形科独活属植物短毛独活Heracleum moellendorffii Hance.、渐尖叶独活Heracleum acuminatum Franch.及独活Heracleum hemsleyanum Diels.的干燥根及根茎, 具有祛风除湿、散寒止痛的功效, 可用来治疗风湿痹痛、腰膝酸软等症[1, 2]。目前, 植物渐尖叶独活的名称已被修改为: 尖叶独活Heracleum franchetii[3]。药材牛尾独活基源较多且不明确, 在形态上容易与其他药材相混, 尤其在干燥或炮制之后。经查证, 市场上牛尾独活常常成为当归、独活以及羌活等药材的混用品[4-7]。当归、独活、羌活为我国常用中药, 用药历史悠久。药材当归(Radix Angelicae Sinensis) 为伞形科植物当归Angelica sinensis (Oliv.) Diels的干燥根, 具有补血活血、调经止痛、润肠通便之功效[8]; 药材独活(Radix Angelicae Pubescentis) 为伞形科植物重齿毛当归Angelica pubescens Maxim.f. biserrata Shan et Yuan的干燥根, 可以祛风除湿、通痹止痛[8]; 药材羌活(Rhizoma et Radix Notopterygii) 为伞形科植物羌活Notopterygium incisum Ting ex H.T.Chang或宽叶羌活Notopterygium franchetii H. de Boiss.的干燥根茎和根, 具有解表散寒、祛风除湿、止痛的功效[8]。牛尾独活与这些药材的功效主治不同, 不能作为这些药材的代用品。目前对于以上药材的研究主要集中在性状鉴别、显微鉴别、薄层色谱、液相色谱和化学成分等方面[5-7], 然而上述几种方法不能有效地应用于中药正品与伪品的鉴定实践中, 传统的鉴别方法还要求鉴别者具备系统的分类学知识和长期的鉴别经验, 为中药材的真伪鉴别增加了难度。因此对于药材牛尾独活以及其成为当归、独活和羌活混伪品的鉴定, 亟需一种快速、客观、准确的基于DNA水平的新方法。
伴随分子生物技术日益发展, 越来越多的新技术、新方法出现, 对传统的鉴别手段进行有效的补充。其中DNA条形码技术成为中药材鉴定的热门方法; DNA条形码鉴定技术是利用基因组上一段标准的、具有足够变异的、易扩增且相对较短的DNA序列来鉴别物种的一种分子生物学技术[9, 10]。ITS2作为DNA条形码中的一种, 是位于5.8S和28S rDNA片段之间的间隔区序列, 长度一般在200~300 bp, 适用于种属的鉴别[11, 12]。ITS2二级结构较为保守, 是由RNA单链自身回折形成的茎环结构, 配对碱基形成茎干, 未配对碱基则形成环[13]。ITS2二级结构不仅包含系统发育信息, 而且通过二级结构的预测可以避免或排除旁系同源或假基因对于系统发育树构建的误导。在物种鉴定研究时, ITS2二级结构信息是对一级结构的有效补充[14, 15]。因此本研究将基于ITS2一级结构和二级结构对中药材牛尾独活、当归、独活和羌活进行鉴别, 为临床安全用药提供理论指导, 为中药材DNA条形码的选择提供参考。
材料与方法材料来源 本研究材料包含牛尾独活、当归、独活和羌活共4种中药材。牛尾独活3种基源植物短毛独活、尖叶独活、独活的样本序列分别有3、3、4条。当归基源植物当归样本序列为5条。独活基源植物重齿毛当归样本序列为3条。药材羌活2种基源植物羌活和宽叶羌活的样本序列均为4条。所有样本序列均来自GenBank数据库, 详细信息见表 1。
| Table 1 GenBank download sequence information |
序列的获得、筛选和比对 从NCBI GenBank下载7种分类群, 即当归、重齿毛当归、短毛独活、尖叶独活、独活、羌活和宽叶羌活, 利用Blast功能筛除可疑序列, 共获得28条序列。应用ITS2 Database (http://its2.bioapps.Biozen trum.uni-wuerzburg.de/) 基于隐马尔可夫模型的HMMer[16]注释方法对数据进行分析, 去除两端5.8S和28S rDNA区段, 获得标准的ITS2片段。利用ClustalW软件将所有ITS2序列进行比对。利用ITS2 Database预测获得各物种的ITS2二级结构。
遗传距离、一级结构进化树构建 采用MEGA软件[17]分析比对所得序列, 基于K2P (Kimura 2-parameter) 算法计算种内、种间遗传距离, 构建NJ聚类树, Bootstrap 1 000次重复检验分支系统发育的可靠性。统计GC含量、变异位点、单倍型等序列特征信息。
一级和二级结构系统发育树的构建 利用ITS2 Database[18]获得核酸序列和二级结构的联合矩阵, 将所有样本的联合矩阵输入4Sale软件[19]中进行比对, 将比对好的结果导入ProfDIstS软件[20]构建剖面邻接PNJ (profile neighbor joining) 系统发育树, bootstrap 1 000次检验各分支置信度。
结果 1 序列比对本研究中26个样本的ITS2序列长度为224~230 bp, GC含量为52.9%~59.2%。MEGA5.2多重序列比对后, 所有样本ITS2序列长234 bp。药材牛尾独活的3种不同基源植物种间共存在8个变异位点, 短毛独活、尖叶独活、独活分别具有1种单倍型。药材当归基源即植物当归, 存在1种单倍型。药材独活的基源植物重齿毛当归3个样本共有6个变异位点, 获得3种单倍型。药材羌活的2种基源植物羌活和宽叶羌活分别有4个样本, 2个种种间变异位点数为14, 植物羌活的样本种内存在1个变异位点, 获得2种单倍型, 植物宽叶羌活样本种内有4个变异位点, 共获得3种单倍型(表 2)。
| Table 2 ITS2 sequences characteristics included in this study |
所有样本ITS2序列的种内遗传距离为0~0.018 3, 种内平均遗传距离0.004 2, 种间遗传距离为0.018 1~0.282 7, 种间平均遗传距离为0.182 4; 其中种间最小遗传距离为短毛独活和尖叶独活0.018 1, 小于种内最大距离0.018 3, 因此植物短毛独活和尖叶独活的区分需进一步分析, 各种的种内和种间遗传距离见表 3。如表 4所示将比对后的ITS2序列按药材分为当归、独活、牛尾独活和羌活共4组: 各组药材内遗传距离为0~0.030 6, 药材内平均距离为0.016 8, 药材间遗传距离为0.131 3~0.267 2, 药材间平均遗传距离为0.201 6, 药材间最小距离显著大于药材内最大遗传距离, 药材之间存在一个明显的“Barcoding Gap”。因此ITS2序列不仅可以区分开药材牛尾独活、当归、独活和羌活, 还可以区分药材的不同种基源。
| Table 3 Genetic distances of all samples |
| Table 4 Genetic distance of each traditional Chinese medicinal materials group |
基于ITS2序列(一级结构) 和基于ITS2一级结构与二级结构整合矩阵, 分别构建系统发育树, 所得拓扑关系基本一致。在基于ITS2序列构建的NJ邻接树如图 1A中显示, 药材牛尾独活、独活、当归和羌活分别聚为一支, 自展支持率分别为100%、99%、100%和100%, 4种药材可明显区分开。其中药材牛尾独活的3种基源植物短毛独活、尖叶独活和独活, 药材羌活的2种基源植物羌活和宽叶羌活, 分别形成独立的亚分支。如图 1B所示, 基于ITS2一级结构与二级结构联合矩阵所构建的进化树不仅包含了单独基于一级结构信息所得的结论, 而且末端分支分类水平的准确率和支系的支持率都有所提高。
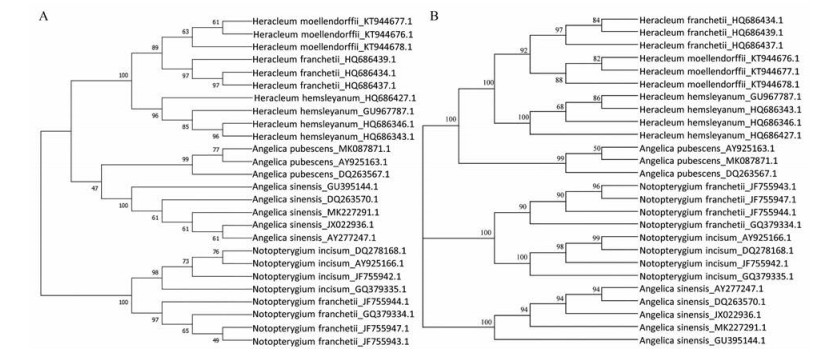
|
Figure 1 Phylogenetic tree of Rhizoma et Radix Heraclei, Radix Angelicae Sinensis, Radix Angelicae Pubescentis and Rhizoma et Radix Notopterygii. A: Neighbor-joining (NJ) tree was built based on the ITS2 sequence; B: Profile neighbor joining (PNJ) phylogenetic tree based on ITS2 sequence and secondary structure joint matrix. Bootstrap 1 000 duplicate test, displaying only 50% ≥ self-expanding support ratings |
药材牛尾独活、独活、当归和羌活的基源植物ITS2二级结构如图 2所示, 所有物种ITS2二级结构均呈“一环四臂”形态, 其中臂III最长。同时, 各个种间ITS2二级结构均有较大差异。牛尾独活基源短毛独活和尖叶独活均包含10个内环、4个多分支环、4个发卡环结构, 但二者内环位置、大小和分支环的大小不同; 第三种基源植物独活有11个内环、4个多分支环、4个发卡环结构, 相比前两种植物多出的1个内环在臂III上; 三种基源植物的相同之处是臂I和臂IV之间有一段单链结构。药材独活的ITS2二级结构与药材牛尾独活差别显著, 药材独活的基源重齿毛当归具有1个较大的分支环、6个内环、1突环、4个发卡环结构。药材羌活的两种基源宽叶羌活和羌活, 以及药材当归的ITS2二级结构均各不相同, 差异主要体现在主环大小、旋臂长度、旋臂间夹角、茎环数目大小及位置等方面。

|
Figure 2 ITS2 secondary structure of all species |
药材当归、独活、羌活为我国常用中药, 用药历史悠久。药材牛尾独活常常作为前三位药材的易混品出现在市场上。药材当归、独活、羌活的基源植物及药用部位已在2015版《中国药典》中被明确规定[8]。当归的用途广泛且需求量大, 优质当归供不应求。独活在各地习用品种较多, 各类古籍对独活来源的记载也并不一致[21]。药材羌活近年资源短缺, 现有资源采挖困难, 价格有逐年上升的趋势[22]。药材牛尾独活, 属地方习用品, 现并未被《中国药典》收录。而不同文献对牛尾独活的药用部位描述也并不完全一致[1]。传统的鉴定方法在牛尾独活、当归、独活和羌活的鉴别及用药安全方面存在一定局限性。ITS2作为牛尾独活、当归、独活和羌活的DNA条形码无疑是比较理想的选择。本研究结果显示牛尾独活、当归、独活和羌活药材之间的平均K2P遗传距离(0.201 6) 远大于药材内部的平均遗传距离(0.016 8), 并且各药材的基源植物种间平均遗传距离(0.182 4) 远大于种内平均遗传距离(0.004 2); 达到种间遗传距离超过种内遗传距离的10倍[23], 即已远远满足序列差异的标准阈值, Barcoding Gap显著存在于不同药材之间以及同一药材的不同基源植物之间。此外, 由系统发育树可以看出, 不仅不同药材可明显区分开, 而且药材的不同基源植物也独立分支。因此ITS2可以作为区分牛尾独活、当归、独活和羌活的DNA条形码, 为上述药材基源植物鉴定以及安全临床用药提供分子依据。
本文在ITS2一级核酸序列的基础上, 对每种药材的基源植物二级结构进行了预测, 并构建了基于联合ITS2一级序列与二级结构的系统进化树, 研究结果显示, 各个基源植物的二级结构符合大多数真核生物的一环四臂的拓扑结构特点。三种牛尾独活基源植物具有基本一致的二级结构, 但在内环数目、位置、大小和分支环的大小存在差异; 与其他种植物的二级结构相比较在主环大小、旋臂长度、旋臂间夹角、茎环数目及位置等存在较大差异, 正是因为这些差异为不同药材及其基源植物的鉴定提供了额外的系统分类信息。另外, 本研究中联合ITS2序列与二级结构共同构建的剖面邻接(PNJ) 发育树, 除了得到与单独ITS2序列构建的NJ树基本一致的拓扑结构外, 还对同一药材不同来源植物的亲缘关系得到了进一步的体现, 将其单独分为不同的支系, 使得系统发育树末端支系的支持率上有所提高(图 1)。因此, 在利用ITS2鉴定中药材物种时, 兼顾ITS2二级结构包含的信息位点, 将有效提升ITS2条形码的物种分辨率。本研究不仅为牛尾独活、当归、独活和羌活的鉴定提供新的方法与思路, 而且对中药材DNA条形码鉴定有重要的理论与实践价值。
作者贡献: 郑梦迪(第一作者兼通讯作者) 直接参与酝酿和设计实验, 实施研究, 分析、解释数据, 起草、撰写文章工作。孙咪咪参与实施研究, 采集、分析数据。贺紫涵参与数据采集, 统计分析工作。李怀珠参与审阅文章等指导工作。
利益冲突: 本文不存在任何利益冲突。文章内容不涉及相关利益冲突, 该研究未涉及任何厂家及相关雇主或其他经济组织直接或间接的经济或利益的赞助。
| [1] |
Institute of Materia Medica. Chinese Academy of Medical Sciences. Annals of Chinese Medicine (Volume 2) (中药志)[M]. Beijing: People's Medical Publishing House, 1982: 453, 459, 461.
|
| [2] |
Li YC, Lao JH, Wu PE. Primary Chromatogram of Genuine Drugs and Local Standard Drugs (道地药和地方标准药原色图谱)[M]. Chengdu: Sichuan Science and Technology Press, 2002: 155.
|
| [3] |
Editorial Board of Flora of China. Chinese Academy of Sciences. Flora of China (中国植物志)[M]. Beijing: Science Press, 1993.
|
| [4] |
Mu HY. Identification of Angelica pubescens and its adulterants[J]. J Pract Tradit Chin Med (实用中医药杂志), 2018, 34: 1537. |
| [5] |
Chen JF. The difference between Angelica sinensis and its adulterants[J]. Lab Med Clin (检验医学与临床), 2010, 7: 2554-2555. |
| [6] |
Li ZP. Identification of several common traditional Chinese medicines and their adulterants[J]. Chin J Clin Ration Drug Use (临床合理用药杂志), 2013, 6: 57-58. |
| [7] |
Zhou Y, Jin PF, Zhao YJ. Comparative identification of Angelicae Pubescentis Radix and its adulterants-roots of Heracleum Hemsleyanum Diels and roots of Heracleum Moellendorffii Hance[J]. Chin J Mod Appl Pharm (中国现代应用药学), 2013, 30: 863-866. |
| [8] |
Chinese Pharmacopoeia Committee. Pharmacopoeia of the People's Republic of China (中华人民共和国药典)[S]. Part 1. Beijing: China Medical Science Press, 2015: 302.
|
| [9] |
Hebert PDN, Cywinska A, Ball SL, et al. Biological identifications through DNA barcodes[J]. Proc Biol Sci, 2003, 270: 313-321. DOI:10.1098/rspb.2002.2218 |
| [10] |
Wu ZT, Zhao XH, Hu XY, et al. Application progress on DNA barcoding technology of traditional Chinese medicine[J]. Guid J Tradit Chin Med Pharm (中医药导报), 2019, 25: 125-130. |
| [11] |
Chen SL, Yao H, Han JP, et al. Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species[J]. PLoS One, 2010, 5: e8613. DOI:10.1371/journal.pone.0008613 |
| [12] |
Li DZ, Gao LM, Li HT, et al. Comparative analysis of a large dataset indicates that internal transcribed spacer (ITS) should be incorporated into the core barcode for seed plants[J]. Proc Natl Acad Sci U S A, 2011, 108: 19641-19646. DOI:10.1073/pnas.1104551108 |
| [13] |
Leontis NB, Westhof E. Geometric nomenclature and classification of RNA base pairs[J]. RNA, 2001, 7: 499-512. DOI:10.1017/S1355838201002515 |
| [14] |
Keller A, Förster F, Müller T, et al. Including RNA secondary structures improves accuracy and robustness in reconstruction of phylogenetic trees[J]. Biol Direct, 2010, 5: 4. DOI:10.1186/1745-6150-5-4 |
| [15] |
Prasad PK, Tandon V, Biswal DK, et al. Phylogenetic reconstruction using secondary structures and sequence motifs of ITS2 rDNA of Paragonimus westermani (Kerbert, 1878) Braun, 1899(Digenea: Paragonimidae) and related species[J]. BMC Gen, 2009, 10(Suppl 3): S25. |
| [16] |
Keller A, Schleicher T, Schultz J, et al. 5.8S-28S rRNA interaction and HMM-based ITS2 annotation[J]. Gene, 2009, 430: 50-57. DOI:10.1016/j.gene.2008.10.012 |
| [17] |
Kumar S, Stecher G, Tamura K. MEGA7:molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Mol Biol Evol, 2016, 337: 1870-1874. |
| [18] |
Ankenbrand MJ, Keller A, Wolf M, et al. ITS2 database V: twice as much[J]. Mol Biol Evol, 2015, 32: 3030-3032. DOI:10.1093/molbev/msv174 |
| [19] |
Seibel PN, Müller T, Dandekar T, et al. 4SALE-A tool for synchronous RNA sequence and secondary structure alignment and editing[J]. BMC Bioinf, 2006, 7: 498. DOI:10.1186/1471-2105-7-498 |
| [20] |
Wolf M, Ruderisch B, Dandekar T, et al. ProfdistS: (Profile-) distance based phylogeny on sequence-structure alignments[J]. BMC Bioinf, 2008, 24: 2401-2402. DOI:10.1093/bioinformatics/btn453 |
| [21] |
Zhu WJ, Ding XQ, Li Q, et al. Study on the botanical origin of Radix Angelicae Pubescentis[J]. Agric Sci Technol Inf (农业科技与信息), 2018(10): 20-23. |
| [22] |
Yao L, Yu J, Zhang Y, et al. Research progress and trend for Qianghuo (Notopterygii Rhizoma et Radix) in China[J]. Guid J Tradit Chin Med Pharm (中医药导报), 2020, 26: 93-98. |
| [23] |
Hebert PD, Stoeckle MY, Zemlak TS, et al. Identification of birds through DNA barcodes[J]. PLoS Biol, 2004, 2: e312. DOI:10.1371/journal.pbio.0020312 |
 2021, Vol. 56
2021, Vol. 56


