2. 西藏藏医药大学, 西藏 拉萨 850000;
3. 北京中医药大学, 北京 100029
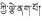 )基原植物之一,含有龙胆苦苷等环烯醚萜类活性成分,具有重要的药用价值。本文对粗茎秦艽转录组进行测序分析,分别构建其根、茎、叶、花转录组数据库,并对可能参与编码环烯醚萜苷类合成通路中关键酶的部分unigene进行qRT-PCR验证。结果如下:①共得到159 534条unigene,平均长度为679。根据GO功能分类,可将unigene分为3大类67分支;基于KOG功能可将unigene分为25类;② KEGG代谢通路分析发现粗茎秦艽中215条unigene参与到20个次生代谢标准通路中,有305条unigene参与编码环烯醚萜苷类合成通路中的28个关键酶,且在不同部位中的表达存在差异;③ qRT-PCR与RNA-Seq结果基本吻合,实验涉及的HMGS、DXS、MCS、GPPS、G10H、7-DLNGT及STR 7个基因皆为地上部位(茎、叶、花)的相对表达量高于地下部位(根)。环烯醚萜类为中药秦艽、龙胆、当药及青叶胆等药材的指标性成分及活性成分之一,本工作可为应用生物技术方法获取环烯醚萜类活性成分或其中间体,探讨药用植物次生产物累积规律、不同生态型品种的质量评价及药材道地性形成机制研究等提供基础科学资料。
)基原植物之一,含有龙胆苦苷等环烯醚萜类活性成分,具有重要的药用价值。本文对粗茎秦艽转录组进行测序分析,分别构建其根、茎、叶、花转录组数据库,并对可能参与编码环烯醚萜苷类合成通路中关键酶的部分unigene进行qRT-PCR验证。结果如下:①共得到159 534条unigene,平均长度为679。根据GO功能分类,可将unigene分为3大类67分支;基于KOG功能可将unigene分为25类;② KEGG代谢通路分析发现粗茎秦艽中215条unigene参与到20个次生代谢标准通路中,有305条unigene参与编码环烯醚萜苷类合成通路中的28个关键酶,且在不同部位中的表达存在差异;③ qRT-PCR与RNA-Seq结果基本吻合,实验涉及的HMGS、DXS、MCS、GPPS、G10H、7-DLNGT及STR 7个基因皆为地上部位(茎、叶、花)的相对表达量高于地下部位(根)。环烯醚萜类为中药秦艽、龙胆、当药及青叶胆等药材的指标性成分及活性成分之一,本工作可为应用生物技术方法获取环烯醚萜类活性成分或其中间体,探讨药用植物次生产物累积规律、不同生态型品种的质量评价及药材道地性形成机制研究等提供基础科学资料。2. Tibetan Traditional Medical College, Lhasa 850000, China;
3. Beijing University of Chinese Medicine, Beijing 100029, China
粗茎秦艽Gentiana crassicaulis Duthie ex Burk.隶属于龙胆科Gentianaceae龙胆属秦艽组(Sect. Cruciata), 我国特有物种, 自然分布区涉及西藏、云南、四川、贵州、青海及甘肃等省区, 多见于海拔2 100~4 500 m的高山草甸、林下及林缘[1]。该植物具有重要的药用价值, 作为中药秦艽的4种基原植物之一, 根可祛风湿, 清湿热, 止痹痛, 退虚热[2]; 同时, 在传统藏医药学中, 其根或花以“解吉那保”(
环烯醚萜类(iridoids) 广泛分布于龙胆科(Gentianaceae) 及玄参科(Scrophulariaceae) 等植物中, 具有抗病毒、抗炎、抗氧化、抗肿瘤、抗菌及保肝等多种药理作用[5-7]。其中龙胆苦苷、马钱苷酸、当药苷及獐牙菜苦苷等常作为中药秦艽、龙胆、当药及青叶胆等药材的指标性成分, 如2020版《中华人民共和国药典》秦艽项下“龙胆苦苷和马钱苷酸”的含量测定等[2]。
药用植物转录组作为现今研究药用植物基因组最活跃的领域之一, 可从整体水平了解细胞中基因表达情况及其调控规律[8]。通过转录组测序可以在没有参考基因组的情况下发现与活性成分密切相关的功能基因, 为研究中药原植物生长发育及次生代谢的分子机制提供基因资源文库[9]。环烯醚萜类具有多样的药理活性, 同时也是中药秦艽等的重要指标性成分。但目前尚不清楚秦艽、龙胆、当药及青叶胆等药材中环烯醚萜类成分的代谢通路及调控机制, 无法科学阐明药材品质形成的内在生物学规律。对转录组的研究将有助于探讨基因进化、生物转化及药材道地性形成机制等诸多方面的问题, 并为种质资源评价、遗传育种及药材栽培学研究等工作提供新思路, 具有重要的科学意义。
课题组前期已开展藏药“解吉那保”品种整理与分类学鉴定等工作, 测定粗茎秦艽近缘物种长梗秦艽Gentiana waltonii Burk.和粗壮秦艽Gentiana robusta King ex Hook. f.转录组序列, 并进行环烯醚萜苷类相关基因的初步挖掘[10-15], 在此基础上, 拟进一步开展粗茎秦艽G. crassicaulis转录组及其环烯醚萜类生物合成相关基因分析与qRT-PCR验证工作, 深入解析秦艽组植物环烯醚萜类生物合成通路及表达模式, 寻找物种DNA分子标记, 以期为粗茎秦艽药材客观、准确的品种鉴定, 种质资源评价, 龙胆科药用植物次生产物累积规律、不同生态型品种的质量评价及药材道地性形成机制研究等提供科学资料。
材料与方法样品采集及分类学鉴定 采集日期: 2019-07-30; 西藏波密县, 地理坐标N 29°47.94′, E95°49.382′, 海拔2 841 m; 采集号: 赵志礼, 倪梁红2019XZ04 (图 1)。取正常生长无病害植株, 连土采挖后鲜活状态下运至实验室, 纯水清洗, 无菌吸水纸吸干表面水分, 迅速将根、茎、叶、花各部位分别切成小块, 液氮速冻, -80 ℃保存待RNA提取。原植物鉴定为龙胆科龙胆属Gentiana秦艽组(Sect. Cruciata) 粗茎秦艽Gentiana crassicaulis Duthie ex Burk.; 凭证标本存放于上海中医药大学中药学院药物植物标本室。

|
Figure 1 Gentiana crassicaulis (Zhao ZL, et al. 2019XZ04, alt. 2 841 m, Bomi, Tibet). A: Habit; B: Inflorescence; C: Flower. Scale bar = 1 mm |
RNA提取分离、文库构建和转录组测序 Total RNA Extractor (Trizol) 试剂盒(生工, 中国上海) 分别提取粗茎秦艽样品根、茎、叶、花的总RNA, 检测RNA浓度及完整性。磁珠法分离mRNA, 将mRNA片段化, 完成双链cDNA合成、末端修复、末端dA-Tailing、接头连接、连接产物纯化和片段大小分选、文库扩增(15个循环)。回收DNA后精确定量, 按数据比例混合上机。在Illumnina HiseqTM X Ten测序平台进行2×150 bp测序。
测序数据处理及生物信息学分析 原始图像数据文件经碱基识别(base calling) 分析转化为原始测序序列(sequenced reads), 以“fastq”文件格式存储。对原始数据质量值等信息进行统计, 并使用FastQC 0.11.2软件对样本的测序数据质量进行可视化评估, 使用Trimmomatic 0.36软件[16]过滤除去含有带接头的、低质量的序列, 得到clean数据。使用Trinity 2.4.0软件将clean数据de novo组装成转录本(transcript)[17], 对转录本去冗余, 取每个转录本聚类中最长的转录本作为unigene, 以此作为后续分析的参考序列。
转录数据使用NCBI Blast+ 2.60软件(e-value < 1×10-5)[18]将unigene序列与CDD、Nr、Nt、Swiss-Prot、KOG、Pfam、TrEMBL 7个数据库比对, 进行功能注释和分类; 再对unigene序列进行GO (gene ontology) 功能注释和分类, 并使用KAAS 2.1软件[19]与KEGG (Kyoto encyclopedia of genes and genomes) 数据库进行比对, 分析相关代谢通路。
环烯醚萜类生物合成基因筛选 参考文献中环烯醚萜苷类化合物的生物合成代谢通路[20-22], 结合9个数据库注释结果, 挖掘本转录组中与环烯醚萜苷类合成相关的unigene, 以常用的基因表达水平估算方法中TPM (transcripts per million) 进行表达量统计。一个基因表达水平的直接体现是其转录本的丰度情况, 转录本丰度越高, 则基因表达水平越高。不采用RPKM值[23]。
qRT-PCR (quantitative real-time PCR) 定量表达分析与验证 TPM值分析后, 从环烯醚萜生物合成途径上、中、下游分别挑选部分基因的unigene, 以β-tubulin为内参基因, Primer Premier 5设计引物用于qPCR反应。cDNA合成试剂盒: PrimeScriptTM RT reagent Kit with gDNA Eraser (Perfect Real Time) (TaKaRa, 中国大连); qPCR反应: 试剂盒为TB Green® Premix Ex TaqTM (Tli RNaseH Plus) (TaKaRa, 中国大连), 仪器为ABI StepOnePlus Real-Time System(Thermo Fisher, 美国)。以根为参考部位, Livak法(2-ΔΔCT法)[24]进行根、茎、叶、花4个部位的相对表达量验证; StepOne Software v2.3软件进行结果分析。共3次生物学重复, 每次进行技术性重复3次。
结果与分析 1 转录组数据组装及样本间相关性粗茎秦艽根、茎、叶和花4个部位共12个样品(每个部位3个生物学重复) 分别获得根41 820 380、42 872 292、40 940 202, 茎40 864 200、42 263 446、41 228 880, 叶41 039 970、42 358 876、41 387 782, 花44 693 684、43 646 718、42 372 014个高质量reads片段。序列上传SRA数据库, 登录号: PRJNA682437。将质控后得到的所有高质量序列进行de novo拼接。共拼接得到粗茎秦艽unigene 159 534条, 平均长度为679 bp (表 1)。3个生物学重复样本数据之间相似程度以样本间距离热图表示(图 2), 由图可见, 各样本间数据相似程度高。
| Table 1 Summary of the transcript statistics generated from G. crassicaulis |

|
Figure 2 Heatmap of distance between samples. The color block represents the distance value. The grayer the color is, the closer the distance between samples is, and the higher the similarity is. The darker the yellow color is, the farther the distance is |
将unigene与Nr、Nt、GO、KEGG、Swiss-Prot及KOG等9个数据库通过NCBI Blast+ 2.60进行比对与注释, 粗茎秦艽有46.60%的unigene在至少一种数据库中得到注释(表 2)。
| Table 2 Unigene functional annotation |
Unigene在Nr数据库比对时, 同源性最高的物种为中粒咖啡Coffea canephora (22.82%); 其次是葡萄Vitis vinifera (5.94%)、芝麻Sesamum indicum (4.31%) 和大麦Hordeum vulgare (7.08%); 其余均小于3%。
共有59 229条、27 969条unigene在GO、KOG数据库中分别得到成功注释, 注释到的类型与占比与近缘种长梗秦艽Gentiana waltonii和粗壮秦艽Gentiana robusta的相关数据类似[14]。
与长梗秦艽、粗壮秦艽数据相比, 粗茎秦艽unigene参与的KEGG代谢通路可分别分为5个分支, 多出了一个分支: 有机过程(organismal systems) (图 3)。包括33个中类、298条代谢通路, 获得注释量前5的通路也略有不同: 为核糖体(ribosome) 313条(2.05%), 胞吞作用(endocytosis)芳239条(1.57%), PI3K-Akt信号通路237条(1.55%), 嘌呤代谢(purine metabolism) 187条(1.23%), 碳代谢(carbon metabolism) 182条(1.19%); 其余主要富集于RNA转运(RNA transport)、MAPK信号通路(MAPK signaling pathway)、内质网蛋白加工(protein processing in endoplasmic reticulum) 等代谢通路。高等植物含有多种次生代谢产物, 与其药用功效密切相关。在粗茎秦艽转录组中发现215条unigene参与到20个KEGG次生代谢标准通路中, 其中, 条数最多的通路为烟酸和烟酰胺代谢(nicotinate and nicotinamide metabolism), 有36条。其次为萜类化合物骨架合成(terpenoid backbone biosynthesis), 有31条(表 3)。
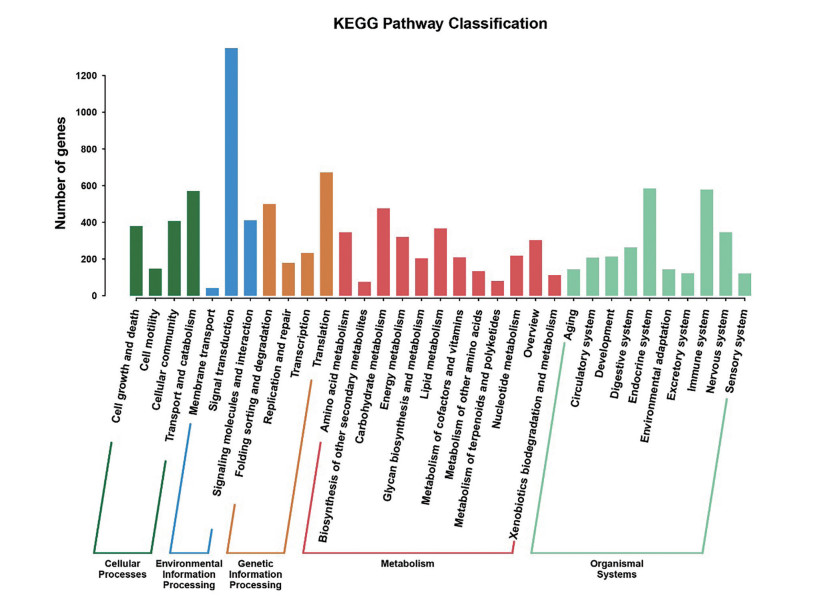
|
Figure 3 KEGG classification of assembled unigenes |
| 表 3 Secondary metabolism KEGG pathway analysis of transcriptomic unigenes |
环烯醚萜类化合物的生物合成代谢通路可通过中间体的生成、环烯醚萜类骨架合成及修饰等步骤完成[20-22]。分析结果显示: 粗茎秦艽转录组中有305条unigene参与编码其中28个关键酶(表 4)。相关基因在根、茎、叶、花中的表达量用热图表示(TPM值为标尺), 3个生物学重复的数据取算术平均数(图 4)。
| Table 4 Unigenes involved in biosynthesis of iridoids |
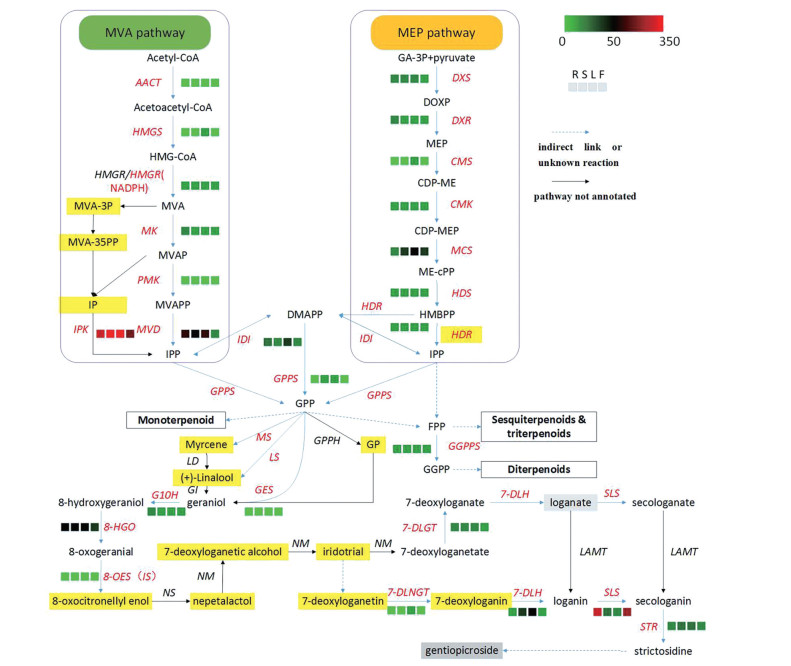
|
Figure 4 Putative pathways for biosynthesis of iridoids. Genes annotated in this study are shown in red color. The expression levels of unigenes in the root (R), stem (S), leaf (L) and flower (F) is shown by heatmap, mapped using TPM values. Boxes highlighted in yellow show the new putative pathways in the paper |
现有研究表明, 种类繁多、结构多样的萜类化合物都起始于共同的前体异戊烯基焦磷酸(isopentenyl diphosphate, IPP) 和二甲基丙烯基焦磷酸(dimethylallyl diphosphate, DMAPP)。其生成途径有两条, 一条为在细胞质中进行的甲羟戊酸(mevalonic acid, MVA) 途径, 关键酶有乙酰-CoA乙酰基转移酶(AACT)、HMG-CoA合酶(HMGS)、HMG-CoA还原酶(HMGR)、MVA激酶(MK)、MVAP脱羧酶、异戊烯基磷酸(IP) 激酶(IPK)、MVP激酶(PMK) 及MVAPP脱羧酶(MVD); 另一条为在质体中进行的2-C-甲基-D-赤藓糖醇-4-磷酸(2-C-methyl-D-erythritol-4-phosphate, MEP) 途径, 关键酶包括1-脱氧-D-木酮糖-5-磷酸合酶(DXS)、1-脱氧-D-木酮糖-5-磷酸还原异构酶(DXR)、2-C-甲基-D-赤藓糖醇-4-磷酸胞苷酰基转移酶(CMS)、4-(胞苷-5'-二磷酸基)-2-C-甲基-D-赤藓糖醇激酶(CMK)、2-C-甲基-D-赤藓糖醇-2, 4-环焦磷酸合酶(MCS)、4-羟基-3-甲基-2-丁烯基焦磷酸合酶(HDS) 及4-羟基-3-甲基-2-丁烯基焦磷酸还原酶(HDR); 两条途径的产物IPP和DMAPP之间的可逆转换由IPP δ异构酶(isopentenyl-diphosphate δ-isomerase, IDI) 来完成[25]。在粗茎秦艽转录组中, 除MVA途径中的一条支路未被注释完全外(图 4黑色箭头), 其他以上相关基因均有注释。未被注释到的支路主要生成含3, 5-二磷酸基的中间体, 然后由对应的脱羧酶催化脱去羧基和磷酸基生成IP, 或是由MVAP脱羧生成IP, 最后IP在IPK的催化下生成IPP; 而注释到的支路生成含5-焦磷酸基的产物, 即两条支路的重要不同点在于磷酸基加成的位置不同。结果提示, 粗茎秦艽的MVA合成路径选择可能是单一的, 磷酸基不加在C-3位。查阅文献知, 未被注释全的途径是从古菌(archaea) 中发现的[26], 与绿色植物(Viridiplantae) 亲缘关系“极远”, 故有理由认为粗茎秦艽不采用此通路进行MVP→IP的合成; 但这个途径中后续IPK又得到了注释。IPP主要由MEP途径生成, 研究对象为长春花Cantharanthus roseus[20]。本文通过分析, 基于TPM值, MVP与MEP两个途径皆有基因表达, 且表达量并无明显差异, 提示两个途径(发生场所分别位于细胞质与质体) 都能产生IPP, IPP的合成路径选择并非有偏好性。基于TPM值统计, 以上基因表达量存在较明显的差异, 其中MVA途径中的MVD, MEP途径中的MCS表达量相对较高。值得注意的是, IPK表达量非常高。
3.2 环烯醚萜的骨架合成IPP和DMAPP在香叶基焦磷酸合成酶(GPPS) 催化下缩合成香叶基焦磷酸(geranyl diphosphate, GPP), GPP是重要的分界点, 不同的萜类合成自GPP开始通过不同的代谢方向流向单萜、二萜、三萜等合成通路。对于环烯醚萜的合成(图 4), 首先, GPP通过三条路径脱去焦磷酸基生成香叶醇(geraniol), 第一条路径为: GPP在月桂烯合酶(myrcene synthase, MS) 的催化下脱去焦磷酸基生成月桂烯, 接着在芳樟醇脱水酶(linalool dehydratase, LD) 的作用下加水生成S-(+)-芳樟醇, 同时, 此产物也可直接由GPP在S-芳樟醇合酶(S-linalool synthase, LS) 催化下脱去焦磷酸基得到; 然后S-(+)-芳樟醇在香叶醇异构酶(geraniol isomerase, GI) 的催化下生成香叶醇。第二条路径为: GPP在GPP水解酶(geranyl diphosphate phosphohydrolase, GPPH) 催化下脱去一分子磷酸生成香叶基磷酸(geranyl phospate, GP), 然后GP脱去磷酸基转化成香叶醇, 但其关键酶暂时未知[27]。第三条路径为: GPP在香叶醇合酶(geraniol synthase, GES) 的催化下水解生成香叶醇和焦磷酸。只有第三条路径的相关基因全部得到了注释, 另外两条途径未被注释完全。然后, 香叶醇在香叶醇8-羟化酶(geraniol 8-hydroxylase, G10H) (多数文献写为10-羟化酶, 为不符合IUPAC命名法的讹误[28])、8-羟基香叶醇氧化还原酶(8-hydroxy-geraniol oxidoreductase, 8-HGO)、8-羰基香茅基烯醇合酶(8-oxocitronellyl enol synthase, IS) 催化下经过多步反应, 最终在nepetalactol合酶(nepetalactol synthase, NS) 催化下由链状单萜生成环状单萜nepetalactol。8-HGO、8-羰基香茅基烯醇合酶、NM涉及的三步反应为一个酶催化, 称之为环烯醚萜合酶(iridoid synthase, IS)。最新研究表明, IS实际上已被拆分为8-羰基香茅基烯醇合酶和NS, 并且发现了新的关键酶NS[29]。此后, nepetalactol在nepetalactol单加氧酶(nepetalactol monooxygenase, NM) 的作用下经过两步反应氧化、异构生成琉蚁二醛(iridodial)[30], 自此, 得到环烯醚萜的骨架。8-羰基香叶醛(8-oxogeranial) 在环烯醚萜合酶(IS) 一个酶的催化下生成琉蚁二醛[14], 中间步骤虽有烯醇化的中间体[20], 但总体不甚明了。本文根据最新文献[29, 30], 挖掘出这个步骤的3个关键酶, 使合成通路更加明确。
3.3 修饰——环烯醚萜苷的合成环烯醚萜主要成苷存在于植物体内。琉蚁二醛在NM催化下氧化为7-去氧马钱苷酸苷元(7-deoxyloganetate), 通过暂时未明确的通路生成7-去氧马钱苷苷元(7-deoxyloganetin); 两者分别通过各自的葡萄糖基转移酶(7-deoxyloganetic acid glucosyltransferase, 7-DLGT; 7-deoxyloganetin glucosyltransferase, 7-DLNGT) 生成对应的糖苷[31], 接着共同在7-去氧马钱苷酸7-羟化酶(7-deoxyloganate 7-hydroxylase, 7-DLH) 催化下生成产物马钱苷酸(loganic acid) 和马钱苷(loganin), 前者可由马钱苷酸O-甲基转移酶(loganic acid O-methyltransferase, LAMT) 催化生成后者; 紧接着二者在裂环马钱苷合酶(secologanin synthase, SLS) 催化下开环。最后, 裂环马钱苷和色胺(tryptamine) 在异胡豆苷合酶(strictosidine synthase, STR) 催化下生成异胡豆苷, 然后经过暂不明确的后续反应生成龙胆苦苷(gentiopicroside)。
在粗茎秦艽转录组中, LD、GI、GPPH、NS、NM、LAMT未被注释到。其他相关基因均有不同数量的unigene成功注释。基于TPM平均值统计, 表达量较高的为根、茎、叶中的IPK; 4个部位的DXR; 根和花中的SLS。本文得到的通路与环烯醚萜类经典的代谢通路大致相同, 为中间体生成、环烯醚萜类骨架合成、修饰3个步骤。粗茎秦艽转录组中有305条unigene参与编码其中28个关键酶, 与之前报道的近缘种长梗秦艽、粗壮秦艽的80条、57条相比[14], 由于挖掘的数据库更多, 资料得到了补充。就不同器官的表达情况看, 各基因在根、茎、叶、花不同部位的表达量具有较明显的差异, 情况较为复杂。有的基因在根、花中表达较高, 茎、叶中低, 如SLS; 有的基因恰好相反, 茎、叶中表达量较高, 如MCS, 有的基因4个部位表达量相差不大, 如MVD、IDI等。
4 部分基因的qRT-PCR验证RNA-Seq数据计算是以数学方法模拟计算表达量, 筛选的基因较多, 体现的是整体趋势准确, 具体到每个基因, 准确性没有那么高, 从mean ± SD值也可看出各样本间离散程度大; 而qRT-PCR是不同原理的实验性方法, 是对特异基因表达量的检测, 特异性相对更高。两种方法互相验证后结果会更具说服力。
RNA-Seq数据计算结果(TPM值) (图 5) 与实际测定结果(相对表达量, RQ, relative quantity) (图 6) 皆使用3个生物学重复的算术平均数计算log2值。结果表明, qRT-PCR与RNA-Seq结果基本吻合, HMGS、DXS、MCS、GPPS、G10H、7-DLNGT及STR 7个基因皆为地上部位(茎、叶、花) 的相对表达量高于地下部位(根); 证实了RNA-Seq数据的可靠性。
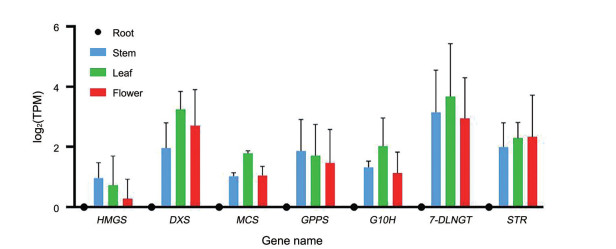
|
Figure 5 RNA-Seq data of DEGs related to biosynthesis of iridoids pathways. Results were mean ± SD for three individual experiments which, for each condition, were performed in triplicate. The mean ± SD of each gene according to the order of stem, leaf and flower (The root as the reference site is equal to 0): HMGS: 0.97 ± 0.51, 0.73 ± 0.97, 0.28 ± 0.65; DXS: 1.96 ± 0.84, 3.24 ± 0.60, 2.71 ± 1.20; MCS: 1.03 ± 0.12, 1.79 ± 0.09, 1.05 ± 0.31; GPPS: 1.87 ± 1.04, 1.71 ± 1.04, 1.47 ± 1.12; G10H: 1.32 ± 0.20, 2.02 ± 0.94, 1.13 ± 0.69; 7-DLNGT: 3.15 ± 1.40, 3.67 ± 1.75, 2.95 ± 1.35; STR: 2.00 ± 0.80, 2.29 ± 0.52, 2.34 ± 1.38 |
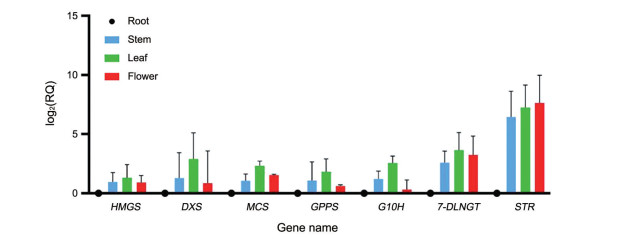
|
Figure 6 qRT-PCR validation of DEGs related to biosynthesis of iridoids pathways. Results were mean ± SD for three individual experiments which, for each condition, were performed in triplicate. The mean ± SD of each gene according to the order of stem, leaf and flower (The root as the reference site is equal to 0): HMGS: 0.97 ± 0.79, 1.31 ± 1.12, 0.92 ± 0.59; DXS: 1.30 ± 2.13, 2.90 ± 2.21, 0.85 ± 2.72; MCS: 1.07 ± 0.56, 2.32 ± 0.40, 1.54 ± 0.06; GPPS: 1.08 ± 1.58, 1.82 ± 1.09, 0.61 ± 0.10; G10H: 1.22 ± 0.65, 2.56 ± 0.59, 0.32 ± 0.81; 7-DLNGT: 2.59 ± 0.98, 3.64 ± 1.49, 3.25 ± 1.58; STR: 6.46 ± 2.16, 7.24 ± 1.90, 7.63 ± 2.34 |
本文首次测定粗茎秦艽G. crassicaulis的转录组序列, 并对其环烯醚萜类生物合成相关基因进行深入分析与qRT-PCR验证, 获得新的资料。根据KEGG数据库注释到20个次生代谢通路的unigene, 其中条数最多的通路为烟酸和烟酰胺代谢, 但近缘种长梗秦艽、粗壮秦艽中[14]皆为苯丙素生物合成; 粗茎秦艽不仅通路不同且对应的苯丙素生物合成unigene数量较少。苯丙素类化合物研究相对较少, 对于植物自身的调节作用更是鲜为人知。同时。长梗秦艽、粗壮秦艽及粗茎秦艽均有各自不同的自然分布区[1], 且粗壮秦艽为一自然杂交种[32]。不同物种、不同生境的影响以及植物对环境的适应性等, 可能导致基因有不同的表达[33, 34]。
就unigene而言, 与长梗秦艽、粗壮秦艽的相关报道相比较, 粗茎秦艽unigene数目较多。是否为样本量较大(每个部位平行3个样本), 导致尚未得到注释的部分增多?长度较短未得到有效注释、非编码序列或新的未知功能基因的存在等?有待进一步研究[35]。
环烯醚萜类作为一类龙胆科等来源的药用植物活性成分及指标性成分, 其经典通路研究工作多来自于长春花Cantharanthus roseus[20-22]。本文参考9个数据库(CDD、KOG、Nt、Nr、Swiss-Prot、Pfam、TrEBML、GO、KEGG) 进行基因挖掘, 发现MVP→IPP, GPP→香叶醇, 琉蚁二醛→马钱苷三条合成通路有支路。其中某条支路或多或少有未被注释到的基因, 支路未被“打通”; 其中, LAMT仍未被注释到。推测原因: ①如NS是2019年新发表成果, 测序时还没有相应数据; ②与植物的生长发育状态有关[35], 取样的时间不同导致基因表达情况不同; ③居群生存环境胁迫; ④物种间差异; ⑤有新的合成通路或酶; ⑥相关基因表达水平可能低于Illumina HiSeq XTM Ten平台的检测限[36]; ⑦相关基因与龙胆科植物中基因同源性很低等。同时, 龙胆苦苷等后续裂环环烯醚萜苷的合成通路仍旧未明, 认识停留在前体异胡豆苷。对于尚未注释的基因, 可结合代谢组学研究, 筛选诱导子, 刺激植物产生相应化合物, 从而通过对转录组的比较分析来发现相关基因。
秦艽类中药的药用部位为根, 但在传统藏医药中, 藏药“解吉那保”基原植物, 其地上部位(茎、叶、花) 也作为用药部位[3]。有文献报道解吉类药材的地上部位质量可能更优[10]。本文在环烯醚萜类合成通路上游挑选了MVA和MEP的起始步骤关键酶基因HMGS、DXS, 中游“中间体合成”与“骨架合成”相关关键步骤GPPS、GES、G10H基因, 马钱苷酸与龙胆苦苷(终产物) 合成前段7-DLNGT、STR基因进行了qRT-PCR验证实验, 尝试对导致此现象的原因进行初步探究。结合qRT-PCR与RNA-Seq的结果, 部分有代表性的环烯醚萜类生物合成相关基因在地上部位有较高表达, 提示地上部分的质量可能优于地下部分。植物不同器官间环烯醚萜类的表达量可能有所不同[37], 如何结合不同部位次生产物合成及积累特点进行新药用部位的开发研究, 合理利用有限的药用植物资源, 同样值得深入探究。
龙胆属为龙胆科第一大属, 国产类群近250种, 大部分物种分布于西南高山地区[1], 具有重要的药用价值。就目前已报道的龙胆属植物转录组分析工作而言, 仅仅是起步阶段, 有必要开展更为广泛的相关研究。同时, 植物不同器官的环烯醚萜类等次生产物表达量亦受到植物生长发育阶段及所处生态环境等因素的影响, 转录组测序、qRT-PCR验证与同时测定各器官龙胆苦苷等环烯醚萜类成分实际含量相结合, 将为进一步揭示植物环烯醚萜类次生产物累积规律提供更为全面的科学证据。
致谢: 分子生物学部分实验得到上海中医药大学中药研究所陈敏同学的帮助, 特此感谢。
作者贡献: 康恒是本文的第一作者, 负责分子生物学实验及稿件撰写; 倪梁红负责样品采集工作; 李尉涛参与数据分析工作; 赵淑娟参与分子生物学部分实验指导; 刘铜华参与研究工作总体设计; 赵志礼为本文的通讯作者, 负责样品分类学鉴定, 研究工作总体设计及稿件修改等工作。
利益冲突: 本文的研究内容无任何利益冲突。
| [1] |
Ho TN. Gentianaceae in Flora Reipublicae Popularis Sinicae: Vol 62 (中国植物志: 62卷)[M]. Beijing: Science Press, 1988: 14-75.
|
| [2] |
State Pharmacopoeia Commission. Pharmacopoeia of the People's Republic of China: Vol I (中华人民共和国药典: 一部)[S]. Beijing: China Medical Science Press, 2020: 99, 100, 140, 204, 205, 282.
|
| [3] |
Yang YC. Tibetan Medicine (藏药志)[M]. Xining: Qinghai People Press, 1991: 11-12.
|
| [4] |
Zhou XJ, Xu HF, Shun QS. Resource Science of Chinese Medicinal Materials (中药资源学)[M]. Shanghai: Shanghai Scientific and Technological Literature Publishing House, 2007: 370.
|
| [5] |
Ma LN, Tian CW, Zhang TJ, et al. Advances in study on iridoids in plants of Swertia L. and their pharmacological activity[J]. Chin Tradit Herb Drugs (中草药), 2008, 39: 790-795. |
| [6] |
Zheng LS, Liu XQ. Advances in the research of iridoids[J]. Nat Prod Res Dev (天然产物研究与开发), 2009, 21: 702-711, 725. |
| [7] |
Zheng HR, Chu Y, Li W, et al. Research progress on pharmacokinetics of natural iridoids[J]. Drug Eval Res (药物评价研究), 2018, 41: 1147-1153. |
| [8] |
Qi YX, Liu YB, Rong WH. RNA-Seq and its applications: a new technology for transcriptomics[J]. Hereditas, 2011, 33: 1191-1202. |
| [9] |
Wang YL, Huang LQ, Yuan Y, et al. Research advances on analysis of medicinal plants transcriptome[J]. China J Chin Mater Med (中国中药杂志), 2015, 40: 2055-2061. |
| [10] |
Zhao ZL, Gaawe Dorje, Wang ZT. Identification of medicinal plants used as Tibetan traditional medicine Jie-Ji[J]. J Ethnopharmacol, 2010, 132: 122-126. DOI:10.1016/j.jep.2010.07.051 |
| [11] |
Ni LH, Zhao ZL. A morphometric comparison of three closely related species of Gentiana (Gentianaceae), endemic to the region of the Qinghai-Tibet Plateau[J]. Botany, 2018, 96: 209-215. DOI:10.1139/cjb-2017-0166 |
| [12] |
Ni LH, Zhao ZL, Xu HX, et al. Chloroplast genome structures in Gentiana (Gentianaceae), based on three medicinal alpine plants used in Tibetan herbal medicine[J]. Curr Genet, 2017, 63: 241-252. DOI:10.1007/s00294-016-0631-1 |
| [13] |
Ni LH, Zhao ZL, Xiong B, et al. A strategy for identifying six species of Sect. Cruciata (Gentiana) in Gansu using DNA barcode sequences[J]. Acta Pharm Sin (药学学报), 2016, 51: 821-827. |
| [14] |
Ni LH, Zhao ZL, Wu JR, et al. Analysis of transcriptomes to explore genes contributing to iridoid biosynthesis in Gentiana waltonii and Gentiana robusta (Gentianaceae)[J]. Acta Pharm Sin (药学学报), 2019, 54: 944-953. |
| [15] |
Zong FF, Zhao ZL, Ni LH, et al. DNA fingerprinting and genetic diversity in a threatened alpine medicinal plant, Gentiana crassicaulis (Gentianaceae) using AFLP[J]. Acta Pharm Sin (药学学报), 2020, 55: 1941-1950. |
| [16] |
Bolger AM, Lohse M, Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data[J]. Bioinformatics, 2014, 30: 2114-2120. DOI:10.1093/bioinformatics/btu170 |
| [17] |
Haas BJ, Papanicolaou A, Yassour M, et al. De novo transcript sequence reconstruction from RNA-seq using the Trinity platform for reference generation and analysis[J]. Nat Protoc, 2013, 8: 1494-1512. DOI:10.1038/nprot.2013.084 |
| [18] |
Altschul SF, Madden TL, Schäffer AA, et al. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs[J]. Nucleic Acids Res, 1997, 25: 3389-3402. DOI:10.1093/nar/25.17.3389 |
| [19] |
Moriya Y, Itoh M, Okuda S, et al. KAAS: an automatic genome annotation and pathway reconstruction server[J]. Nucleic Acids Res, 2007, 35: W182-W185. DOI:10.1093/nar/gkm321 |
| [20] |
Contin A, Heijden RVD, Lefeber AWM, et al. The iridoid glucoside secologanin is derived from the novel triose phosphate/pyruvate pathway in a Catharanthus roseus cell culture[J]. FEBS Lett, 1998, 434: 413-416. DOI:10.1016/S0014-5793(98)01022-9 |
| [21] |
Wu XY, Liu XL. Progress of biosynthetic pathway and the key enzyme genes of iridoids[J]. Chin J Ethnomed Ethnopharm (中国民族民间医药), 2017(08): 44-48. |
| [22] |
Yang R, Fang L, Li J, et al. Research progress on biosynthetic pathways and related enzymes of iridoid glycosides[J]. Chin Tradit Herb Drugs (中草药), 2018, 49: 2482-2488. |
| [23] |
Wagner GP, Kin K, Lynch VJ. Measurement of mRNA abundance using RNA-seq data: RPKM measure is inconsistent among samples[J]. Theory Biosci, 2012, 131: 281-285. DOI:10.1007/s12064-012-0162-3 |
| [24] |
Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT Method[J]. Methods, 2001, 25: 402-408. DOI:10.1006/meth.2001.1262 |
| [25] |
Capell T, Christou P. Progress in plant metabolic engineering[J]. Curr Opin Biotechnol, 2004, 15: 148-154. DOI:10.1016/j.copbio.2004.01.009 |
| [26] |
Vinokur JM, Korman TP, Cao Z, et al. Evidence of a novel mevalonate pathway in archaea[J]. Biochemistry, 2014, 53: 4161-4168. DOI:10.1021/bi500566q |
| [27] |
Magnard JL, Roccia A, Caissard JC, et al. Biosynthesis of monoterpene scent compounds in roses[J]. Science, 2015, 349: 81-83. DOI:10.1126/science.aab0696 |
| [28] |
IUPAC-IUB Joint Commission on Biochemical Nomenclature (JCBN). Prenol nomenclature[J]. Eur J Biochem, 1987, 167: 181-184. DOI:10.1111/j.1432-1033.1987.tb13321.x |
| [29] |
Lichman BR, Kamileen MO, Titchiner GR, et al. Uncoupled activation and cyclization in catmint reductive terpenoid biosynthesis[J]. Nat Chem Biol, 2019, 15: 71-79. DOI:10.1038/s41589-018-0185-2 |
| [30] |
Miettinen K, Dong L, Navrot N, et al. The seco-iridoid pathway from Catharanthus roseus[J]. Nat Commun, 2014, 5: 3606. DOI:10.1038/ncomms4606 |
| [31] |
Asada K, Salim V, Masada-Atsumi S, et al. A 7-deoxyloganetic acid glucosyltransferase contributes a key step in secologanin biosynthesis in Madagascar periwinkle[J]. Plant Cell, 2013, 25: 4123-4134. |
| [32] |
Xiong B, Zhao ZL, Ni LH, et al. DNA-based identification of Gentiana robusta and related species[J]. China J Chin Mater Med (中国中药杂志), 2015, 40: 4680-4685. |
| [33] |
Kreps JA, Simon AE. Environmental and genetic effects on circadian clock-regulated gene expression in Arabidopsis[J]. Plant Cell, 1997, 9: 297-304. |
| [34] |
Smith EN, Kruglyak L, Mackay T. Gene-environment interaction in yeast gene expression[J]. PLoS Biol, 2008, 6: e83. DOI:10.1371/journal.pbio.0060083 |
| [35] |
Li H, Zhang N, Li YM, et al. High-throughput transcriptomic sequencing of Rheum palmatum L. seedlings and elucidation of genes in anthraquinone biosynthesis[J]. Acta Pharm Sin (药学学报), 2018, 53: 152-161. |
| [36] |
Tian M, Zhang X, Zhu Y, et al. Global transcriptome analyses reveal differentially expressed genes of six organs and putative genes involved in (iso) flavonoid biosynthesis in Belamcanda chinensis[J]. Front Plant Sci, 2018, 9: 1160. DOI:10.3389/fpls.2018.01160 |
| [37] |
Gu CY, Zhao ZL, Wu JR, et al. Pharmacognostical study on Gentiana lhassica Burk. and Gentiana waltonii Burk.[J]. Chin J Inf Tradit Chin Med (中国中医药信息杂志), 2010, 17: 41-43. |
 2021, Vol. 56
2021, Vol. 56


