1982年, 研究者们[1-4]在人类膀胱癌细胞中发现了突变的RAS (rat sarcoma) 基因, 这使RAS成为首个被发现的人类肿瘤基因[5, 6]。研究发现RAS主要有HRAS、NRAS和KRAS三种亚型[7]。
RAS在细胞信号转导过程中发挥核心作用[8]: 在分子水平上可以调控转录、翻译等过程, 在细胞水平上可以调节细胞增殖、分化、衰老、凋亡等等[9-11]。RAS的突变致癌约占人类癌症的30%[12], 因此, RAS一直被视为肿瘤治疗的重要靶点。
RAS蛋白(图 1) 是一种低分子质量(21 kDa)、位于细胞膜上、拥有GTPase酶活性的鸟嘌呤核苷酸结合蛋白。RAS主要由一个GTPase催化结构域(残基1~166) 和紧随其后的高度可变区域(highly variable region, HVR; 残基167~188) 组成[13]。各个亚型中GTPase结构域的序列和结构几乎相同, 而HVR的序列存在显著差异[14, 15]。

|
Figure 1 Structure of RAS. (A) Protein structure of RAS with GDP-bound (PDB: 4obe). HVR is not fully displayed in the structure (Residues 1-169 were presented in this protein structure). (B) 2D depiction of the secondary structure of RAS |
RAS通过在非活性状态(与GDP结合) 和活性状态(与GTP结合) 之间循环, 发挥着二元开关的作用[13, 16]: 在没有信号刺激时, 这种循环十分缓慢。一旦接收到信号, RAS就会在细胞膜上与被募集过来的鸟嘌呤核苷酸交换因子(guanine nucleotide-exchange factor; GEF) 结合, 释放GDP, 并迅速与GTP结合[17], 进入"开"的阶段; 随后在GTP酶激活蛋白(GTPase-activating protein; GAP) 的作用下, GTPase的活性被大幅度增强[18], 与RAS结合的GTP被水解为GDP, 重新进入"关"的状态。绝大多数的RAS突变会导致GTP的水解受到抑制, RAS的活性无法被淬灭, 进而引起RAS的功能被异常激活。因此GTP的水解成为了二元开关过程中的关键步骤。
根据RAS的结构和调控机制, 研究者们发现, 在治疗RAS方面存在种种难题[19, 20]: ① RAS蛋白上的GTPase催化结构域高度保守, 各个亚型之间很难做到高度选择性; ② RAS与GDP/GTP的亲和力常数能达到皮摩尔级别, 而细胞中的GTP浓度是毫摩尔级别, 因此小分子药物很难与底物进行竞争性结合; ③ RAS蛋白表面缺少有利于小分子结合的腔。
但是, 随着新技术的发展, 近年来针对RAS的抑制剂又重新回到了人们的视野中。研究者们首先将目标放在了RAS家族的亚型之一KRAS上面。KRAS基因突变占RAS基因突变总数的85% (NRAS占12%, HRAS占3%)[21]。在结肠癌、肺癌等多种人类癌症当中, 经常能观察到KRAS基因突变[22-24], 而且在超过90% 的胰腺癌患者体内KRAS基因都发生了突变[25]。因此, KRAS成为了有效治疗RAS的重要突破点。
1 KRAS抑制剂的研究进展按抑制剂的作用模型将KRAS抑制剂分为直接靶向KRAS的抑制剂和间接作用于KRAS的抑制剂两类。前者是直接以KRAS为靶点的抑制剂, 后者主要是通过抑制KRAS信号通路的上下游部分, 间接地对KRAS起抑制作用。
1.1 直接靶向KRAS的抑制剂绝大多数的RAS致癌突变集中在Gly12、Gly13、Gln61这三个氨基酸残基上[12, 13, 26, 27]。其中, KRASG12C突变占所有非小细胞肺癌(NSCLC) 的病例超过59%, 占所有KRAS突变驱动的癌症的12%[12, 19, 28]。而基于半胱氨酸(Cys) 共价结合抑制剂的成功研制, 让KRASG12C成为了治疗RAS变异的理想靶点: 在KRASG12C中, 12号位的甘氨酸突变为半胱氨酸, 而Cys是一种易于形成共价键的氨基酸, 如果小分子药物能够与这种Cys结合, 这类抑制剂将减少对结合腔的依赖, 也可以获得相对较高的特异性。因此, 特异性针对KRASG12C的共价抑制剂, 为靶向KRAS药物的开发提供了一个重要的参考方向。
1.1.1 6H05系列化合物KRASG12C共价抑制剂的发现是通过一项名为"tethering"的技术[19, 29]。"tethering"技术的基本原理是: 针对含有Cys的靶点, 利用含双硫键的小分子碎片库进行筛选——小分子碎片的双硫键与KRASG12C蛋白上Cys的巯基发生交换后, 小分子碎片以Cys为"桥梁"与KRAS蛋白相连接, 小分子碎片在蛋白上找到合适的结合腔后便能附着下来, 随后通过还原反应将作为"桥梁"的Cys给清除掉。
通过"tethering"技术, 在480个化合物中筛选出了两个先导物, 显示出对突变KRASG12C的修饰程度最大, 被命名为6H05和2E07[29] (图 2)。随后通过对6H05的构效关系进行研究, 进一步优化确定了两个有效的化合物(化合物3和化合物4, 图 2), 这些化合物被称为6H05系列[30]。
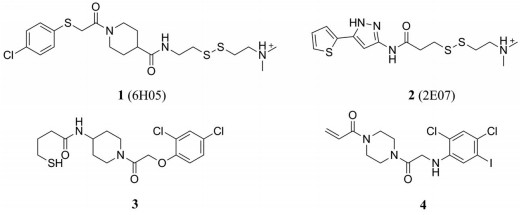
|
Figure 2 The structures of 6H05 series compounds |
6H05系列的化合物与KRASG12C结合, 发现了一个被称为Switch-Ⅱ pocket (S-ⅡP) 的新结合位点[31]。晶体结构显示, S-ⅡP位于KRAS中央的β折叠和Switch-Ⅱ (SW2) 之间, 是通过抑制剂结合并诱导SW2重新排列而形成的[30]。结果证明6H05系列让KRASG12C共价抑制剂的尝试获得了首次成功。而S-ⅡP的发现意味着KRAS蛋白中有了额外的结合位点, 成为了后来药物设计的一个关键点。
1.1.2 喹唑啉系列化合物KRASG12C共价抑制剂发展进程中的另一个突破是喹唑啉系列化合物的发现。研究者[32]设计了一组喹唑啉化合物能够结合在S-IIP空腔中, 并在此基础上进一步的进行结构优化(图 3)。结果表明, 酰胺取代基的加入能够提高药物活性: 除了增强抗增殖能力和诱导凋亡能力以外, 酰胺基的修饰还增强了对KRASG12C癌细胞的选择性。S-IIP的发现以及喹唑啉结构的发现与优化, 为后续KRASG12C抑制剂的发展奠定了坚实的基础。

|
Figure 3 The structures of quinazoline series compounds |
ARS系列化合物是Wellspring公司研发的一系列KRASG12C抑制剂。其中比较有代表性的是ARS-853[33]和ARS-1620[34] (图 4)。

|
Figure 4 The structures of ARS-853 and ARS-1620 |
研究发现, KRASG12C是ARS-853在2 700多种细胞蛋白质中最能有效进行共价结合的靶点[33]。ARS-853与KRASG12C的结合速率常数为76 L·mol-1·s-1, 细胞结合IC50在6 h内为1.6 μmol·L-1。细胞实验表明, ARS-853对多种肿瘤细胞系均具有抑制活性, 表现出对肿瘤细胞凋亡的促进以及对肿瘤细胞生长的抑制。ARS-853仅与非活性状态的KRASG12C结合, 表明KRASG12C不是停留在异常活跃的状态, 而是在与GDP/GTP结合态之间快速循环[33]。
ARS-853化合物虽然在低微摩尔范围内仍表现出较好的细胞活性, 但存在致命的缺点: 血浆代谢稳定性差(t1/2 < 0.2 h人, t1/2 < 0.3 h小鼠), 而且小鼠口服生物利用度低(F < 2%)[34]。这导致ARS-853无法用于进一步的体内研究。
Wellspring公司基于喹唑啉的结构基础, 对喹唑啉母核周围的取代基进行系统优化后, 到得了几个活性较高的化合物, 其中引人注目的是ARS-1620[34]。ARS-1620是一种对KRASG12C具有高效价和高选择性的共价化合物, 与KRASG12C的结合速率相比于ARS-853提高了10倍。细胞实验表明, 在KRASG12C细胞系(H358、MIA-PaCa2、LU65) 中, ARS-1620的IC50能达到150 nmol·L-1, 在其他KRAS突变的细胞系(H441、A549、HCT116) 中也表现出良好的效果。在动物模型的进一步研究中, ARS-1620在小鼠体内表现出极好的口服生物利用度(F > 60%)和足够的血液稳定性, 而且在人源肿瘤异种移植瘤(patient-derived tumor xenograft, PDX) 模型中也证明了ARS-1620对KRASG12C具有高度选择性, 相比于ARS-853有了显著的提高。实验数据也说明ARS-1620可快速地实现体内靶标占据, 抑制RAS信号的传导, 以此消灭掉肿瘤。
ARS-3248是在ARS-1620基础上研发的新一代KRASG12C抑制剂, 但其结构目前尚未被披露。目前, ARS-3248正在开展临床Ⅰ期试验, 选择KRASG12C阳性晚期实体瘤患者入组。
1.1.4 MRTX849MRTX849 (图 5) 是由Mirati Therapeutics公司开发的针对KRASG12C的高度选择性的共价抑制剂, 能够稳定地与处于非活性状态的KRASG12C结合[35]。在100 mg·kg-1·d-1的最大有效剂量下, MRTX849针对不同的肿瘤模型均显示出剂量依赖性的抗肿瘤作用。相比于野生型KRAS以及其他蛋白中的Cys来说, MRTX849针对KRASG12C的选择性高了1 000倍以上。MRTX849口服生物利用度能达到30%, 单次给药后半衰期长达25 h[30]。

|
Figure 5 The structure of MRTX849 |
临床试验显示[36], MRTX849临床试验招募了17名患者, 其中包括10名NSCLC患者、4名结肠直肠癌(CRC) 患者和3名其他癌症类型患者。他们接受不同剂量的治疗, 分别为150、300、600、1 200 mg每天口服一次, 以及600 mg每天口服两次。在接受所有剂量治疗的患者中, 3/6的NSCLC患者, 以及1/4的CRC患者能实现了部分缓解(partial response, PR); 在接受最高剂量治疗的患者中, 3/5例的NSCLC患者和1/2例的CRC患者达到部分缓解, 其他患者病情稳定(stable disease, SD)。
1.1.5 AMG 510AMG 510 (图 6) 是Amgen公司开发的一种小分子抑制剂, 也是第一个进入临床阶段的KRASG12C共价抑制剂[37]。AMG 510通过将KRAS锁定在非活性的状态(与GDP结合), 特异性地抑制KRASG12C。

|
Figure 6 The structure of AMG 510 |
在肿瘤模型中, AMG 510能选择性地靶向KRASG12C, 并且单药治疗就可引起KRASG12C肿瘤小鼠模型的肿瘤消退[38]。在临床试验中[39], AMG 510依然表现出良好的耐受性, 且没有剂量限制的毒性。在招募的34位NSCLC患者中, 有23位患者的疗效可被评估。其中有13名患者接受了每天一次960 mg目标剂量的治疗, 其中7名实现了部分缓解, 6名患者的病情稳定, 控制率达到100%。
1.1.6 SML系列以GDP/GTP结合位点进行共价激酶抑制剂的研发[40-42], 为KRASG12C抑制剂的研究提供了新的思路, 得到了一系列核苷酸类的KRASG12C共价抑制剂。
基于GDP的结构首先设计了一系列针对催化位点的、底物竞争相关的共价抑制剂, SML-8-73-1 (图 7) 被确定为主要候选物[43]。模拟细胞条件, 在1 mmol·L-1的GDP/GTP存在的情况下, 对SML-8-73-1的结合效率进行测定, 结果表明, 在孵育2 h后, SML-8-73-1进行底物竞争性结合的KRASG12C超过95%。
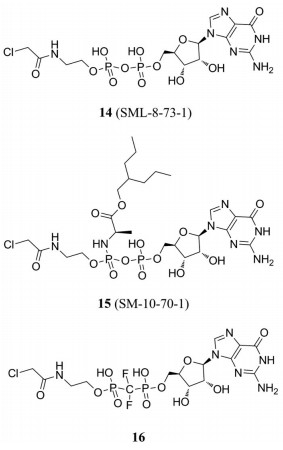
|
Figure 7 The structures of SML series compounds |
然而, SML-8-73-1含有两个带负电荷的磷酸基团, 导致难以穿过细胞膜。"caging"技术, 也即合成各种修饰磷酸, 如磷酸酯, 是一种有效缓解甚至解决该问题的常见策略, 它能通过化学修饰将带电离子部分所掩盖, 从而使细胞能被动吸收[44]。在此基础上, 对SML-8-73-1的磷酸基团进行修饰, 合成出了SM-10-70-1 (图 7)。SM-10-70-1表现为细胞通透性增强, 并通过共价结合竞争性地抑制KRASG12C。此外, KRAS依赖的信号通路, 如Akt和ERK通路也被抑制。不仅如此, 在几个表达KRAS G12C突变的癌细胞株中, 证实了SM-10-70-1具有抗增殖活性的能力。但其有效率和选择特异性仍有待进一步提高。因此, 新的SARs研究得以继续, 获得了有希望的化合物16[45] (图 7)。通过亚甲基取代SML-8-73-1和SM-10-70-1磷酸酐键中的中心氧, 化合物16的化学稳定性和酶稳定性有了很大的提高。尽管相比于SML-8-73-1降低了40倍的亲和力, 化合物16仍然可能是一个有发展潜力的化合物。
1.2 间接作用于KRAS的抑制剂 1.2.1 靶向KRAS的mRNA肿瘤疫苗mRNA肿瘤疫苗的治疗思路是: 在肿瘤细胞发生基因突变后, 会特异性表达新的抗原, 通过对肿瘤细胞进行DNA或RNA测序, 寻找出这类新抗原, 随后以相对应的mRNA为基础完成疫苗的制备, 输回体内激活免疫细胞, 进而杀灭带有上述新抗原的肿瘤细胞[46]。其中, 构建的疫苗被称为mRNA肿瘤疫苗。
有研究发现, 从一种转基因小鼠体内分离并改造后的T细胞, 对多种类型肿瘤患者体内的KRAS突变体均有识别作用[47], 在后续实验中, 有研究者[48]成功地从一名KRAS突变的癌症患者身上移植培育出可被该T细胞识别的细胞系。在此基础上, Moderna Therapeutics公司与Merck & Co.合作开发了一种名为mRNA-5671的mRNA肿瘤疫苗, mRNA-5671能在体内产生大量的抗原, 引导T细胞寻找并攻击表达4种关键KRAS突变体(G12C、G12D、G12V和G13C) 的细胞[49]。mRNA-5671可以直接在宿主细胞中表达KRAS抗原, 比基于DNA的疫苗更容易降解, 减少了不良反应, 比基于肽的疫苗也能更有效地促进T细胞做出免疫反应[46, 50]。目前, 为了更加充分地利用免疫系统能力, mRNA-5671在与PD1抑制剂pembrolizumab联合使用[49]。
1.2.2 SOS1抑制剂GEF承担着介导KRAS与GTP结合的重任, 因此, 抑制GEF可以减少KRAS与GTP结合的机会, 可能是靶向RAS驱动肿瘤的一种可行方法[51]。而且, GEF抑制剂类化合物阻断的是GEF与KRAS的相互作用, 与KRAS是何种突变无太大关联。
SOS (son of sevenless) 是常见的GEF之一, 包括SOS1和SOS2两种常见的亚型[52]。作为一种核苷酸交换因子, SOS首先会与RAS结合。RAS-SOS复合物会改变RAS蛋白的构象, 改变后的构象与缺失镁辅因子(magnesium cofactor) 的RAS (非活性状态) 构象十分相似, 因此阻断了镁辅因子与GDP中的磷酸盐部分进行亲水性的相互作用[17], 进而降低了RAS与GDP的亲和力, 有助于GDP的释放。然后GTP的鸟嘌呤核苷和核糖部分开始与RAS结合, 随后GTP的γ-磷酸基团与RAS的镁辅因子相互作用, 导致RAS的构象再次发生变化, SOS从络合物中脱离出来。
由此可见, 抑制SOS的活性可降低RAS蛋白对GTP的结合能力, 进而抑制RAS的异常激活。基于这个思路, Boehringer Ingelheim (BI) 公司找出了一些能够结合SOS1并阻止SOS1与RAS相互作用的苗头化合物。随后BI公司报道了BI-3406 (IC50 = 6 nmol·L-1, 图 8)[53]。BI-3406通过与SOS1的催化位点结合, 阻止KRAS-GDP的相互作用。在单药治疗的情况下, BI-3406能够有效地减少KRAS依赖性的肿瘤细胞中活性形式(与GTP结合) KRAS的形成, 并抑制MAPK信号通路, 引起肿瘤消退[53]。
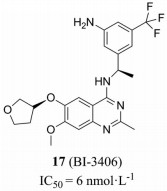
|
Figure 8 The structure of BI-3406 |
随后BI 1701963进入了临床开发阶段[54] (目前未公布结构)。BI 1701963与BI-3406密切相关, 均能广泛针对KRAS等位基因(G12D、G12V、G12C以及G13D) 表现出活性, 同时保留了KRAS与SOS2的相互作用[55]。
此外, Bayer公司也发现了一款SOS1抑制剂[51], 命名为BAY-293 (IC50 = 21 nmol·L-1, 图 9), 可以选择性的抑制SOS1活性, 下调肿瘤细胞中RAS的活性水平。BAY-293在野生型KRAS细胞中能够有效抑制RAS-RAF-MEK-ERK通路, 且BAY-293与ARS-853的联用对于KRASG12C细胞系的抗增殖活性表现出协同作用[51]。
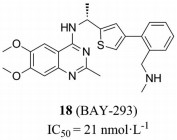
|
Figure 9 The structure of BAY-293 |
除了新药的研制, 在"老药新用"方面, 靶向KRAS的研究也有重大突破: 最近有研究团队发现, 降糖一线药物二甲双胍(metformin, 图 10) 也可用于靶向KRAS的治疗, 且二甲双胍的抑癌机制可能不依赖于其降糖作用[56]。研究发现, 相比于其他降糖药, 服用二甲双胍以后, 携带KRAS突变的转移性结直肠癌(metastatic colorectal cancer, mCRC) 患者总生存时间延长了37.8个月, 然而二甲双胍不能提高野生型KRAS患者的生存率。细胞实验发现, KRAS的突变会导致膜通道蛋白MATE1的表达水平下降, 二甲双胍无法从肿瘤细胞中排出, 进而在细胞中富集。上调癌细胞中MATE1表达水平后二甲双胍的抑癌作用消失也证明了这一点。而高浓度的二甲双胍则通过抑制RAS/ERK和AKT/mTOR这两条通路, 抑制了KRAS突变癌细胞的增殖[56]。目前研究团队已经安排了前瞻性的临床研究。

|
Figure 10 The structure of metformin |
KRAS与GTP结合后会将效应蛋白分泌到质膜(plasma membrane, PM) 上, 随后激活细胞中的增殖和存活信号[57, 58]。而PDEdelta (phosphodiesterase 6 delta) 作为一种膜转运蛋白, 通过相互作用控制KRAS蛋白的转运[59, 60]。因此, 干扰PDEdelta与KRAS的结合为靶向KRAS突变癌症提供了一种新方法。
Zimmermann等[61]首先研发得到了一种小分子抑制剂deltarasin (图 11), deltarasin通过结合PDEdelta的异戊二烯结合腔, 抑制了KRAS-PDEdelta的相互作用, 干扰KRAS在PM上的定位, 进而抑制下游通路信号, 减少癌细胞的增殖。然而当浓度高于9 μmol·L-1时, deltarasin在所有实验用的细胞系中都表现出细胞毒性[61], 并且在NSCLC中deltarasin会诱导自噬[62]。第二种PDEdelta抑制剂deltazinone 1 (图 11) 有效避免了细胞毒性的问题, 且对PDEdelta有较高的亲和力, 但是在体内实验中deltazinone 1会被快速代谢掉[63]。目前, 仍有NHTD (图 11) 等PDEdelta抑制剂处于临床前研究[64], 并在考虑与联合治疗的策略。

|
Figure 11 The structures of deltarasin, deltazinone 1 and NHTD |
目前来看, 针对G12C突变的抑制剂仍是靶向KRAS治疗肿瘤的主流方向。而且, 针对KRASG12C抑制剂临床表现不够完美的情况来看, 不少研究者也采用了联合用药的策略[35, 38, 49, 65]。例如, 在小鼠体内使用AMG 510会引起促进发炎性的肿瘤微环境, 采用AMG 510和PD1阻断剂pembrolizumab能够有效缓解这个问题[38], 并且对KRASG12D肿瘤细胞也有一定的排斥作用。此外, 研究者将AMG 510与MEK抑制剂、MAPK抑制剂以及SHP2抑制剂等多种抑制剂分别组合使用, 均表现出不错的疗效[49]; MRTX849与HER2抑制剂、CDK4抑制剂以及SHP2抑制剂等小分子抑制剂的组合[49], 在增强MRTX849的反应活性以及减弱耐药性方面, 也具有良好的前景; BI 1701963也在寻求与MEK抑制剂的联合使用, 以增强更好的疗效[54, 55]。
此外, 蛋白降解靶向嵌合体(proteolysis targeting chimera, PROTAC) 技术凭借其独有的优势, 在治疗KRAS方面也有着不错的应用[66, 67]。但是PROTAC本身依然面临着一些难题, 例如分子质量较大导致的药代动力学性质不佳以及口服生物利用度不高[68], 可用于PROTAC的泛素连接酶E3过少(不到10种)[68, 69]等问题。
总之, 这些新的在研药物(表 1)[49]不仅开拓了KRAS治疗的新领域, 为不同类型的KRAS突变患者带来了更多的治疗方法, 也为研究RAS基因提供了有价值的方向, 有助于将历史上"不可成药的"RAS的相关药物引入临床。
| Table 1 KRAS-related inhibitors under clinical trials currently |
无论是新技术的研发, 还是新药物的发现, 亦或是如二甲双胍在内的"老药新用"开辟的新天地, 都表明研究者通过有策略地靶向RAS治疗肿瘤取得了越来越好的临床结果。如同KRAS抑制剂的研究正在如火如荼地展开一样, 人们对于RAS机制的研究也没有停止脚步。随着技术的发展, RAS也会更加清晰地被展现在我们面前, 我们也终将会攻克这个癌症领域里的难关。
作者贡献: 许俨钊撰写论文初稿; 文辉、崔华清修改论文并定稿。
利益冲突: 本文无相关利益冲突。
| [1] |
Der CJ, Krontiris TG, Cooper GM. Transforming genes of human bladder and lung carcinoma cell lines are homologous to the ras genes of Harvey and Kirsten sarcoma viruses[J]. Proc Natl Acad Sci U S A, 1982, 79: 3637-3640. DOI:10.1073/pnas.79.11.3637 |
| [2] |
Parada LF, Tabin CJ, Shih C, et al. Human EJ bladder carcinoma oncogene is homologue of Harvey sarcoma virus ras gene[J]. Nature, 1982, 297: 474-478. DOI:10.1038/297474a0 |
| [3] |
Santos E, Tronick SR, Aaronson SA, et al. T24 human bladder carcinoma oncogene is an activated form of the normal human homologue of BALB-and Harvey-MSV transforming genes[J]. Nature, 1982, 298: 343-347. DOI:10.1038/298343a0 |
| [4] |
Taparowsky E, Suard Y, Fasano O, et al. Activation of the T24 bladder carcinoma transforming gene is linked to a single amino acid change[J]. Nature, 1982, 300: 762-765. DOI:10.1038/300762a0 |
| [5] |
Stephen AG, Esposito D, Bagni RK, et al. Dragging ras back in the ring[J]. Cancer Cell, 2014, 25: 272-281. DOI:10.1016/j.ccr.2014.02.017 |
| [6] |
Cox AD, Der CJ. Ras history: the saga continues[J]. Small GTPases, 2010, 1: 2-27. DOI:10.4161/sgtp.1.1.12178 |
| [7] |
Barbacid M. Ras genes[J]. Annu Rev Biochem, 1987, 56: 779-827. DOI:10.1146/annurev.bi.56.070187.004023 |
| [8] |
Li S, Jang H, Zhang J, et al. Raf-1 cysteine-rich domain increases the affinity of K-Ras/Raf at the membrane, promoting MAPK signaling[J]. Structure, 2018, 26: 513-525. DOI:10.1016/j.str.2018.01.011 |
| [9] |
Rojas AM, Fuentes G, Rausell A, et al. The Ras protein superfamily: evolutionary tree and role of conserved amino acids[J]. J Cell Biol, 2012, 196: 189-201. DOI:10.1083/jcb.201103008 |
| [10] |
Quinlan MP, Settleman J. Isoform-specific ras functions in development and cancer[J]. Future Oncol, 2009, 5: 105-116. DOI:10.2217/14796694.5.1.105 |
| [11] |
Van Hattum H, Waldmann H. Chemical biology tools for regulating RAS signaling complexity in space and time[J]. Chem Biol, 2014, 21: 1185-1195. DOI:10.1016/j.chembiol.2014.08.001 |
| [12] |
Forbes SA, Bindal N, Bamford S, et al. COSMIC: mining complete cancer genomes in the catalogue of somatic mutations in cancer[J]. Nucleic Acids Res, 2011, 39: D945-950. DOI:10.1093/nar/gkq929 |
| [13] |
Lu S, Jang H, Gu S, et al. Drugging Ras GTPase: a comprehensive mechanistic and signaling structural view[J]. Chem Soc Rev, 2016, 45: 4929-4952. DOI:10.1039/C5CS00911A |
| [14] |
Prior IA, Hancock JF. Compartmentalization of Ras proteins[J]. J Cell Sci, 2001, 114: 1603-1608. DOI:10.1242/jcs.114.9.1603 |
| [15] |
Hancock JF. Ras proteins: different signals from different locations[J]. Nat Rev Mol Cell Biol, 2003, 4: 373-385. DOI:10.1038/nrm1105 |
| [16] |
Bos JL, Rehmann H, Wittinghofer A. GEFs and GAPs: critical elements in the control of small G proteins[J]. Cell, 2007, 129: 865-877. DOI:10.1016/j.cell.2007.05.018 |
| [17] |
Boriack-Sjodin PA, Margarit SM, Bar-Sagi D, et al. The structural basis of the activation of Ras by SOS[J]. Nature, 1998, 394: 337-343. DOI:10.1038/28548 |
| [18] |
Settleman J, Albright CF, Foster LC, et al. Association between GTPase activators for Rho and Ras families[J]. Nature, 1992, 359: 153-154. DOI:10.1038/359153a0 |
| [19] |
Cox AD, Fesik SW, Kimmelman AC, et al. Drugging the undruggable RAS: mission possible?[J]. Nat Rev Drug Discov, 2014, 13: 828-851. DOI:10.1038/nrd4389 |
| [20] |
Ledford H. The ras renaissance[J]. Nature, 2015, 520: 278-280. DOI:10.1038/520278a |
| [21] |
Hunter JC, Manandhar A, Carrasco MA, et al. Biochemical and structural analysis of common cancer-associated KRAS mutations[J]. Mol Cancer Res, 2015, 13: 1325-1335. DOI:10.1158/1541-7786.MCR-15-0203 |
| [22] |
Van Cutsem E, van de Velde H, Karasek P, et al. Phase Ⅲ trial of gemcitabine plus tipifarnib compared with gemcitabine plus placebo in advanced pancreatic cancer[J]. J Clin Oncol, 2004, 22: 1430-1438. DOI:10.1200/JCO.2004.10.112 |
| [23] |
Rao S, Cunningham D, De Gramont A, et al. Phase Ⅲ double-blind placebo-controlled study of farnesyl transferase inhibitor R115777 in patients with refractory advanced colorectal cancer[J]. J Clin Oncol, 2004, 22: 3950-3957. DOI:10.1200/JCO.2004.10.037 |
| [24] |
Johnson BE, Heymach JV. Farnesyl transferase inhibitors for patients with lung cancer[J]. Clin Cancer Res, 2004, 10: 4254s-4257s. DOI:10.1158/1078-0432.CCR-040016 |
| [25] |
Xu Q, Zhou Z, Xiang M. Progress in the study of signal pathways for chronic inflammation-associated pancreatic carcinogenesis[J]. Acta Pharm Sin (药学学报), 2016, 51: 174-181. |
| [26] |
Lu S, Banerjee A, Jang H, et al. GTP binding and oncogenic mutations may attenuate hypervariable region (HVR)-catalytic domain interactions in small GTPase K-Ras4B, exposing the effector binding site[J]. J Biol Chem, 2015, 290: 28887-28900. DOI:10.1074/jbc.M115.664755 |
| [27] |
Prior IA, Lewis PD, Mattos C. A comprehensive survey of Ras mutations in cancer[J]. Cancer Res, 2012, 72: 2457-2467. DOI:10.1158/0008-5472.CAN-11-2612 |
| [28] |
Visscher M, Arkin MR, Dansen TB. Covalent targeting of acquired cysteines in cancer[J]. Curr Opin Chem Biol, 2016, 30: 61-67. DOI:10.1016/j.cbpa.2015.11.004 |
| [29] |
Erlanson DA, Braisted AC, Raphael DR, et al. Site-directed ligand discovery[J]. Proc Natl Acad Sci U S A, 2000, 97: 9367-9372. DOI:10.1073/pnas.97.17.9367 |
| [30] |
Ni D, Li X, He X, et al. Drugging K-RasG12C through covalent inhibitors: mission possible?[J]. Pharmacol Ther, 2019, 202: 1-17. DOI:10.1016/j.pharmthera.2019.06.007 |
| [31] |
Ostrem JM, Peters U, Sos ML, et al. K-Ras(G12C) inhibitors allosterically control GTP affinity and effector interactions[J]. Nature, 2013, 503: 548-551. DOI:10.1038/nature12796 |
| [32] |
Zeng M, Lu J, Li L, et al. Potent and selective covalent quinazoline inhibitors of KRAS G12C[J]. Cell Chem Biol, 2017, 24: 1005-1016e1003. DOI:10.1016/j.chembiol.2017.06.017 |
| [33] |
Patricelli MP, Janes MR, Li LS, et al. Selective inhibition of oncogenic KRAS output with small molecules targeting the inactive state[J]. Cancer Discov, 2016, 6: 316-329. DOI:10.1158/2159-8290.CD-15-1105 |
| [34] |
Janes MR, Zhang J, Li LS, et al. Targeting KRAS mutant cancers with a covalent G12C-specific inhibitor[J]. Cell, 2018, 172: 578-589e517. DOI:10.1016/j.cell.2018.01.006 |
| [35] |
Hallin J, Engstrom LD, Hargis L, et al. The KRAS(G12C) inhibitor MRTX849 provides insight toward therapeutic susceptibility of KRAS-mutant cancers in mouse models and patients[J]. Cancer Discov, 2020, 10: 54-71. DOI:10.1158/2159-8290.CD-19-1167 |
| [36] |
Papadopoulos KP, Ou SHI, Johnson ML, et al. A phase Ⅰ/Ⅱ multiple expansion cohort trial of MRTX849 in patients with advanced solid tumors with KRAS G12C mutation[C]. Chicago, IL: Annual Meeting of the American-Society-of-Clinical-Oncology, 2019.
|
| [37] |
Govindan R, Fakih MG, Price TJ, et al. Phase Ⅰ study of AMG 510, a novel molecule targeting KRAS G12C mutant solid tumours[J]. Ann Oncol, 2019, 30: 163-164. |
| [38] |
Canon J, Rex K, Saiki AY, et al. The clinical KRAS(G12C) inhibitor AMG 510 drives anti-tumour immunity[J]. Nature, 2019, 575: 217-223. DOI:10.1038/s41586-019-1694-1 |
| [39] |
Safety GP, Sheets SD. Amgen announces new clinical data evaluating novel investigational KRAS (G12C) inhibitor in larger patient group at WCLC 2019[EB/OL]. 2019-09-08[2020-08-13]. https://investors.amgen.com/news-releases/news-release-details/amgen-announces-new-clinical-data-evaluating-novel.
|
| [40] |
Zhang T, Inesta-Vaquera F, Niepel M, et al. Discovery of potent and selective covalent inhibitors of JNK[J]. Chem Biol, 2012, 19: 140-154. DOI:10.1016/j.chembiol.2011.11.010 |
| [41] |
Zhou W, Ercan D, Chen L, et al. Novel mutant-selective EGFR kinase inhibitors against EGFR T790M[J]. Nature, 2009, 462: 1070-1074. DOI:10.1038/nature08622 |
| [42] |
Zhou W, Hur W, McDermott U, et al. A structure-guided approach to creating covalent FGFR inhibitors[J]. Chem Biol, 2010, 17: 285-295. DOI:10.1016/j.chembiol.2010.02.007 |
| [43] |
Lim SM, Westover KD, Ficarro SB, et al. Therapeutic targeting of oncogenic K‐Ras by a covalent catalytic site inhibitor[J]. Angew Chem, 2014, 126: 203-208. DOI:10.1002/ange.201307387 |
| [44] |
Adams SR, Tsien RY. Controlling cell chemistry with caged compounds[J]. Annu Rev Physiol, 1993, 55: 755-784. DOI:10.1146/annurev.ph.55.030193.003543 |
| [45] |
Xiong Y, Lu J, Hunter J, et al. Covalent guanosine mimetic inhibitors of G12C KRAS[J]. ACS Med Chem Lett, 2017, 8: 61-66. DOI:10.1021/acsmedchemlett.6b00373 |
| [46] |
McNamara MA, Nair SK, Holl EK. RNA-based vaccines in cancer immunotherapy[J/OL]. J Immunol Res, 2015: Article ID 794528. [2020-08-24]. https://www.hindawi.com/journals/jir/2015/794528.
|
| [47] |
Wang QJ, Yu Z, Griffith K, et al. Identification of T-cell receptors targeting KRAS-mutated human tumors[J]. Cancer Immunol Res, 2016, 4: 204-214. DOI:10.1158/2326-6066.CIR-15-0188 |
| [48] |
Tran E, Robbins PF, Lu YC, et al. T-cell transfer therapy targeting mutant KRAS in cancer[J]. N Engl J Med, 2016, 375: 2255-2262. DOI:10.1056/NEJMoa1609279 |
| [49] |
Mullard A. Cracking KRAS[J]. Nat Rev Drug Discov, 2019, 18: 887-891. DOI:10.1038/d41573-019-00195-5 |
| [50] |
Fotin-Mleczek M, Duchardt KM, Lorenz C, et al. Messenger RNA-based vaccines with dual activity induce balanced TLR-7 dependent adaptive immune responses and provide antitumor activity[J]. J Immunother, 2011, 34: 1-15. DOI:10.1097/CJI.0b013e3181f7dbe8 |
| [51] |
Hillig RC, Sautier B, Schroeder J, et al. Discovery of potent SOS1 inhibitors that block RAS activation via disruption of the RAS-SOS1 interaction[J]. Proc Natl Acad Sci U S A, 2019, 116: 2551-2560. DOI:10.1073/pnas.1812963116 |
| [52] |
Vigil D, Cherfils J, Rossman KL, et al. Ras superfamily GEFs and GAPs: validated and tractable targets for cancer therapy?[J]. Nat Rev Cancer, 2010, 10: 842-857. DOI:10.1038/nrc2960 |
| [53] |
Hofmann MH, Gmachl M, Ramharter J, et al. Abstract C133: Effective targeting of KRAS-driven tumors by the first-in-class, orally bioavailable SOS1: KRAS inhibitor BI-3406[C]. Boston, MA: International Conference on Molecular Targets and Cancer Therapeutics, 2019.
|
| [54] |
Gort E, Johnson ML, Hwang JJ, et al. A phase Ⅰ, open-label, dose-escalation trial of BI 1701963 as monotherapy and in combination with trametinib in patients with KRAS mutated advanced or metastatic solid tumors[C]. Chicago, IL: American Society of Clinical Oncology Congress (ASCO) Annual Meeting, 2020.
|
| [55] |
Gerlach D, Gmachl M, Ramharter J, et al. BI-3406 and BI 1701963: potent and selective SOS1: KRAS inhibitors induce regressions in combination with MEK inhibitors or irinotecan[C]. Philadelphia, PA: AACR Annual Meeting 2020, 2020.
|
| [56] |
Xie J, Xia L, Xiang W, et al. Metformin selectively inhibits metastatic colorectal cancer with the KRAS mutation by intracellular accumulation through silencing MATE1[J]. Proc Natl Acad Sci U S A, 2020, 117: 13012-13022. DOI:10.1073/pnas.1918845117 |
| [57] |
Karnoub AE, Weinberg RA. Ras oncogenes: split personalities[J]. Nat Rev Mol Cell Biol, 2008, 9: 517-531. DOI:10.1038/nrm2438 |
| [58] |
Pylayeva-Gupta Y, Grabocka E, Bar-Sagi D. RAS oncogenes: weaving a tumorigenic web[J]. Nat Rev Cancer, 2011, 11: 761-774. DOI:10.1038/nrc3106 |
| [59] |
Chandra A, Grecco HE, Pisupati V, et al. The GDI-like solubilizing factor PDEδ sustains the spatial organization and signalling of Ras family proteins[J]. Nat Cell Biol, 2012, 14: 148-158. DOI:10.1038/ncb2394 |
| [60] |
Ismail SA, Chen YX, Rusinova A, et al. Arl2-GTP and Arl3-GTP regulate a GDI-like transport system for farnesylated cargo[J]. Nat Chem Biol, 2011, 7: 942-949. DOI:10.1038/nchembio.686 |
| [61] |
Zimmermann G, Papke B, Ismail S, et al. Small molecule inhibition of the KRAS–PDEδ interaction impairs oncogenic KRAS signalling[J]. Nature, 2013, 497: 638-642. DOI:10.1038/nature12205 |
| [62] |
Leung ELH, Luo LX, Liu ZQ, et al. Inhibition of KRAS-dependent lung cancer cell growth by deltarasin: blockage of autophagy increases its cytotoxicity[J]. Cell Death Dis, 2018, 9: 1-15. DOI:10.1038/s41419-017-0012-9 |
| [63] |
Papke B, Murarka S, Vogel HA, et al. Identification of pyrazolopyridazinones as PDEδ inhibitors[J]. Nat Commun, 2016, 7: 11360. DOI:10.1038/ncomms11360 |
| [64] |
Leung ELH, Luo LX, Li Y, et al. Identification of a new inhibitor of KRAS‐PDEδ interaction targeting KRAS mutant nonsmall cell lung cancer[J]. Int J Cancer, 2019, 145: 1334-1345. DOI:10.1002/ijc.32222 |
| [65] |
Ruess DA, Heynen GJ, Ciecielski KJ, et al. Mutant KRAS-driven cancers depend on PTPN11/SHP2 phosphatase[J]. Nat Med, 2018, 24: 954-960. DOI:10.1038/s41591-018-0024-8 |
| [66] |
Zeng M, Xiong Y, Safaee N, et al. Exploring targeted degradation strategy for oncogenic KRASG12C[J]. Cell Chem Biol, 2020, 27: 19-31.e16. DOI:10.1016/j.chembiol.2019.12.006 |
| [67] |
Minko T. Nanoformulation of BRD4-degrading PROTAC: improving druggability to target the 'undruggable' MYC in pancreatic cancer[J]. Trends Pharmacol Sci, 2020, 41: 684-686. DOI:10.1016/j.tips.2020.08.008 |
| [68] |
Duan YC, Zhai XY, Qin WP, et al. Advances in the treatment of cancer by PROTACs[J]. Acta Pharm Sin (药学学报), 2017, 52: 1801-1810. |
| [69] |
Guo ZR. Examples of biology-driven drug design[J]. Acta Pharm Sin (药学学报), 2020, 55: 1707-1725. |
 2021, Vol. 56
2021, Vol. 56


