2. 中国中医科学院中药研究所, 北京 100700
2. Institute of Chinese Meteria Medica, China Academy of Chinese Medical Sciences, Beijing 100700, China
C2H2型基因家族是目前研究最为详尽的锌指家族之一, 在真核生物中分布广泛并能调控基因的表达[1]。C2H2型锌指蛋白通常含有X2-Cys-X(2-4)-Cys-X12-His-X(3-5)-His构成的保守结构域(Cys: 半胱氨酸, His: 组氨酸, X: 任意氨基酸), 根据氨基酸残基数量和位置的差异, 可以将锌指蛋白转录因子分为C2H2、C2HC、C2HC5、C2C2、CCCH、C3HC4、C4、C4HC3、C6和C8等类型[2-4]。Takatsuji等[5]首次在矮牵牛中发现植物C2H2型锌指蛋白EPF1。根据研究报道, 目前已在多种植物中进行了C2H2基因家族成员的分析, 如拟南芥中有176个C2H2基因, 烟草、番茄、黄瓜、水稻、杨树中分别含有118、92、120、189、109个C2H2家族成员[6-11]。植物C2H2型转录因子在植物生长发育、干旱胁迫、盐胁迫、冷胁迫、外源激素胁迫等方面发挥作用, 也在DNA识别、RNA包装、转录激活、凋亡调控以及蛋白质折叠和组装中发挥重要的作用[1, 12]。拟南芥中的RBE基因参与花原基的早期分化, 影响花瓣的生长发育过程[13]。水稻中的OsMSR15基因诱导其生长发育过程中温度胁迫及干旱胁迫下的基因表达[14]。番茄中的SICZFP1基因调控冷胁迫下的基因表达, 是提高作物耐寒性的一个关键基因; SlZF2基因通过影响番茄中花青素和丙二醛的积累, 进而改变叶和花的形态[15, 16]。
大麻(Cannabis sativa L.) 是大麻科(Cannabinaceae) 大麻属(Cannabis) 一年生草本植物, 是一种重要的药用经济作物[17]。大麻的种植已有数千年的历史, 被一些亚洲和欧洲国家广泛栽培, 在治疗慢性神经性疼痛等方面发挥重要作用[18, 19]。大麻中含有许多酚萜类、萜类以及黄酮类等药用成分[20]。大麻素为大麻特有的酚萜类化合物, 主要通过脂肪酸途径和萜类途径(图 1) 生成大麻素生物合成的主要前体大麻萜酚酸(cannabigerolic acid, CBGA), 再经酶催化作用合成终产物大麻二酚(cannabidiol, CBD) 和四氢大麻酚(tetrahydrocannabinol, THC)[21]。
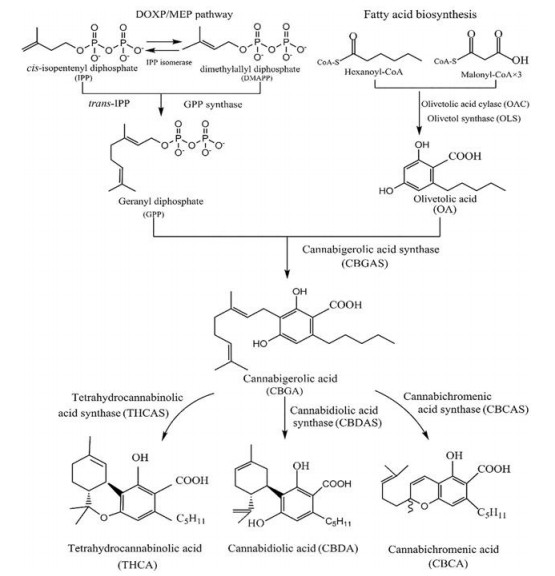
|
Figure 1 Synthetic pathways of cannabinoids |
THC具有较强的致瘾性和致幻性, 具有抗癫痫、抗炎及镇痛的作用, 但也会引发焦虑、抑郁等精神疾病[22-26]。CBD类大麻素主要富集在雌性大麻的花及苞片的有柄腺毛的囊腔中, 可以抑制高剂量THC引发的焦虑, 降低认知障碍和不适感, 并有治疗精神分裂症的潜力[27-30]。
大麻种植历史悠久, 具有较高的经济价值和药用价值[31]。因其含有非致幻性成分CBD等和致幻性成分THC等, 不同行业及学者对其进行不同程度的划分, Li等[32]明确了大麻种质资源管理三级分类体系, 即医用大麻(THC > 0.3%)、药用大麻(THC < 0.3%, CBD含量高) 和工业大麻(THC < 0.3%, CBD含量低), 其中我国法律明令禁止种植THC > 0.3%的大麻。以基因家族角度解析大麻次生代谢产物合成途径, 挖掘功能基因对大麻素生物合成的调控机制, 继而培育低THC高CBD含量的大麻品种是现阶段生物信息学研究热点。本文基于大麻的基因组数据及转录组数据, 对其C2H2转录因子的基因信息、染色体定位情况、进化关系、保守基序、共线性关系等进行分析, 探讨C2H2型转录因子对次生代谢的调控作用, 并对可能参与大麻素合成的基因进行了预测, 为大麻与C2H2型转录因子的后续研究及培育高CBD低THC含量的优质大麻品种提供理论依据。
材料与方法材料本研究所应用的大麻的基因组数据(GCA_900626175.1) 来源于NCBI (https://www.ncbi.nlm.nih.gov/) 数据库[33], 品种为CRBRx的雌株。拟南芥C2H2基因的数据来自Englbrecht等[6]的研究, 拟南芥基因组数据(GCA_000005425.2) 来自NCBI数据库。9个不同品种雌花同一批次的转录组数据(PRJNA498707) 获取于NCBI数据库[34], 这9个品种分别为Mama Thai (MT)、White Cookies (WC)、Canna Tsu (CT)、Black Lime (BL)、Terple (T)、Cherry Chem (CC)、Black Berry Kush (BB)、Sour Diesel (SD) 和Valley Fire (VF)。大麻品种Diku (名称为Dinamed Kush CBD Autoflowering) 是Purple Kush和Dinamed Autoflowering CBD杂交而得的雌性植株, 其苞片、茎、花、和叶的转录组数据(NCBI Accession No: 苞片: SAMN16122880~SAMN16122882; 茎: SAMN16122883~SAMN16122885; 花: SAMN16122886~SAMN16122888; 叶: SAMN16122889~SAMN16122891) 来自本课题组。番茄SlZF2转录因子数据来自NCBI数据库(HQ738351)。
C2H2基因家族成员获取及理化性质分析 利用TBtools软件[35]提取大麻全部蛋白序列, 将蛋白序列文件在PlantTFDB网站进行C2H2基因家族成员的预测, 并通过HMMSCAN (https://www.ebi.ac.uk/Tools/hmmer/search/phmmer) 网站去除不含C2H2锌指结构域的家族成员, 通过TBtools软件得到大麻C2H2基因家族的全部蛋白序列。利用ExPASy (http://web.expasy.org/protparam/) 在线工具对大麻C2H2锌指蛋白的分子质量、氨基酸大小、等电点、不稳定系数、蛋白质疏水性等理化性质进行预测。采用WoLFPSORT网站(https://wolfpsort.hgc.jp/) 在线预测大麻C2H2锌指蛋白的亚细胞定位状况。利用TBtools软件确定不同的大麻C2H2基因家族成员在染色体上的分布情况, 并进行可视化分析。
C2H2基因家族成员基因结构及Motif分析 利用NCBI (https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi) 网站的CD-search功能对大麻C2H2基因家族成员的保守结构域进行分析。通过MEME (http://meme-suite.org/tools/meme) 网站进行保守基序(Motif) 分析, 识别的Motif数设置为10。将大麻C2H2基因家族成员的进化树、Motif以及保守结构域利用TBtools软件合并处理并可视化分析。
C2H2基因家族成员系统进化分析及共线性分析 利用MEGA-X软件采用邻接法(Neigh-Joining, NJ), Bootstrap值设置为1 000, 将大麻与拟南芥的C2H2型锌指蛋白合并构建NJ树, 并用Evolview (https://www.evolgenius.info/evolview) 在线网站对其美化。利用TBtools软件对大麻、拟南芥的共线性关系进行可视化分析。
C2H2基因家族成员顺式作用元件分析 利用在线网站PlantCARE (http://bioinformatics.psb.ugent.be/webtools/plantcare/html/) 对大麻C2H2基因启动子上游2 000 bp的序列进行顺式作用元件分析, 并将结果用TBtools可视化。
C2H2基因家族成员表达模式分析 利用课题组自测的杂交品种Diku的雌花、叶、苞片、茎的转录组数据以及9个不同品种雌花的数据[34]进行大麻C2H2基因的表达模式分析, 并利用TBtools软件绘制热图, 进行可视化分析。
实时荧光定量PCR分析 本实验对转录组数据中表达量差异的CsC2H2-1、CsC2H2-5、CsC2H2-19、CsC2H2-24进行实时荧光定量PCR (quantitative real-time PCR, qRT-PCR) 验证, 内参基因选择EF1a, 利用NCBI设计引物(表 1)。实验样品为大麻Diku品种的花、苞片、叶和茎, 每组实验3个重复, 采用WARYONG试剂盒提取样品RNA, 用TransGen试剂盒进行cDNA合成, 以及FOREVER STAR试剂盒进行实时荧光定量PCR分析。反应程序为: 95 ℃酶激活5 min, 95 ℃变性30 s, 56 ℃退火20 s, 72 ℃延伸1 s, 循环次数40。仪器为Rotor-Gene Q (QIAGEN, 德国)。CsC2H2s的相对表达量采用2–ΔΔCt法计算。
| Table 1 Quantitative primers of CsC2H2s |
通过PlantTFDB数据库和HMMSCAN网站的对比及结果分析, 共从大麻基因组中鉴定出30个大麻C2H2基因家族成员。根据TBtools软件将大麻C2H2基因家族成员进行染色体分布可视化分析(图 2), 并根据基因在染色体上的分布情况, 依次将30个大麻C2H2基因家族成员命名为CsC2H2-1~CsC2H2-30。观察发现, 经鉴定出的大麻C2H2基因家族成员在大麻的9条染色体上(NC1~9) 呈现出不均匀分布的情况, 其中在NC1号染色体上分布的成员较多, 共包含6个基因, 在NC5和NC8号染色体上分布最少, 仅有一个家族成员。有5组基因簇呈现在大麻的4条染色体上, 依次为NC1号中的CsC2H2-3和CsC2H2-4, NC2号中的CsC2H2-7和CsC2H2-8, NC3号中的CsC2H2-11和CsC2H2-12, NC7号中的CsC2H2-22和CsC2H2-23以及CsC2H2-25和CsC2H2-26。
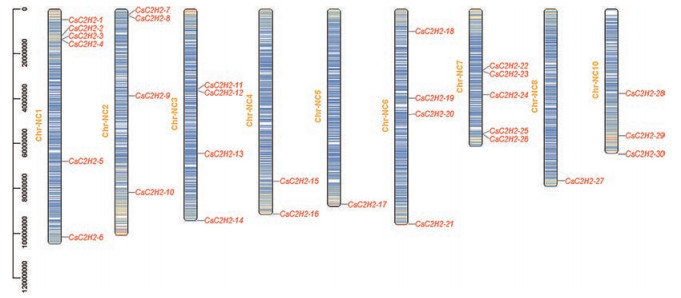
|
Figure 2 Chromosome locations of CsC2H2 genes. NC1, 2, 3, 4, 5, 6, 7, 8 and 10 of chromosomes represent NC_044370.1, NC_044371.1, NC_0443 72.1, NC_044373.1, NC_044374.1, NC_044375.1, NC_044376.1, NC_044377.1, NC_044379.1 |
大麻C2H2基因家族的信息特征及理化性质分析(表 2、3) 显示, 氨基酸大小介于138~635个氨基酸之间, 理论等电点介于5.85~9.52, 蛋白质分子质量介于15 909.48~68 445.53 Da。不稳定系数均大于40, 且GRAVY均为负值, 该结果表明大麻的C2H2蛋白均为不稳定的亲水蛋白。亚细胞定位分析显示, 所有成员均定位在细胞核中, 但一些成员有可能也在细胞质、细胞外基质、叶绿体以及过氧化物酶体中发挥作用。
| Table 2 Information of CsC2H2 gene family |
| Table 3 Physical and chemical properties of CsC2H2 gene family. N indicates nucleus, C indicates cytoplasm, E indicates extracell, H indicates chloroplast, and P indicates peroxisome |
结合大麻C2H2基因家族的Motif、UTR (untranslated region, 非编码区)、CDS、内含子以及进化关系, 对基因结构进行可视化分析(图 3)。其中, 所有CsC2H2成员中均存在Motif 1, 其次分别有22个、16个和15个成员中存在Motif 2、Motif 5和Motif 7。CsC2H2-16、CsC2H2-22、CsC2H2-23、CsC2H2-24只含有Motif 1, CsC2H2-13所含保守基序最多。CsC2H2-2、CsC2H2-6、CsC2H2-20、CsC2H2-25和CsC2H2-26所含保守基序相似, 以上5个成员在系统进化关系上(图 4) 同属A1a亚家族, 这可能与其行使某些特定的功能有关。在被研究的30个大麻C2H2基因家族成员中, 10个CsC2H2成员含有内含子, 内含子数目为1~5, 内含子数目较少。
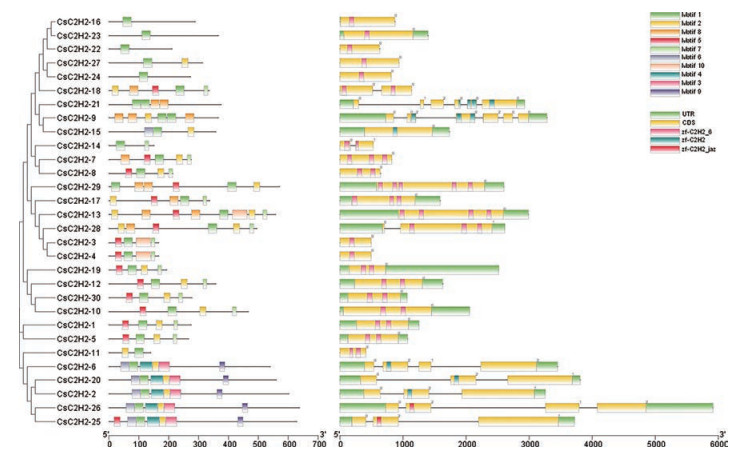
|
Figure 3 Genetic structure and conserved motif analysis of CsC2H2 |
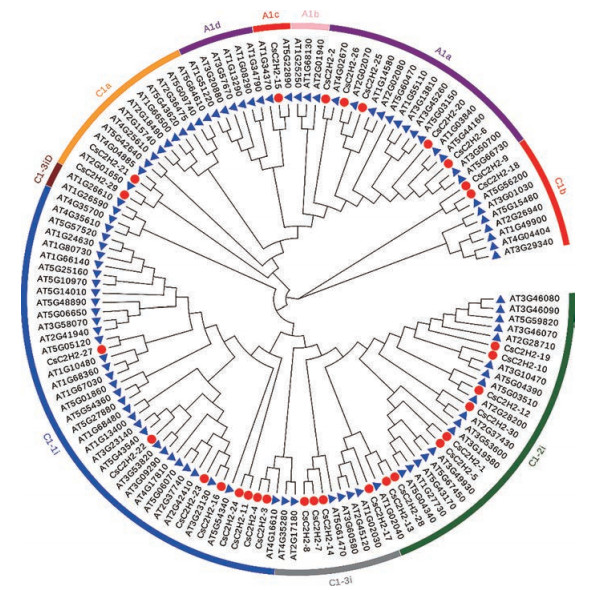
|
Figure 4 Phylogenetic tree of the C2H2 zinc finger protein of C. sativa (●) and Arabidopsis thaliana (▲) |
通过MEME网站对30个大麻C2H2锌指蛋白保守基序进行研究, 将10个Motif区块进行可视化分析(图 5), 其中Motif 1含有CX2CX3FX5LX2HX3H结构域, 分布于大麻C2H2锌指家族的所有成员中, 表明Motif 1是CsC2H2锌指结构域发挥作用的关键, 其次Motif 2、Motif 5和Motif 7保守基序分布较多, 丰富了CsC2H2锌指结构域的种类。

|
Figure 5 Ten conserved motifs of the CsC2H2 zinc finger protein |
通过对大麻和拟南芥的C2H2家族成员的系统进化研究, 利用MEGA-X软件以邻接法(neighbor joining, NJ) 构建系统进化树(图 4), 根据Englbrecht等[6]的分类方法进行分类, 系统进化关系显示大麻的C2H2家族可分为10个亚家族, 分别为A1a、A1b、A1c、A1d、C1a、C1b、C1-1i、C1-2i、C1-3i和C1-3iD, 其中A1a、A1c、C1a、C1b、C1-1i、C1-2i和C1-3i中分别含有5、1、2、2、8、8、4个大麻C2H2家族成员, 其他亚家族中无CsC2H2成员。大麻C2H2型转录因子与拟南芥C2H2型转录因子在系统进化上的同源关系表明, 两者可能在某些生物学功能上起着相同或相似的作用。番茄SlZF2蛋白与CsC2H2-1、CsC2H2-5同源关系较近(图 6)。

|
Figure 6 Unrooted neighbor-joining phylogenetic tree of SlZF2 and CsC2H2 |
大麻和拟南芥中, 共发现13条存在共线性关系的基因(图 7), 分别为CsC2H2-1/AT3G19580、CsC2H2-3/AT2G17180、CsC2H2-3/AT4G35280、CsC2H2-6/AT5G66730、CsC2H2-10/AT3G10470、CsC2H2-10/AT5G04390、CsC2H2-12/AT5G03510、CsC2H2-16/AT3G23130、CsC2H2-17/AT4G16610、CsC2H2-19/AT5G59820、CsC2H2-25/AT1G14580、CsC2H2-25/AT2G02070和CsC2H2-29/AT5G15480。大麻与拟南芥在共线性分析中有同源关系的基因, 在系统发育中同样为近缘关系, 表明基因在进化的过程中具有一定的保守性, 其可能以相同或相似的遗传形式作用于植物体。

|
Figure 7 The collinear relationship between C. sativa and Arabidopsis thaliana. Red indicates the distribution of CsC2H2 genes on chromosomes, and blue indicates the collinearity between C. sativa and Arabidopsis thaliana |
对基因家族成员的启动子序列(基因上游2 000 bp) 进行顺式作用元件预测, 结果(图 8) 显示, 大麻C2H2基因家族30个成员中全部含有光响应元件, 且光响应元件数目最多, 数目最少的为缺氧特异性诱导响应元件, 只存在于CsC2H2-20, 其次为类黄酮生物合成基因调控元件, 存在于CsC2H2-7和CsC2H2-8。该基因家族还含有茉莉酸甲酯诱导响应元件、赤霉素响应元件、脱落酸响应元件等多种顺式作用元件, 可见该基因家族顺式作用元件种类繁多、功能多样。
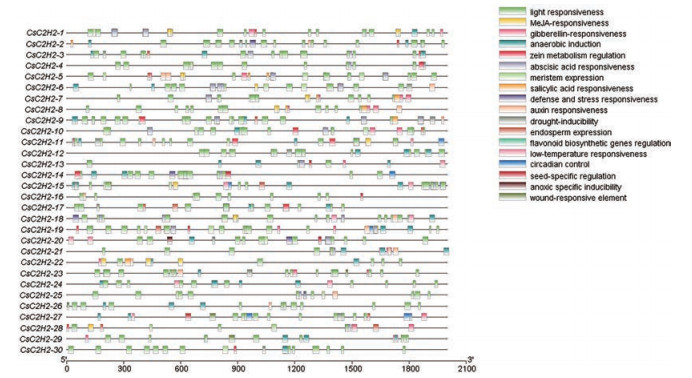
|
Figure 8 The cis-acting elements within the promoters of CsC2H2 |
通过对课题组自测的Diku品种以及源自数据库中的9个不同品种雌花的转录组数据进行聚类分析, 构建大麻中C2H2基因表达模式的热图(图 9A、B), 在Diku品种的聚类分析中(图 9A), CsC2H2-1、CsC2H2-5和CsC2H2-19表达量高于其他基因, 说明以上三者可能参与了Diku品种的生长发育过程, 并且其在苞片中表达量均为最高, 说明其可能在Diku品种苞片的生长发育过程中发挥关键作用。CsC2H2-24在Diku品种的叶中特异性表达, 说明该基因可能只调控叶的生长发育。CsC2H2-3、CsC2H2-4、CsC2H2-7、CsC2H2-8、CsC2H2-11、CsC2H2-16、CsC2H2-18、CsC2H2-23、CsC2H2-27在花、叶、苞片和茎中均没有表达, 推测以上未表达的基因可能是环境依赖型转录因子或者在Diku品种某一特定的生长发育过程中参与表达。CsC2H2在9个不同品种及大麻Diku品种雌花的表达中(图 9B), CsC2H2-1、CsC2H2-5和CsC2H2-19同样表达量最高; CsC2H2-3、CsC2H2-4、CsC2H2-11、CsC2H2-18、CsC2H2-27未参与表达, 其他基因表达量相对低水平呈现。
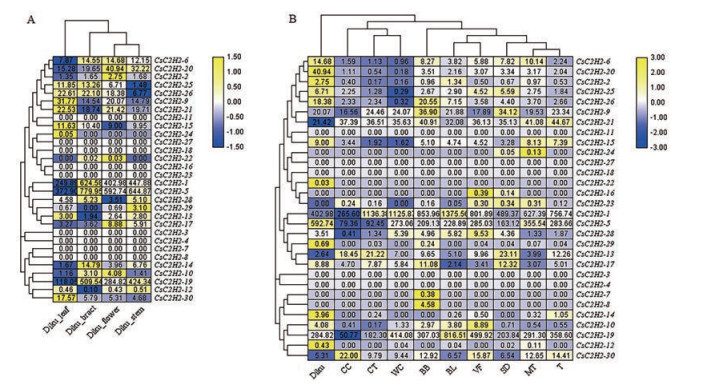
|
Figure 9 Expression analysis of CsC2H2 genes. A: CsC2H2 gene expression patterns in leaf, bract, flower, and stem of Diku variety; B: Expression of CsC2H2 in female flowers of ten different varieties. (Diku: Dinamed Kush CBD Autoflowering; CC: Cherry Chem; CT: Canna Tsu; WC: White Cookies; BB: Black Berry Kush; BL: Black Lime; VF: Valley Fire; SD: Sour Diesel; MT: Mama Thai; T: Terple) |
利用qRT-PCR检测CsC2H2-1、CsC2H2-5、CsC2H2-19、CsC2H2-24在大麻Diku品种不同组织部位的表达情况, 结果(图 10) 显示, CsC2H2-1、CsC2H2-5、CsC2H2-19在大麻Diku品种各组织部位的表达情况与转录组趋势略有不同, 但整体来说这三个基因在花、苞片和茎中的表达量高于在叶中的表达, 且CsC2H2-1、CsC2H2-5、CsC2H2-19在花和苞片中表达量极高, CsC2H2-24表达量极低甚至不表达, 证明qRT-PCR结果与转录组结果一致。

|
Figure 10 The relative expression levels of the CsC2H2s in different tissues |
本研究基于生物信息学分析, 对大麻C2H2基因家族的染色体定位情况、进化关系、保守基序、共线性关系等可视化构图并分析。C2H2型转录因子广泛存在于真核生物中, 研究表明, C2H2基因可能在植物的生长发育、水分胁迫及次生代谢等方面发挥重要的作用, 基于大麻基因组数据分析, 共鉴定出30个CsC2H2基因家族成员, 分布在大麻的9条染色体上, 各成员之间理化性质存在差异。保守基序分析显示, Motif 1在各个成员中均有体现, 在大麻Motif 1及Motif 2结构中, 存在QALGGH基序, 该基序主要存在于植物蛋白中, 在C2H2锌指结构域中相对较为保守, QALGGH基序对DNA结合活性至关重要, 并且也在植物的生长发育过程中发挥关键作用[4, 36, 37]。
在大麻染色体中, 有5组基因簇串联重复, 表明了C2H2基因在进化过程中自身发生了复制。大麻与拟南芥间的共线性分析显示, 大麻的某些C2H2基因在进化的过程中与拟南芥的基因存在着同源关系, 可见C2H2基因在进化的过程中存在一定的功能保守性。AZF2 (AT3G19580) 基因与拟南芥水分胁迫反应有关, 可见其同源基因CsC2H2-1也可能在大麻中发挥相似作用[38]。
转录组结果分析显示, CsC2H2-1、CsC2H2-5和CsC2H2-19在Diku品种及9个不同品种雌花中均表达显著, 表明其为大麻生命活动过程中的关键转录因子, 可能参与次生代谢产物的合成过程。qRT-PCR结果验证了以上三者在花及苞片中表达显著, 其他未参与表达的基因可能是环境依赖型转录因子或者在大麻某一特定的生长发育过程中参与表达, 但具体表达形式还有待进一步分析。大麻素属于酚萜类化合物, 其生物合成途径包括酚类化合物与萜类化合物。酚类化合物中的黄酮类及其衍生物具有抗肿瘤、抗衰老、提高机体免疫等药理作用[39, 40]。花青素是酚类中的类黄酮物质, 是广泛存在于植物体内的一类次级代谢产物[41]。拟南芥中的ZAT6 (AT5G04340) 基因[42]正向调节花青素和总黄酮的浓度; SlZF2基因[16]在番茄的生长发育进程中广泛表达, 易受盐胁迫及干旱胁迫的诱导, 该C2H2基因通过影响花青素和丙二醛的积累, 进而改变叶和花的形态。在系统进化关系上, CsC2H2-1、CsC2H2-5与ZAT6及SlZF2同源关系较高且在大麻苞片的腺毛中表达量最高, 由此表明CsC2H2-1、CsC2H2-5可能参与大麻次生代谢过程中酚类化合物的合成, 进而影响大麻素的合成。SlZF2基因亦有通过脱落酸生物信号传导影响植株发育的作用, 且对基因成员上游2 000 bp的启动子序列的顺式作用元件预测显示, CsC2H2-1、CsC2H2-5均含有脱落酸响应元件, 说明大麻可能受脱落酸信号介导次生代谢产物的合成[16]。
本研究利用生物信息学对大麻的C2H2基因家族进行分析, 验证了CsC2H2-1、CsC2H2-5、CsC2H2-19在大麻Diku品种花及苞片中表达显著, 证明以上基因在植物的生长发育过程及次生代谢调控中发挥重要作用, 有助于更加全面地了解基因家族的生物学功能, 为培育高CBD、低THC的优质大麻品种及C2H2型基因家族的功能研究提供理论支撑。
作者贡献: 刘美琦负责文章撰写及修改; 孙伟负责文章的选题; 孟祥霄、万会花负责实验材料的收集; 刘廷霞负责qRT-PCR实验部分; 孙嘉莹、王震指导文章撰写并提出修改意见; 米要磊负责实验设计及论文审阅; 马伟指导论文写作及修改。
利益冲突: 所有作者均声明不存在利益冲突。
| [1] |
Zhang J, Liu JF, Zhao TT, et al. Research progress of C2H2 zinc finger protein in plant[J]. Mol Plant Breed (分子植物育种), 2018, 16: 427-433. |
| [2] |
Pabo CO, Peisach E, Grant RA. Design and selection of novel Cys2 His2 zinc finger proteins[J]. Annu Rev Biochem, 2001, 70: 313-340. DOI:10.1146/annurev.biochem.70.1.313 |
| [3] |
Moore M, Ullman C. Recent developments in the engineering of zinc finger proteins[J]. Brief Funct Genomic Proteomic, 2003, 1: 342-355. DOI:10.1093/bfgp/1.4.342 |
| [4] |
Berg JM, Shi Y. The galvanization of biology: a growing appreciation for the roles of zinc[J]. Science, 1996, 271: 1081-1085. DOI:10.1126/science.271.5252.1081 |
| [5] |
Takatsuji H, Mori M, Benfey PN, et al. Characterization of a zinc finger DNA-binding protein expressed specifically in Petunia petals and seedlings[J]. EMBO J, 1992, 11: 241-249. DOI:10.1002/j.1460-2075.1992.tb05047.x |
| [6] |
Englbrecht CC, Schoof H, Böhm S. Conservation, diversification and expansion of C2H2 zinc finger proteins in the Arabidopsis thaliana genome[J]. BMC Genomics, 2004, 5: 39. DOI:10.1186/1471-2164-5-39 |
| [7] |
Yang ML, Chao JT, Wang DW, et al. Genome-wide identification and expression profiling of the C2H2-type zinc finger protein transcription factor family in tobacco[J]. Hereditas (遗传), 2016, 38: 337-349. |
| [8] |
Hu JH, Wang DW, Yang ML, et al. Identification and bioinformatics analysis of C2H2 zinc finger protein in tomato[J]. Jiangsu Agric Sci (江苏农业科学), 2018, 46: 23-27. |
| [9] |
Zhang S. Genome-wide Identification and Characterization of Zinc Finger Proteins in Cucumber (黄瓜基因组内锌指蛋白的鉴定及特征分析)[D]. Beijing: Chinese Academy of Agricultural Sciences, 2017.
|
| [10] |
Agarwal P, Arora R, Ray S, et al. Genome-wide identification of C2H2 zinc-finger gene family in rice and their phylogeny and expression analysis[J]. Plant Mol Biol, 2007, 65: 467-485. DOI:10.1007/s11103-007-9199-y |
| [11] |
Liu Q, Wang Z, Xu X, et al. Genome-wide analysis of C2H2 zinc-finger family transcription factors and their responses to abiotic stresses in Poplar (Populus trichocarpa)[J]. PLoS One, 2015, 10: e0134753. DOI:10.1371/journal.pone.0134753 |
| [12] |
Laity JH, Lee BM, Wright PE. Zinc finger proteins: new insights into structural and functional diversity[J]. Curr Opin Struct Biol, 2001, 11: 39-46. DOI:10.1016/S0959-440X(00)00167-6 |
| [13] |
Takeda S, Matsumoto N, Okada K. Rabbit ears, encoding a superman-like zinc finger protein, regulates petal development in Arabidopsis thaliana[J]. Development, 2004, 131: 425-434. DOI:10.1242/dev.00938 |
| [14] |
Zhang X, Zhang B, Li MJ, et al. OsMSR15 encoding a rice C2H2-type zinc finger protein confers enhanced drought tolerance in transgenic Arabidopsis[J]. J Plant Biol, 2016, 59: 271-281. DOI:10.1007/s12374-016-0539-9 |
| [15] |
Zhang X, Guo X, Lei C, et al. Overexpression of SlCZFP1, a novel TFIIIA-type zinc finger protein from tomato, confers enhanced cold tolerance in transgenic Arabidopsis and rice[J]. Plant Mol Biol Rep, 2011, 29: 185-196. DOI:10.1007/s11105-010-0223-z |
| [16] |
Hichri I, Muhovski Y, Žižková E, et al. The solanum lycopersicum zinc finger2 cysteine-2/histidine-2 repressor-like transcription factor regulates development and tolerance to salinity in tomato and Arabidopsis[J]. Plant Physiol, 2014, 164: 1967-1990. DOI:10.1104/pp.113.225920 |
| [17] |
Li J, Zhu XW, Wan HH, et al. Progress on chemical constituents and analytical methods of cannabinoids in Cannabis sativa[J]. Chin Tradit Herb Drugs (中草药), 2020, 51: 6414-6425. |
| [18] |
Schultes RE, Klein WM, Plowman T. Cannabis: an example of taxonomic neglect[J]. Bot Museum Leaflets, Harvard Univ, 1974, 23: 337-367. |
| [19] |
Ware MA, Wang T, Shapiro S, et al. Smoked cannabis for chronic neuropathic pain: a randomized controlled trial[J]. CMAJ, 2010, 182: E694-E701. DOI:10.1503/cmaj.091414 |
| [20] |
Flores-Sanchez IJ, Choi YH, Verpoorte R. Metabolite analysis of Cannabis sativa L. by NMR spectroscopy[J]. Methods Mol Biol, 2012, 815: 363-375. |
| [21] |
Preedy V. The Biosynthesis of Cannabinoids[M]//Handbook of Cannabis and Related Pathologies.Chapter 2. San Diego: Academic Press, 2017: 13-23.
|
| [22] |
Chang XW, Chen WQ, Sun Y, et al. Potential medical use and risks of cannabis[J]. Chin J Drug Depend (中国药物依赖性杂志), 2020, 29: 161-168. |
| [23] |
Rácz I, Nent E, Erxlebe E, et al. CB1 receptors modulate affective behaviour induced by neuropathic pain[J]. Brain Res Bull, 2015, 114: 42-48. DOI:10.1016/j.brainresbull.2015.03.005 |
| [24] |
Romero-Sandoval EA, Kolano AL, Alvarado-Vázquez PA. Cannabis and cannabinoids for chronic pain[J]. Curr Rheumatol Rep, 2017, 19: 67. DOI:10.1007/s11926-017-0693-1 |
| [25] |
Cristino L, Bisogno T, Di Marzo V. Cannabinoids and the expanded endocannabinoid system in neurological disorders[J]. Nat Rev Neurol, 2019, 16: 9-29. |
| [26] |
Chopra K, Arora V. An intricate relationship between pain and depression: clinical correlates, coactivation factors and therapeutic targets[J]. Expert Opin Ther Targets, 2014, 18: 159-176. DOI:10.1517/14728222.2014.855720 |
| [27] |
Zhang JQ, Chen SL, Wei GF, et al. Cultivars breeding and production of non-psychoactive medicinal cannabis with high CBD content[J]. China J Chin Mater Med (中国中药杂志), 2019, 44: 4772-4780. |
| [28] |
Rodriguez de Fonseca F, Cebeira M, Hernández ML, et al. Changes in brain dopaminergic indices induced by perinatal exposure to cannabinoids in rats[J]. Dev Brain Res, 1990, 51: 237-240. DOI:10.1016/0165-3806(90)90280-C |
| [29] |
Bergamaschi MM, Queiroz RH, Chagas MH, et al. Cannabidiol reduces the anxiety induced by simulated public speaking in treatment-naive social phobia patients[J]. Neuropsychopharmacology, 2011, 36: 1219-1226. DOI:10.1038/npp.2011.6 |
| [30] |
Levin R, Peres FF, Almeida V, et al. Effects of cannabinoid drugs on the deficit of prepulse inhibition of startle in an animal model of schizophrenia: the SHR strain[J]. Front Pharmacol, 2014, 5: 10. |
| [31] |
Luo X, Reiter MA, d'Espaux L, et al. Complete biosynthesis of cannabinoids and their unnatural analogues in yeast[J]. Nature, 2019, 567: 123-126. DOI:10.1038/s41586-019-0978-9 |
| [32] |
Li QS, Meng Y, Chen SL. A new Cannabis germplasm classification system and research strategies of non-psychoactive medicinal cannabis[J]. China J Chin Mater Med (中国中药杂志), 2019, 44: 4309-4316. |
| [33] |
Grassa CJ, Wenger JP, Dabney C, et al. A complete Cannabis chromosome assembly and adaptive admixture for elevated cannabidiol (CBD) content[J]. BioRxiv, 2018. DOI:10.1101/458083 |
| [34] |
Zager JJ, Lange I, Srividya N, et al. Gene networks underlying cannabinoid and terpenoid accumulation in Cannabis[J]. Plant Physiol, 2019, 180: 1877-1897. DOI:10.1104/pp.18.01506 |
| [35] |
Chen C, Xia R, Chen H, et al. TBtools, a toolkit for biologists integrating various HTS-data handling tools with a user-friendly interface[J]. BioRxiv, 2018. DOI:10.1101/289660 |
| [36] |
Lee BM, Xu J, Clarkson BK, et al. Induced fit and "lock and key" recognition of 5 S RNA by zinc fin-gers of transcription factor IIIA[J]. J Mol Biol, 2006, 357: 275-291. DOI:10.1016/j.jmb.2005.12.010 |
| [37] |
Takatsuji H. Zinc-finger transcription factors in plants[J]. Cell Mol Life Sci, 1998, 54: 582-596. DOI:10.1007/s000180050186 |
| [38] |
Sakamoto H, Araki T, Meshi T, et al. Expression of a subset of the Arabidopsis Cys2/His2-type zinc-finger protein gene family under water stress[J]. Gene, 2000, 248: 23-32. DOI:10.1016/S0378-1119(00)00133-5 |
| [39] |
Li XP, Yu MX, Kuang TR, et al. Research progress on the antitumor effect of flavonoid derivatives[J]. Acta Pharm Sin (药学学报), 2021, 56: 913-923. |
| [40] |
Li MR, Zhou YZ, Du GH, et al. Research progress about the anti-aging effect and mechanism of flavonoids from traditional Chinese medicine[J]. Acta Pharm Sin (药学学报), 2019, 54: 1382-1391. |
| [41] |
Harborne JB, Williams CA. Anthocyanins and other flavonoids[J]. Nat Prod Rep, 2001, 18: 310-333. DOI:10.1039/b006257j |
| [42] |
Shi H, Liu G, Wei Y, et al. The zinc-finger transcription factor ZAT6 is essential for hydrogen peroxide induction of anthocyanin synthesis in Arabidopsis[J]. Plant Mol Biol, 2018, 97: 165-176. DOI:10.1007/s11103-018-0730-0 |
 2021, Vol. 56
2021, Vol. 56


