线粒体(mitochondria) 常被称为细胞的“能量工厂”, 是真核生物细胞进行能量合成的主要场所, 通过自身分裂实现增殖。线粒体参与细胞内电解质稳态平衡的调控、离子跨膜转运、信号转导和细胞凋亡等重要的生物学过程。
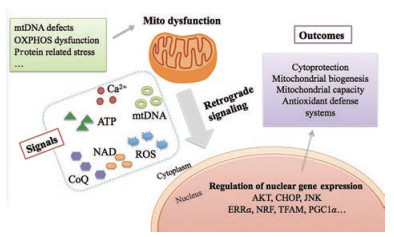
线粒体基因突变、呼吸链缺陷、氧化磷酸化失调和线粒体膜的改变等因素均会造成线粒体功能障碍, 从而导致病变。线粒体的生物发生和线粒体稳态需要核基因的表达及线粒体-细胞核对话来进行调控。一方面取决于核基因转录及核至线粒体信号(正向信号, anterograde signaling) 调控途径, 另一方面线粒体则通过细胞内信号分子, 例如Ca2+、线粒体DNA (mitochondrial DNA, mtDNA)、活性氧(reactive oxygen species, ROS)、腺苷三磷酸(adenosine triphosphate, ATP)、辅酶Q (coenzyme Q, CoQ) 以及烟酰胺腺嘌呤二核苷酸(nicotinamide adenine dinucleotide, NAD) 等将线粒体异常以及细胞代谢变化信号呈递给细胞核(逆行信号, retrograde signaling), 细胞核通过动员一系列核转录因子引起重要信号通路的激活、线粒体转录及线粒体生物合成等过程(图 1)。因此, 线粒体-细胞核相互作用对于维持细胞稳态至关重要。
|
Figure 1 General view of mitochondrial signals and outcomes of retrograde signaling pathways. Mitochondrial nuclear retrograde signaling is mainly mediated by Ca2+, mtDNA, ROS, and other signal molecules, while the nucleus mobilizes a series of nuclear transcription factors to implement response measures. Diverse mitochondrial signals elicit varied responses. Ca2+: Calcium ion; ROS: Reactive oxygen species; mtDNA: Mitochondrial DNA; AKT: Protein kinase B; CHOP: C/EBP homologous protein; JNK: C-Jun N-terminal kinase; ERRα: Estrogen receptor-related receptor α; NRF: Nuclear respiratory factor; TFAM: Mitochondrial transcription factor A; PGC-1α: Peroxisome proliferator-activated receptor γ coactivator-1α |
细胞核基因组通过编码大量的线粒体蛋白在维持线粒体正常功能中发挥着重要作用, 线粒体基因组则编码部分构成氧化磷酸化组件的重要蛋白与细胞核蛋白共同作用[1]。由于细胞核基因组编码生命活动中的主要蛋白质, 之前关于信号调控的研究多针对细胞核向线粒体, 最近的研究发现线粒体也可以逆向调控核内基因的表达而调控生命活动, 被称为线粒体逆行信号(mitochondrial retrograde signaling), 简称逆行信号。
最早关于线粒体影响核基因表达的研究是在1987年, 研究[2]发现两种核基因表达受mtDNA缺陷影响, 因而确定了从线粒体到细胞核信号传导的存在。目前识别的主要逆行信号是柠檬酸合酶2 (citrate synthase 2, CIT2), 在缺乏mtDNA的细胞中CIT2的表达明显增加[3]。使用CIT2启动子的报告基因, 发现多种调控因子共同参与调控逆行信号通路[4-8], 这些发现大大加快了研究者对于线粒体逆行信号的理解。
1.2 线粒体逆行信号的传导机制线粒体是目前发现的哺乳动物细胞中唯一含有独立基因组的细胞器, 自身可以表达多种蛋白。其基因组长度只有16.5 kb, 共包含37个基因, 其中22个编码转移核糖核酸(tRNA), 2个编码核糖体核糖核酸(12S和16S rRNA), 13个编码多肽[9]。由于其蛋白合成能力有限, 主要线粒体蛋白由细胞核基因编码, 在细胞质中合成, 定向转运到线粒体。线粒体逆行信号的发生存在于线粒体-细胞核之间, 其中包括线粒体RNA、DNA的迁移及蛋白质的转运。
1.2.1 线粒体RNA及DNA的分布和迁移最早认为核酸不能透过线粒体外周的双层膜, 而较新研究显示某些RNA可以透过线粒体。MicroRNA (miRNA) 是一种碱基较少的非编码RNA分子, 一般约为18~25个核苷酸。研究显示, miRNA分子能够进入线粒体并与线粒体内的mRNA分子相互作用, 从而降低其稳定性, 影响线粒体基因的表达。计算机分析也揭示了mtDNA编码miRNA的前体序列[10, 11]。Barrey等[10]报道了存在于线粒体中的4种胞质miRNA, 包括Let-7b、miR-365、preLet-b和pre-miR-302a等。Sripada等[11]基于小RNA测序确定了人细胞系HEK293线粒体中的428个已知miRNA和196个新miRNA, 以及HeLa细胞线粒体中的327个已知miRNA和13个未知miRNA。RNA的线粒体转位说明存在一种能够跨线粒体膜转运RNA的系统, 研究显示该系统的关键要素是多核苷酸磷酸化酶(polynucleotide phosphorylase, PNPase)[12]。PNPase和人花斑抑制因子3 (human suppressor of variegation 3, hSuv3) 解旋酶在线粒体内形成了降解复合物, 参与RNA代谢[13, 14]。此外, 线粒体也可以输出由线粒体基因组编码的RNA。
线粒体DNA在进化过程中产生了多种核转位途径, 使核基因组中的许多位点被大小不等的线粒体DNA片段占据。在人类基因组中, 有755个这样的基因座, 称为核线粒体DNA (nuclear mitochondrial DNA, NUMT)[15]。对抗番木瓜环斑病毒(Papaya ringspot virus, PRSV) 转基因番木瓜的基因组变异研究中, 发现2 764个NUMT连接位点[16]。有观点认为NUMT是无活性的假基因, 然而研究显示NUMT也出现在癌细胞中[15], 具体作用仍需进一步探索。
1.2.2 线粒体蛋白质的合成与转运多数线粒体蛋白质由核基因组编码, 在细胞质中合成, 然后被转运进线粒体。这些蛋白质大多数在N端具有数十个氨基酸形成的信号序列, 可被线粒体蛋白质输入系统识别, 依靠线粒体外膜上的转位酶(translocase of outer mitochondrial membrane, TOM) 转运及线粒体内膜上的转位酶(translocase of inner mitochondrial membrane, TIM) 定位到内膜和基质[17]。某些蛋白质以前仅仅被认为存在于胞质或细胞核, 没有线粒体信号序列, 但是现有研究显示它们也存在于线粒体中, 如TP53蛋白和端粒酶z蛋白2 (Ago2) 等[18]。
近年来, 人们对mtDNA编码蛋白表达的调控机制提出了许多新见解。在酵母中, 缺少用于所有基因表达的通用调节途径, 随着线粒体的不断进化, 线粒体核糖体上合成的单个蛋白质的表达由特定的调控因子调节。通过分析呼吸缺陷型酵母菌发现翻译的激活与复合物Ⅳ (细胞色素氧化酶) 的亚基环氧化酶(cyclooxygenase, Cox) 1~3和复合物Ⅲ的细胞色素b (cytochrome b, Cytb) 相关[19]。在Cox亚基的合成中共鉴定了7种激活剂, 在Cytb合成中共鉴定了4种激活剂, 它们的功能是多种多样且重叠的, 其中一些稳定mRNA, 一些与核糖体结合, 另一些支持装配。线粒体热休克蛋白70 (mitochondrial heat shock protein 70, mtHsp70) 和血红素b进一步参与包括转录物的结合、与核糖体的结合、稳定和保护新生链、释放到膜双层中以及折叠与其他亚基组装的过程中。新的研究提出了线粒体调控基因表达(mitochondrial organization of gene expression, MIOREX) 复合体[19]是由大量蛋白质和RNA形成的一个超分子结构, 介导了从转录后RNA成熟到蛋白质翻译的众多过程, 用于协调线粒体基因的表达以及外部和自我调节控制, 这一复合体为研究线粒体基因表达开辟了新的视野。
2 线粒体逆行信号触发因素、传导途径及调控因子 2.1 线粒体逆行信号触发因素逆行信号传导途径由特定的线粒体信号触发, 继而传递给一个或多个转录因子, 最终到达细胞核。作为线粒体的主要产物, ATP是逆行信号通路显著的触发因子。在酵母中, 逆行信号蛋白(retrograde signaling protein, Rtg) 通路依赖于ATP水解并具有ATP特异性, 这表明mtDNA缺失可能通过降低ATP浓度激活了逆行信号, 使梅克尔综合征蛋白1 (Meckel syndrome type 1 protein, Mks1p) 和逆行信号蛋白2p (retrograde signaling protein 2p, Rtg2p) 缔合, Rtg1p和Rtg3p发生核易位[20]。在衰老过程中, 线粒体膜电位下降会触发逆行信号[21], 但其电位下降引发信号传递的机制尚不明确。在哺乳动物细胞中, 线粒体膜电位的破坏也是逆行信号转导的主要诱因, 损害线粒体Ca2+的吸收并导致细胞质中游离Ca2+的升高[22], 继而激活Ca2+依赖性蛋白激酶C (protein kinase C, PKC)、钙调蛋白依赖性蛋白激酶Ⅳ (calcium/calmodulin-dependent protein kinase Ⅳ, CamK Ⅳ)、cAMP反应元件结合蛋白(cAMP response element binding protein, CREB) 和C/EBP同源蛋白(CHOP)[23]。Arnold等[24]发现线粒体内膜上ATP酶与多种细胞活动相关(intermembrane space-ATPases associated with diverse cellular activities, i-AAA) 的蛋白酶结构域, 存在酵母线粒体逃逸蛋白1 (yeast mitochondrial escape protein 1, YME1) 基因的缺失, 造成膜间的肽生成障碍, 引发呼吸链的生物发生并诱导具有调控线粒体基因表达功能的核基因表达。因而在某些情况下, 线粒体衍生肽也参与逆行信号的激活。
2.2 线粒体逆行信号传导途径及调控因素 2.2.1 酵母中线粒体逆行信号传导途径及调控因子在酵母中, 线粒体功能障碍导致细胞内ATP浓度降低, 继而引发线粒体逆行信号传导途径。最早在酵母的研究中发现存在线粒体到细胞核的逆行信号分子, 包括细胞色素氧化酶6 (COX Ⅵ)、CIT2和Rtg[25], 细胞可以通过调节核基因的表达来影响线粒体的功能。Rtg途径的信号传导主要是通过Rtg1、Rtg2和Rtg3来完成, Rtg1和Rtg3是具有遗传保守性的碱性螺旋环螺旋亮氨酸拉链(bHLH/Zip) 结构的转录因子, 它们形成异二聚体并与靶基因的启动子区域结合, Rtg3p是一种磷蛋白, Rtg3p的激活与其部分脱磷酸作用与Rtg1p的核易位有关[26], Rtg2p在N末端具有ATP结合域的细胞质蛋白, 属于肌动蛋白/Hsp70/糖激酶超家族[27]。Rtg3p去磷酸化和Rtg1/3p的核转位需要Rtg2p, Rtg2p的ATP结合域的完整性对其功能至关重要[7]。Rtg途径作为线粒体与细胞核间信号的“传感器”, 调控细胞内各种生物合成及生理代谢活动, 进而维持酵母正常生长与繁殖。
2.2.2 哺乳动物中线粒体逆行信号传导途径及调控因子目前线粒体逆行信号对mtDNA突变、线粒体未折叠蛋白反应(mitochondrial unfolded protein response, UPRmt) 等刺激的响应只是在酿酒酵母和线虫得到了较好诠释[28]。越来越多的相关研究已经在各种动物体内和体外细胞模型展开, 这些研究证明在哺乳动物细胞逆行信号也是一种活跃的、重要的信号转导机制[29]。Marusich等[30]在哺乳动物细胞中首次识别了逆行信号介导蛋白, 包括细胞色素C氧化酶亚基Va (COX Va) 和琥珀酸脱氢酶(SDH)。
线粒体逆行信号途径存在于细胞代谢过程中, 并且发挥调控作用使细胞能够适应环境变化。正常情况下, 机体对营养成分的吸收通过顺行信号通路调控, 细胞参与氧化代谢, 细胞质中发生糖酵解, 将葡萄糖代谢成丙酮酸, 然后通过线粒体三羧酸循环(tricarboxylic acid cycle, TCA) 将丙酮酸氧化为二氧化碳, 经过电子传递等一系列呼吸作用分解产生能量。研究表明, 在营养过剩情况下, 细胞会增加对营养的吸收, 发生有氧糖酵解, 将丙酮酸转化为乳酸, 细胞通过激活过氧化物酶体增殖物受体共激活因子-1α (peroxisome proliferator-activated receptor γ coactivator-1α, PGC-1α) 影响表观遗传动力学来调控细胞代谢, 协调生化反应和线粒体活动[31]。
线粒体中间代谢产物通过逆行信号传导和转录程序激活调控核基因表达, 其表观遗传学发生改变, 包括DNA的甲基化和组蛋白的翻译后修饰。在动物细胞中, 核基因的转录取决于组蛋白的乙酰化修饰, 该修饰所需要的乙酰辅酶A来自于线粒体。Wellen等[32]发现, 组蛋白乙酰化需要三磷酸腺苷柠檬酸裂解酶(adenosine triphosphate citrate lyase, ACL) 的参与, 敲除ACL大大减少了细胞核中乙酰辅酶A的储备, 降低了组蛋白乙酰化水平, 并且在糖酵解基因周围降低明显, 导致其转录下调抑制糖酵解。组蛋白的脱乙酰化也表现出代谢影响。组蛋白的脱乙酰化通过Ⅰ类和Ⅱ类组蛋白脱乙酰基酶(histone deacetylases, HDACs) 和Ⅲ类组蛋白脱乙酰基酶(sirtuins) 来实现。Sirtuins以NAD作为脱乙酰的辅助因子, 通过发生还原反应生成还原型烟酰胺腺嘌呤二核苷酸(nicotinamide adenine dinucleotide, NADH), 二者比值可影响sirtuins活性。在富含碳水化合物的饮食中, 生长因子刺激细胞对葡萄糖摄取, 能量的产生通过糖酵解进行。在这种情况下, NAD/NADH比值降低, 抑制细胞核中Sirt1、6、7表达, Sirt1和Sirt6活性降低会促进组蛋白乙酰化。作为线粒体逆行信号介导蛋白和转录激活因子, G蛋白途径抑制因子2 (G-protein pathway suppressor 2, GPS2) 可以通过在细胞核中募集目的基因启动子调节组蛋白H3K9去甲基化和RNA POL2激活发挥作用。
尽管在哺乳动物中还没有找到酵母Rtg基因的直系同源物, 但是一些转录因子包括核因子-κB (nuclear transcription factor-κB, NF-κB)、CREB、叉头状转录因子O1 (forkhead box O 1, FoxO1)、激活转录因子2、4、6 (activating transcription factor 2、4、6, ATF2、4、6)、缺氧诱导因子1α (hypoxia inducible factor-1α, HIF-1α) 和E2F转录因子1 (E2F1) 等在功能上与酵母中Rtg1/Rtg3、线虫中ATFS-1存在部分重叠, 说明逆行信号在很多物种都是普遍存在的, 其传导途径存在功能保守性[33]。目前认为参与逆行信号通路的转录因子有X盒结合蛋白1 (X-box binding protein 1, XBP1)、早期生长反应因子1 (early growth response 1, EGR1)、线粒体转录因子A (mitochondrial transcription factor A, TFAM)、哺乳动物雷帕霉素靶蛋白(mammalian target of rapamycin, mTOR)、C端结合蛋白1 (C-terminal-binding proteins 1, CtBP1) 活化T细胞的核因子(nuclear factor of activated T cell, NFAT)、PGC-1α、PGC-1相关辅助因子(PGC-1 related coactivator, PRC)、蛋白激酶B (protein kinase B, AKT)、细胞外信号调节激酶(extracellular signal-regulated kinase, ERK)、c-Jun N末端激酶(c-Jun N-terminal kinase, JNK)、视黄醇X受体α基因(retinoid X receptor alpha gene, RXRA) 和磷脂酰肌醇-3-羟激酶(phosphatidylinositol-3-hydroxykinase, PI3K) 等。参与逆向信号传导信号通路有Ras-ERK-ETS、c-JUN-JNK、PI3K-AKT、PGC-1α-PRC等[34-36]。这些发现揭示了线粒体-核通讯的多种调节途径, 表明逆行信号传导途径是哺乳动物线粒体应激反应的关键组成部分。
3 线粒体逆行信号可能为多种疾病的治疗提供新策略维持线粒体正常功能对于细胞的存活至关重要, 线粒体产生ATP为细胞供能, 参与生化信号的传递, 是细胞自然凋亡和代谢的核心。研究显示, 线粒体逆行信号通路存在于氧化应激、电子传递链损伤、细胞凋亡与自噬、血管紧张和炎症反应等病理过程中, 而线粒体功能障碍作为线粒体逆行信号通路的关键启动信号之一, 与多种疾病的发生发展关系密切, 包括心血管疾病、代谢性疾病、神经退行性疾病和肿瘤等。
3.1 肿瘤线粒体功能障碍与多种恶性肿瘤的发生有密切关系, 线粒体代谢紊乱、Ca2+含量异常和线粒体基因缺陷等均与肿瘤的生物学行为相关。线粒体逆行信号传导通路参与并影响肿瘤发生发展。研究发现, 存在线粒体功能障碍的肿瘤细胞中, 线粒体功能表现为明显的补偿机制上调[37], 过量的超氧化物产生, 电子传递链的超载可能导致转移性肿瘤产生[38]。
正常细胞中miR-663介导线粒体至细胞核的逆行信号, 并受ROS的调控, 其中线粒体调节miR-663启动子甲基化, miR-663调节核编码的氧化磷酸化系统(oxidative phosphorylation system, OXPHOS) 亚基及装配因子的表达和超复合物的稳定性。然而, 功能异常的线粒体会诱导氧化应激, 增加甲基转移酶的活性, 导致miR-663启动子超甲基化, miR-663表达降低, OXPHOS基因的表达也降低, 超复合物的稳定性被破坏, 并降低OXPHOS的酶促活性, 这些共同促进了肿瘤的发生, 因而miR-663是作为控制线粒体至细胞核逆行信号调控细胞癌变发展的关键[39]。
研究表明, 转移性表型肿瘤受mtDNA突变的影响, mtDNA突变由于ROS信号增强而导致mtDNA耗竭[40, 41]。线粒体基因组编码的新多肽——humanin (HN) 具有细胞保护、抗凋亡作用, HN表达水平在皮肤T细胞淋巴瘤中显著增加[42], 表明其在癌细胞形成和存活中发挥作用。
线粒体RNA在线粒体外的功能研究发现, 线粒体16S rRNA分子[43, 44]在17种不同肿瘤类型的肿瘤细胞中显著下调, 有研究者[45]提出是RNA依赖性RNA聚合酶改变基质使这种杂交RNA下调导致肿瘤细胞凋亡, 具体机制尚不明确。但通过差异表达可将正常细胞与肿瘤细胞区分开来, 为肿瘤生物学和诊断提供新思路。
3.2 神经退行性疾病线粒体稳态的破坏与神经退行性疾病的发生发展关系密切。C型12S核糖体核糖核酸的线粒体开放阅读框(mitochondrial open reading frame of the 12S ribosomal ribonucleic acid type c, MOTS-c) 是2015年被鉴定出的一种线粒体衍生肽, 具有独特的生物活性, 能通过自分泌和旁分泌的方式发挥多种生理作用。MOTS-c可在代谢应激(如葡萄糖缺乏和氧化应激) 时依赖5'-腺苷单磷酸激活蛋白激酶(AMP-activated kinase, AMPK) 转位到细胞核, 并直接调节核基因表达以促进细胞内稳态[46, 47], 它由线粒体12S rRNA基因内的ORF编码, 在日本人群中的研究观察到其序列变异与寿命呈相关性[48]。
HN是由24个氨基酸组成的残基肽, 由编码16S核糖体RNA的线粒体基因内的短ORF编码, 于2001年在一名患有阿尔茨海默病(Alzheimer's disease, AD) 的患者中被发现, 后来研究发现该肽存在于许多组织和体液中。HN具有减少氧化应激、抑制神经变性和炎症的发生、增加细胞对胰岛素的敏感性以及抑制细胞凋亡、细胞保护等作用, 还直接作用于线粒体, 促进ATP合成增加[49, 50]。
在AD患者的脑组织中观察到UPRmt转录反应, 表明该线粒体应激反应途径被激活[51]。散发性和家族性AD患者额叶前皮质中UPRmt基因、热休克蛋白10 (Hsp10)、热休克蛋白60 (Hsp60) 以及酪蛋白水解蛋白酶蛋白水解亚基(CLpP) 的mRNA表达均增加。此外, AD患者有AKT和ROS信号改变以及HIF-1α水平降低的报道[52], 研究显示靶向线粒体逆行信号传导机制可以改善神经元功能[33]。
研究发现, Ras-ERK-ETS信号为果蝇神经系统中的一种新型线粒体逆行信号通路, 抑制Ras-ERK-ETS信号可以部分抑制逆转录反应, 以减轻神经元线粒体功能障碍[36]。HIF-1α是线粒体功能障碍的关键调节因子, 通过敲低HIF-1α靶向逆向信号可恢复帕金森病(Parkinson's disease, PD) 患者的神经元功能和寿命[53, 54]。研究显示, 通过逆行信号mTOR通路靶向自噬治疗PD具有极好的应用前景[55]。基于PD小鼠模型探讨UPRmt和综合应激反应(integrated stress response, ISR) 之间的串扰关系, 发现敲除靶点CHOP对神经具有保护作用。神经系统疾病模型和临床研究明确证实, 线粒体逆行信号的组成性激活足可诱发神经元功能障碍和死亡, 抑制逆行信号能减轻神经元功能缺陷或神经退行性病变。
3.3 心血管疾病线粒体是细胞的能量代谢中心, 其通过氧化磷酸化产生能量以满足心脏的高能量需求。以呼吸链功能异常和ATP合成障碍为特征的线粒体功能紊乱是许多心血管疾病的发病原因。线粒体的代谢与ROS、钙离子、一氧化氮及激素等信号分子相关, 这些信号分子发生变化, 如ROS增加和ATP减少等, 影响线粒体及细胞功能进而引发应激反应, 因此, 在疾病早期通过对逆行信号的调节进行线粒体质量控制、减少线粒体功能障碍和氧化应激成为缺血性心血管疾病治疗的新思路。
线粒体融合蛋白2 (mitofusin2, Mfn2) 广泛分布于线粒体外膜和线粒体与内质网结合膜上, 负责参与调控线粒体形态和功能, 是具有多重功能的动力素相关蛋白。同时, Mfn2作为一个重要的细胞内信号分子, 参与细胞的代谢、增殖及凋亡等过程, Mfn2与高血压、冠状动脉腔内成形术后再狭窄、动脉粥样硬化、心肌肥厚及心肌氧化损伤等多种心血管疾病的病理生理过程密切相关[56], 推测Mfn2可能是心血管疾病的一个重要的分子标志和治疗靶点。
研究显示, 在大鼠心肌细胞中, miR-181c特异性抑制COX1基因mRNA的翻译[57]。在鼠成肌细胞中, miR-1可刺激线粒体基因ND1和COX1的翻译, 增加基因表达[58], 表明胞质RNA能够调节线粒体基因的表达, 但这种现象的机制尚未明确。线粒体是ROS的主要来源, 细胞质ROS的升高经ROS/人硫氧还蛋白相互作用蛋白(thioredoxin-interacting protein, TXNIP)/核苷酸结合寡聚化结构域样受体蛋白3 (NOD-like receptor family pyrin domain-containing 3, NLRP3) 炎症小体通路诱导NLRP3炎症小体激活, 抑制线粒体活性氧的产生可以抑制NLRP3炎症小体的活化[59]。有证据表明, mtDNA进入细胞浆后通过环磷酸鸟苷-腺苷酸合成酶(cyclic GMP-AMP synthase, cGAS)-干扰素刺激基因(stimulator of interferon gene, STING) 信号通路促进NLRP3炎症小体的活化[60]。
长链非编码RNA预测心脏重构(long intergenic non-coding RNA predicting cardiac remodeling, LIPCAR) 作为重要的线粒体转录物之一, 它是2个反义线粒体RNA片段的融合体, 来源于CytB和COX2基因。LIPCAR由781个核苷酸组成, 线粒体DNA上编码它的基因座距离大约8 kb。LIPCAR出现在缺血性心脏病患者的外周血中, 推断可能是重要的预后因素[61]。
3.4 代谢性疾病常见的代谢性疾病包括超重/肥胖、糖调节受损/糖尿病、高血压、血脂异常、代谢综合征和高尿酸血症等。研究显示, 线粒体逆行信号可通过多种调控因子参与胰岛素抵抗、肥胖和2型糖尿病等代谢性疾病病理过程中。目前, 提高胰岛素敏感性和干预生活方式的临床措施都是基于促进线粒体生物发生和氧化代谢来进行治疗。
研究显示, GPS2下调与人类肥胖以及肥胖脂肪组织内炎症的形成有关, 通过调节脂质储存和能量消耗之间的平衡, 促进线粒体生物发生, 以支持组织特定的功能。此外, GPS2在通过抑制泛素信号传导来调节细胞生长, 在炎症、代谢和应激抗性的多种途径中起重要作用[29]。MOTS-c肽不仅在神经退行性疾病中发挥影响作用, 同时也具有调节细胞对胰岛素的敏感性作用[48]。Mfn2不仅与多种心血管疾病的病理生理过程密切相关, 还可以通过调节物质代谢影响糖尿病和胰岛素抵抗[62]。
4 结论与展望线粒体和细胞核两个遗传系统在生命活动中紧密协调。随着研究的不断深入, 证明线粒体-核信号在线粒体疾病、代谢综合征和退行性疾病等病理进程中扮演着重要角色。线粒体逆行信号基于复杂的信号通路影响多种调控因子, 从而调控细胞核基因表达, 协调多种质量控制机制, 包括蛋白质质量控制、线粒体动力学、线粒体生物发生和凋亡等来维持细胞稳态, 参与调控细胞生长、新陈代谢和衰老等生命活动。
目前对于线粒体逆行信号的研究仍处于起步阶段, 认识比较局限, 有待进一步深入探索。借助体内逆行信号调节因子功能缺失和功能获得模型对包括人类在内的不同种属进行研究, 分析相关调控因素, 为研究者提供了对逆行信号调节机制深入研究的思路。确定启动线粒体信号的调节因子, 以及这些信号是如何被线粒体基因调控影响核基因表达, 对于针对线粒体功能障碍的疾病开发潜在的生物标志物有着重要意义。在不远的将来, 干预线粒体逆行信号的关键调节因子可能成为疾病治疗的一种新策略, 尤其对于治疗线粒体功能障碍相关的疾病具有广阔的前景。
作者贡献: 姜瑜负责文章的资料收集整理、撰写以及修改完善工作; 王守宝对文章结构和具体内容进行审阅和指导以及获取研究经费; 杜冠华对文章内容及示意图进行指导。
利益冲突: 本文所有作者无任何利益冲突。
| [1] |
Liu Z, Butow RA. Mitochondrial retrograde signaling[J]. Annu Rev Genet, 2006, 40: 159-185. DOI:10.1146/annurev.genet.40.110405.090613 |
| [2] |
Parikh VS, Morgan MM, Scott R, et al. The mitochondrial genotype can influence nuclear gene expression in yeast[J]. Science, 1987, 235: 576-580. DOI:10.1126/science.3027892 |
| [3] |
Liao XS, Small WC, Srere PA, et al. Intramitochondrial functions regulate nonmitochondrial citrate synthase (CIT2) expression in Saccharomyces cerevisiae[J]. Mol Cell Biol, 1991, 11: 38-46. DOI:10.1128/MCB.11.1.38 |
| [4] |
Jia Y, Rothermel B, Thornton J, et al. A basic helix-loop-helix-leucine zipper transcription complex in yeast functions in a signaling pathway from mitochondria to the nucleus[J]. Mol Cell Biol, 1997, 17: 1110-1117. DOI:10.1128/MCB.17.3.1110 |
| [5] |
Liao X, Butow RA. RTG1 and RTG2:two yeast genes required for a novel path of communication from mitochondria to the nucleus[J]. Cell, 1993, 72: 61-71. DOI:10.1016/0092-8674(93)90050-Z |
| [6] |
Liu Z, Sekito T, Epstein CB, et al. RTG-dependent mitochondria to nucleus signaling is negatively regulated by the seven WD-repeat protein Lst8p[J]. EMBO J, 2001, 20: 7209-7219. DOI:10.1093/emboj/20.24.7209 |
| [7] |
Liu Z, Sekito T, Spirek M, et al. Retrograde signaling is regulated by the dynamic interaction between Rtg2p and Mks1p[J]. Mol Cell, 2003, 12: 401-411. DOI:10.1016/S1097-2765(03)00285-5 |
| [8] |
Sekito T, Liu Z, Thornton J, et al. RTG-dependent mitochondria-to-nucleus signaling is regulated by MKS1 and is linked to formation of yeast prion[URE3][J]. Mol Biol Cell, 2002, 13: 795-804. |
| [9] |
Sharma N, Pasala MS, Prakash A. Mitochondrial DNA: epigenetics and environment[J]. Environ Mol Mutagen, 2019, 60: 668-682. DOI:10.1002/em.22319 |
| [10] |
Barrey E, Saint-Auret G, Bonnamy B, et al. Pre-microRNA and mature microRNA in human mitochondria[J]. PLoS One, 2011, 6: e20220. DOI:10.1371/journal.pone.0020220 |
| [11] |
Sripada L, Tomar D, Prajapati P, et al. Systematic analysis of small RNAs associated with human mitochondria by deep sequencing: detailed analysis of mitochondrial associated miRNA[J]. PLoS One, 2012, 7: e44873. DOI:10.1371/journal.pone.0044873 |
| [12] |
Wang G, Shimada E, Koehler CM, et al. PNPASE and RNA trafficking into mitochondria[J]. Biochim Biophys Acta, 2012, 1819: 998-1007. DOI:10.1016/j.bbagrm.2011.10.001 |
| [13] |
Borowski LS, Dziembowski A, Hejnowicz MS, et al. Human mitochondrial RNA decay mediated by PNPase-hSuv3 complex takes place in distinct foci[J]. Nucleic Acids Res, 2013, 41: 1223-1240. DOI:10.1093/nar/gks1130 |
| [14] |
Szczesny RJ, Borowski LS, Brzezniak LK, et al. Human mitochondrial RNA turnover caught in flagranti: involvement of hSuv3p helicase in RNA surveillance[J]. Nucleic Acids Res, 2010, 38: 279-298. DOI:10.1093/nar/gkp903 |
| [15] |
Ramos A, Barbena E, Mateiu L, et al. Nuclear insertions of mitochondrial origin: database updating and usefulness in cancer studies[J]. Mitochondrion, 2011, 11: 946-953. DOI:10.1016/j.mito.2011.08.009 |
| [16] |
Fang J, Wood AM, Chen Y, et al. Genomic variation between PRSV resistant transgenic SunUp and its progenitor cultivar Sunset[J]. BMC Genomics, 2020, 21: 398. DOI:10.1186/s12864-020-06804-7 |
| [17] |
Liu AY, Hou XS, Ding Y. Advances of antineoplastic agents based on mitochondrial targeting mechanism[J]. Acta Pharm Sin (药学学报), 2017, 52: 879-887. |
| [18] |
Yogev O, Pines O. Dual targeting of mitochondrial proteins: mechanism, regulation and function[J]. Biochim Biophys Acta, 2011, 1808: 1012-1020. DOI:10.1016/j.bbamem.2010.07.004 |
| [19] |
Neupert W. Mitochondrial gene expression: a playground of evolutionary tinkering[J]. Annu Rev Biochem, 2016, 85: 65-76. DOI:10.1146/annurev-biochem-011116-110824 |
| [20] |
Zhang F, Pracheil T, Thornton J, et al. Adenosine triphosphate (ATP) is a candidate signaling molecule in the mitochondria-to-nucleus retrograde response pathway[J]. Genes (Basel), 2013, 4: 86-100. DOI:10.3390/genes4010086 |
| [21] |
Borghouts C, Benguria A, Wawryn J, et al. Rtg2 protein links metabolism and genome stability in yeast longevity[J]. Genetics, 2004, 166: 765-777. DOI:10.1093/genetics/166.2.765 |
| [22] |
Amuthan G, Biswas G, Ananadatheerthavarada HK, et al. Mitochondrial stress-induced calcium signaling, phenotypic changes and invasive behavior in human lung carcinoma A549 cells[J]. Oncogene, 2002, 21: 7839-7849. DOI:10.1038/sj.onc.1205983 |
| [23] |
Butow RA, Avadhani NG. Mitochondrial signaling: the retrograde response[J]. Mol Cell, 2004, 14: 1-15. DOI:10.1016/S1097-2765(04)00179-0 |
| [24] |
Arnold I, Wagner-Ecker M, Ansorge W, et al. Evidence for a novel mitochondria-to-nucleus signalling pathway in respiring cells lacking i-AAA protease and the ABC-transporter Mdl1[J]. Gene, 2006, 367: 74-88. DOI:10.1016/j.gene.2005.09.044 |
| [25] |
Butow RA, Docherty R, Parikh VS. A path from mitochondria to the yeast nucleus[J]. Philos Trans R Soc Lond B Biol Sci, 1988, 319: 127-133. DOI:10.1098/rstb.1988.0037 |
| [26] |
Sekito T, Thornton J, Butow RA. Mitochondria-to-nuclear signaling is regulated by the subcellular localization of the transcription factors Rtg1p and Rtg3p[J]. Mol Biol Cell, 2000, 11: 2103-2115. DOI:10.1091/mbc.11.6.2103 |
| [27] |
Bork P, Sander C, Valencia A. An ATPase domain common to prokaryotic cell cycle proteins, sugar kinases, actin, and hsp70 heat shock proteins[J]. Proc Natl Acad Sci U S A, 1992, 89: 7290-7294. DOI:10.1073/pnas.89.16.7290 |
| [28] |
da Cunha FM, Torelli NQ, Kowaltowski AJ. mitochondrial retrograde signaling: triggers, pathways, and outcomes[J]. Oxid Med Cell Longev, 2015, 2015: 482582. |
| [29] |
Cardamone MD, Tanasa B, Cederquist CT, et al. Mitochondrial retrograde signaling in mammals is mediated by the transcriptional cofactor GPS2via direct mitochondria-to-nucleus translocation[J]. Mol Cell, 2018, 69: 757-772.e7. DOI:10.1016/j.molcel.2018.01.037 |
| [30] |
Marusich MF, Robinson BH, Taanman JW, et al. Expression of mtDNA and nDNA encoded respiratory chain proteins in chemically and genetically-derived Rho0 human fibroblasts: a comparison of subunit proteins in normal fibroblasts treated with ethidium bromide and fibroblasts from a patient with mtDNA depletion syndrome[J]. Biochim Biophys Acta, 1997, 1362: 145-159. DOI:10.1016/S0925-4439(97)00061-6 |
| [31] |
Martinez-Pastor B, Cosentino C, Mostoslavsky R. A tale of metabolites: the cross-talk between chromatin and energy metabolism[J]. Cancer Discov, 2013, 3: 497-501. DOI:10.1158/2159-8290.CD-13-0059 |
| [32] |
Wellen KE, Hatzivassiliou G, Sachdeva UM, et al. ATP-citrate lyase links cellular metabolism to histone acetylation[J]. Science, 2009, 324: 1076-1080. DOI:10.1126/science.1164097 |
| [33] |
Hunt RJ, Bateman JM. Mitochondrial retrograde signaling in the nervous system[J]. FEBS Lett, 2018, 592: 663-678. DOI:10.1002/1873-3468.12890 |
| [34] |
Jones AW, Yao Z, Vicencio JM, et al. PGC-1 family coactivators and cell fate: roles in cancer, neurodegeneration, cardiovascular disease and retrograde mitochondria-nucleus signalling[J]. Mitochondrion, 2012, 12: 86-99. DOI:10.1016/j.mito.2011.09.009 |
| [35] |
Granat L, Hunt RJ, Bateman JM. Mitochondrial retrograde signalling in neurological disease[J]. Philos Trans R Soc Lond B Biol Sci, 2020, 375: 20190415. DOI:10.1098/rstb.2019.0415 |
| [36] |
Duncan OF, Granat L, Ranganathan R, et al. Ras-ERK-ETS inhibition alleviates neuronal mitochondrial dysfunction by reprogramming mitochondrial retrograde signaling[J]. PLoS Genet, 2018, 14: e1007567. DOI:10.1371/journal.pgen.1007567 |
| [37] |
Gasparre G, Romeo G, Rugolo M, et al. Learning from oncocytic tumors: why choose inefficient mitochondria[J]. Biochim Biophys Acta, 2011, 1807: 633-642. DOI:10.1016/j.bbabio.2010.08.006 |
| [38] |
Porporato PE, Payen VL, Perez-Escuredo J, et al. A mitochondrial switch promotes tumor metastasis[J]. Cell Rep, 2014, 8: 754-766. DOI:10.1016/j.celrep.2014.06.043 |
| [39] |
Carden T, Singh B, Mooga V, et al. Epigenetic modification of miR-663 controls mitochondria-to-nucleus retrograde signaling and tumor progression[J]. J Biol Chem, 2017, 292: 20694-20706. DOI:10.1074/jbc.M117.797001 |
| [40] |
Ishikawa K, Hashizume O, Koshikawa N, et al. Enhanced glycolysis induced by mtDNA mutations does not regulate metastasis[J]. FEBS Lett, 2008, 582: 3525-3530. DOI:10.1016/j.febslet.2008.09.024 |
| [41] |
Kulawiec M, Owens KM, Singh KK. Cancer cell mitochondria confer apoptosis resistance and promote metastasis[J]. Cancer Biol Ther, 2009, 8: 1378-1385. |
| [42] |
Hartmann TB, Mattern E, Wiedemann N, et al. Identification of selectively expressed genes and antigens in CTCL[J]. Exp Dermatol, 2008, 17: 324-334. DOI:10.1111/j.1600-0625.2007.00637.x |
| [43] |
Villegas J, Burzio V, Villota C, et al. Expression of a novel non-coding mitochondrial RNA in human proliferating cells[J]. Nucleic Acids Res, 2007, 35: 7336-7347. DOI:10.1093/nar/gkm863 |
| [44] |
Burzio VA, Villota C, Villegas J, et al. Expression of a family of noncoding mitochondrial RNAs distinguishes normal from cancer cells[J]. Proc Natl Acad Sci U S A, 2009, 106: 9430-9434. DOI:10.1073/pnas.0903086106 |
| [45] |
Bianchessi V, Badi I, Bertolotti M, et al. The mitochondrial lncRNA ASncmtRNA-2 is induced in aging and replicative senescence in endothelial cells[J]. J Mol Cell Cardiol, 2015, 81: 62-70. DOI:10.1016/j.yjmcc.2015.01.012 |
| [46] |
Benayoun BA, Lee C. MOTS-c: a mitochondrial-encoded regulator of the nucleus[J]. Bioessays, 2019, 41: e1900046. DOI:10.1002/bies.201900046 |
| [47] |
Kim KH, Son JM, Benayoun BA, et al. The mitochondrial-encoded peptide MOTS-c translocates to the nucleus to regulate nuclear gene expression in response to metabolic stress[J]. Cell Metab, 2018, 28: 516-524.e7. DOI:10.1016/j.cmet.2018.06.008 |
| [48] |
Fuku N, Pareja-Galeano H, Zempo H, et al. The mitochondrial-derived peptide MOTS-c: a player in exceptional longevity[J]. Aging Cell, 2015, 14: 921-923. DOI:10.1111/acel.12389 |
| [49] |
Bodzioch M, Lapicka-Bodzioch K, Zapala B, et al. Evidence for potential functionality of nuclearly-encoded humanin isoforms[J]. Genomics, 2009, 94: 247-256. DOI:10.1016/j.ygeno.2009.05.006 |
| [50] |
Gong Z, Tas E, Muzumdar R. Humanin and age-related diseases: a new link[J]. Front Endocrinol (Lausanne), 2014, 5: 210. |
| [51] |
Beck JS, Mufson EJ, Counts SE. Evidence for mitochondrial UPR gene activation in familial and sporadic Alzheimer's disease[J]. Curr Alzheimer Res, 2016, 13: 610-614. DOI:10.2174/1567205013666151221145445 |
| [52] |
Tramutola A, Triplett JC, Di Domenico F, et al. Alteration of mTOR signaling occurs early in the progression of Alzheimer disease (AD): analysis of brain from subjects with pre-clinical AD, amnestic mild cognitive impairment and late-stage AD[J]. J Neurochem, 2015, 133: 739-749. DOI:10.1111/jnc.13037 |
| [53] |
Cagin U, Duncan OF, Gatt AP, et al. Mitochondrial retrograde signaling regulates neuronal function[J]. Proc Natl Acad Sci U S A, 2015, 112: E6000-E6009. DOI:10.1073/pnas.1505036112 |
| [54] |
Duncan OF, Bateman JM. Mitochondrial retrograde signaling in the Drosophila nervous system and beyond[J]. Fly (Austin), 2016, 10: 19-24. DOI:10.1080/19336934.2016.1174353 |
| [55] |
Ouyang L, Zhang L, Liu B. Autophagy pathways and key grug targets in Parkinsons's disease[J]. Acta Pharm Sin (药学学报), 2016, 51: 9-17. |
| [56] |
Zuchner S, De Jonghe P, Jordanova A, et al. Axonal neuropathy with optic atrophy is caused by mutations in mitofusin 2[J]. Ann Neurol, 2006, 59: 276-281. DOI:10.1002/ana.20797 |
| [57] |
Das S, Ferlito M, Kent OA, et al. Nuclear miRNA regulates the mitochondrial genome in the heart[J]. Circ Res, 2012, 110: 1596-1603. DOI:10.1161/CIRCRESAHA.112.267732 |
| [58] |
Zhang X, Zuo X, Yang B, et al. MicroRNA directly enhances mitochondrial translation during muscle differentiation[J]. Cell, 2014, 158: 607-619. DOI:10.1016/j.cell.2014.05.047 |
| [59] |
Sorbara MT, Girardin SE. Mitochondrial ROS fuel the inflammasome[J]. Cell Res, 2011, 21: 558-560. DOI:10.1038/cr.2011.20 |
| [60] |
Riley JS, Quarato G, Cloix C, et al. Mitochondrial inner membrane permeabilisation enables mtDNA release during apoptosis[J]. EMBO J, 2018, 37: e99238. |
| [61] |
Dorn GW. LIPCAR: a mitochondrial lnc in the noncoding RNA chain[J]. Circ Res, 2014, 114: 1548-1550. DOI:10.1161/CIRCRESAHA.114.304028 |
| [62] |
Zhang X, Wang C, Song G, et al. Mitofusion-2-mediated alleviation of insulin resistance in rats through reduction in lipid intermediate accumulation in skeletal muscle[J]. J Biomed Sci, 2013, 20: 45. DOI:10.1186/1423-0127-20-45 |
 2021, Vol. 56
2021, Vol. 56


