蛋白水解靶向嵌合体(proteolysis targeting chimeras, PROTAC)的概念最早是由Crews和Deshaies实验室于2001年提出的, 该团队成功通过PROTAC的方法实现了对甲硫氨酰氨肽酶2 (Met Ap-2)的降解[1]。此后, 针对雄激素受体(AR)和雌激素受体(ER)的PROTAC陆续出现[2]。2004年, 一种通过HIF1-α上的多肽片段招募肿瘤抑制蛋白VHL的PROTAC分子在膜透性方面有了较大的提升, 可以在细胞中较为稳定地存在[3]。在早期, 基于多肽的PROTAC分子尚未具备成药条件, 直到2008年, 第一种基于小分子E3泛素酶配体的PROTAC问世[4], 标志着小分子PROTAC研究的开端, PROTAC研究也进入了发展期。此后几年, 包括VHL配体在内的几种重要小分子配体被陆续开发, 相应的PROTAC也被成功合成。之后, 结合了卤素的卤代PROTAC (halo PROTAC)[5]以及在细胞内通过“点击技术”自组装生成的CLIPTAC分子(clickformed proteolysis targeting chimeras)[6]相继出现, 对PROTAC的研究也愈发深入且进入了分子层面。
PROTAC是一种双功能分子, 其由三部分组成: 一端为靶蛋白结合配体, 另一端为E3泛素连接酶配体, 中间则为连接链。在细胞中, PROTAC分子能够识别并选择性地结合靶蛋白, 招募特定的E3泛素连接酶, 形成“靶蛋白-PROTAC-E3泛素连接酶”三元复合物。之后, E3泛素连接酶与E2泛素结合酶共同作用, 对靶蛋白进行泛素化。由于泛素-蛋白酶体系统(ubiquitinproteasome system, UPS)是人体细胞内蛋白降解的重要途径[7], 参与细胞内80%以上蛋白质的降解, 故靶蛋白被泛素化标记后, PROTAC分子与靶蛋白、E3泛素酶分离, 泛素标记的靶蛋白就会被蛋白酶体识别并降解(图 1)。通过借助于人体本身存在的泛素化-降解蛋白生理过程, PROTAC分子可以高效且特异性地催化致病蛋白的降解, 从而达到治疗疾病的目的。
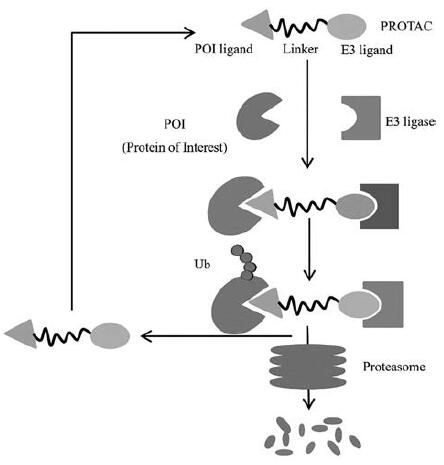
|
Figure 1 Schematic representation for targeted protein degradation by PROTAC through hijacking the E3 ubiquitin ligase |
与传统的蛋白小分子抑制剂不同, PROTAC的作用机制是事件驱动的, 无需长时间占据靶点。PROTAC在完成对一个靶蛋白的泛素化标记后, 即可脱离靶蛋白与E3泛素连接酶, 继续对下一个蛋白进行标记。因此, 在发挥降解效应过程中, 少量的PROTAC分子就可以实现对大量靶蛋白诱导降解的目的。此外, PROTAC可以产生对不同种类靶蛋白以及E3酶的选择性, 其分子设计的开放性使得PROTAC对目标蛋白降解的选择性进一步提高。更为重要的是, 与激酶类抑制剂相比, PROTAC分子对于目标蛋白的亲和力要求较低, 通过合理设计PROTAC的分子结构, 使得它在针对一些目前不可成药的靶点有着广泛的应用前景[8, 9]。
2 “靶蛋白-PROTAC-E3泛素连接酶”三元复合物的形成在PROTAC诱导靶蛋白降解的过程中, “靶蛋白-PROTAC-E3泛素连接酶”三元复合物的形成作为一个核心生物学事件, 是PROTAC对靶蛋白进行多泛素化修饰中必经的步骤。对于PROTAC诱导降解效率而言, 形成三元复合物的稳定性远比PROTAC分子与靶蛋白或是E3泛素酶结合的牢固程度重要。实验发现, 通过PROTAC分子稳定一种p38α‒PROTAC‒VHL的三元复合物, 可以成功的诱导靶蛋白的降解; 相比之下, 对照实验中的PROTAC分子单独引入具有高结合力的靶蛋白配体, 未能诱导三元复合物的形成, 没有得到良好的降解效果[10]。有研究人员通过建立数学模型来分析PROTAC诱导生成的三元复合物效应的浓度关系, 结果显示PROTAC的浓度分布是符合正态分布曲线的[11]。而这样的数学模型结果也是与实验中所观察到的“HOOK效应”[12]相一致的。“HOOK效应”即钩状效应, 本意是指由于抗原抗体比例不合适导致的假阴性现象, 即只有在两者分子比例合适的情况下反应最为充分。与此类似, 在PROTAC研究中, PROTAC分子浓度过高或过低时, 三元复合物的生成都会受到影响。
有意思的是, 靶蛋白与E3泛素酶之间的相互作用也会影响三元复合物的生成。两者之间可以产生使三元复合物更加稳定的吸引作用, 或是破坏三元复合物稳定性的排斥作用。有实验研究表明, PROTAC分子通过构成“自我折叠”, 能使得靶蛋白与E3酶配体之间产生蛋白质-蛋白质相互作用(protein-protein interaction, PPI), 形成更为稳定的三元复合物[13], 从而将“HOOK效应”对三元复合物的浓度的负面影响降到最低, 一定程度上提升PROTAC的降解效率[14]。
因此, PROTAC分子诱导降解靶蛋白的过程更多地与其催化形成的三元复合物有关。近年来, 研究人员对于三元复合物形成过程中的一些关键要素, 如靶蛋白配体、E3酶配体和连接链等, 开展了大量的研究工作并取得了重要进展, 能否形成三元复合物也逐渐成为判断PROTAC分子是否具有降解能力的重要判断标准。鉴于PROTAC分子中连接链对于三元复合物的稳定性以及PROTAC结合的选择性越来越受到关注, 本文将从连接链的长度、连接链与配体的结合位点以及连接链的化学结构三个角度来总结和讨论连接链对PROTAC降解效率影响的研究进展。
3 连接链长度对PROTAC分子的影响 3.1 连接链长度对PROTAC分子降解效率的影响在PROTAC分子连接靶蛋白和E3酶发挥作用时, 两者间会产生基于蛋白的相互作用, 这种相互作用不仅会影响PROTAC分子本身的稳定性, 还会影响PROTAC分子的降解效率。Nowak团队的研究表明, 可以通过改变PROTAC的分子结构使蛋白的弱相互作用变得更强。该研究团队以CRBN与BRD4的相互作用为例, 通过改变PORTAC分子的连接链的长度和位置, 来改变三元复合物的底物构象从而改变其相互作用强度(图 2)。实验以dBET23 (1)以及dBET6 (2)这两种连接链结合位点不同的PROTAC为起始研究对象, 分别改变了连接链的长度, 将得到的PROTAC分子与蛋白结合后进行时间分辨荧光共振能量转移分析(time-resolved fluorescence resonance energy transfer, TR-FRET), 观测蛋白间的相互作用受到的影响。结果发现, 连接链长度较短的PROTAC分子dBET57 (3)相较于dBET6 (2), 通过改变蛋白接近时的构象, 完全改变了CRBN与BRD4相互作用的残基, 极大地削弱了两者的蛋白相互作用。此外, 该研究团队在dBET23 (1)的基础上改变了连接链的原子数, 成功得到了对于一系列高度同源的BRD蛋白具有选择性的PROTAC小分子化合物ZXH-3-26 (4)。结果表明, 具有较短连接链的PROTAC分子选择性更高, 其原因是连接链长度适当缩短后, 具有较短连接链的ZXH-3-26 (4)与蛋白结合时, 产生了不同于该组设计的其他PROTAC分子的构象, 限制了BRD与CRBN间产生稳定性相互作用的构象数目, 使得三元合物形成的条件变得苛刻, 从而具有更好的选择性[15]。

|
Figure 2 Structures of PROTAC molecules with linker length-dependent degradation efficacy.(1: dBET23;2: dBET6;3: dBET57;4: ZXH-3-26;5: SNIPER (ER)-type PROTAC, n=2, 3, 4;6: Halo PROTAC3-8, n=3, 5, 0, 1, 2, 4) |
类似地, 日本的Ohoka团队合成了一系列包含不同ER配体或不同连接链长度的SNIPER (specific and non-genetic IAP-dependent protein eraser)(5), 同样观察到只有连接链中聚乙二醇聚合度为3 (n=3)的SNIPER(5)才能成功诱导蛋白间相互作用[16]。此外, Buckley研究团队在研究中合成的Halo PROTAC系列PROTAC(6)拥有不同长度的连接链。研究结果发现, 连接链更长的Halo PROTAC (n=4, 5)降解效率更高, 据推测, 连接链长度较短时(n=0, 1, 2), 会导致PROTAC分子与其底物发生负协同作用, 从而减低降解效率; 此外该组还对该现象进行进一步的推测, 他们认为PROTAC在连接链长度达到某一中间值后开始产生正协同作用, 促进蛋白间相互作用, 提高PROTAC分子的降解效率。该团队在实验中还观察到具有较长的连接链的PROTAC分子更容易发生自抑制作用, 而具有适中的长度连接链的PROTAC分子则不会, 推测在适中的长度上, 降解效果最佳的Halo PROTAC3 (n=3)与E3酶以及靶蛋白的结合可能具有较强的正面的协同作用[5]。
上述的实验研究表明, PROTAC的连接链长度变化时, 会对靶蛋白和E3酶间的相互作用产生正面或负面的影响, 过短的连接链会限制两者接近的构象, 而过长的连接链则可能会导致PROTAC分子的自抑制作用。而连接链长度可能会从某个中间值开始对蛋白间相互作用有正向的影响, 这或是PROTAC连接链优化中的一个关键点。
3.2 连接链长度对PROTAC分子选择性的影响一般而言, 改变PROTAC分子选择性的方法是替换靶蛋白配体, 但是靶蛋白配体的发现以及合成面临着较大的挑战, 并且针对同家族蛋白时, 配体的选择性欠佳。近年来的研究发现, 合理调整连接链的长度, 可以达到在同族的靶蛋白之间实现选择性的目标(图 3), 因此备受关注。例如, 在Burslem和Smith领导的科研小组利用PROTAC降解受体酪氨酸激酶(receptor tyrosine kinase, RTK)的案例中, 他们对连接链的长度对靶蛋白配体的选择性影响设计了变量实验。该实验采用了不同长度连接链的lapatinib-based PROTAC分子(7), 得到了降解EGFR (epidermal growth factor receptor)与HER2 (human epidermal growth factor receptor 2)的选择性数据。实验结果表明, 聚乙二醇聚合度为2 (n=2)的lapatinib-based PROTAC可以无差别降解EGFR以及HER2, 而聚合度为3 (n=3)者却选择性降解了EGFR而对HER2没有明显降解效果[17]。该组推测具有不同的连接链长度的PROTAC分子, 其以不同的构象与靶蛋白结合时会有不同数量和位置的相互作用位点, 进而影响PROTAC分子选择性。
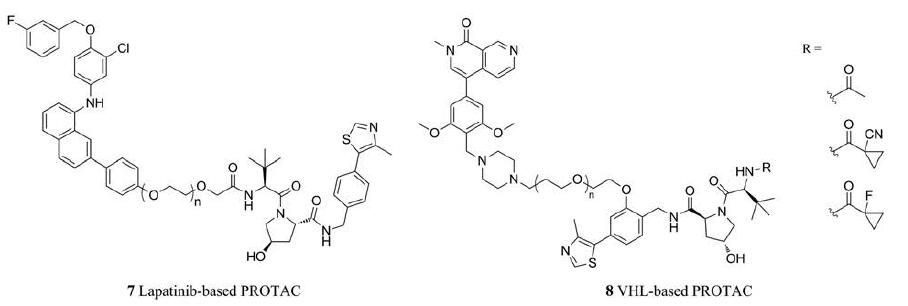
|
Figure 3 Structures of PROTAC molecules with substrate speicificity dictated by the linker length.(7: Lapatinib-based EGFR and HER2 targeting PROTAC, n=2, 3;8: VHL-based BRD7 and BRD9 targeting PROTAC, n=2, 4) |
类似地, Zoppi等[18]在设计降解BRD7和BRD9两种蛋白的PROTAC时, 发现一系列VHL-based PROTAC分子(8)对BRD9比BRD7有多至9倍的选择性降解。通过分析实验数据, 他们发现连接链的长度会影响靶蛋白泛素化降解的模式, 具有更短的连接链的PROTAC(n=2)可以更好地降解BRD9。他们推测, 其中的原因可能是连接链长度使得不同的PROTAC分子拥有不同的空间结构, 而这些不同的空间结构恰好能构成与BRD9蛋白更为匹配的空间关系, 从而产生更佳的适配性。以上来自于不同实验室的独立实验共同研究表明, 连接链长度的改变可以影响“靶蛋白-PROTAC-E3泛素连接酶”三元复合物产生的构象等因素, 影响分子对于靶蛋白和配体的选择性。
上述实验研究表明, PROTAC的连接链长度变化时, 会直接影响到PROTAC分子的空间结构和选择活性。针对特定的靶蛋白和E3酶, 只有找到最适的连接链长度后, PROTAC分子才能使蛋白间的有利相互作用最大化, 顺利地诱导三元复合物的形成, 进而完成靶蛋白泛素化。连接链长度的改变, 影响了PROTAC对不同靶蛋白泛素化的效率, 从而影响其选择性。
4 连接链结合位点对PROTAC分子的影响 4.1 连接链结合位点对PROTAC降解效率及选择性的影响除连接链长度以外, 连接链的结合位点对于PROTAC靶蛋白的选择性也存在影响(图 4)。通常, 配体上潜在的连接链结合位点的数量有限。对于与靶蛋白结合的配体的晶体结构的研究表明, 当配体与目标蛋白结合时, 暴露在溶剂中的只有部分位点。例如, 在对于VHL配体的结构性研究中, Buckley团队[19]调整VHL配体上的取代基时, 得到了将碳末端(R1)或是氮末端(R2)暴露在溶剂中的VHL配体(9), 而这些暴露在溶剂中的末端都是潜在的连接链结合位点。这种基于对配体与靶蛋白的共晶结构的研究, 是当下寻找潜在连接链结合位点的常用方法[20, 21]。而在不同的位点引入连接链, 则会影响PROTAC分子对靶蛋白的选择性。Donoghue等[22]在优化靶向p38α蛋白的PROTAC分子的连接链的过程中, 合成出两组与CRBN配体沙利度胺(thalidomide)的结合位点不相同的NR3c (10)与NR4 (11)。在两种细胞内的实验结果表明, 在仅改变与沙利度胺结合位点的情况下, 两种PROTAC的降解能力出现显著的差异。
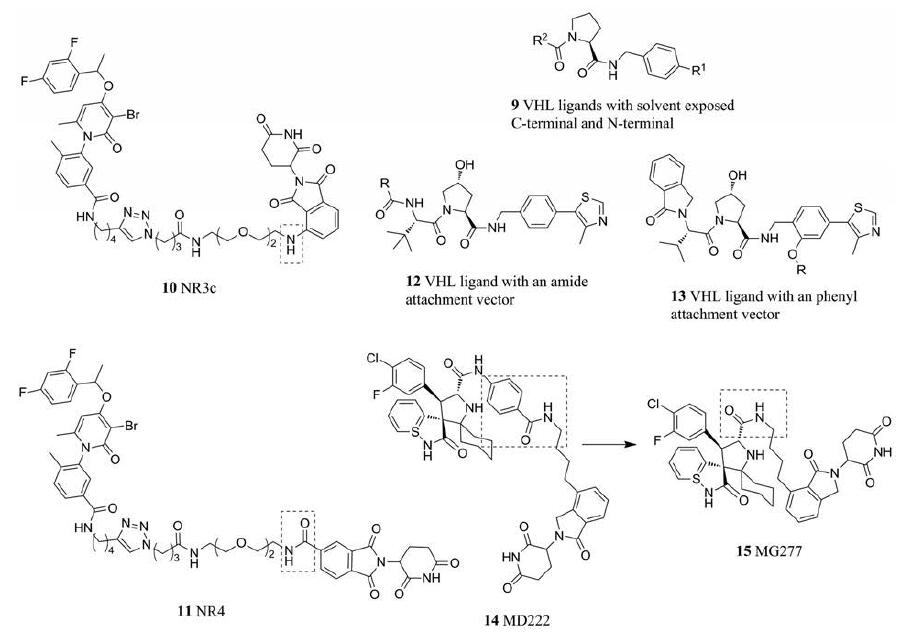
|
Figure 4 Structures of PROTAC molecules with different solvent-exposed terminus |
类似地, Smith团队在VHL配体的不同位置引入连接链, 通过氮原子连接而成的称为“酰胺系列”(12), 通过氧原子连接至苯基上合成的称为“苯基系列”(13), 从而形成两种结合位点不同的PROTAC分子。两种PROTAC系列均分别使用10、11、12和13个原子的连接链长度。随后, 研究人员应用这8种不同的PROTAC分子去降解α, β, γ和δ四种p38MAPK激酶亚型, 以检测连接链结合位点对于PROTAC降解选择性的影响。有趣的是, 实验结果表明“酰胺系列”中, 具有10、11原子连接链的PROTAC分子没有选择性, 会同时降解p38α和p38δ; 而具有较长连接链—12、13原子连接链的PROTAC分子对于p38α具有较强选择性。在“苯基系列”中, 较长连接链的PROTAC几乎对4种p38亚型均不产生降解, 但连接链的长度缩短至10个原子时, 就具有了很好的选择性降解p38δ能力。这一现象产生的原因尚未完全阐明, 但该研究小组认为存在两方面的潜在因素。首先, 三元复合物的亲和力差异可能是不同结合位点的PROTAC分子具有不同选择性带来的。因为, 不同的结合位点会导致PROTAC分子对于不同的靶蛋白的结合能力产生差异, 从而导致了三元复合物亲和力产生差异, 降解效率也随之相应变化, 故而可能对某种靶蛋白产生特定选择性。例如, p38δ中的特定残基, 在结合位点在酰胺基上的10原子长度PROTAC环境下, 与VHL的作用被大大削弱, 而结合位点在苯基上的13原子长度的PROTAC, 则使得残基与VHL的作用被加强。其次, 结合位点的变化会产生不同的空间效应。苯基连接位点使得PROTAC不需要弯曲自身, 而基于酰胺连接的PROTAC结构则产生了扭结连接链构象, 增加了产生相互作用的接触位点数量, 从而使得p38α上的残基更容易与其产生相互作用[23]。而Yang团队[24]改变了PROTAC分子MD-222 (14)中MDM2配体的分子结构, 将连接链的结合位点从苯甲酸转移到了脂肪酸上, 得到MG-227分子(15)。后者虽然在降解MDM2的效率上不如前者, 但是被实验证实是一种“分子胶水”, 可以通过不同的机制诱导翻译终止因子GSPT1的降解, 具有强大的抗癌活性。
众多实验结果表明, 改变连接链在配体上的结合位点会对PROTAC分子的降解效率、选择性产生显著影响, 通过改变结合位点来改善“靶蛋白-PROTAC-E3泛素连接酶”三元复合物的稳定性以及亲和力, 对于PROTAC分子的设计以及降解效率的改善都将带来一定的帮助以及启发。
4.2 连接链结合位点对PROTAC分子稳定性的影响研究发现, 连接链结合位点的位置同样会影响PROTAC分子的稳定性, 进而影响其降解能力。以靶向雌激素受体(ER)的PROTAC为例(图 5), 最初的方法是利用酯键将连接链连接到雌二醇的O-17位置(16), 但是该附着位点存在两个问题: 一是易受酯酶的影响灭活PROTAC, 释放雌二醇; 二是17-OH基团的丢失不利于E2与ER的结合。因此, 研究人员随后尝试了其他连接位点, 如C-16位置(17)和C-7位置(18), 结果表明通过碳碳键连接配体与连接链, 能够得到更具稳定的PROTAC分子。Cyrus小组经过实验, 进一步印证了这一观点, 与基于O-17和C-16的PROTAC相比, C-7的PROTAC具有更强的ER降解能力[25]。类似地, Cromm等[26]通过调整配体结构, 将连接链的结合位点从苯甲酰胺替代为苯酚。此举不仅提高了PROTAC分子的化学稳定性, 还通过减少分子内的酰胺键的数量增加了PROTAC的细胞通透性。鉴于连接链在配体结合位点的变化会对PROTAC分子的稳定性产生显著影响, 在进行PROTAC分子设计时, 应当充分考虑对应的结构的化学性质, 寻找最佳的连接位置, 实现PROTAC降解效率的最大化, 以寻求最优的PROTAC分子设计方案。
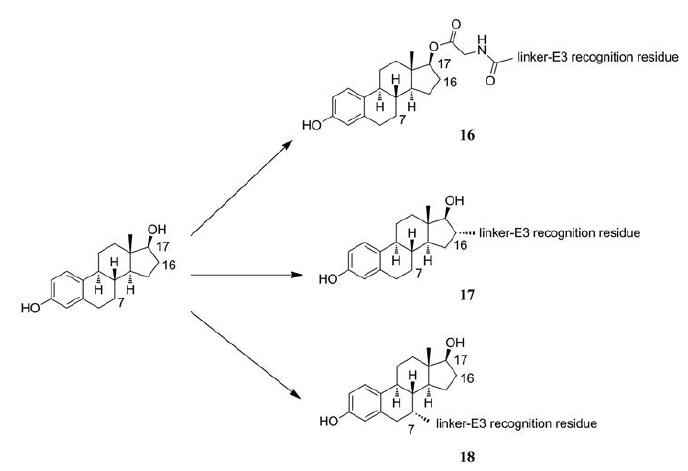
|
Figure 5 Linker location and orientation studies in the development of an ER-targeting PROTAC |
连接链的化学性质也是在连接链研究中备受关注的焦点之一。其不同的化学性质一方面可以影响PROTAC分子的稳定性以及刚性, 从而对整体的结合效率产生影响; 另一方面, 连接链中一些特殊的分子结构可以和靶蛋白或E3酶之间产生特定的分子间作用力, 从而影响“靶蛋白-PROTAC-E3泛素连接酶”三元复合物的总体稳定性以及自由能。此外, 在合成PROTAC分子时, 连接链的分子组成也会影响到合成的难度以及成本。
从小分子PROTAC开始被报道起, 许多PROTAC分子(图 6)的连接链都采用了聚乙二醇(PEG)的结构来调整连接链的长度(19~21)[27]。这种PEG类连接链(22, 23), 在合成上十分便捷, 不仅易于调节其整体长度, 也利于通过形成酯基或者醚键使两端更易设计合适的与E3配体或是靶蛋白配体连接的结合位点。与此同时, PEG类连接链中的氧原子可以与水分子形成氢键, 因此具有一定的亲水性[28]。除此之外, 针对PROTAC的共晶结构研究中也表明, 在稳定“靶蛋白-PROTAC-E3泛素连接酶”三元复合物的时, PEG类连接链起到了关键的作用。PEG类连接链与靶蛋白或E3酶的连接链-蛋白间相互作用, 限制了三元复合物的构象, 使得三元复合物更加稳定。不仅如此, 针对特定的靶蛋白以及E3酶, 通过在PEG类连接链上引入亲油性基团或可以产生π-π堆积的芳香环(图 7)也可以降低PROTAC分子的的极性, 产生额外的连接链-蛋白相互作用, 从而增加这种稳定性[29]。类似地, 为了降低PROTAC分子的自由能, 限制连接链的构象, Testa等[30]开创性地采用分子内大环化的策略, 基于MZ1(26)改进设计了具有两条PEG连接链的Macro PROTAC-1 (27)。后者对于BRD蛋白的降解效力以及在一系列同源蛋白中的选择性都有显著提升。
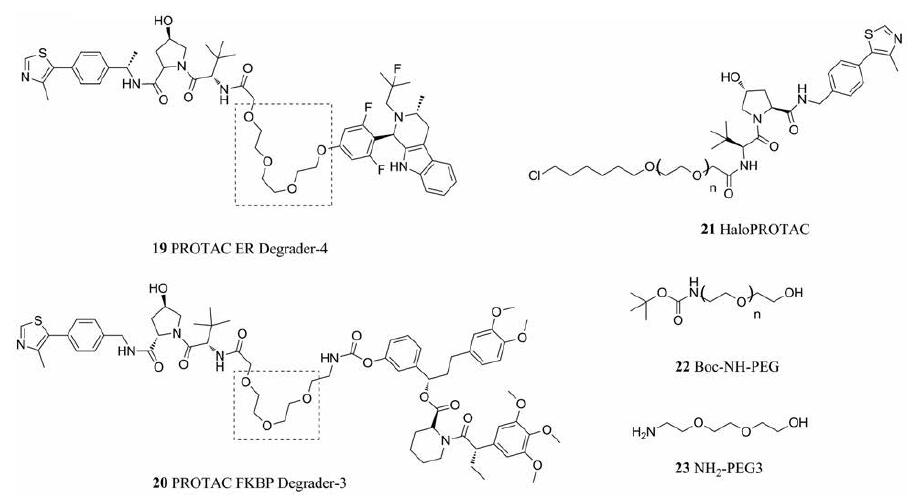
|
Figure 6 A collection of PROTAC and Halo PROTAC molecules with PEG-type linkers (19-21, the structure of PEG linker is boxed by dotted line); Synthetic precursors of NH2-PEG-OH type linkers (22 and 23) |
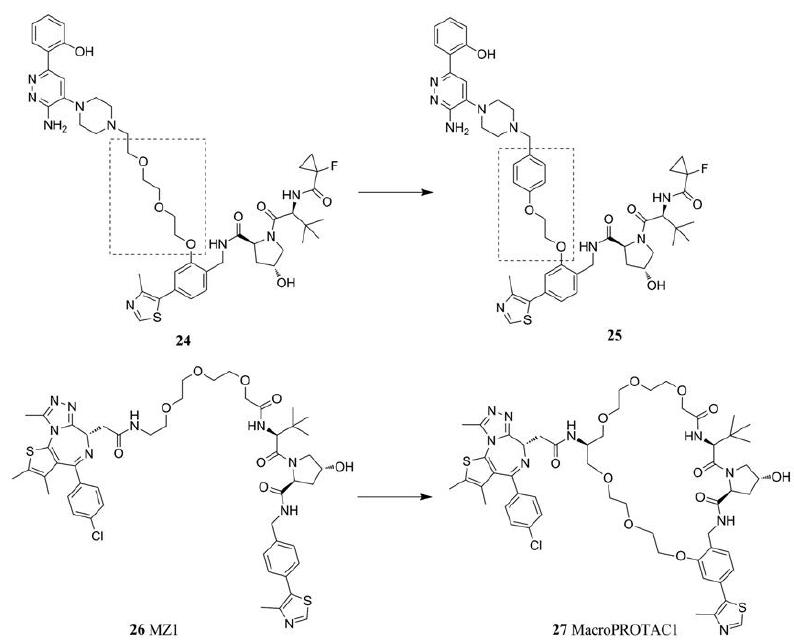
|
Figure 7 Replacement of part of PEG linker with a phenol group increases conformational restraint and generates new linker-protein interaction (24 and 25); Linker is optimized from MZ1 (26) to Macro PROTAC-1 (27) through macrocyclization |
除了基于PEG之外, 也有基于全碳链的连接链(图 8)。例如NH2-Cn-NH-Boc (n=2, 4, 5)系列与BrC10-methyl ester (28~30)。这一类连接链通常由全碳链以及末端的酰胺或者酯构成, 因其碳链在反应中呈惰性, 相较于PEG类连接链, 这些连接链在PROTAC的合成中具有更加好的稳定性。全碳的连接链具有的疏水性, 也会对PROTAC的作用效果有所影响[31], 可以通过在连接链上引入杂原子、用卤素修饰来改善。由于全碳链与配体、蛋白可能产生的相互作用较少, 故在形成三元复合物时其构象受限较少, 因此较长的全碳链也会带来连接链柔性过强的问题, 而这一点则可以通过在连接链中引入炔键[32]、螺环(32、33)来改善[33]。除此之外, 合理设计连接链的末端, 可以明显降低连接链与E3酶/靶蛋白配体串联的难度, 也是当下全碳连接链结构设计中的关键点。

|
Figure 8 Synthetic precursors of flexible all-carbon linkers (28-31); Modification of an all-carbon linker (32) to a rigid spirocyclic pyrimidine linker (33) |
为了经济地调节全碳连接链的长度, 一些通过将两条较短的碳链通过酰胺或酯连接为一条更长的连接链的方式被应用, 例如Boc-NH-C6-amido-C4-acid (31)通过一个酰胺键连接了两段碳链。虽然这样的方式在合成上可以更方便地用现成的连接链组合成目标长度的连接链, 但是这样不可避免地对连接链的化学稳定性带来了一定的影响。因为不仅在后续的合成中要保护酰胺键, 还要考虑人体内环境中下酰胺键可能的水解, 使得PROTAC的分子结构被破坏。在合成中, 连接链上额外的酰胺键也会对反应的产率产生影响[34]。另一方面, 引入酰胺键或可以作为一种改善整体分子的水溶性的方法, 但引入酰胺键后PROTAC分子的降解效率有可能受到影响[22]。
研究者优化连接链过程中, 往往需要尝试多种不同类型的连接链, 并且在连接链上引入不同的取代基来改善其水溶性、提升与靶蛋白的相互作用的强度以及限制连接链的构象(图 9)。在Hu等[35]对靶向ER的PROTAC分子(34)的研究中, 他们比较了全碳链、不同聚合度的PEG链、以及不同杂环取代的连接链对于PROTAC分子降解ER的效率的影响。其得到降解效率最佳的两种PROTAC, 分别含一种九碳连接链(34, a)和一种单氧原子取代的九碳连接链(34, e)。类似地, 在Zhou等[36]对靶向BET的PROTAC分子(35)的研究中, 他们在确定连接链的最适长度之后, 对连接链的化学组成进行了进一步的优化。他们先后尝试了将连接链中的氧原子替换为氮原子(a与b, c与d)、酰胺键替换为等长的碳链(a与c, b与d), 最终确定了具有最优降解效率的连接链(35, b), 进行进一步的优化。

|
Figure 9 Linker optimization on ER (34a-34j) and BET (35a-35d) targeting PROTACs |
对于PROTAC中连接链的研究, 早期大多是针对某种特定的PROTAC分子设计不同的连接链, 通过实验选择降解效率以及选择性最好的一种。近几年, 得益于对PROTAC作用机制、靶蛋白与E3酶的蛋白-蛋白相互作用以及“靶蛋白-PROTAC-E3泛素连接酶”三元复合物的研究逐步深入, 连接链在PROTAC中的作用逐步被重视。在今后, 通过对“靶蛋白-PROTAC-E3泛素连接酶”三元复合物中蛋白质-蛋白质相互作用、蛋白质-小分子相互作用模式的深入研究, 有望发现通过改变连接链的性质可控地改变三元配合物的空间构象, 以及连接链是如何在分子层面影响三元配合物形成的规律, 进而更加全面地认识连接链在PROTAC分子中的作用。
系统地研究PROTAC连接链—蛋白质间的作用模式, 对于连接链的设计有指导意义。连接链—蛋白的作用模式与PROTAC结合的蛋白质间的蛋白质-蛋白质相互作用关系密切, 可以影响PROTAC分子的结合稳定性以及降解效率。此方面的研究, 或可以通过建立PROTAC分子连接链的数据库, 对同类连接链的作用模式进行归纳, 通过机器学习等人工智能技术得到通用的结论。使得从连接链化学结构、长度, 蛋白质的结构等信息来预测连接链—蛋白的作用模式成为可能。这样或将减少为配体寻找连接链的时间, 从而缩短PROTAC分子的发现期。
目前优化PROTAC的连接链结构的方法, 主要有合理化结构设计(rational structure-based design)、骨架跃迁(scaffold hopping)及构效关系(structure activity relationship)等理论, 大多高度依赖经验且易混用各种理论, 缺乏通用的优化流程。寻找PROTAC连接链通用的优化策略乃至连接链定量化设计的方法, 或是该领域发展的下一阶段。一方面, 依赖经验的连接链优化策略会在PROTAC分子的合成上耗费大量的时间与精力, 而通用的优化策略可以避免大量的重复实验, 显著降低PROTAC合成的压力。另一方面, 定量的药物设计技术, 例如定量构效关系(quantitative structureactivity relationship)以及虚拟筛选(virtual screening)与骨架跃迁结合, 正在逐渐成熟, 这些技术的发展或对于PROTAC连接链的定量化设计有所帮助。
作者贡献: 曹戟和吴昊姝确定综述主题和论文框架; 沈心远、吴诗慧、李保林、李徐诺和曹戟共同查阅资料、撰写论文并绘制了插图; 沈心远和曹戟最后校对并确定论文终稿。
利益冲突: 本论文在撰写和发表过程中不存在利益冲突。
| [1] |
Sakamoto KM, Kim KB, Kumagai A, et al. Protacs: chimeric molecules that target proteins to the Skp1-Cullin-F box complex for ubiquitination and degradation[J]. Proc Natl Acad Sci U S A, 2001, 98: 8554-8559. DOI:10.1073/pnas.141230798 |
| [2] |
Sakamoto KM, Kim KB, Verma R, et al. Development of protacs to target cancer-promoting proteins for ubiquitination and degradation[J]. Mol Cell Proteomics, 2003, 2: 1350-1358. DOI:10.1074/mcp.T300009-MCP200 |
| [3] |
Schneekloth JS, Fonseca FN, Koldobskiy M, et al. Chemical genetic control of protein levels: selective in vivo targeted degradation[J]. J Am Chem Soc, 2004, 126: 3748-3754. DOI:10.1021/ja039025z |
| [4] |
Schneekloth AR, Pucheault M, Tae HS, et al. Targeted intracellular protein degradation induced by a small molecule: en route to chemical proteomics[J]. Bioorg Med Chem Lett, 2008, 18: 5904-5908. DOI:10.1016/j.bmcl.2008.07.114 |
| [5] |
Buckley DL, Raina K, Darricarrere N, et al. HaloPROTACS: use of small molecule PROTACs to induce degradation of halotag fusion proteins[J]. ACS Chem Biol, 2015, 10: 1831-1837. DOI:10.1021/acschembio.5b00442 |
| [6] |
Lebraud H, Wright DJ, Johnson CN, et al. Protein degradation by in-cell self-assembly of proteolysis targeting chimeras[J]. ACS Central Sci, 2016, 2: 927-934. DOI:10.1021/acscentsci.6b00280 |
| [7] |
Ciechanover A. Proteolysis: from the lysosome to ubiquitin and the proteasome[J]. Nat Rev Mol Cell Biol, 2005, 6: 79-87. DOI:10.1038/nrm1552 |
| [8] |
Cyrus K, Wehenkel M, Choi EY, et al. Two-headed PROTAC: an effective new tool for targeted protein degradation[J]. Chembiochem, 2010, 11: 1531-1534. DOI:10.1002/cbic.201000222 |
| [9] |
Paiva SL, Crews CM. Targeted protein degradation: elements of PROTAC design[J]. Curr Opin Chem Biol, 2019, 50: 111-119. DOI:10.1016/j.cbpa.2019.02.022 |
| [10] |
Chan KH, Zengerle M, Testa A, et al. Impact of target warhead and linkage vector on inducing protein degradation: comparison of bromodomain and extra-terminal(BET)degraders derived from triazolodiazepine(JQ1)and tetrahydroquinoline(IBET726)BET inhibitor scaffolds[J]. J Med Chem, 2018, 61: 504-513. DOI:10.1021/acs.jmedchem.6b01912 |
| [11] |
Hughes SJ, Ciulli A. Molecular recognition of ternary complexes: a new dimension in the structure-guided design of chemical degraders[J]. Essays Biochem, 2017, 61: 505-516. DOI:10.1042/EBC20170041 |
| [12] |
Miles LE. Properties, variants, and applications of the immunoradiometric assay method[J]. Ric Clin Lab, 1975, 5: 59-72. |
| [13] |
Gadd MS, Testa A, Lucas X, et al. Structural basis of PROTAC cooperative recognition for selective protein degradation[J]. Nat Chem Biol, 2017, 13: 514-521. DOI:10.1038/nchembio.2329 |
| [14] |
Roy RD, Rosenmund C, Stefan MI. Cooperative binding mitigates the high-dose hook effect[J]. BMC Syst Biol, 2017, 11: 74-84. DOI:10.1186/s12918-017-0447-8 |
| [15] |
Nowak RP, DeAngelo SL, Buckley D, et al. Plasticity in binding confers selectivity in ligand-induced protein degradation[J]. Nat Chem Biol, 2018, 14: 706-714. DOI:10.1038/s41589-018-0055-y |
| [16] |
Ohoka N, Okuhira K, Ito M, et al. In vivo knockdown of pathogenic proteins via specific and nongenetic inhibitor of apoptosis protein(IAP)-dependent protein erasers(SNIPERs)[J]. J Biol Chem, 2017, 292: 4556-4570. DOI:10.1074/jbc.M116.768853 |
| [17] |
e63.
|
| [18] |
Zoppi V, Hughes SJ, Maniaci C, et al. Iterative design and optimization of initially inactive proteolysis targeting chimeras(PROTACs)identify VZ185 as a potent, fast, and selective von hippel-lindau(VHL)based dual degrader probe of BRD9 and BRD7[J]. J Med Chem, 2019, 62: 699-726. DOI:10.1021/acs.jmedchem.8b01413 |
| [19] |
Buckley DL, Gustafson JL, Van Molle I, et al. Small-molecule inhibitors of the interaction between the E3 ligase VHL and HIF1alpha[J]. Angew Chem Int Ed Engl, 2012, 51: 11463-11467. DOI:10.1002/anie.201206231 |
| [20] |
Scott DE, Rooney TPC, Bayle ED, et al. Systematic investigation of the permeability of androgen receptor PROTACs[J]. ACS Med Chem Lett, 2020, 11: 1539-1547. DOI:10.1021/acsmedchemlett.0c00194 |
| [21] |
Jaime-Figueroa S, Buhimschi AD, Toure M, et al. Design, synthesis and biological evaluation of proteolysis targeting chimeras(PROTACs)as a BTK degraders with improved pharmacokinetic properties[J]. Bioorg Med Chem Lett, 2020, 30: 126877. DOI:10.1016/j.bmcl.2019.126877 |
| [22] |
Donoghue C, Cubillos-Rojas M, Gutierrez-Prat N, et al. Optimal linker length for small molecule PROTACs that selectively target p38alpha and p38beta for degradation[J]. Eur J Med Chem, 2020, 201: 112451. DOI:10.1016/j.ejmech.2020.112451 |
| [23] |
Smith BE, Wang SL, Jaime-Figueroa S, et al. Differential PROTAC substrate specificity dictated by orientation of recruited E3 ligase[J]. Nat Commun, 2019, 10: 131. DOI:10.1038/s41467-018-08027-7 |
| [24] |
Yang J, Li Y, Aguilar A, et al. Simple structural modifications converting a bona fide MDM2 PROTAC degrader into a molecular glue molecule: a cautionary tale in the design of PROTAC degraders[J]. J Med Chem, 2019, 62: 9471-9487. DOI:10.1021/acs.jmedchem.9b00846 |
| [25] |
Cyrus K, Wehenkel M, Choi EY, et al. Jostling for position: optimizing linker location in the design of estrogen receptortargeting PROTACs[J]. ChemMedChem, 2010, 5: 979-985. DOI:10.1002/cmdc.201000146 |
| [26] |
Cromm PM, Samarasinghe KTG, Hines J, et al. Addressing kinase-independent functions of Fak via PROTAC-mediated degradation[J]. J Am Chem Soc, 2018, 140: 17019-17026. DOI:10.1021/jacs.8b08008 |
| [27] |
Itoh Y, Ishikawa M, Naito M, et al. Protein knockdown using methyl bestatin-ligand hybrid molecules: design and synthesis of inducers of ubiquitination-mediated degradation of cellular retinoic acid-binding proteins[J]. J Am Chem Soc, 2010, 132: 5820-5826. DOI:10.1021/ja100691p |
| [28] |
Lai AC, Toure M, Hellerschmied D, et al. Modular PROTAC design for the degradation of oncogenic BCR-ABL[J]. Angew Chem Int Ed Engl, 2016, 55: 807-810. DOI:10.1002/anie.201507634 |
| [29] |
Farnaby W, Koegl M, Roy MJ, et al. BAF complex vulnerabilities in cancer demonstrated via structure-based PROTAC design[J]. Nat Chem Biol, 2019, 15: 672-680. DOI:10.1038/s41589-019-0294-6 |
| [30] |
Testa A, Hughes SJ, Lucas X, et al. Structure-based design of a macrocyclic PROTAC[J]. Angew Chem Int Ed, 2020, 59: 1727-1734. DOI:10.1002/anie.201914396 |
| [31] |
Wang Z, He N, Guo Z, et al. Proteolysis targeting chimeras for the selective degradation of Mcl-1/Bcl-2 derived from nonselective target binding ligands[J]. J Med Chem, 2019, 62: 8152-8163. DOI:10.1021/acs.jmedchem.9b00919 |
| [32] |
Qin C, Hu Y, Zhou B, et al. Discovery of QCA570 as an exceptionally potent and efficacious proteolysis targeting chimera(PROTAC)degrader of the bromodomain and extra-terminal(BET)proteins capable of inducing complete and durable tumor regression[J]. J Med Chem, 2018, 61: 6685-6704. DOI:10.1021/acs.jmedchem.8b00506 |
| [33] |
Nunes J, McGonagle GA, Eden J, et al. Targeting IRAK4 for degradation with PROTACs[J]. ACS Med Chem Lett, 2019, 10: 1081-1085. DOI:10.1021/acsmedchemlett.9b00219 |
| [34] |
Krajcovicova S, Jorda R, Hendrychova D, et al. Solid-phase synthesis for thalidomide-based proteolysis-targeting chimeras(PROTAC)[J]. Chem Commun(Camb), 2019, 55: 929-932. DOI:10.1039/C8CC08716D |
| [35] |
Hu J, Hu B, Wang M, et al. Discovery of ERD-308 as a highly potent proteolysis targeting chimera(PROTAC)degrader of estrogen receptor(ER)[J]. J Med Chem, 2019, 62: 1420-1442. DOI:10.1021/acs.jmedchem.8b01572 |
| [36] |
Zhou B, Hu J, Xu F, et al. Discovery of a small-molecule degrader of bromodomain and extra-terminal(BET)proteins with picomolar cellular potencies and capable of achieving tumor regression[J]. J Med Chem, 2018, 61: 462-481. DOI:10.1021/acs.jmedchem.6b01816 |
 2021, Vol. 56
2021, Vol. 56


